A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
خلية واحدة القياس الكمي لمعدلات تحلل البروتين بالأسفار الوقت الفاصل بين الفحص المجهري في ثقافة الخلية ملتصقة
In This Article
Summary
ويصف هذا البروتوكول أسلوب لتحديد البروتين التنصيف في الخلايا الحية واحدة ملتصقة، استخدام وسم نبض وتصوير الوقت الفاصل بين الأسفار المفاجئة، العلامة الانصهار البروتينات.
Abstract
البروتينات في حالة ديناميكية من التوليف ويمكن ضبط التدهور والنصف حياتهم تحت ظروف مختلفة. ولكن الأكثر استخداماً النهج لتحديد عمر البروتين أما أن تكون محدودة إلى متوسطات السكان من تفكيك الخلايا أو تتطلب استخدام مثبطات توليف البروتين. ويصف هذا البروتوكول وسيلة لقياس البروتين التنصيف في الخلايا الحية واحدة ملتصقة، استخدام الأداة الإضافية-العلامة الانصهار البروتينات في تركيبة مع الأسفار الوقت الفاصل بين الفحص المجهري. يمكن ربط أي البروتين من الفائدة التي تنصهر فيها علامة مبكرة تساهمي بصبغة نفاذية خلية فلورسنت، ويقترن بمشتق بينزيلجوانيني، ويمكن رصد انحلال السكان البروتين المسمى بعد تبييض للصبغة المتبقية. اللاحقة خلية تتبع والتحديد الكمي لشدة الأسفار المتكاملة على مر الوقت النتائج في منحنى اضمحلال أسي لكل خلية يتم تعقبها، مما يسمح لتحديد معدلات تحلل البروتين في الخلايا المفردة بملائمة منحنى. يوفر هذا الأسلوب تقديراً لعدم تجانس التنصيف في عدد أن خلايا المزروعة، التي لا يمكن تقييمها بسهولة بطرق أخرى. النهج الذي قدم هنا تنطبق على أي نوع من خلايا ملتصقة مثقف معربا عن وتين فائدة التي تنصهر فيها علامة مبكرة. هنا نستخدم الخلايا الجذعية الجنينية (ES) الماوس نمت على ألواح الخلايا المغلفة من كادهيرين ه الثقافة لتوضيح تردي خلية واحدة كيف يمكن تحديد معدلات البروتينات مع طائفة واسعة من إنصاف.
Introduction
من المعروف جيدا أن البروتينات الخلوية الخضوع دوران واسعة النطاق، مع معدلات التوليف وتدهور يجري محددة لكل البروتين وتخضع للتنظيم الفسيولوجي. تقليديا، كانت معدلات تحلل البروتين يقاس باستخدام أساليب السائبة، مثل تحليل تشيس نبض المشعة، أو إشراك مثبطات توليف البروتين مثل سيكلوهيكسيميدي1. في الآونة الأخيرة، أنشأ النظائر المستقرة وسم مع الأحماض الأمينية في الخلية والثقافة (سيلك) في تركيبة مع الطيف الكتلي لقياس معدل دوران البروتين على نطاق عالمي2. بيد أن هذه الأساليب هي محدودة من السكان في المتوسط ولذلك يتم فقدان المعلومات حول تقلب خلية بخلية. وعلاوة على ذلك، لا يمكن تحديد تغيرات عابرة في تدهور البروتين التي هي غير متزامنة عبر سكان الخلية.
بدلاً من ذلك، يمكن أيضا تحديد البروتين التنصيف بالنهج المستندة إلى الأسفار، التي غالباً ما تتميز بتقديم القرار خلية واحدة. على سبيل المثال، استخدمت بروتين فلوري أخضر فوتواكتيفاتابل (باجفب) لتحديد نصف عمر Oct4 في جنين الثدييات المبكر3. هناك طريقة أخرى لرصد تحلل البروتين في الخلايا الحية هو استخدام علامة مبكرة في تركيبة مع الأسفار الوقت الفاصل بين التصوير. علامة مبكرة هو نسخة متحولة من الحمض النووي إصلاح إنزيم س6-الكيلجوانيني الحمض النووي--الكيلترانسفيراسي (AGT) الذي يتفاعل مع بينزيلجوانيني على وجه التحديد المشتقات (حرس الحدود)، الذي يمكن أن يكون بالإضافة إلى المسابر الجزيئية4،5، 6. ولذلك، لا رجعة فيه المسمى البروتين الانصهار أي علامة مبكرة مع صبغة نفاذية خلية فلورسنت،. نبض وسم من بروتين فائدة التي تنصهر فيها الأداة العلامة، تليها تبييض للصبغة المتبقية، يسمح لرصد تدهور البروتين المسمى السكان وبالتالي لتحديد عمر البروتين. العلامات الإضافية التي استخدمت بنجاح لمطاردة نبض وسم البروتينات وتحديد البروتين الخلية التنصيف ملتصقة في الثقافة و في فيفو5،7،،من89. مجموعة كبيرة ومتنوعة من ركائز علامة مبكرة تغطي استخداماً أطياف فلورسنت متوفرة تجارياً، مما يتيح اختيار صبغ المثلى لكل تطبيق معين. وهكذا، يمكن أيضا استخدام العلامات الإضافية للتصوير متعدد الألوان بالاقتران مع غيرها من البروتينات الفلورية الانصهار أو الأصباغ. خلية كتيمة الأصباغ تصلح لوسم البروتينات الغشاء المربوطة، بينما الأصباغ نفاذية الخلية قابلة للتطبيق لرصد البروتينات داخل الخلية والغشاء زمنياً. وعلاوة على ذلك، بعض هذه المسابر يحمل تقريبا لا الأسفار القاعدية وتبدأ فقط التي تنبعث منها إشارة نيون قوية عند الربط ب الأداة إضافية-علامة10.
ويصف هذا البروتوكول كيفية قياس معدلات تدهور البروتينات المختلفة ذات الاهتمام في الخلايا المفردة باستخدام الأداة إضافية-علامة. هنا يمكننا تطبيق هذا الأسلوب للخلايا الجذعية الجنينية (ES) الماوس المستزرعة في ه-كادهيرين، ولكن ينبغي أن يكون من الممكن استخدامها مع أي نوع من الخلايا المستزرعة ملتصقة. نحن تبين أن نبض وسم البروتينات الانصهار علامة مبكرة تليها fluorescence الوقت الفاصل بين التصوير يسمح لتحديد خلية مفردة التنصيف من البروتينات المختلفة ذات الاهتمام ويوفر تقدير لتباين التنصيف في خلية إلى خلية سكان خلايا المستزرعة.
Access restricted. Please log in or start a trial to view this content.
Protocol
ملاحظة: في هذه الدراسة، تم استخدام خط الخلية E14 ES. بيد أن هذا البروتوكول لا تنطبق مباشرة على أي أخرى الماوس ES الخلية خط معربا عن وتين فائدة التي تنصهر فيها علامة مبكرة، أما بوضع علامات البروتين الذاتية أو باستخدام أوفيريكسبريشن. الأمثلة المعروضة في مقطع النتائج، استخدمت الدوكسي إيندوسيبلي خطوط الخلايا في الانصهار المفاجئة-العلامة (علامة مبكرة تنصهر فيها البروتينات التالية: نانج، Oct4، Srsf11، أو mOrange2 البروتينات الفلورية وسفجفب، ووضعها تحت السيطرة مروج الدوكسيسيكلين إيندوسيبلي. انظر11 للحصول على مزيد من المعلومات عن والبلازميدات المستخدمة لإنشاء خطوط خلية الانصهار المفاجئة، العلامة الدوكسيسيكلين إيندوسيبلي). النظام الدوكسي إيندوسيبلي يمكن أن تكون مفيدة بشكل خاص، كما أنه يسمح لأحكام السيطرة على توقيت وكثافة التعبير عن البروتين للفائدة. ج-محطة لتحديد المواقع من علامة مبكرة ويوصي، كتغيير N--المحطة الطرفية تسلسل الأحماض الأمينية أكثر احتمالاً لتغيير حياة نصف البروتين المستهدف (N-نهاية القاعدة12).
1-ه-كادهيرين طلاء وبذر الخلية
- إنشاء خط خلية ES معربا عن وتين فائدة التي تنصهر فيها الأداة الإضافية-العلامة4.
- معطف 100 ملم خلية ثقافة الأطباق مع 4 مل جيلاتين 0.1% (مخففة في الفوسفات مخزنة المالحة (PBS) دون Ca2 +/Mg2 +) حاء 1 إزالة الجيلاتين واسمحوا جافة حاء 1 الاستخدام فورا أو تخزين الأطباق المغلفة لمدة تصل إلى شهرين.
- تنمو خط الخلية من الفائدة في وفاق خلية الثقافة المتوسطة (غلاسكو الحد الأدنى الأساسية المتوسطة، وتستكمل مع 10% وفاق مؤهل بخلية الجنين البقري مصل، بيروفات صوديوم 2 مم، والأحماض الأمينية غير الأساسية 1%، 1% البنسلين/ستربتوميسين، 2 مم ل الجلوتامين، 100 ميكرومتر 2- ميركابتوثانول، اللوكيميا العوامل المثبطة (LIF)، 3 ميكرومتر CHIR99021 و 0.8 ميكرومتر PD184352) على أطباق الجيلاتين المغلفة في 37 درجة مئوية و 5% CO2 لمدة يومين حتى وصلت إلى الجمع بين 10-20 ميو الخلايا في الطبق.
ملاحظة: هنا، ليف أنتجت عابر تعداء الخلايا 293T HEK، تليها مجموعة طافية والتصفية. تم اختبار كل دفعة من ليف لقدرتها على الحفاظ على بلوريبوتينسي، ولكن تم تحديد تركيزات لا. ومع ذلك، ليف المؤتلف أيضا متوفرة تجارياً وتركيز استخداماً للماوس دا خلية ثقافة 1000 وحدة/مل13. - معطف صفيحة 96-كذلك مناسبة للتصوير مع 30 ميليلتر من الماوس المؤتلف Fc ه-كادهيرين الوهم البروتين (E-كندي-Fc) للبئر (5 نانوغرام/ميليلتر، تضعف في برنامج تلفزيوني مع Ca2 +/Mg2 +. استخدم الأسهم تركيزات 100 نانوغرام/ميليلتر، المخزنة في-80 درجة مئوية). تجنب بيبيتينج واسعة النطاق، كما ﻫ-كندي-Fc هش للغاية. احتضان في 37 درجة مئوية ح 1.5.
ملاحظة: اعتماداً على المجهر، قد تستخدم تنسيقات أخرى. - نضح ه-كندي-لجنة التيسير ويغسل مرة واحدة مع 100 ميليلتر من برنامج تلفزيوني مع Ca2 +/Mg2 +وإضافة 100 ميليلتر ES المعالجون مسبقاً خلية الثقافة المتوسطة.
- يغسل خلايا المصلحة مع 5 مل من برنامج تلفزيوني دون Ca2 +/Mg2 +. نضح وإضافة 2 مل يدتا التربسين 0.25%. احتضان لمدة 4 دقيقة عند 37 درجة مئوية. إضافة 4 مل من ES خلية الثقافة المتوسطة، ريسوسبيند وتدور إلى أسفل في 1000 غ س لدقيقة 4 Aspirate المادة طافية وريسوسبيند بيليه في 2 مل من الخلية وفاق جديدة الثقافة المتوسطة.
ملاحظة: يجب استخدام برنامج تلفزيوني دون Ca2 +/Mg2 + للغسيل الخلايا قبل تريبسينيزيشن بغية إزالة Ca2 + الأيونات، التي ضرورية لالتصاق الخلايا. في المقابل، لتمييع ه-كندي-Fc وطلاء (الخطوة 1، 4)، استخدم برنامج تلفزيوني مع Ca2 +/Mg2 +، منذ انضمام دإط الخلايا إلى ه-كندي-نادي الأطباق المغلفة هو Ca2 +-تعتمد14. - تضعف الخلايا حراكه 01:10 في 1 مل ES خلية الثقافة المتوسطة والعد، استخدام دائرة عد (تحميل 10 ميليلتر لدائرة من 0.1 مم).
- البذور 30,000 الخلايا/سم2 في ه-كندي-Fc المغلفة صفيحة التصوير. ملء يصل إلى 200 ميليلتر الواحدة وكذلك مع وفاق خلية الثقافة المتوسطة. لخطوط الخلايا الدوكسيسيكلين إيندوسيبلي، حمل بجرعة مناسبة من الدوكسي (تحسين جرعة وتوقيت التعريفي مسبقاً. تم علاج خطوط الخلايا المستخدمة هنا مع 500 نانوغرام/مليلتر الدوكسي 24 ساعة قبل التصوير). احتضان في 37 درجة مئوية و 5% CO2 والمضي قدما مع نبض وسم والتصوير 24 ساعة بعد البذر الخلية.
2-نبض العلامات الإضافية-العلامة
ملاحظة: لإجراء التجارب على تحلل البروتين المهم لاستخدامه بتركيز صبغة إضافية كافية. ينبغي أن يكون التركيز عالية بما فيه الكفاية لتعطي إشارة ساطعة في بداية الوقت الفاصل، كما ستنخفض الأسفار على مر الزمن. ومع ذلك، قد يتسبب استخدام تركيزات عالية جداً صبغ صبغ المتبقية يتم ترك في الأجل المتوسط أو في الخلايا حتى بعد الغسيل. قد ربط المنتجة حديثا علامة مبكرة جزيئات الصبغة الحرة فيما بعد على مدار الفيلم، الذي سوف يشوه منحنى الاضمحلال. إشارة fluorescence ملاحظتها سيعتمد على خصائص الصبغ، وخط الخلية المستخدمة، فضلا عن مستوى التعبير البروتين المقابل. ولذلك، من الأهمية بمكان لتحسين تركيز الصبغ عن طريق اختبار تخفيف مختلفة، بدءاً من تمييع اقترحتها الشركة المصنعة لتصوير الخلايا الحية. واستخدمت لهذه الدراسة، استعملنا الركازة فلورسنت. بتركيز أمثل من 12 نانومتر مصممة لخطوط الخلايا overexpression الدوكسيسيكلين إيندوسيبلي.
- تمييع صبغ المفاجئة إلى تركيز مناسب في الخلية وفاق قبل حرارة الثقافة المتوسطة. استخدام ماصة إزالة بلطف المتوسطة من الخلايا المصنفة سابقا في لوحة 96-جيدا وإضافة 50 ميليلتر المخفف المفاجئة صبغ كل بئر. احتضان لمدة 30 دقيقة عند 37 درجة مئوية.
- نضح الصبغة المخفف مع ماصة، وتغسل x 3 مع 200 ميليلتر من برنامج تلفزيوني المعالجون مسبقاً (دون Ca2 +/Mg2 +). إضافة 200 ميليلتر من ES خلية الثقافة المتوسطة واحتضان لمدة 15 دقيقة عند 37 درجة مئوية.
- كرر الخطوات السابقة الغسيل مرتين أكثر.
ملاحظة: المهم الغسيل واسعة النطاق بغية إزالة أي صبغة غير منضم. خطوات الغسيل المتكرر مع برنامج تلفزيوني إزالة الصبغة خارج الخلية المتبقية في المتوسط، بينما إينكوبيشنز 15 دقيقة تكفل وسم قوي من البروتينات الانصهار علامة مبكرة قبل البدء بالتجربة التصوير. ومع ذلك، نفذ الخطوات غسالة من بيبيتينج لطيف ولا تستخدم المضخة تلقائي، خلايا المصنف في ه-كندي-نادي تميل إلى فصل بسهولة. - إضافة 200 ميليلتر للتصوير المتوسطة. للحد من الخلفية الفلورية، واستخدام الفينول الحمراء المتوسطة الحرة (تستكمل مع 10% وفاق مؤهل بخلية الجنين البقري المصل، بيروفات صوديوم 2 مم، والأحماض الأمينية غير الأساسية 1%، 1% البنسلين/ستربتوميسين، 2 مم ل الجلوتامين، 2 ميكرومتر 100-ميركابتوثانول، ليف، 3 ميكرومتر CHIR99021 و 0.8 ميكرومتر PD184352).
3-الوقت الفاصل بين الفحص المجهري
- استخدام مجهر مناسب لتصوير حية، مما يتيح التحكم في درجة الحرارة وأول أكسيد الكربون2. تعيين درجة الحرارة إلى 37 درجة مئوية، و أول أكسيد الكربون2 إلى 5% مسبقة استخدام ويسمح لحجته على ح 1-2.
- ضع اللوحة في المجهر والبحث عن نقاط مناسبة للتصوير. حدد المواقع حيث يتم الخلايا موزعة بالتساوي، ولكنها ليست كثيفة جداً، كما سيسهل هذا التحليل. ضبط عدد النقاط المحددة لعدد الخلايا المطلوبة للتحليل. وسجلت في هذه الدراسة، 3-5 البقع كل خط الخلية.
- حدد الهدف. هدف X 20 المناسب لحجم الخلايا ES.
- حدد إعدادات الإضاءة الفلورسنت قناة (ق) التي سيتم تسجيلها. ضبط قوة الليزر والتعرض وفقا لكثافة إشارة الأسفار من الخلايا للفائدة. تأكد من الحصول على إشارة أولية قوية، حيث أنه سوف ينخفض على مدار الفيلم، بل تجنب القوى ليزر أعلى من 30% للتقليل من الضيائية وفوتوبليتشينج. لهذه الدراسة، عامل تصفية إثارة 632-22 نانومتر (الطول الموجي/ممر الموجه)، وتصفية الانبعاثات من 679/34 استخدمت نانومتر (الطول الموجي/ممر الموجه)، السلطة الليزر من الساعة 10% والتعرض من 100 مللي ثانية.
- حدد فترات ومدة التجربة الوقت الفاصل بين التصوير. للبروتينات مع إنصاف المتوقعة من ح 2-20، اختر مرات الحصول على 12-24 ساعة مع فواصل زمنية لمدة 15 دقيقة. للبروتينات، ويقلل من فترات وأوقات اكتساب، لزيادة البروتينات أطول عمرا تبعاً لذلك.
- بدء التصوير.
4-تجهيز وتحليل الصور
- جمع الصور المكتسبة مكدس في تنسيق tiff. للاستمرار في معالجة وتحليل، استخدام البرمجيات فيجي15. إذا كان لا يتم جمع البيانات الوقت الفاصل بين مكدس بالبرنامج المجهر، فتح الإطارات كل الوقت الفاصل بين التجربة في البرنامج (أما بواسطة النقر فوق الملف | فتح... أو سحب وإسقاط الملفات المقابلة على شريط الأدوات) وانقر فوق الصورة | مكدسات | الصور المكدس.
- استخدم الدالة خلفية استقطاع لإزالة الخلفية من كافة الصور الموجودة في بنية تخزين العناصر. للقيام بذلك، انقر فوق عملية | طرح الخلفية. قم بتحديد نصف قطر الكرة المتداول في إطار منبثق. استخدام دائرة نصف قطرها على الأقل حجم الخلايا المصورة. لتطبيق الطرح الخلفية على كافة الخلايا في بنية تخزين العناصر، حدد نعم في عملية مكدس؟ إطار منبثق.
- حدد خلية للفائدة ومنطقة للفائدة (ROI) حوله باستخدام شريط الأدوات رسم. لإشارات النووية، استخدم التحديد البيضاوي بالنقر فوق علامة التبويب المناظرة في شريط الأدوات الأولى وثم النقر فوق نواة للفائدة وضبط البيضاوي حوله. لإشارات هيولى أو مناطق كثيفة جداً استخدام التحديد اليدوي ليتمكن من متابعة عن كثب الخطوط الرئيسية الخلية. تجنب بما في ذلك مناطق كبيرة جداً من الخلفية، على الرغم من أنه ليس من الضروري أن تتبع الخطوط العريضة الخلية تحديداً بسبب الطرح اللاحقة لكل بكسل خلفية (الخطوة 4، 8-4.9).
- إضافة منطقة الاهتمام إلى إدارة العائد على الاستثمار بواسطة النقر فوق تحليل | أدوات | إدارة العائد على الاستثمار | إضافة، أو عن طريق الضغط على T على لوحة المفاتيح.
- المضي قدما إلى الإطار التالي بالانتقال علامة التبويب في إطار المكدس أو بواسطة ضغط shift + < على لوحة المفاتيح، ثم كرر الإجراءات السابقة. تتبع الخلية الاهتمام طوال فترة الفيلم وإضافة العائد على الاستثمار لكل إطار لإدارة العائد على الاستثمار. حفظ دوروا النهائية المحددة لكل خلية يتم تعقبها بواسطة النقر فوق مزيد | حفظ... في إدارة العائد على الاستثمار.
ملاحظة: بعد انقسامات الخلية، تتبع الخلايا ابنه كلا على حدة ومجموع كثافات المتكاملة بعد الطرح الخلفية (انظر الخطوات 4.6 4.9) حساب إضعاف البروتين خلال انقسام الخلية. حفظ مجموعات العائد على الاستثمار لكل من الخليتين ابنه. - حالما يتم تعقب خلية الفائدة طوال الفيلم، انقر فوق، التدبيرالحصول على قيم لمتوسط كثافة ومجال رويس. نسخ القيم التي تم الحصول عليها في برنامج جداول بيانات إلكترونية وحساب كثافة الخلية المتكاملة الخام لكل نقطة الوقت كما يلي:

حيث تعنيجهو متوسط كثافة والمنطقةجمجال دوروا المقابلة. - لتقدير الخلفية المحلية، إضافة عائد قريبة من خلية الفائدة لكل إطار زمني. تجنب بما في ذلك أي إشارة fluorescence الخلوية. استخدام عائد تعميم هو تقريبا حجم الخلية ونقل أو يتقلص تبعاً لذلك إذا لزم الأمر، مثلاً إذا كان التدخل في الخلايا المجاورة. المضي قدما كما هو موضح مسبقاً مع الخلوي تعيين مجموعة من أجل الحصول على خلفية دوروا دوروا ونسخ قيم الكثافة يقاس إلى جدول البيانات.
- للحصول على قيمة أساسية لتصحيح لكثافة الخلية المتكاملة، أولاً حساب كثافة الخلفية المتكاملة لكل نقطة الوقت:

حيث تعنيحرس الحدود هو متوسط كثافة الإشارات الخلفية و منطقةج هو مجال دوروا تطويق الخلية. لا تستخدم منطقة خلفية العائد على الاستثمار، ما لم يكن لكلا المجالين بنفس الحجم. - حساب كثافة المتكاملة النهائية طرح الخلفية الخلية لكل نقطة الوقت:

- لتطبيع منحنيات تسوس خلية مفردة، تقسيم قيمة كثافة كل نقطة الوقت بقيمة الكثافة لأول المرة-النقطة.
ملاحظة: تركيب منحنى (راجع الخطوة 4.11) يمكن أما أن يؤديها كل خلية مفردة أو على متوسط السكان. مطلوب تطبيع إذا تم حساب متوسط سكان على أساس بغية تجنب التحيز من كثافات مختلفة من الأسفار بين الخلايا. وهكذا يضمن التطبيع أن يساهم كل خلية مع نفس الوزن لمنحنى الاضمحلال النهائي. وبالإضافة إلى ذلك، يمكن أن تكون مفيدة لتصور يضمحل خلية مفردة مستقلة عن كثافات fluorescence المطلقة بهم (انظر الأرقام 3A-3 ج) التطبيع. بيد إلا إذا كان يتم تحديد خلية واحدة إنصاف ولا هو متوسط البيانات، قد يكون تم حذف خطوة تطبيع ويمكن إجراء تركيب منحنى مباشرة على البيانات الخام. - لتقدير فترة التنصيف البروتين، استخدم أداة ملائمة منحنى. واستخدمت في هذه الدراسة، منحنى MATLAB تركيب أدوات 3.4.1، الذي يوجد بشكل افتراضي في المقطع تطبيقات واجهة المستخدم MATLAB. استيراد شدة الأسفار وقيم الوقت من جدول البيانات الإلكترونية في MATLAB بالنقر فوق علامة التبويب بيانات الاستيراد فتح منحنى تركيب أدوات وحدد النقاط الزمنية والأسفار تسوس البيانات في البيانات X و Y البيانات التبويب اختر المعادلة مخصص في تركيب منحنى علامة التبويب وأدخل معادلة تحلل أسي:
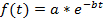
حيث f(t) هو شدة الأسفار عند نقطة وقت معين، كثافة الأولية و ب معدل الانحلال. في خيارات تناسب... علامة التبويب، حدد 0 للحد الأدنى لكل و ب. ثم سوف تظهر القيم المقدرة و ب في إطار نتائج. حساب فترة نصف العمر على النحو التالي:
Access restricted. Please log in or start a trial to view this content.
النتائج
ويوفر البروتوكول وصف تقديراً لتقلب خلية إلى خلية في فترة نصف العمر لأي بروتين معين تنصهر فيها علامة مبكرة. يسمح استخدام المؤتلف ه-كادهيرين-نادي لطلاء اللوحة تصوير لخلية مفردة القرار دإط الخلايا، التي لولاها لتنمو في مستعمرات. يمكن تعقب الخلايا المفردة بشكل منفصل طوال الفيلم ( ?...
Access restricted. Please log in or start a trial to view this content.
Discussion
أن الخطوة الأكثر أهمية عند استخدام علامة مبكرة لرصد تحلل البروتين لضمان أن أي صبغة غير منضم المتبقية متروك في الأجل المتوسط أو في الخلايا بعد الغسيل، على خلاف ذلك قد ربط حديثا إنتاج الجزيئات الإضافية-الوسم في وقت لاحق أثناء التجربة، وهناك أبي الحل الوسط منحنى الاضمحلال. وهذا يتحقق بتنفيذ ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
وأجريت تجارب الوقت الفاصل بين الفحص المجهري في الجزيئية البيولوجية فحص مرفق (قوات حرس الحدود)، لوزان. ونحن نشكر مارك ديلاتشاوكس (خدمة البصري السمعي، لوزان) بالفيديو وتحرير الفيلم.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
References
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745(2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156(2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15(2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved