A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
תא בודד כימות של חלבון השפלה המחירים על-ידי קרינה פלואורסצנטית זמן לשגות מיקרוסקופ בתרבות תא חסיד
In This Article
Summary
פרוטוקול זה מתאר שיטה לקביעת חלבון מחצית החיים של תאים חסיד חי אחד, באמצעות הדופק תיוג פלורסצנטיות הדמיה בצילום מואץ של חלבונים פיוז'ן SNAP-תג.
Abstract
חלבונים הם במצב דינמי של סינתזה, השפלה, החצי חייהם יכול להיות מותאם תחת נסיבות שונות. עם זאת, הנפוץ ביותר גישות לקביעת חלבון מחצית חיים האם מישהו מוגבל ממוצעים באוכלוסייה מתאי lysed או מחייבות שימוש מעכבי סינתזת חלבון. פרוטוקול זה מתאר שיטה למדוד חלבון מחצית החיים של תאים חסיד חי אחד, באמצעות SNAP-תג פיוז'ן חלבונים בשילוב עם מיקרוסקופ זמן לשגות זריחה. כל חלבון עניין דבוקה תג SNAP יכולות להיות covalently קשורות על ידי תא פלורסנט, חדיר לצבוע את זה משולב נגזרת benzylguanine, ניתן לנטר את הדעיכה של האוכלוסייה חלבון שכותרתו לאחר שטיפה של לצבוע שיורית. תא עוקבות מעקב, כימות עוצמת קרינה פלואורסצנטית משולב מעל הזמן יוצרת מעגל דעיכה מעריכית עבור כל תא מסומנים, מתן אפשרות לקביעת חלבון השפלה בבתי תאים בודדים על ידי עקומת התאמה. שיטה זו מספקת הערכה הטרוגניות של מחצית החיים בקרב אוכלוסיה של תאים בתרבית, אשר לא יכול בקלות להיות מוערך על ידי שיטות אחרות. הגישה המוצגת כאן היא ישימה לכל סוג של בתרבית תאים חסיד ביטוי חלבון עניין דבוקה תג SNAP. כאן אנו משתמשים בתאי גזע עובריים (ES) העכבר גדל על צלחות תרבות תא E קדהרין-מצופים להמחשת השפלה תא יחיד איך המחירים של חלבונים עם מגוון רחב של מחצית החיים יכול להיקבע.
Introduction
זה ידוע כי החלבונים עוברים תחלופת נרחב, עם המחירים סינתזה והשפלה להיות ספציפיים עבור כל חלבון ובכפוף תקנה פיזיולוגיים. באופן מסורתי, חלבון השפלה המחירים כבר נמדד בשיטות בתפזורת, כגון ניתוח צ'ייס הדופק רדיואקטיביים, או מעורבים מעכבי סינתזה חלבונים כגון cycloheximide1. לאחרונה, איזוטופ יציב תיוג עם חומצות אמינו בתרבות תא (SILAC) בשילוב עם ספקטרומטר מסה הוקם לכמת חלבון מחזור על מידה עולמי2. עם זאת, שיטות אלה מוגבלים על ידי ממוצע של אוכלוסייה, מידע על השתנות לתא ולכן הוא אבוד. יתר על כן, אין אפשרות לזהות שינויים חולף ב השפלה חלבון זה הם מסונכרן על פני האוכלוסייה תא.
לחלופין, חלבון מחצית החיים יכול להיקבע גם על-ידי קרינה פלואורסצנטית גישות, אשר לעיתים קרובות יש את היתרון של מתן רזולוציה תא בודד. למשל, חלבון פלואורסצנטי ירוק photoactivatable (paGFP) שימש כדי לקבוע Oct4 half-life של יונקים העובר מוקדם3. שיטה נוספת לעקוב אחר דעיכה חלבונים בתאים חיים היא השימוש תג הצמד בשילוב עם פלורסצנטיות הדמיה בצילום מואץ. SNAP-התג הוא גרסה מוטציה של ה-DNA תיקון אנזים O6- alkylguanine DNA-alkyltransferase (של AGT) זה במיוחד מגיב עם benzylguanine (BG) נגזרים, אשר יכול להיות מצמידים הגששים מולקולרית4,5, 6. לכן, כל חלבון כימרי SNAP-תג יכול להיות בלתי הפיך עם תוויות צבע חדיר פלורסנט, תא. הדופק תיוג של חלבון עניין התמזגו SNAP-תג, ולאחריה שטיפה של לצבוע שיורית, מאפשר לניטור ההשפלה של האוכלוסייה שכותרתו חלבון ובכך לקביעת חלבון half-life. SNAP-tags שימשו בהצלחה במשך. צ'ייס הדופק תיוג של חלבונים, לקביעת חלבון מחצית החיים ב מחסידי תא תרבות, אין ויוו5,7,8,9. ספקטרה פלורסנט מגוון גדול של מצעים SNAP-תג כיסוי נפוץ זמינים מסחרית, המאפשרת הבחירה של לצבוע האופטימלי עבור כל יישום ספציפי. לפיכך, SNAP-tags יכול לשמש גם עבור הדמיה ססגוניות בשילוב עם חלבונים פיוז'ן פלורסנט או צבעים אחרים. תא אטום צבעי מתאימים עבור תיוג קשורה קרום חלבונים, ואילו צבעי תא חדיר הינם ישימים עבור ניטור חלבונים תאיים והן קרום מאוגד. יתר על כן, חלק רגשים אלו שהפגינו כמעט אין קרינה פלואורסצנטית הבזליים, רק להתחיל לשדר אות ניאון חזק על קשירה SNAP-תג10.
פרוטוקול זה מתאר כיצד למדוד את שיעורי השפלה של חלבונים שונים עניין בתאים בודדים באמצעות תג SNAP. כאן אנו מיישמים שיטה זו לתאי גזע עובריים (ES) העכבר בתרבית על E-קדהרין, אבל זה צריך להיות ניתן להשתמש בה עם כל סוג תאים בתרבית חסיד. אנחנו מראים כי הדופק תיוג של חלבונים פיוז'ן SNAP-תג ואחריו קרינה פלואורסצנטית הדמיה בצילום מואץ מאפשר לקביעת תא בודד מחצית החיים של חלבונים שונים של עניין, ומספק הערכה ההשתנות לתא של מחצית החיים ב אוכלוסיית תאים בתרבית.
Access restricted. Please log in or start a trial to view this content.
Protocol
הערה: במחקר זה, הקו תא E14 ES שימש. עם זאת, פרוטוקול זה ישימה ישירות על כל העכבר ES תא קו אחר ביטוי חלבון עניין דבוקה תג SNAP, על-ידי תיוג החלבון אנדוגני או באמצעות ביטוי. על הדוגמאות המוצגת במקטע תוצאות, שימשו דוקסיציקלין-inducible SNAP-תג פיוז'ן תא קווים (SNAP-תג דבוקה החלבונים הבאות: Nanog, Oct4, Srsf11, או חלבונים פלורסנט mOrange2, sfGFP ו- put תחת השליטה של דוקסילין-inducible יזם. ראה11 למידע נוסף פלסמידים משמש את הדור של הקווים תא דוקסיציקלין-inducible SNAP-תג fusion). דוקסילין-inducible המערכת יכול להיות שימושי במיוחד, שכן היא מאפשרת לפיקוח הדוק על התזמון והעוצמה של הביטוי של החלבון עניין. C-מסוף מיצוב של הצמד-התג מומלץ, כמו שינוי של N-מסוף רצף חומצות אמיניות סביר יותר לשנות את half-life של החלבון היעד (N-end כלל12).
1. E-קדהרין ציפוי ו תא זריעה
- ליצור קו תא ES ביטוי חלבון עניין דבוקה SNAP-תג4.
- מעיל 100 מ מ תא תרבות מנות עם 4 מ"ל של ג'לטין 0.1% (מדולל בפוספט buffered תמיסת מלח (PBS) ללא Ca2 +/Mg2 +) עבור ה 1 הסר ג'לטין לתת להתייבש במשך ה 1 השתמש באופן מיידי או לאחסן את הכלים מצופים עד 2 חודשים.
- לגדל את שורת התאים עניין ב- ES תא תרבות בינוני (גלאזגו מינימום הכרחי בינוני, בתוספת 10% ES מוסמך התא העוברי שור סרום, פירובט נתרן 2 מ מ, חומצות אמינו שאינן הכרחיות 1%, 1% פניצילין/סטרפטומיצין, 2 מ מגלוטמין, 100 מיקרומטר 2- mercaptoethanol, לוקמיה מעכבות פקטור (LIF), מיקרומטר 3 CHIR99021, מיקרומטר 0.8 PD184352) על מנות מצופים ג'לטין-37 מעלות צלזיוס ו-5% CO2 2 ימים עד שהגיע זרימה של 10-20 תאים Mio לכל תבשיל.
הערה: כאן, LIF הופק על ידי תרביות תאים ארעית של תאים 293T HEK, ואחריו supernatant איסוף וסינון. כל אצווה של LIF נבדקה הפוטנציאל לשמור על pluripotency, אך לא נקבעו ריכוזים. עם זאת, LIF רקומביננטי גם זמינים מסחרית, ריכוז משמש בדרך כלל תרבית תאים העכבר ES הוא 1000 יחידות/mL13. - מעיל צלחת 96-ובכן מתאים הדמיה עם 30 µL של העכבר רקומביננטי E-קדהרין Fc כמירה חלבון (E-cad-Fc) ליום טוב (5 ng/µL, מדולל ב- PBS עם Ca2 +/Mg2 +. השתמש מניות ריכוזי 100 ng/µL, המאוחסנים ב-80 מעלות צלזיוס). הימנע pipetting מקיף, כמו E-cad-Fc שברירית מאוד. דגירה ב 37 ° C עבור 1.5 h.
הערה: בהתאם המיקרוסקופ, פורמטים אחרים עשויים לשמש. - האחות של E-cad-Fc, לשטוף פעם עם µL 100 ל- PBS עם Ca2 +/Mg2 +ולהוסיף 100 µL מראש ומחוממת ES תא תרבות בינוני.
- לשטוף את התאים עניין עם 5 מ של PBS ללא Ca2 +/Mg2 +. האחות ולהוסיף 2 מ של טריפסין-EDTA 0.25%. תקופת דגירה של 4 דקות ב 37 º C. להוסיף 4 מיליליטר ES תא תרבות בינוני, resuspend ספין למטה ב- 1000 g x עבור 4 דק לשאוב תגובת שיקוע, resuspend בגדר ב 2 מיליליטר טריים ES תא תרבות בינוני.
הערה: PBS ללא Ca2 +/Mg2 + אמור לשמש לניקוי התאים לפני trypsinization על מנת להסיר Ca2 + יונים, אשר נדרשים עבור תא אדהזיה. לעומת זאת, ה-cad-Fc דילול ציפוי (שלב 1.4), ולהשתמש PBS עם Ca2 +/Mg2 +, מאז הידבקות תאים ES E-cad-Fc מנות מצופה היא Ca2 +-תלויים14. - לדלל את התאים resuspended 1:10 ב 1 מ"ל של ES תא תרבות בינונית, ספירה, באמצעות חדר הספירה (עומס 10 µL עבור תא של 0.1 מ מ עומק).
- זרע תאים 30,000/cm2 ב- E-cad-Fc מצופה צלחת הדמיה. למלא עד 200 µL לכל טוב עם ES תא תרבות בינוני. עבור שורות תאים דוקסיציקלין-inducible, זירוז עם המינון המתאים של דוקסיציקלין (מיטוב מינון ותזמון של מראש את תנאי הגיוס. הקווים התא המשמש כאן טופלו ng/mL 500 דוקסיציקלין 24 שעות לפני הדמיה). דגירה-37 מעלות צלזיוס ו-5% CO2 והמשך עם דופק תיוג והדמיה 24 שעות לאחר תא זריעה.
2. דופק תיוג של הצמד-תג
הערה: לניסויים דעיכה חלבון חשוב להשתמש ריכוז לצבוע נאותה של הצמד. הריכוז צריך להיות גבוה מספיק להניב אות בהיר בתחילת מואץ, כמו ידי קרינה פלואורסצנטית יתמעטו עם הזמן. עם זאת, שימוש ריכוזי צבעים גבוה מדי עלול לגרום לצבוע שיורית להישאר בטווח הבינוני או בתאים גם לאחר כביסה. לצבוע חינם אולי לאחר מכן לאגד החדש מיוצר מולקולות SNAP-תג במהלך הסרט, יעוות את עקומת דעיכה. האות פלורסצנטיות שנצפה תלויות מאפייני לצבוע, את שורת התאים בשימוש, כמו גם את רמת הביטוי של החלבון המתאים. לכן, זה הכרחי למטב את הריכוז צבע על-ידי בדיקת דילולים שונים, החל מדילול הציע עבור הדמיה תא בשידור חי על ידי היצרן. במחקר זה, שימשה מצע פלורסנט מרחיקת אדום. ריכוז האופטימלית 12 ננומטר נקבע עבור שורות תאים דוקסיציקלין-inducible ביטוי.
- לדלל לצבוע SNAP כדי הריכוז המתאים במדיום התרבות תאים ES ומחוממת מראש. השתמש פיפטה להסיר בעדינות את המדיום התאים בעבר נזרע בצלחת 96-ובכן ולהוסיף 50 µL של צבע מדולל SNAP לכל טוב. תקופת דגירה של 30 דקות ב- 37 מעלות צלזיוס.
- וארוקן את צבע מדולל עם פיפטה ולשטוף 3 x עם µL 200 ל- PBS ומחוממת מראש (ללא Ca2 +/Mg2 +). להוסיף 200 µL ES תא תרבות בינוני ולאחר תקופת דגירה של 15 דקות ב 37 º C.
- חזור על השלבים הקודמים כביסה עוד פעמיים.
הערה: כביסה רב חשוב כדי להסיר את כל צבע לא מאוגד. השלבים כביסה חוזרות עם PBS להסיר את שאריות הצבע חוץ-תאית במדיום, ואילו incubations 15 דקות להבטיח תיוג חזקים של החלבונים פיוז'ן SNAP-תג לפני הפעלת הניסוי הדמיה. עם זאת, בצע את השלבים כביסה מאת pipetting עדין, אל תשתמש של העצמות אוטומטיים, כמו תאים נזרע על E-cad-Fc נוטים לנתק בקלות. - להוסיף 200 µL הדמיה בינוני. כדי להפחית את הרקע פלורסצנטיות, השתמש פנול אדום בינוני חינם (בתוספת 10% ES מוסמך התא העוברי שור סרום פירובט נתרן 2 מ מ, חומצות אמינו שאינן הכרחיות 1%, 1% פניצילין/סטרפטומיצין, 2 מ מגלוטמין, 100 מיקרומטר מרקפטואתנול, LIF, 3 מיקרומטר CHIR99021 ו- 0.8 מיקרומטר PD184352).
3. זמן לשגות מיקרוסקופ
- השתמש מיקרוסקופ מתאים הדמיה בשידור חי, ומאפשר טמפרטורה מבוקרת ו- CO2. הגדר את הטמפרטורה 37 ° C ו CO2 כדי 5% מראש להשתמש ולאפשר כדי equilibrate עבור h 1-2.
- למקם את הצלחת לתוך המיקרוסקופ, למצוא מקומות מתאימים עבור הדמיה. בחר כתמים התאים איפה המפוזרים באופן שווה, אבל לא יותר מדי צפוף, כמו זה יקל את הניתוח. להתאים את מספר הנקודות שנבחרו על מספר התאים הנדרש לניתוח. במחקר זה, נרשמו 3-5 נקודות לכל שורת התאים.
- בחר את המטרה. מטרה X 20 מתאים לגודל של תאים ES.
- בחר את הגדרות תאורה עבור פרופיל פלורסנט שיירשמו. להתאים את עוצמת הלייזר ואת חשיפה על פי עוצמת האות פלורסצנטיות מתאי עניין. הקפד להשיג קליטה ראשונית חזקה, מאז זה להקטין במהלך הסרט, אבל להימנע לייזר כוחות גבוה מ 30% על מנת למזער את phototoxicity ואת photobleaching. במחקר זה, מסנן עירור של 632/22 ננומטר (גל/bandpass), מסנן פליטה של 679/34 nm (גל/bandpass), עוצמת הלייזר של 10% וזמן חשיפה של 100 ms שימשו.
- בחר את מרווחי והמשך הדמיה של הניסוי זמן לשגות. החלבונים עם מחצית החיים הצפוי של h 2-20, בחר רכישת פעמים של 12-24 h עם מרווחי זמן של 15 דקות. חלבונים shorter-lived, להפחית את מרווחי ואת רכישת פעמים, להגדלת חלבונים longer-lived בהתאם.
- להתחיל הדמיה.
4. התמונה עיבוד וניתוח
- לאסוף את התמונות הנרכש ככל בערימה בתבנית tiff. ניתוח ועיבוד נוסף, לשימוש של תוכנות פיג'י15. אם זמן לשגות לא איסוף הנתונים כמו ערימה על ידי תוכנת מיקרוסקופ, פתח כל המסגרות של הניסוי זמן לשגות בתוכנה (או על ידי לחיצה על קובץ | לפתוח... או גרירה של יורד הקבצים המתאים לסרגל הכלים) ולחצו על התמונה | ערימות | תמונות לערום.
- השתמש בפונקציה רקע הפחת כדי להסיר את הרקע של כל התמונות במחסנית. לשם כך, לחץ על תהליך | להחסיר את הרקע. בחר את רדיוס הכדור מתגלגל בחלון המוקפץ. השתמש רדיוס זה לפחות בגודל של התאים עם תמונה. כדי להחיל רקע חיסור על כל התאים במחסנית, בחר כן ב תהליך מחסנית? חלון מוקפץ.
- בחר תא עניין וצייר אזור עניין (ROI) סביב זה באמצעות סרגל הכלים. עבור אותות גרעינית, השתמש הבחירה אליפסה מאת הראשון לחיצה על הלשונית המתאימה בסרגל הכלים ואז לחיצה על הגרעין של עניין, התאמת את האליפסה סביבו. עבור אותות cytoplasmic או אזורים צפופים מאוד להשתמש הבחירה ביד חופשית כדי להיות מסוגל לעקוב מקרוב את קווי המתאר של התא. הימנע כולל אזורים גדולים מדי של רקע, למרות שזה לא הכרחי לעקוב אחר קווי המתאר תא בדיוק בגלל חיסור עוקבות של רקע כל הפיקסלים (שלב 4.8 4.9).
- להוסיף את האזור של ריבית למנהל רועי בלחיצה נתח | כלים | רועי מנהל | להוסיף, או על-ידי הקשה על T על המקלדת.
- המשך למסגרת הבאה על-ידי הזזת הכרטיסיה בחלון מחסנית או על-ידי הקשה על shift + < במקלדת, ואז לחזור על הפעולות הקודמות. בצע את התא של עניין לאורך כל הקורס של הסרט ולהוסיף ההחזר של כל מסגרת מנהל רועי. להציל את רועי הסופי להגדיר עבור כל תא מסומנים על-ידי לחיצה על יותר | שמור. במנהל ROI.
הערה: לאחר חלוקות תאים, לעקוב אחר שני תאי הבת בנפרד, לסכם את עוצמות משולב שלהם לאחר רקע חיסור (ראה שלבים 4.6-4.9) לקחת בחשבון חלבון דילול במהלך חלוקת התא. שמור את ערכות ROI עבור שני תאי הבת. - לאחר התא עניין מתבצע לאורך כל הסרט, לחץ על מידה, כדי לקבל ערכים של ROIs עוצמת רשע ואזור. להעתיק את הערכים שהושג לתוכנית גליון נתונים אלקטרוניים, לחשב את עוצמת משולב raw של התא עבור כל הזמן-נקודה כדלקמן:

איפה מתכווןcזה את העוצמה רשע,שטחCהאזור של רועי המתאימים. - כדי להעריך את הרקע מקומיים, להוסיף רועי ליד התא עניין לכל מסגרת זמן. הימנע כולל קליטה סלולרית קרינה פלואורסצנטית. השתמש רועי מעגלי זה בערך בגודל של תא, להזיז או לכווץ את זה בהתאם. במידת הצורך, למשל אם התאים הסמוכים להתערב. המשך כמתואר קודם לכן עם הטלפון הסלולרי רועי להגדיר כדי לקבל רקע רועי להגדיר ולהעתיק את ערכי העוצמה נמדד הגיליון.
- כדי לקבל ערך מתוקן-רקע על עוצמת משולב של התא, תחילה לחשב את עוצמת משולב של הרקע עבור כל נקודת-זמן:

מתכווןBG איפה העוצמה אכזרי של רקע אות ו שטחC הוא האזור של רועי המקיפות את התא. אל תשתמש/י את האזור של רקע רועי, אלא אם כן בשני התחומים יש באותו הגודל. - חישוב סופי המופחת-רקע משולב האינטנסיביות של התא עבור כל נקודת זמן:

- לנרמל את עקומות דעיכה של תא בודד, לחלק את הערך בעוצמה של כל הזמן-נקודה לפי הערך בעוצמה של נקודת-בפעם הראשונה.
הערה: ההתאמה עקומה (ראה שלב 4.11) ניתן גם לבצע על כל תא בודד או על ממוצע האוכלוסייה. נורמליזציה נדרש אם מחושב ממוצע של אוכלוסייה בהתבסס על מנת למנוע הטיות של עוצמות קרינה פלואורסצנטית שונה בין תאים. נירמול ובכך מבטיחה כי כל תא תורם עם אותו משקל, עיקול דעיכה הסופי. בנוסף, נירמול יכול להיות שימושי להמחיש את נרקב תא בודד ללא תלות עוצמות קרינה פלואורסצנטית המוחלט שלהם (ראה דמויות 3A-3 C). אולם, אם רק מתא בודד מחצית החיים נקבעים, הנתונים לא הממוצע היה הצעד נרמול עשוי להיות מושמט, ההתאמה עקומה יכולה להתבצע ישירות על הנתונים הגולמיים. - על ההערכה של מחצית חיים חלבון, להשתמש בכלי המדידה עקומה. במחקר זה, העקומה MATLAB התאמת הכלים 3.4.1 שימש, אשר ממוקם כברירת מחדל במקטע יישומים של ממשק המשתמש של MATLAB. לייבא את עוצמת קרינה פלואורסצנטית, ערכי הזמן מהגיליון האלקטרוני לתוך MATLAB על ידי לחיצה על הכרטיסיה ייבוא נתונים פתח את עקומת התאמה בארגז הכלים ובחר את נקודות זמן והנתונים דעיכה פלורסצנטיות נתונים X ו Y הנתונים טאב בחר מותאמת אישית המשוואה פריסטלטיות טאב, הזן את המשוואה עבור דעיכה מעריכית:
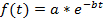
היכן f(t) עוצמת קרינה פלואורסצנטית בכל נקודת זמן נתונה, העוצמה הראשונית ו b קצב הדעיכה. בתיבת הדו-שיח אפשרויות להתאים. בכרטיסיה, בחר 0 עבור הגבול התחתון של גם וגם b . בין הערכים המשוערים עבור ו- b יופיע בחלון תוצאות. לחשב את זמן מחצית החיים כדלקמן:
Access restricted. Please log in or start a trial to view this content.
תוצאות
הפרוטוקול המתואר מספקת הערכה של מידת ההשתנות לתא בתוך מחצית חיים עבור כל חלבון נתון דבוקה תג SNAP. השימוש רקומביננטי E-קדהרין-Fc לציפוי של צלחת הדמיה מאפשרת לתא בודד רזולוציה ES תאים, אשר אחרת צומחים במושבות. תאים בודדים ניתן לעקוב בנפרד לאורך כל הקורס של הסרט ( איור 1 א').
Access restricted. Please log in or start a trial to view this content.
Discussion
השלב המכריע ביותר כאשר היא בעזרת תג SNAP לפקח חלבון דעיכה כדי להבטיח כי אין צבע לא מאוגד שיורית שנשאר במדיום או בתאים אחרי כביסה, אחרת זה עלול להיקשר החדש מיוצר מולקולות SNAP-תג מאוחר יותר במהלך ניסוי של שם פשרה eby העקומה דעיכה. זה מצד אחד, מושגת על ידי ביצוע בקפידה את כל השלבים המתוארים כביסה. מצ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
המחברים אין לחשוף.
Acknowledgements
מיקרוסקופ זמן לשגות הניסויים בוצעו ב למערכות ביולוגיות הקרנת מתקן (BSF), EPFL. אנו מודים מארק Delachaux (שירות Audiovisuel, EPFL) של צילום וידאו ועריכה של הסרט.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
References
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745(2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156(2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15(2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved