Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Sola célula cuantificación de las tasas de degradación de proteína por microscopia de fluorescencia Time-lapse en cultivo de células adherentes
En este artículo
Resumen
Este protocolo describe un método para determinar proteína vida media en las células adherentes vivas sola, con pulso de etiquetado e imagen Time-lapse de fluorescencia complemento etiqueta de proteínas de fusión.
Resumen
Las proteínas están en un estado dinámico de la síntesis y degradación y su vida media se pueden ajustar bajo diversas circunstancias. Sin embargo, más utilizados enfoques para determinar la vida media de la proteína pueden ser limitadas a medias de la población de las células sometidas a lisis o requieren el uso de inhibidores de la síntesis de proteínas. Este protocolo describe un método para medir la proteína vida media en las células adherentes solo vivas, utilizando proteínas de fusión SNAP tag en combinación con Time-lapse de microscopía de fluorescencia. Cualquier proteína de interés unida a una etiqueta de cierre puede ser covalentemente por, célula permeable colorante fluorescente que se une a un derivado de benzylguanine, y el decaimiento de la población proteínas etiquetados puede controlarse después de lavado del tinte residual. Celda posterior seguimiento y cuantificación de la intensidad de fluorescencia integrada por los resultados del tiempo en una curva de decaimiento exponencial para cada celda de orugas, lo que permite determinar las tasas de degradación de proteínas en las células por ajuste de curvas. Este método proporciona una estimación de la heterogeneidad de la vida media de una población de células cultivadas, que no se puede evaluar fácilmente por otros métodos. El enfoque presentado aquí es aplicable a cualquier tipo de cultivos de células adherentes expresando una proteína de interés unida a una etiqueta de cierre. Aquí utilizamos células madre embrionarias (ES) de ratón cultivadas en placas de cultura de E-cadherin-revestida de la célula para ilustrar cómo única degradación celular tipos de proteínas con una amplia gama de vida media pueden ser determinadas.
Introducción
Es bien sabido que las proteínas celulares someterse a rotación extensa, con tasas de síntesis y degradación es específica para cada proteína y conforme a la regulación fisiológica. Tradicionalmente, las tasas de degradación de proteína han sido medidos utilizando métodos a granel, tales como el análisis de chase de pulso radiactivo, ni recurrir a inhibidores de la síntesis de proteínas como la cicloheximida1. Más recientemente, Isótopo estable que etiqueta con aminoácidos en cultura de célula (SILAC) en combinación con la espectrometría de masas se ha establecido para cuantificar el volumen de la proteína en una escala global2. Sin embargo, estos métodos están limitados por un promedio de población, y por lo tanto se pierde la información sobre la variabilidad de célula a célula. Además, no se puede identificar cambios transitorios en la degradación de las proteínas que son sincronización entre la población de la célula.
Como alternativa, proteínas de vida media también puede ser determinado por los enfoques basados en la fluorescencia, que a menudo tienen la ventaja de proporcionar resolución unicelular. Por ejemplo, una proteína fluorescente verde photoactivatable (paGFP) se ha utilizado para determinar Oct4 Half-Life en el mamífero embrión temprano3. Otro método para controlar la descomposición de proteína en las células vivas es el uso de una etiqueta de cierre en combinación con imágenes de Time-lapse de fluorescencia. La etiqueta de cierre es una versión mutante de la DNA reparación enzimática O6- alkylguanine ADN-Alquilotransferasa (AGT) que reacciona específicamente con benzylguanine derivados (BG), que se pueden acoplar a sondas moleculares4,5, 6. por lo tanto, cualquier proteína de la fusión de SNAP tag puede etiquetarse irreversible con un colorante fluorescente, de permeable de la célula. Pulso de etiquetado de una proteína de interés unida a la etiqueta de complemento, seguida de lavado del tinte residual, permite monitoreo de la degradación de la población de proteínas marcadas y así para determinar la vida media de la proteína. Etiquetas de cierre se han utilizado con éxito para el pulso-persiga el etiquetado de proteínas y para la determinación de proteína vida media en adherente celular cultivo e in vivo5,7,8,9. Una gran variedad de sustratos de SNAP tag cubriendo comúnmente utilizado los espectros fluorescentes están disponibles en el mercado, lo que permite la selección del colorante óptimo para cada aplicación específica. Por lo tanto, también pueden utilizarse SNAP-etiquetas para imágenes multicolor en combinación con otras proteínas de fusión fluorescentes o colorantes. Tintes de celda impermeable son adecuados para el etiquetado de las proteínas de membrana atada, mientras que tintes permeable a la célula son aplicables para el control de proteínas intracelulares y de membrana-limitan. Además, algunas de estas puntas de prueba no exhiben casi ninguna fluorescencia basal y sólo empezar a emitir una fuerte señal fluorescente sobre Unión SNAP tag10.
Este protocolo describe cómo medir las tasas de degradación de diversas proteínas de interés en las células usando una etiqueta de cierre. Aquí aplicamos este método a células madre embrionarias (ES) de ratón cultivadas en la E-cadherina, pero debería ser posible usarlo con cualquier tipo de cultivo de células adherentes. Demostramos que pulso etiquetado complemento etiqueta proteínas de fusión seguidas por imágenes de Time-lapse de fluorescencia permite determinar la vida media de unicelular de diferentes proteínas de interés y proporciona una estimación de la variabilidad de célula a célula de vida media en un población de células cultivadas.
Protocolo
Nota: En este estudio, se utilizó la línea celular de E14 ES. Sin embargo, este protocolo es directamente aplicable a cualquier otra línea de células de ratón ES expresar una proteína de interés unida a una etiqueta de cierre, ya sea por selección de la proteína endógena o con sobreexpresión. Para los ejemplos mostrados en la sección de resultados, se utilizaron líneas de células de fusión de SNAP tag doxiciclina-inducible (SNAP-etiqueta unida a las proteínas siguientes: Oct4, Nanog, Srsf11, o a las proteínas fluorescentes mOrange2 y sfGFP y puesto bajo el control de un promotor inducible por la doxiciclina. Ver11 para obtener más información sobre la plásmidos utilizada para la generación de las líneas de células de fusión SNAP tag doxiciclina-inducible). El sistema inducible por la doxiciclina puede ser particularmente útil, ya que para controlar bien el tiempo y la intensidad de la expresión de la proteína de interés. C-terminal de posicionamiento de la etiqueta de complemento se recomienda, como cambiar el N-terminal del aminoácido secuencia es más probable que altera la vida media de la proteína diana (N-end regla12).
1. E-cadherin revestimiento y siembra de células
- Generar una línea de celular ES expresar una proteína de interés fusionado a la etiqueta de cierre4.
- Capa 100 mm celular cultura platos con 4 mL de 0.1% gelatina (diluido en tampón fosfato salino (PBS) sin Ca2 +/Mg2 +) para 1 h. retirar la gelatina y dejaron secas por 1 h. uso inmediatamente o almacenan los platos cubiertos para hasta 2 meses.
- Crecer la línea celular de interés en medio de cultivo celular ES (Glasgow mínimo medio esencial, suplementado con 10% ES calificado de celular suero bovino fetal, piruvato de sodio 2 m m, los de aminoácidos no esenciales 1%, 1% de penicilina/estreptomicina, 2 mM L-glutamina, 100 μm 2- mercaptoetanol, factor inhibidor de leucemia (LIF), 3 μm CHIR99021 y 0,8 μm PD184352) platos recubiertos de gelatina a 37 ° C y 5% de CO2 durante 2 días hasta llegar a la confluencia de Mio de 10-20 células por plato.
Nota: Aquí, LIF fue producido por transitorios de la transfección de células de HEK 293T, seguido del sobrenadante colección y filtración. Cada lote de LIF fue probado por su capacidad para mantener la pluripotencialidad, pero no se determinaron las concentraciones. Sin embargo, LIF recombinante también está disponible en el mercado y la concentración comúnmente utilizada para el cultivo de células de ratón ES 1000 unidades/mL13. - Cubrir una placa de 96 pocillos adecuada para la proyección de imagen con 30 μl de ratón recombinante proteína quimera de Fc E-cadherin (E-cad-Fc) por pozo (5 ng/μl, diluido en PBS con Ca2 +/Mg2 +. Utilizar concentraciones de valores de 100 ng/μl, almacenados a-80 ° C). Evitar pipetear extensa, como E-cad-Fc es muy frágil. Incubar a 37 ° C durante 1,5 horas.
Nota: Dependiendo del microscopio, podrían utilizarse otros formatos. - Aspirar E-cad-Fc, lavar una vez con 100 μl de PBS con Ca2 +/Mg2 +y añadir 100 μl de medio de cultivo de células ES precalentado.
- Lavar las células de interés con 5 mL de PBS sin Ca2 +/Mg2 +. Aspire y añadir 2 mL de tripsina-EDTA 0.25%. Incubar durante 4 min a 37 ° C. Añadir 4 mL de medio de cultivo celular ES, resuspender y desactivación a 1000 x g durante 4 min aspirar el sobrenadante y resuspender el precipitado en 2 mL de medio de cultivo de células ES fresco.
Nota: Puede usarse para el lavado de las células antes de tripsinización PBS sin Ca2 +/Mg2 + para eliminar el Ca2 + iones, que son necesarios para la adhesión celular. En cambio, para la dilución E-cad-Fc y la capa (paso 1.4), uso de PBS con Ca2 +/Mg2 +, desde la adhesión de células madre embrionarias a E-cad-Fc platos recubiertos es Ca2 +-dependiente14. - Diluir las células resuspendidos 1:10 en 1 mL de medio de cultivo ES celular y recuento, con una cámara de conteo (carga 10 μL para una cámara de 0,1 mm de profundidad).
- Chapa de proyección de imagen con semilla 30.000 células/cm2 en el E-cad-Fc. Completar hasta 200 μL por pozo con medio de cultivo celular ES. Para líneas celulares doxiciclina-inducible, inducir una dosis adecuada de doxiciclina (optimizar la dosis y el momento de la inducción de antemano. Las líneas celulares usadas aquí fueron tratadas con doxiciclina 500 de ng/mL 24 h antes de la proyección de imagen). Incubar a 37 ° C y 5% CO2 y continuar con el pulso etiquetado e imagen 24 h después de la siembra de la célula.
2. etiquetado de la etiqueta de presión de pulso
Nota: Para experimentos de descomposición de proteínas es crucial utilizar una concentración adecuada de tinte SNAP. La concentración debe ser suficientemente alta como para producir una señal brillante en el comienzo de Time-lapse, como la fluorescencia disminuye con el tiempo. Sin embargo, el uso de tinte demasiado altas concentraciones podría causar tinte residual quedando en el medio o en las células incluso después del lavado. El colorante libre posteriormente podría enlazar nuevamente producidas moléculas de complemento etiqueta a lo largo de la película, que se distorsione la curva de decaimiento. La señal de fluorescencia observada dependerá de las propiedades de la tintura, la línea celular utilizada, así como el nivel de expresión de la proteína correspondiente. Por lo tanto, es crucial optimizar la concentración del tinte probando diferentes diluciones, a partir de la dilución sugerida para la proyección de imagen de células vivas por el fabricante. Para este estudio, se utilizó un sustrato fluorescente far-red. Una concentración óptima de 12 nM se determinó para las líneas celulares sobreexpresión inducible por la doxiciclina.
- Diluir el tinte de complemento a la concentración adecuada en medio de cultivo de células ES precalentado. Utilice una pipeta para eliminar suavemente el medio de las células previamente sembradas en la placa de 96 pocillos y añadir 50 μl de colorante de complemento diluido por pozo. Incubar durante 30 min a 37 ° C.
- Aspirar el medio de contraste diluido con una pipeta y lavar 3 x con 200 μL de PBS precalentada (sin Ca2 +/Mg2 +). Añadir 200 μL de medio de cultivo celular ES e incubar por 15 min a 37 ° C.
- Repita los pasos anteriores de lavado dos veces más.
Nota: Lavado amplia es importante para eliminar cualquier tinte. Los pasos de varias lavadas con PBS Quite el tinte residual extracelular en el medio, considerando que las incubaciones de 15 min garantizar robusto etiquetado las etiqueta complemento de proteínas de fusión antes de comenzar el experimento de la proyección de imagen. Sin embargo, realice los pasos de lavado mediante pipeteo suave y no utilice un aspirador automático, ya que las células sembradas en E-cad-Fc tienden a soltar fácilmente. - Añadir 200 μL de medio de la proyección de imagen. Para reducir el fondo de la fluorescencia, utilice medio libre de rojo de fenol (complementado con piruvato de sodio 2 m m, los de aminoácidos no esenciales 1%, 10% ES calificado de celular suero fetal bovino, penicilina/estreptomicina de 1%, 2 mM L-glutamina, 100 μm 2-Mercaptoetanol, LIF, 3 μm CHIR99021 y 0,8 μm PD184352).
3. Time-lapse microscopía
- Utilizar un microscopio adecuado para la proyección de imagen vivo, permitiendo a temperatura controlada y CO2. Ajuste la temperatura a 37 ° C y el CO2 al 5% antes de usar y permite equilibrar para 1-2 h.
- Coloque la placa en el microscopio y encontrar lugares adecuados para la proyección de imagen. Seleccionar lugares donde las células están uniformemente distribuidos, pero no demasiado denso, como esto facilitará el análisis. Ajustar el número de puntos seleccionados para el número de células necesarias para el análisis. En este estudio, se registraron 3-5 puntos por línea celular.
- Seleccione el objetivo. Objetivo 20 X es apropiado para el tamaño de células madre embrionarias.
- Seleccione los ajustes de iluminación para el canal fluorescente para ser grabada. Ajustar la potencia del láser y exposición según la intensidad de la señal de fluorescencia de las células de interés. Asegúrese de obtener una señal inicial fuerte, ya que disminuyen en el transcurso de la película, pero evitar láser poderes superiores al 30% con el fin de minimizar la fototoxicidad y photobleaching. Para este estudio, un filtro de la excitación del 632 22 nm (longitud de onda/banda), filtro de emisión de 679/34 nm (longitud de onda/banda), energía del laser de 10% y exposición de 100 ms se utilizaron.
- Seleccione los intervalos de proyección de imagen y duración del experimento Time-lapse. Proteínas con vida media esperada de 2-20 h, seleccione tiempos de adquisición de 12-24 h con intervalos de 15 minutos. Shorter-lived proteínas, reducir los intervalos y tiempos de adquisición, aumento de proteínas de larga vida en consecuencia.
- Iniciar la proyección de imagen.
4. procesamiento y análisis de imágenes
- Recoger las imágenes adquiridas como una pila en formato tiff. Para posterior procesamiento y análisis, utilice el software de FIJI15. Si los datos Time-lapse no es recopilados como una pila por el software del microscopio, abrir todos los marcos de la experiencia de lapso de tiempo en el software (ya sea haciendo clic en archivo | Abrir... o arrastrar y soltar los archivos correspondientes a la barra de herramientas) y haga clic en imagen | Pilas | Imágenes a la pila.
- Utilice la función de fondo de restar para quitar el fondo de todas las fotografías de la pila. Para ello, haga clic en proceso | Sustraer el fondo. Seleccionar el radio balanceo de la bola en la ventana emergente. Utilizar un radio por lo menos el tamaño de las células reflejada. Para aplicar la sustracción de fondo a todas las celdas de la pila, seleccione sí en la pila proceso? ventana emergente.
- Seleccionar una celda de interés y dibuja una región de interés (ROI) alrededor de ella usando la barra de herramientas. Para señales nucleares, uso la selección Oval primero clic en la pestaña correspondiente en la barra de herramientas haciendo clic en el núcleo de interés y ajustar el óvalo alrededor de él. Para señales citoplasmáticas o regiones muy densas, utilice la selección a mano alzada para poder seguir de cerca los contornos de la célula. Evitar incluyendo regiones demasiado grandes de fondo, aunque no es necesario seguir los contornos de la célula precisamente debido a la posterior sustracción de los píxeles del fondo (paso 4.8-4.9).
- Añadir la región de interés para el gestor del retorno de la inversión haciendo clic analizar | Herramientas | Gerente de ROI | Añadir, o pulsando T en el teclado.
- Continúe con el siguiente fotograma moviendo la pestaña en la ventana de la pila o presionando shift + < en el teclado, repita las acciones anteriores. Siga la célula de interés en el transcurso de la película y agregue el ROI de cada fotograma con el Gerente de ROI. Guardar el ROI final establezca para cada celda seguimiento haciendo clic en más | Guardar... en el administrador de ROI.
Nota: Después de divisiones de célula, seguimiento por separado de ambas células hijas y en definitiva sus intensidades integradas después de fondo (ver pasos 4.6-4.9) para tener en cuenta para la dilución de la proteína durante la división celular. Guardar los conjuntos de retorno de la inversión para ambas células hijas. - Una vez que la célula de interés se realiza un seguimiento a lo largo de la película, haga clic en medida, para obtener los valores de la intensidad media y el área de los ROIs. Copiar los valores obtenidos en un programa de hoja electrónica de cálculo y calcular la intensidad integrada cruda de la célula para cada punto del tiempo como sigue:

que significaces la intensidad media yzonaCla zona del retorno de la inversión correspondiente. - Para estimar el fondo local, añadir un retorno de la inversión cerca de la célula de interés para cada plazo. Evitar incluyendo cualquier señal de fluorescencia celular. Utilizar un ROI circular que es aproximadamente del tamaño de una célula y mover o reducir por consiguiente si es necesario, por ejemplo, si interfieren de células vecinas. Proceda como se describe anteriormente con el celu para obtener un fondo ROI ROI establecido y copiar los valores de intensidad medidos en la hoja de cálculo.
- Para obtener un valor corregido de fondo para la intensidad integrada de la célula, calcular primero la intensidad integrada de fondo para cada momento:

donde decirBG es la intensidad media de la señal de fondo y el áreaC es el área de la ROI que rodea la célula. No utilice el área del fondo del retorno de la inversión, a menos que ambas áreas tienen el mismo tamaño. - Calcular la intensidad integrada final restada por el fondo de la célula para cada momento:

- Para normalizar las curvas de decaimiento de unicelular, divida el valor de la intensidad de cada momento por el valor de la intensidad del primer tiempo-punto.
Nota: El ajuste de curvas (ver paso 4.11) puede ya realizarse en cada célula o en el promedio de la población. Una normalización es necesaria si se calcula un promedio de la población base para evitar sesgos de intensidades de fluorescencia diferentes entre las células. La normalización garantiza así que cada célula contribuye con el mismo peso a la curva de decaimiento final. Además, la normalización puede ser útil para visualizar las caries unicelular independientemente de sus intensidades de fluorescencia absoluta (ver Figuras 3A-3C). Sin embargo, si sólo se determina la vida media de unicelular y no es un promedio de los datos, puede omitir el paso de la normalización y el ajuste de curvas se puede realizar directamente en los datos en bruto. - Para la estimación de la vida media de la proteína, utilice una herramienta de ajuste de curva. En este estudio, se utilizó el ajuste toolbox 3.4.1 de la curva MATLAB, que se encuentra por defecto en la sección de aplicaciones de la interfaz de usuario MATLAB. Importar la intensidad de fluorescencia y los valores de tiempo de la hoja de cálculo electrónica en MATLAB haciendo clic en la ficha de Datos de importación abren la caja de herramientas de ajuste de la curva y seleccione los puntos de tiempo y datos de decaimiento de la fluorescencia en los datos de X y Y datos tabla elija Custom ecuación en la guarnición de curva ficha y escriba la ecuación para un decaimiento exponencial:
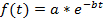
donde f (t) es la intensidad de fluorescencia en un momento dado, una intensidad inicial y b la velocidad de descomposición. En las Opciones de ajuste... ficha, seleccione 0 para el límite inferior de a y b. Los valores estimados de a y b aparecerán en la ventana resultados. Calcular la vida media como sigue:
Resultados
El protocolo descrito proporciona una estimación de la variabilidad de célula a célula en Half-Life para cualquier proteína dada sobre una etiqueta de cierre. El uso de E-cadherin-Fc recombinante para el recubrimiento de la placa de imagen permite resolución de célula en células madre embrionarias, que crecen en colonias. Las células pueden ser rastreadas por separado en el transcurso de la película ( figura 1A).
Para determinar la vida media de proteína...
Discusión
El paso más crucial cuando se utiliza una etiqueta de cierre para controlar la descomposición de la proteína asegurar que ningún tinte residual queda en el medio o en las células después del lavado, como si no lo puede enlazar a recién producido moléculas de complemento etiqueta más adelante en el curso de la experiencia y compromiso la curva de decaimiento de la Eby. Se trata por un lado logrado realizando cuidadosamente todos los pasos de lavado descrito. Por otro lado, la concentración de tinte se debe mante...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Se realizaron experimentos de microscopía de Time-lapse en la Biomolecular Screening Facility (BSF), EPFL. Agradecemos a Marc Delachaux (Service Audiovisuel, EPFL) para la grabación y edición de la película.
Materiales
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
Referencias
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745 (2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156 (2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15 (2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados