Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tek hücreli miktar Protein yıkımı oranları hızlandırılmış Floresans mikroskobu yapisan hücre kültüründe tarafından
Bu Makalede
Özet
Bu iletişim kuralı protein yarı-tek yaşam yapisan hücrelerde, hayatını darbe etiketleme ve floresan hızlandırılmış görüntüleme ek-etiket füzyon proteinlerin kullanarak belirlemek için bir yöntem açıklanır.
Özet
Protein sentezi dinamik bir durumda olduğundan ve bozulması ve yarı hayatlarını çeşitli şartlar altında ayarlanabilir. Ancak, en sık protein half-life ya olduğunu belirlemek için yaklaşımlar kullanılan lysed hücrelerden popülasyon ortalamaları için sınırlı veya protein sentez inhibitörleri kullanılmasını gerektirir. Bu iletişim kuralı protein yarı-hızlandırılmış Floresans mikroskobu ile birlikte ek-etiket füzyon protein kullanarak hayatımızda tek yaşam yapışık hücreleri, ölçmek için bir yöntem açıklanır. Bir ek etiketi için erimiş ilgi herhangi bir protein kovalent birleştiğinde bir Floresan, hücre geçirgen boya tarafından benzylguanine türev bağlanabilir ve etiketli protein nüfus çürüme kalan boya fiyasko sonra izlenebilir. Sonraki hücre izleme ve miktar bir üstel çürüme eğri eğri uydurma tarafından protein yıkımı oranları Tek Kişilik hücrelerde belirlemek için izin izlenen her hücre için zaman sonuçlar üzerinde tümleşik Floresans yoğunluğunu. Bu yöntem için yarı-hayat kolayca diğer yöntemlerle ölçülen olamaz kültürlü hücrelerinin bir popülasyondaki heterojen bir tahmin sağlar. Burada sunulan yaklaşım kültürlü yapışık hücreleri bir protein bir ek etiketi için erimiş ilgi ifade, her türlü için geçerlidir. Burada E-cadherin kaplı hücre kültür Tabaklarda yetiştirilen fare embriyonik kök (ES) hücre protein oranları geniş bir yarı-hayat ile belirlenebilir nasıl tek hücre yıkımı göstermek için kullanın.
Giriş
İyi hücresel protein sentezi ve yıkımı oranları belirli her protein ve fizyolojik düzenleme tabi olmak ile geniş ciro, tabi olduğu bilinmektedir. Geleneksel olarak, protein yıkımı oranları ölçülen radyoaktif darbe chase analizi gibi toplu yöntemleri kullanarak veya sikloheksimit1gibi protein sentez inhibitörleri içeren olmuştur. Daha yakın zamanlarda, kararlı izotop kütle spektrometresi ile birlikte (SILAC) hücre kültüründe amino asitler ile etiketleme bir küresel ölçekte2protein ciro ölçmek için kurulmuştur. Ancak, bu yöntemlerin nüfus sayı ortalaması alınarak sınırlıdır ve hücre-hücre değişkenlik hakkında bilgi bu nedenle kaybolur. Ayrıca, hücre nüfus arasında iseEşitlenmemiş protein yıkımı geçici değişimler tanımlanamıyor.
Alternatif olarak, protein yarı-hayat da hangi sık sık tek hücreli çözünürlük sağlama avantajına sahip floresan tabanlı yaklaşımlar tarafından belirlenebilir. Örneğin, bir photoactivatable yeşil flüoresan protein (paGFP) erken memeli embriyo3Oct4 half-life belirlemek için kullanılır. Canlı hücreler içinde protein çürüsün izlemek için başka bir yöntem bir ek etiketi Floresans hızlandırılmış görüntüleme ile birlikte kullanmaktır. EK-etiket DNA onarım enzim O6- alkylguanine DNA-alkyltransferase (AGT) özellikle moleküler probları4,5' e, birleştiğinde (BG) türevleri ile benzylguanine tepki verir, mutasyona uğramış bir sürümüdür 6. bu nedenle, herhangi bir ek-etiket füzyon proteini bir Floresan, hücre geçirgen boya ile geri dönüşümsüz olarak etiketlenmiş. Darbe bir protein ek etiketi artık boya fiyasko tarafından takip, erimiş ilgi etiketlerine göre etiketli protein nüfus düşüşü izleme ve dolayısıyla protein half-life belirlemek için izin verir. Nabız-chase proteinlerin etiketleme için ek-etiketleri başarıyla kullanılmaktadır ve protein belirlemek için yarı-yapışık hayatımızda hücre kültür ve in vivo5,7,8,9. Yaygın olarak kullanılan çeşitli kapsayan ek-etiket yüzeylerde floresan spectra her özel uygulama için en iyi boya seçimi etkinleştirmek ticari olarak kullanılabilir. Böylece, ek-etiketleri diğer floresan füzyon protein veya boya ile çok renkli görüntüleme için de kullanılabilir. Hücre geçirgen boyalar hücre içi hem de membran bağlı proteinler izlemek için geçerlidir, ancak hücre geçirimsiz boyalar proteinler membran hayvan zinciri, etiketleme için uygundur. Ayrıca, bazı Bu sondalar neredeyse hiçbir Bazal Floresans sergi ve sadece ek-etiket10bağlama üzerine güçlü bir floresan sinyal yayan başlar.
Bu iletişim kuralı tek hücrelere ek etiketini kullanarak ilgi farklı protein yıkımı oranları ölçmek nasıl açıklar. Burada E-cadherin kültür fare embriyonik kök (ES) hücrelere bu yöntemi uygulamak, ama herhangi bir yapisan kültürlü hücre türü ile kullanmak mümkün olmalıdır. Biz ek-etiket füzyon protein Floresans hızlandırılmış görüntüleme tarafından takip darbe etiketleme tek hücre yarı-hayat ilgi çeşitli proteinlerin belirlemek için izin verir ve yarı-hayatımızda hücre hücre çeşitliliği için bir tahmin sağlar gösterir bir nüfus kültürlü hücre.
Access restricted. Please log in or start a trial to view this content.
Protokol
Not: Bu çalışmada, E14 ES hücre kültürünü kullanıldı. Ancak, bu iletişim kuralını doğrudan diğer fare ES hücre kültürünü bir protein endojen protein etiketleme veya overexpression kullanarak bir ek etiketi için erimiş ilgi ifade etmek için geçerlidir. Sonuçlar bölümünde gösterilen örnekler için Doksisiklin indüklenebilir ek-etiket füzyon hücre satırları kullanılmıştır (ek-etiket için aşağıdaki proteinler erimiş: MicroRNAs, Oct4, Srsf11, veya floresan proteinler mOrange2 ve sfGFP ve kontrolü altında koymak için bir Doksisiklin indüklenebilir organizatörü. Daha fazla bilgi için11 Doksisiklin indüklenebilir ek-etiket füzyon hücre satırları oluşturulmasında kullanılan plazmid bakınız). Sıkı zamanlama ve protein ilgi ifade yoğunluğunu kontrol için izin verdiği Doksisiklin indüklenebilir sistemi özellikle yararlı olabilir. C-terminal ek-etiket-in konumlandırma tavsiye edilir, N-terminal değiştirme olarak amino asit dizisi half-life hedef protein (N-son kural12) değiştirmek daha yüksektir.
1. E-cadherin kaplama ve hücre tohumlama
- Bir protein için ek-tag4erimiş ilgi ifade bir ES hücre satırı oluşturmak.
- Kat 100 mm hücre kültür yemekleri (fosfat tamponlu tuz (PBS) olmadan Ca2 +/Mg2 +içinde seyreltilmiş) % 0,1 jelatin 1 h. için 4 mL ile jelatin kaldırmak ve hemen 1 h. kullanım kurumaya bırakın veya kaplamalı yemekleri 2 aya kadar saklayabilirsiniz.
- Hücre kültürünü ES hücre kültür orta (Glasgow en az temel takıma % 10 ES hücre nitelikli fetal sığır serum, 2 mM sodyum pyruvate, %1 non-gerekli amino asitler, % 1 penisilin/streptomisin, 2 mM L-glutamin, 100 µM 2 - Orta, ilgi büyümek mercaptoethanol, lösemi inhibitör faktörü (LIF), 3 µM CHIR99021 ve 0.8 µM PD184352) jelatin kaplı yemekleri yemek başına 10-20 Mio hücre bir izdiham ulaşan kadar 2 gün 37 ° C ve % 5 CO2 .
Not: Burada, LIF süpernatant toplama tarafından takip ve filtreleme HEK 293T hücreleri, geçici transfection tarafından üretildi. LIF her toplu iş iş potansiyelini pluripotency korumak test edildi, ama konsantrasyonlarının tespit. Ancak, rekombinant LIF da piyasada bulunan ve sık kullanılan fare ES hücre kültürü için konsantrasyon 1000 adet/mL13. - 96-şey plaka başına iyi (5 ng/PBS içinde Ca2 +/Mg2 +ile seyreltilmiş µL,. E-cadherin Fc chimera protein (E-cad-Fc) rekombinant fare 30 µL ile görüntüleme için uygun ceket Hisse senedi-80 ° C'de depolanan 100 ng/µL konsantrasyonları kullanın.) E-cad-Fc çok kırılgan olduğu gibi geniş pipetting, kaçının. 1,5 saat için 37 ° C'de kuluçkaya.
Not: mikroskop bağlı olarak, diğer formatlar kullanılabilir olur. - E-cad-Fc Aspire edin, bir kez PBS 100 µL Ca2 +/Mg2 +ile yıkayın ve Önceden ısıtılmış ES hücre kültür ortamının 100 µL ekleyin.
- PBS 5 mL olmadan Ca2 +/Mg2 +faiz hücrelerle yıkayın. Aspire edin ve tripsin-EDTA 2 mL % 0.25 ekleyin. 4 dk. 37 ° C'de için kuluçkaya ES hücre kültür orta 4 mL ekleyin, resuspend ve 1000 x g 4 dk. aspiratı süpernatant de aşağı spin ve Pelet 2 mL taze ES hücre kültür orta resuspend.
Not: PBS olmadan Ca2 +/Mg2 + trypsinization önce hücreleri yıkama için hücre adezyon için gerekli olan Ca2 + iyonları, kaldırmak için kullanılır. Buna ek olarak, E-cad-Fc seyreltme ve kaplama (1.4. adım) kullanmak için PBS Ca2 +/Mg2 +ile E-cad-Fc hücrelere ES yapışma beri kaplamalı yemekleri olduğunu Ca2 +-bağımlı14. - Resuspended hücreleri 1:10 ES hücre kültür orta ve sayım odası (0,1 mm derinlik TMMOB için yük 10 µL) kullanan sayısı, 1 ml seyreltik.
- Tohum 30.000 hücre/cm2 ' E-cad-Fc görüntüleme plaka kaplı. İyi ücret 200 µL ES hücre kültür orta ile doldurun. Doksisiklin indüklenebilir hücre hatlarında Doksisiklin uygun bir doz ile teşvik (doz ve zamanlama indüksiyon önceden optimize edin. Burada kullanılan hücre hatları ile 500 ng/mL Doksisiklin görüntüleme önce 24 saat tedavi edildi). 37 ° C ve % 5 CO2 kuluçkaya ve etiketleme ve hücre tohum sonra 24 h Imaging darbe ile devam edin.
2. nabız ek-etiket etiketleme
Not: protein çürüme deneyler için bu yeterli bir ek boya toplama kullanmak çok önemlidir. Konsantrasyonu hızlandırılmış, başında parlak bir sinyal vermeye yüksek olmalıdır Floresans zamanla azalacak gibi. Ancak, çok yüksek boya konsantrasyonları kullanarak kalan boya orta veya hücreleri yıkamadan sonra bile bırakılmak neden olabilir. Ücretsiz boya daha sonra çürüme eğri tahrif edecek film boyunca yeni üretilen ek-etiket moleküllerine bağlamak. Gözlenen Floresans sinyal boya, kullanılan hücre hattı gibi ifade düzeyi karşılık gelen protein özellikleri üzerinde bağlıdır. Bu nedenle, farklı dilutions, canlı hücre görüntüleme için üretici tarafından önerilen seyreltme itibaren test ederek boya toplama en iyi duruma getirmek çok önemlidir. Bu çalışma için bir far-red floresan substrat kullanıldı. En iyi bir konsantrasyon 12 nM Doksisiklin indüklenebilir overexpression hücre hatlarında kararlıydım.
- Önceden ısıtılmış ES hücre kültürü ortamında uygun toplama için ek boya sulandırmak. Hafifçe daha önce 96-şey plaka numaralı seribaşı hücrelerden orta kaldırmak için bir pipet kullanın ve seyreltilmiş ek boya iyi başına 50 µL ekleyin. 37 ° C'de 30 dk için kuluçkaya
- Bir pipet ile seyreltilmiş boya Aspire edin ve 3 x 200 µL (olmadan Ca2 +/Mg2 +) Önceden ısıtılmış PBS ile yıkayın. ES hücre kültür ortamının 200 µL ekleyin ve 37 ° C'de 15 dakika kuluçkaya
- Önceki çamaşır adımları iki kez daha tekrarlayın.
Not: Geniş çamaşır ilişkisiz herhangi bir boya kaldırmak için önemlidir. 15 dk incubations görüntüleme deney başlamadan önce ek-etiket füzyon proteinlerin sağlam etiketleme sağlamak ise PBS tekrarlanan çamaşır adımlarla ortamda, kalan ekstraselüler boya kaldırın. Ancak, nazik pipetting tarafından çamaşır adımlarını gerçekleştirin ve E-cad-Fc seribaşı hücreleri kolayca ayırmak eğilimi olarak bir otomatik aspiratör kullanmayın. - Orta görüntüleme 200 µL ekleyin. Floresan arka plan azaltmak, fenol red ücretsiz orta kullanın (takıma % 10 ES hücre nitelikli fetal sığır serum, 2 mM sodyum pyruvate, %1 non-gerekli amino asitler, % 1 penisilin/streptomisin, 2 mM L-glutamin, 100 µM 2-mercaptoethanol, LIF, 3 µM CHIR99021 ve 0.8 µM PD184352).
3. hızlandırılmış mikroskobu
- Mikroskop bırakmak ısı kontrollü ve CO2canlı görüntüleme için uygun kullanın. Sıcaklığı 37 ° C, CO%2 5'için kullanın ve 1-2 h için equilibrate için izin vermek için önceden ayarlayın.
- Plaka mikroskop yer ve noktalar görüntüleme için uygun bulmak. Nerede bu analiz kolaylaştıracaktır gibi hücreler eşit olarak dağıtılmış ama çok yoğun, noktalar seçin. Analiz için gerekli hücre sayısı için seçili noktaların sayısını ayarlayın. Bu çalışmada, 3-5 noktalar hücre satır başına kaydedildi.
- Hedefi seçin. 20 X amaç ES hücre boyutu için uygundur.
- Kaydedilecek floresan kanal (lar) aydınlatma ayarlarını seçin. Lazer güç ve ilgi hücrelerden Floresans sinyalin yoğunluğunu göre pozlama ayarlamak. O film boyunca azaltmak ama lazer güçler fototoksisite ve photobleaching en aza indirmek için 30 daha yüksek önlemek bu yana güçlü bir ilk sinyal aldığınızdan emin olun. Bu çalışmada, bir uyarma filtre 632/22 için nm (dalga boyu/bant), emisyon filtre 679/34 nm (dalga boyu/bant), lazer güç % 10 ve pozlama süresi 100 MS kullanılmıştır.
- Görüntüleme aralıkları ve hızlandırılmış deneme süresi seçin. Beklenen yarı-ömrü 2-20 h olan proteinler için 12-24 h edinme zamanlarında 15 dk zaman aralıkları ile seçin. Shorter-Lived proteinler için aralıkları ve longer-lived proteinler artış satın alma saatleri buna göre azaltın.
- Görüntüleme başlatın.
4. görüntü işleme ve analiz
- Alınan görüntüleri TIFF biçiminde bir yığın olarak toplamak. Daha fazla işleme ve analiz için FIJI yazılım15kullanın. Hızlandırılmış veri yığını olarak mikroskop bilgisayar yazılımı tarafından toplanan değil, tüm çerçeveleri hızlandırılmış deneme yazılımı açın ( dosya tıklayarak tarafından | Açık... veya sürükle ve araç çubuğu'na karşılık gelen dosyaları atılan) tıklatın ve resmi | Yığınlar | Görüntü yığını.
- Yığındaki tüm resimlerden arka plan çıkarmak için çıkarma arka plan işlevini kullanın. Bunu yapmak için işlemi ' ı tıklatın | Arka plan çıkarma. Çalışırken topu RADIUS açılır penceresinde seçin. En azından görüntüsü hücreleri büyüklüğünde bir RADIUS kullanın. Evet , arka plan çıkarma yığındaki tüm hücrelere uygulamak için seçin işlem yığın? açılır pencere.
- İlgi bir hücre seçin ve faiz (ROI) etrafında araç çubuğunu kullanarak bir bölgenin çizin. Nükleer sinyalleri için Oval seçim ilk araç çubuğunda karşılık gelen sekmeyi tıklayarak ve sonra faiz çekirdeği üzerinde tıklayarak ve oval etrafında ayarlayarak kullanın. Sitoplazmik sinyalleri veya çok yoğun bölgeler için serbest seçim hücre özetliyor yakından takip edebilmek için kullanın. Tüm arka plan pikseller (Adım 4,8-4,9) sonraki çıkarma nedeniyle tam olarak hücre anahatları takip için gerekli olmadığı halde çok büyük arka plan, bölgelerinde eklemekten kaçının.
- Analiz tıklatarak ROI Yöneticisi faiz bölgesine eklemek | Araçlar | ROI Yöneticisi | Eklemek, veya T klavye tuşlarına basarak.
- Sonraki kareye yığın penceresinde sekmesini taşıyarak veya üst karakter tuşuna basarak devam + < klavye, sonra önceki eylemlerin yineleyin. Hücre film seyri boyunca ilgi izleyin ve yatırım Getirisi her çerçevenin ROI Yöneticisi'ne ekleyin. Son ROI izlenen her hücre için daha fazla tıklatarak ayarlayın kaydetmek | Kaydet... ROI Yöneticisi'nde.
Not: Ayrıca her iki kızı hücreleri hücre bölünmeler sonra izlemek ve onların entegre yoğunluklarda arka plan çıkarma sonra toplamı (protein seyreltme hücre bölünmesi sırasında hesaba 4.6-4,9 adımlara bakın). Her iki kızı hücreleri için yatırım Getirisi setleri kaydedin. - Bir kez faiz hücre film izlenir, ortalama yoğunluğu ve ROIs alan değerlerini elde etmek için, ölçü birimi,'ı tıklatın. Elde edilen değerler bir elektronik tablo programına kopyalama ve hücrenin ham entegre yoğunluk her zaman noktası aşağıdaki gibi hesaplar:

nerede cdemekortalama yoğunluğu vealanCkarşılık gelen yatırım Getirisi alan. - Yerel arka plan tahmin etmek için hücrenin her zaman dilimi için ilgi yakın bir yatırım Getirisi ekleyin. Herhangi bir hücresel Floresans sinyal eklemekten kaçının. Kabaca bir hücre boyutudur dairesel bir yatırım Getirisi kullanın ve taşıma veya buna göre gerekirse, örneğin komşu hücreleri burnunu sokarsan küçültmek. Elektronik tabloya ölçülen yoğunluk değerleri kopyalamak ve yatırım Getirisi bir arka plan yatırım Getirisi elde etmek için ayarla ayarla cep ile daha önce açıklandığı gibi devam edin.
- Entegre yoğunluk hücre için arka plan-düzeltilmiş değer elde etmek için önce her zaman noktası arka entegre yoğunluğunu hesaplamak:

arka plan sinyal ve alanC ortalama yoğunluğu nerede BG demek hücre çevreleyen ROI alanıdır. Her iki alanda aynı boyutta olmadığı sürece arka plan yatırım Getirisi, alanı kullanmayın. - Her zaman için hücrenin son arka plan düşülen entegre yoğunluk Hesapla:

- Tek hücre çürümesine eğrileri normalize etmek her zaman-nokta yoğunluk değeri ilk zaman noktası yoğunluk değeri tarafından bölün.
Not: Eğri uydurma (bkz. Adım 4.11) de her tek hücre veya nüfus ortalama gerçekleştirilebilir. Bir popülasyonun ortalama önyargıları farklı floresan yoğunluklarda hücreler arasında üzerinden önlemek için hesaplanır, bir normalleştirme gerek yoktur. Normalleştirme böylece her hücre son bozunma eğrisi için aynı ağırlık katkıda bulunur sağlar. Buna ek olarak, normalleştirme bağımsız olarak (bkz. Şekil 3A-3 C) onların mutlak Floresans yoğunluklarda tek hücre bozunmaları görselleştirmek yararlı olabilir. Ancak, eğer sadece tek hücreli yarı-hayat belirlenir ve verileri değil ortalama olarak, normalleştirme adım atlanabilir ve eğri uydurma doğrudan ham veriler üzerinde gerçekleştirilir. - Protein half-life tahmini için bir eğri uydurma aracını kullanın. Bu çalışmada, MATLAB kullanıcı arabiriminin Apps bölümünde varsayılan olarak bulunduğu araç kutusu 3.4.1 uygun MATLAB eğrisi kullanıldı. Floresan yoğunluğu ithalat ve saat değerleri Veri al sekmesini tıklatarak elektronik tablo içine MATLAB'araç Montaj eğrisi açın ve floresan çürüme veri ve zaman Puan X veri ve Y seçin veri sekmesini seçin Özel denklem eğri uydurma tab ve üstel bir çürüme denklemi girin:
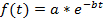
f(t) floresan yoğunluğu zaman-noktada verildi, olduğu yerde bir ilk yoğunluğu ve b çürüme oranı. Uygun seçenekleri... sekmesinde, hem a ve balt sınırı için 0 seçin. A ve b için tahmini değerleri daha sonra sonuçları penceresinde görünür. Yarı ömrü aşağıdaki gibi hesaplar:
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Açıklanan protokol tahmini hücre hücre değişkenlik half-life için bir ek etiketi için erimiş herhangi bir verilen protein sağlar. Aksi takdirde koloniler halinde yetişen ES hücreleri tek hücre çözümlenmek rekombinant E-cadherin-Fc kullanımı görüntü plaka kaplama için sağlar. Tek hücreler ayrı ayrı Film ( şekil 1A) ders boyunca izlenebilir.
Her tek hücre proteini half-life belirlemek için her iki kızı hücreleri entegre yoğunluklarda...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Protein çürüme izlemek için ek etiketi kullanan hiçbir kalıntı ilişkisiz boya orta veya hücreleri olarak aksi takdirde yıkama sonrası bu sol emin olmak için yeni bağlamak olduğunda en önemli adım ek-etiket molekülleri daha sonra sırasında deneme ve orada üretilen Münir uzlaşma bozunma eğrisi. Bir yandan dikkatle açıklanan çamaşır adımları uygulayarak elde bu. Öte yandan, boya konsantrasyon hala iyi bir sinyal-gürültü oranı elde etmek için izin verir bir aralıktaki olurken mümkün old...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Hızlandırılmış mikroskobu deneyler biyomoleküler eleme tesisi (BSF adlı), EPFL gerçekleştirilmiştir. Marc Delachaux (hizmet Audiovisuel, EPFL) videografisi ve film düzenleme için teşekkür ederiz.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
Referanslar
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745(2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156(2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15(2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır