Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Количественная оценка одноклеточных темпы деградации белка с покадровой флуоресцентной микроскопии в культуре адэрентных клеток
В этой статье
Резюме
Этот протокол описывает метод для определения белков полураспада в отдельные живые адэрентных клеток, используя импульсный маркировки и флуоресценции покадровой изображений SNAP-тег синтеза белков.
Аннотация
Белки находятся в динамическом состоянии синтеза и деградации и их половина жизнь может быть скорректирована при различных обстоятельствах. Однако, наиболее часто используемые подходы для определения белков half-life, либо ограничен населения средние от лизированных клетках или требовать использования ингибиторов синтеза белка. Этот протокол описывает метод для измерения белка полураспада в отдельные живые адэрентных клеток, используя SNAP-тег синтез белков в сочетании с покадровой микроскопии флуоресцирования. Любой протеин интереса, сливается с SNAP-тег можно ковалентно обязательность флуоресцентные, клетки проницаемых краситель, который соединен к производным benzylguanine, и распад помечены белка населения может контролироваться после вымывания остатков красителя. Последующие ячейки отслеживания и количественная оценка интенсивности флуоресценции комплексной времени результаты в кривой экспоненциального распада для каждой отслеживаемой ячейки, позволяя определить темпы деградации белка в одиночных клетках кривой. Этот метод обеспечивает оценку для гетерогенности полураспада в популяции культивируемых клеток, которые легко могут оцениваться другими методами. Представленный здесь подход применим к любому типу культивировали адэрентных клеток, выражая протеин интереса, сливается с SNAP-тег. Здесь мы используем клетки мыши эмбриональных стволовых (ES), выращенных на E-Кадгерины покрытием клетки культуры пластин для иллюстрации как одной ячейки деградации ставки белков с широким кругом половина-жизнь может быть определено.
Введение
Хорошо известно, клетчатые протеины проходят обширные оборот, с специфичны для каждого белка и подлежит физиологической регуляции синтеза и деградации ставки. Традиционно темпы деградации белка являются измеренные с помощью массовых такие методы, как анализ Чейз радиоактивных импульса, или с участием ингибиторы синтеза белка, такие как циклогексимида1. Совсем недавно стабильный изотоп маркировки с аминокислоты в клеточной культуре (SILAC) в сочетании с масс-спектрометрии была создана для количественного определения белка оборот на глобальном уровне2. Однако эти методы ограничены путем усреднения населения, и поэтому будут потеряны сведения об изменчивости к ячейке. Кроме того не могут быть определены временные изменения в деградации белков, которые являются несинхронизированные всей популяции клеток.
Кроме того белок полураспада также может определяться на основе флуоресценции подходы, которые часто имеют преимущество предоставления одной ячейки резолюции. Например photoactivatable Зеленый флуоресцентный белок (paGFP) был использован для определения Oct4 half-life в ранних млекопитающих эмбриона3. Другой метод для мониторинга распада белка в живых клетках является использование оснастки тега в сочетании с работой с образами замедленной флуоресценции. SNAP-тег — это мутант версия ДНК6ремонт фермента O - alkylguanine ДНК alkyltransferase (AGT), специально реагирует с benzylguanine (BG) производные финансовые инструменты, которые могут быть связаны с молекулярные датчики4,5, 6. Таким образом, любой протеин сплавливания SNAP-тегов могут необратимо помечены флуоресцентные, проницаемых краска клеток. Пульс маркировки протеина интереса, сливается с SNAP-тег, после вымывания остатков краски, позволяет для контроля за деградацией населения помечены белка и, таким образом, для определения белков half-life. SNAP-теги были успешно использованы для импульса Чейз маркировки белков и для определения белка в приверженца полураспада клеточной культуры и в естественных условиях5,,78,9. Большое разнообразие субстратов SNAP-тег охватывающей обычно используются люминесцентные спектры коммерчески доступных, позволяя выбор оптимального краситель для каждого конкретного приложения. Таким образом SNAP-Теги также может использоваться для многоцветного воображения в сочетании с другими флуоресцентные синтез белков или красителей. Ячейки непроницаемый красители подходят для маркировки привязал мембранных белков, тогда как клетки проницаемой красители применяются для мониторинга внутриклеточные и мембраны прыгните белков. Кроме того некоторые из этих датчиков демонстрируют почти не базальная флуоресценции и только начать, испуская флуоресцентные сигналом на привязку к SNAP-тег10.
Этот протокол описывает как измерять темпы деградации различных протеинов интереса в одиночных клетках, с помощью тега привязки. Здесь мы применяем этот метод к мыши эмбриональных стволовых (ES) клетки культивировали на E-Кадгерины, но она должна быть возможность использовать его с любым типом адэрентных культивируемых клеток. Мы показывают, что пульс маркировки SNAP-тег синтез белков, следуют флуоресценции покадровой изображений позволяет для определения одной ячейки полураспада различных протеинов интереса и дает оценку для ячейки к ячейке изменчивость полураспада в численность населения культивируемых клеток.
протокол
Примечание: В данном исследовании использовалась линия клетки E14 ES. Однако этот протокол является непосредственно применимо к любой другой мыши ES клеток линии выражения протеина интереса, сливается с SNAP-тег, либо путем пометки эндогенного белка, либо с помощью гиперэкспрессия. Примеры, приведенные в разделе результаты, доксициклин индуцибельной SNAP-тег синтеза клеточных линий использовались (SNAP-тег сливается на следующих белки: Nanog, Oct4, Srsf11, или sfGFP и mOrange2 флуоресцентных белков и поставить под контроль Доксициклин индуцибельной промоутер. Смотрите11 для получения дополнительной информации на плазмид, используется для генерации доксициклин индуцибельной SNAP-тег синтеза клеточных линий). Доксициклин индуцибельной система может быть особенно полезным, как он позволяет плотно контролировать сроки и интенсивности выражения протеина интереса. C-терминал позиционирования SNAP-тега рекомендуется, как изменение N-терминальный аминокислотной последовательности более вероятно изменить период полураспада целевого белка (N-end правило12).
1. E-Кадгерины покрытие и заполнения ячеек
- Создание линии клетки ES, выражая протеин интереса, сливается с SNAP-тегов4.
- Пальто 100 мм ячейку культуры блюда с 4 мл 0,1% желатина (разводят в фосфатный буфер (PBS) без Ca2 +/Mg2 +) за 1 ч. удалить желатина и высушить на 1 ч. использование сразу или хранить покрытием блюда на срок до 2 месяцев.
- Растут линии клетки интереса в среде культуры клеток ES (Глазго минимум основных средних, с 10% ES уточненное клеток плода бычьим сывороточным, пируват натрия 2 мм, 1% несущественные аминокислот, пенициллин/стрептомицина 1%, 2 мм L-глютамином, 100 мкм 2- меркаптоэтанол, лейкемия ингибирующего фактора (LIF), 3 мкм CHIR99021 и 0,8 мкм PD184352) на желатин покрытием блюд при 37 ° C и 5% CO2 за 2 дня до достижения слияния клеток 10-20 млн за блюдо.
Примечание: Здесь, LIF был продюсирован переходных transfection клеток 293T ГЭС, следуют супернатанта сбора и фильтрации. Каждый пакет LIF была протестирована на его потенциал для поддержания плюрипотентности, но не были определены концентрации. Однако рекомбинантных LIF также коммерчески доступных и широко используется для культуры клеток мыши ES концентрация составляет 1000 единиц/мл13. - Слой плиты 96-хорошо подходит для изображений с 30 мкл рекомбинантных мыши E-Кадгерины ФК Химера белка (E-cad-Fc) на колодец (5 нг/мкл, разбавляют в PBS Ca2 +/Mg2 +. Использование запасов концентрации 100 нг/мкл, температуре-80 ° C). Избегайте обширные закупорить, как E-cad ФК является очень хрупким. Инкубируйте при 37 ° C для 1,5 ч.
Примечание: В зависимости от Микроскоп, могут использоваться другие форматы. - Аспирационная E-cad ФК, раз промойте 100 мкл PBS с Ca2 +/Mg2 +и 100 мкл подогретым ES клеток питательной среды.
- Вымойте клетки интерес с 5 мл PBS без Ca2 +/Mg2 +. Аспирационная и добавить 0,25% 2 мл трипсина-ЭДТА. Инкубируйте 4 мин при 37 ° C. Добавьте 4 мл ES клеток питательной среды, Ресуспензируйте и спин вниз на 1000 x g 4 мин аспирата супернатант и Ресуспензируйте гранулы в 2 мл свежего ES клеток питательной среды.
Примечание: PBS без Ca2 +/Mg2 + должно использоваться для мытья клетки до trypsinization, чтобы удалить Ca2 + ионы, которые требуются для клеточной адгезии. В противоположность этому, для E-cad ФК разрежения и покрытие (шаг 1.4), используйте PBS с Ca2 +/Mg2 +, с адгезии клеток ES для E-cad ФК покрытием Посуда является Ca2 +-зависимых14. - Разбавьте 1:10 ресуспензированы клеток в 1 мл ES клеток питательной среды и счетчика, используя Счетной палаты (нагрузка 10 мкл для палаты глубина 0,1 мм).
- Семя 30000 клеток/см2 в E-cad ФК покрытием изображений пластины. Заполните до 200 мкл в колодец с ES клеток питательной среды. Для доксициклин индуцибельной клеточных линий, побудить с соответствующей дозой доксициклин (оптимизировать доза и время применения индукции заранее. Клеточных линий, используемых здесь лечились с 500 нг/мл доксициклин 24 ч до изображений). Инкубируйте на 37 ° C и 5% CO2 и продолжить с пульсом маркировки и изображений 24 ч после заполнения ячейки.
2. Пульс маркировки SNAP-тега
Примечание: Для экспериментов распад белков важно пользоваться надлежащей концентрации красителя оснастки. Концентрация должна быть достаточно высокой, чтобы дать яркий сигнал в начале промежуток времени, как флюоресценция будет уменьшаться с течением времени. Однако использование краска слишком высокой концентрации может привести к остаточной красителя остались в среде или в клетках даже после стирки. Бесплатные краситель может впоследствии привязку к недавно произведенные молекулы SNAP-тег на протяжении фильма, который будет искажать кривая распада. Наблюдаемые флуоресценции сигнал будет зависеть от свойства краситель, линии клеток, а также уровень экспрессии соответствующего белка. Таким образом крайне важно оптимизировать концентрации красителя, тестирование различных разведениях, начиная от разрежения, предложенная производителем для живых клеток. Для этого исследования была использована far-red флуоресцентные субстрата. Оптимальная концентрация 12 Нм был определен для доксициклин индуцибельной гиперэкспрессия клеточных линий.
- Разбавьте SNAP красителя соответствующей концентрации в среде культуры клеток подогретым ES. Использование пипетки аккуратно удалить носитель из клеток, ранее посеян в пластину 96-луночных и добавьте 50 мкл разбавленного SNAP красителя на хорошо. Инкубируйте 30 мин при температуре 37 ° C.
- Аспирационная разбавленной краски с пипеткой и мыть 3 x с 200 мкл подогретым PBS (без Ca2 +/Mg2 +). 200 мкл ES клеток питательной среды и Инкубируйте 15 мин при температуре 37 ° C.
- Повторите предыдущие шаги Стиральная вдвое больше.
Примечание: Обширные Стиральная имеет важное значение для того, чтобы удалить любые свободные красителя. Многократную стирку шаги с PBS удалить остаточные внеклеточного красителя в среде, тогда как 15 мин инкубаций обеспечить надежной маркировки SNAP-тег синтез белков до начала обработки изображений эксперимент. Однако выполните шаги Стиральная с нежным закупорить и не использовать автоматическое аспиратор, как клетки посеян на E-cad ФК, как правило, легко отсоединить. - 200 мкл изображений среднего. Чтобы уменьшить флуоресценции фона, использовать средство свободного фенола красного (дополнена 10% ES уточненное клеток плода бычьим сывороточным, пируват натрия 2 мм, 1% несущественные аминокислот, пенициллин/стрептомицина 1%, 2 мм L-глютамином, 100 мкм 2-меркаптоэтанол, LIF, 3 мкм CHIR99021 и 0,8 мкм PD184352).
3. промежуток времени микроскопия
- Используйте микроскопом подходит для живых изображений, позволяя контролируемой температуре и CO2. Установите температуру 37 ° C и CO2 до 5%, до использования и позволяют сбалансировать для 1-2 ч.
- Установите пластину в микроскоп и найти места, подходит для изображений. Выбор места, где клетки равномерно распределенные, но не слишком плотно, как это будет способствовать анализу. Отрегулируйте количество отдельных пятен на количество клеток, необходимых для анализа. В этом исследовании были записаны 3-5 точек на линии клеток.
- Выберите цель. 20 X цель подходит для размера ES клеток.
- Выберите настройки освещения для флуоресцентных каналы для записи. Отрегулируйте мощность лазера и воздействия по интенсивности флуоресценции сигнала от клеток интерес. Убедитесь в том получить сильный первоначальный сигнал, так как он будет уменьшаться в течение фильма, но избежать лазерной полномочий выше 30% для того, чтобы свести к минимуму Фототоксичность и Фотообесцвечивание. Для этого исследования, фильтр возбуждения 632/22 Нм (длина волны/bandpass), фильтр выбросов 679/34 Нм (длина волны/bandpass), мощность лазера 10% и выдержка времени 100 мс были использованы.
- Выбор изображений интервалы и продолжительность покадровой эксперимента. Для белков с ожидаемый период полураспада 2-20 ч выберите приобретение раз 12-24 ч с интервалами времени 15 мин. Для короче белков уменьшите интервалы и время приобретения, для долгоживущих белки увеличение соответственно.
- Начало обработки изображений.
4. обработка и анализ изображений
- Соберите полученные изображения как стек в формате tiff. Для дальнейшей обработки и анализа используйте программное обеспечение Фиджи15. Если промежуток времени данные не собираются как стек программного обеспечения Микроскоп, открыть все кадры покадровой эксперимента в программном обеспечении (либо нажав файл | Открыть... или перетащите и удаление соответствующих файлов на панели инструментов) и нажмите на изображение | Стеки | Изображения в стек.
- Используйте функцию вычитание фона для удаления фона из всех изображений в стеке. Чтобы сделать это, щелкните процесс | Вычитание фона. В всплывающем окне выберите радиусу качения шарика. Использование radius, по крайней мере это размер отображаемого клеток. Чтобы применить вычитание фона ко всем ячейкам в стеке, выберите Да в стека процесса? всплывающее окно.
- Выберите ячейку интерес и нарисуйте региона интерес (ROI) вокруг него с помощью панели инструментов. Для ядерной сигналов используйте Выбор овал , первый нажав на соответствующей закладке на панели инструментов и затем нажав на ядро интерес и регулируя овал вокруг него. Для цитоплазматических сигналов или очень плотный регионов использовать рукописные выбор чтобы иметь возможность внимательно следить за контуры клеток. Избегайте, включая слишком большие регионы происхождения, хотя это не обязательно следовать контуры клеток именно из-за последующего вычитания всех фон пикселей (шаг 4,8-4,9).
- Добавьте области интереса к диспетчеру ROI, нажав анализ | Инструменты | ROI менеджер | Добавить, или нажав T на клавиатуре.
- Перейти к следующему кадру, перемещая на вкладку в окне стека или нажав shift + < на клавиатуре, затем повторите предыдущие действия. Выполните ячейки интерес на протяжении фильма и добавьте ROI каждого кадра к диспетчеру ROI. Сохранить окончательный набор для каждой отслеживаемой ячейки, нажав больше ROI | Сохранить... в менеджере ROI.
Примечание: После клеточных делений, отслеживать обе дочери клетки отдельно и суммы их комплексной света после вычитание фона (см. шаги 4.6-4.9) для учета для растворения белков во время деления клеток. Сохраните рентабельность наборы для обоих дочерних клеток. - После того, как клетки интереса отслеживается на протяжении всего фильма, щелкните меру, чтобы получить значения для средней интенсивности и области ROIs. Скопируйте полученные значения в программу электронных таблиц и вычислить сырья комплексной интенсивность ячейки для каждой точки время следующим:

где означаетcявляется средняя интенсивность ирайонаCобласти соответствующего ROI. - Чтобы оценить местные фон, добавьте ROI близко к клетке интерес для каждого периода времени. Избегайте включения сигнала сотовой флуоресценции. Использовать круговые ROI, это примерно размер ячейки и перемещение или уменьшить его соответственно, при необходимости, например если соседние клетки вмешиваться. Действуйте, как описано ранее, с сотовых, заданные ROI, чтобы получить на фоне ROI и скопировать значения измерений интенсивности в электронную таблицу.
- Для получения фон исправлены значения для комплексного интенсивность ячейки, сначала Рассчитайте интенсивность комплексной фона для каждой точки время:

где означаетBG является средней интенсивности фонового сигнала и районC — это область ROI, обводить ячейки. Не используйте область фона ROI, если обе области имеют одинаковый размер. - Вычислите окончательного фон вычитается комплексной интенсивности ячейки для каждой точки времени:

- Нормализовать кривых распад одной ячейки, разделите значение интенсивности каждой точки времени значение интенсивности первый раз-точки.
Примечание: Кривой (см. шаг 4.11) может быть выполнено на каждую ячейку или на среднем населения. Если во избежание отклонений от интенсивности флуоресценции различных между ячейками вычисляется среднее население на основе нормализации не требуется. Нормализация таким образом гарантирует, что каждая ячейка вносит с такой же вес до окончательного распада кривой. Кроме того нормализации может быть полезным для визуализации одноклеточного распадов независимо от их абсолютной флюоресценция света (см. Показатели 3А-3 C). Однако если только одной ячейки полураспада определяются и данные не в среднем, нормализации шаг может быть опущен и кривой могут выполняться непосредственно на raw-данных. - Для оценки периода полураспада белка используйте средство установки кривой. В этом исследовании использовался MATLAB кривой элементов 3.4.1, который находится в разделе приложения MATLAB пользовательского интерфейса по умолчанию. Импортировать интенсивности флуоресценции и времени значения из электронной таблицы в MATLAB, нажав на вкладку Импорт данных откройте кривой панели инструментов и выберите моменты времени и флуоресценции распада данных в данные X и Y данные tab. Выберите Custom уравнение в кривой вкладку и введите уравнение для экспоненциального распада:
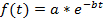
там, где f(t) является интенсивность флуоресценции в данный момент времени, первоначальный интенсивности и b скорость распада. В Параметры подходят... Вкладка, выберите 0 для нижнего предела и b. Расчетные значения для и b будут затем отображаются в окне результаты. Период полураспада Рассчитайте следующим образом:
Результаты
Описывается протокол дает оценку изменчивости к ячейке в период полураспада для любого данного белка, сливается с SNAP-тег. Использование рекомбинантных E-Кадгерины ФК для нанесения изображений плиты позволяет для одной ячейки резолюции ES клеток, которые в противном случае расти в колон...
Обсуждение
Самый важный шаг при использовании тега привязки для мониторинга распада белка обеспечить, что без остаточного несвязанных краска остается в среде или в клетках после мытья, поскольку в противном случае она может связывать недавно подготовила SNAP-тег молекул позднее в ходе эксперимент...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Покадровой микроскопии эксперименты были проведены на биомолекулярных скрининг фонда (ФСБ), EPFL. Мы благодарим Марк Delachaux (служба официально, EPFL) за видеосъемка и монтаж фильма.
Материалы
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
Ссылки
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745 (2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156 (2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15 (2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены