JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
부착 세포 배양에서 시간 경과 형광 현미경 검사 법에 의해 단백질 저하의 단일 셀 정량화
요약
이 프로토콜에서는 단백질 반감기 하나의 살아있는 부착 세포, 펄스 라벨 및 스냅인 태그 융해 단백질의 형광 시간 경과 이미징 사용 하 여 결정 하는 방법을 설명 합니다.
초록
단백질 합성의 동적 상태에 있고 저하 및 그들의 반감기는 다양 한 상황에서 조정 될 수 있다. 그러나, 가장 일반적으로 사용 되 접근 단백질 반감기는 결정 하기 위해 인구 평균 lysed 세포에서 제한 하거나 단백질 합성 억제제의 사용을 필요로. 이 프로토콜 단백질 반감기 하나의 살아있는 부착 세포에 형광 현미경 검사 시간 경과 함께에서 스냅인 태그 융해 단백질을 사용 하 여 측정 하는 방법을 설명 합니다. 스냅인 태그를 융합 하는 관심사의 어떤 단백질 covalently에 바인딩될 수 있는 결합 된 형광, 세포 투과성 염료에 의해 benzylguanine 파생, 그리고 잔여 염료의 세척 후 레이블이 단백질 인구의 감퇴를 모니터링할 수 있습니다. 이후 셀 추적 그리고 통합된 형광 강도 곡선 기준으로 단일 세포에서 단백질 저하 속도 결정 하기 위한 수 있도록 추적 된 각 셀에 대 한 지 수 감퇴 곡선에서 시간 결과 정량화. 이 메서드는 다른 방법으로 쉽게 평가 될 수 없는 경작된 한 세포의 인구에 반감기의이 대 한 견적을 제공 합니다. 여기에 제시 된 접근 스냅인 태그를 융합 하는 관심사의 단백질을 표현 하는 교양된 부착 세포의 모든 종류에 적용 됩니다. 여기 전자 cadherin 코팅 세포 배양 배지에서 자란 마우스 배아 줄기 (ES) 세포 사용 하 여 단백질의 반감기의 광범위 한 범위와 속도 확인할 수 있습니다 어떻게 단일 셀 저하를 설명 하기 위해.
서문
그것은 잘 알려진 세포질 단백질 합성 및 분해 속도 특정 각 단백질에 대 한 및 생리 적 규정이 적용 되 고 광범위 한 회전율, 받 다. 전통적으로, 단백질 저하 속도 측정 된 방사성 펄스 체이스 분석 같은 대량 방법을 사용 하 여 또는 cycloheximide1단백질 합성 억제제와 관련 된 되었습니다. 더 최근에, 안정 동위 원소 질량 분석 함께에서 세포 배양 (SILAC) 아미노산으로 세계적인 규모2단백질 회전율 계량에 설립 되었습니다. 그러나, 이러한 방법은 인구 평균에 의해 제한 되 고 셀 다양성에 대 한 정보 손실 따라서 됩니다. 또한, 세포 인구에서 동기화 되는 단백질 저하의 과도 변화를 확인할 수 수 없습니다.
또는, 단백질 반감기 또한 종종 단일 셀 해상도 제공 하는 이점을 있는 형광-기반 접근에 의해 결정 수 있습니다. 예를 들어 photoactivatable 녹색 형광 단백질 (paGFP) 초기 포유류 배아3Oct4 반감기를 결정 하기 위해 사용 되었습니다. 살아있는 세포에서 단백질 부패를 감시 하는 또 다른 방법은 형광 시간 경과 영상에와 함께 스냅인 태그의 사용 이다. 스냅-태그는 DNA 수리 효소 O6-alkylguanine를 DNA-alkyltransferase (AGT) 특별히 benzylguanine와 반응 분자 프로브4,5, 로 결합 될 수 있다 (BG) 파생 하의 돌연변이 체 버전 6. 따라서, 모든 스냅인 태그 융해 단백질 수 irreversibly 표시 형광, 세포 투과성 염료와. 펄스 잔여 염료의 희미하게 다음 스냅인 태그에 융합 하는 관심사의 단백질의 라벨 표시 단백질 인구의 저하를 모니터링 및 따라서 단백질 반감기를 결정 수 있습니다. 스냅-태그 펄스 체이스 단백질의 라벨에 대 한 성공적으로 사용 된 고 단백질을 결정 하기 위한 점착에 반감기 셀 문화 그리고 vivo에서5,7,,89. 일반적으로 사용 되 포함 하는 스냅인 태그 기판의 큰 다양 한 형광 스펙트럼 상업적으로, 각 특정 응용 프로그램에 대 한 최적의 염료의 선택 사용 사용할 수 있습니다. 따라서, 스냅-태그 다른 형광 성 융해 단백질 또는 염료와 함께 멀티 컬러 이미징에 대 한 또한 사용할 수 있습니다. 셀 스며들 지 않는 염료는 세포 투과성 염료는 세포내, 막 바인딩 단백질을 모니터링에 적용 하는 반면 단백질 막 곁의 라벨 적합 합니다. 또한, 일부 이러한 프로브 거의 없는 기저 형광을 전시 하 고만 스냅인 태그10바인딩 시 강한 형광 신호를 방출 시작 합니다.
이 프로토콜 스냅인 태그를 사용 하 여 단일 셀에 대 한 관심의 다른 단백질의 저하 속도 측정 하는 방법을 설명 합니다. 여기 우리가 마우스 배아 줄기 (ES) 세포 E cadherin에 교양된이 방법 적용 하지만 모든 부착 교양된 셀 종류 사용 가능 해야 한다. 우리 표시 펄스 라벨 형광 시간 경과 영상 다음 스냅인 태그 융해 단백질의 관심의 다양 한 단백질의 단일 셀 반감기를 결정 하기 위한 허용에 반감기의 셀 다양성에 대 한 견적을 제공 한다는 경작된 한 세포의 인구입니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
참고:이 연구에서 E14 ES 세포 라인 사용 되었다. 그러나,이 프로토콜 어떤 다른 마우스 ES 세포 라인 생 단백질을 태그 하거나 overexpression를 사용 하 여 스냅인 태그에 융합 하는 관심사의 단백질을 표현에 직접 적용 됩니다. Doxycycline 유도할 수 있는 스냅인 태그 퓨전 셀 라인 사용한 결과 섹션에 표시 된 예제에 대 한 (다음 단백질을 융합 하는 스냅-태그: Nanog, Oct4, Srsf11, 또는 형광 단백질 mOrange2 및 sfGFP, 및의 통제에 넣어 한 doxycycline 유도할 수 있는 발기인. 참조 대 한 자세한 내용은11 doxycycline 유도할 수 있는 스냅인 태그 퓨전 셀 라인의 생성에 사용 되는 플라스 미드에). Doxycycline 유도할 수 있는 시스템은 특히 유용할 수 있습니다, 그것은 밀접 하 게 제어 하는 타이밍 및 관심사의 단백질의 식의 강도 대 한 수로. 스냅-태그의 C-터미널 위치는 것이 좋습니다, 그리고 N 맨끝 변경 아미노산 시퀀스는 대상 단백질 (N-엔드 규칙12)의 반감기를 변경할 가능성이 더.
1. E-cadherin 코팅 및 셀 시드
- 스냅인 태그4를 융합 하는 관심사의 단백질을 표현 하는 ES 세포 라인을 생성 합니다.
- 코트 100 mm 셀 문화 요리 4 mL의 0.1% 젤라틴 (Ca2 +/Mg2 +없이 버퍼링 인산 염 분 (PBS)에 희석) 1 헤에 대 한 젤라틴을 제거 및 1 h. 사용에 대 한 건조 즉시 하거나 최대 2 개월까지 코팅된 접시를 저장.
- ES 세포 배양 매체 (글래스고 최소 필수 보통 10 %ES 세포 자격 태아 둔감 한 혈 청, 2mm 나트륨 pyruvate, 1% 비 본질적인 아미노산, 1% 페니실린/스, 2 mM L-글루타민, 100 µ M 2-보충의 셀 라인을 성장 mercaptoethanol, 백혈병 금지 요인 (LIF), 3 µ M CHIR99021 및 0.8 µ M PD184352) 젤라틴 코팅 요리 접시 당 10-20 미 오 셀의 합류를 도달할 때까지 2 일 동안 37 ° C, 5% CO2 .
참고: 여기, LIF HEK 293T 세포, 표면에 뜨는 수집 및 필터링의 과도 transfection에 의해 제작 되었다. LIF의 각 배치 pluripotency를 유지 하기 위해 그것의 잠재력에 대 한 테스트를 했다 하지만 농도 결정 하지 했다. 그러나, 재조합 형 LIF 또한 상업적으로 사용할 수 있으며 마우스 ES 세포 배양에 대 한 일반적으로 사용 되는 농도 1000 단위/mL13. - 96 잘 접시 재조합 마우스의 30 µ L로 잘 (5 ng / µ L, 캘리포니아2 +/Mg2 +PBS에 희석 당 전자 cadherin Fc 키메라 단백질 (E-cad-Fc) 이미징에 대 한 적합 한 코트. 사용 하 여 재고 농도 100 ng / µ L,-80 ° C에 저장). 전자 cad Fc은 매우 깨지기 쉬운 광범위 한 pipetting 하지 마십시오. 1.5 h 37 ° C에서 품 어.
참고: 현미경에 따라 다른 형식은 사용할 수 있습니다. - 전자 cad Fc를 발음 하 고 캘리포니아2 +/Mg2 +, PBS의 100 µ L로 한 번 씻어 미리 따뜻하게 ES 세포 배양 매체의 100 µ L를 추가 합니다.
- 없이 Ca2 +/Mg2 +PBS의 5 mL와 관심사의 세포를 씻어. 발음 하 고 0.25% Trypsin EDTA의 2 개 mL를 추가 합니다. 37 ° c.에 4 분 동안 품 어 ES 세포 배양 매체의 4 개 mL를 추가, resuspend 그리고 4 분 Aspirate는 상쾌한 1000 x g에서 스핀 다운 신선한 ES 세포 배양 매체의 2 mL에 펠 릿을 resuspend.
참고: 없이 Ca2 +/Mg2 + PBS 한다 사용할 수 세척 trypsinization 전에 세포에 세포 접착에 필요한 캘리포니아2 + 이온을 제거 하기 위해. 반면, 전자 cad Fc 희석 및 코팅 (1.4 단계), PBS를 사용 하 여 Ca2 +/Mg2 +, 전자 cad Fc를 ES 세포의 접착 이후 코팅된 요리는 캘리포니아2 +-종속14. - ES 세포 배양 매체 및 횟수, 세 실 (0.1 m m 깊이의 챔버에 대 한 부하 10 µ L)를 사용 하 여 1 ml resuspended 셀 1시 10분 희석.
- 전자 cad Fc에서 시드 30000 셀/cm2 이미징 플레이트 코팅. ES 세포 배양 매체에 잘 당 최대 200 µ L 채우십시오. Doxycycline 유도할 수 있는 셀 라인, doxycycline의 적절 한 복용량으로 유도 (복용량과 사전에 유도의 타이밍 최적화. 여기에 사용 되는 셀 라인 치료 했다 500 ng/mL doxycycline 이미징 하기 전에 24 시간으로). 37 ° C, 5% CO2 incubate 고 라벨 및 셀 시드 후 24 h를 이미징 펄스와 함께 진행 합니다.
2. 펄스 스냅인 태그 라벨
참고: 단백질 부패 실험에 대 한 그것은 적절 한 스냅 염료 농도 사용 하 여 중요 한입니다. 농도, 시간 경과의 시작 부분에 밝은 신호를 충분히 높은 해야로 형광 시간이 지남에 저하 됩니다. 그러나, 너무 높은 염료 농도 사용 하 여 세척 후에 매체에 또는 셀에 남아 잔류 염료를 발생할 수 있습니다. 무료 염료 수 이후에 바인딩할 새로 생산된 스냅인 태그 분자 감퇴 곡선 왜곡 됩니다 동영상의 과정을 통해. 관찰 된 형광 신호는 해당 단백질 식 수준 뿐만 아니라 염료, 사용, 셀 라인의 속성에 따라 달라 집니다. 따라서, 테스트 다른 희석, 희석 제조업체가 라이브 셀 이미징에 대 한 제안에서 시작 하 여 염료 농도 최적화 하기 위해 결정적 이다. 이 연구를 위해 빨강 형광 기판 사용 되었다. 12의 최적 농도 nM doxycycline 유도할 수 있는 overexpression 셀 라인에 대 한 결정 했다.
- 미리 데워 진된 ES 세포 배양 매체에 적절 한 농도에 스냅 염료 희석. 부드럽게 96 잘 접시에 시드 이전 세포에서 매체를 제거 하는 피 펫을 사용 하 고 잘 당 희석된 스냅 염료의 50 µ L를 추가 합니다. 37 ° c.에 30 분 동안 품 어
- 피 펫과 희석된 염료를 발음 하 고 미리 데워 공영 (없이 캘리포니아2 +/Mg2 +)의 200 µ L 3 x를 씻어. ES 세포 배양 매체의 200 µ L을 추가 하 고 37 ° c.에 15 분 동안 품 어
- 이전 세 단계를 두 번 더 반복 합니다.
참고: 광범위 한 세척은 어떤 언바운드 염료를 제거 하기 위해 중요 합니다. PBS 가진 반복된 세척 단계 15 분 외피 강력한 이미징 실험을 시작 하기 전에 스냅인 태그 융해 단백질의 라벨을 확인 하는 반면, 매체에서 잔여 extracellular 염료를 제거 합니다. 그러나, 부드러운 pipetting으로 세척 단계를 수행 하 고 셀 전자 cad Fc에 시드 쉽게 분리 하는 경향이 자동 흡 인기를 사용 하지 마십시오. - 영상 매체의 200 µ L를 추가 합니다. 줄이기 위해 형광 배경, 페 놀 레드 무료 매체를 사용 하 여 (보충 10 %ES 세포 자격 태아 둔감 한 혈 청, 2mm 나트륨 pyruvate, 1% 비 본질적인 아미노산, 1% 페니실린/스, 2 m m L-글루타민, 100 µ M 2-mercaptoethanol, LIF, 3 µ M CHIR99021 및 0.8 µ M PD184352).
3. 시간 경과 현미경
- 라이브 영상, 제어 온도 CO2를 위해 적당 한 현미경을 사용 합니다. 설정 온도 37 ° C, 5% CO2 를 사용 하 고 1-2 h에 대 한 equilibrate를 허용 하기 전에.
- 접시를 현미경에 놓고 이미징에 대 한 적합 한 장소를 찾을 합니다. 관광 명소 어디 세포는 균등 하 게 분산, 하지만 너무 조밀한,이 분석을 용이 하 게 합니다를 선택 합니다. 분석에 필요한 셀의 수 선택 된 관광 명소의 수를 조정 합니다. 이 연구에서는 셀 라인 당 3-5 관광 명소를 기록 했다.
- 목표를 선택 합니다. 20 X 목표는 ES 세포의 크기에 적합 합니다.
- 기록 형광 채널에 대 한 조명 설정을 선택 합니다. 레이저 파워 및 관심사의 세포에서 형광 신호 강도 따라 노출 조정 합니다. 하지만, 영화의 과정을 통해 감소, phototoxicity 및 photobleaching을 최소화 하기 위해 30% 보다 높은 레이저 능력을 방지 이후 강한 초기 신호를 얻을 수 있는지 확인 합니다. 이 연구, 632/22 여기 필터에 대 한 nm (파장/대역), 679/34의 방출 필터 nm (파장/대역), 100 ms의 10% 및 노출 시간의 레이저 파워를 사용 했다.
- 이미징 간격 및 시간 경과 실험 기간을 선택 합니다. 2-20 h의 예상된 반감기와 단백질에 대 일 분의 시간 간격으로 12-24 h의 수집 시간을 선택 합니다. Shorter-lived 단백질에 대 한 간격 및 수집 시간을 최소로 단백질 증가에 따라 줄일.
- 이미징 시작 합니다.
4. 이미지 처리 및 분석
- 스택 tiff 형식에서으로 얻은 이미지를 수집 합니다. 추가 처리 및 분석에 대 한 피지 소프트웨어15를 사용 합니다. 저속 데이터 현미경 소프트웨어 스택으로 수집 되지 않습니다, 소프트웨어에서 시간 경과 실험의 모든 프레임을 열고 ( 파일 을 클릭 하 여 | 오픈... 또는 드래그 앤 도구 모음에 해당 하는 파일) 이미지 클릭 | 스택 | 이미지 스택입니다.
- Subtract 배경 함수를 사용 하 여 스택의 모든 그림에서 배경을 제거 하. 이렇게 하려면 프로세스 | 배경 빼기. 팝업 창에서 롤링 볼 radius를 선택 합니다. 반지름은 적어도 몇 군데 셀의 크기를 사용 합니다. 배경 빼기 스택의 모든 셀에 적용 하려면 선택 예 에 프로세스 스택? 팝업 창.
- 관심의 셀을 선택 하 고 도구 모음을 사용 하 여 주위 (ROI) 관심 영역을 그립니다. 핵 신호에 대 한 첫 번째 도구 모음에서 해당 탭을 클릭 하 고 다음 클릭 하 여 관심의 핵 조정 주위 타원형 타원형 선택을 사용 합니다. 세포질 신호 또는 매우 조밀한 지역에 대 한 자유형 선택을 사용 하 여 밀접 하 게 셀 윤곽선을 따를 수 있을. 비록 모든 배경 픽셀 (단계 4.8-4.9)의 후속 빼기 때문에 정확 하 게 셀 윤곽선을 따라 하는 데 필요한 배경, 너무 큰 영역을 포함 하지 마십시오.
- 분석을 클릭 하 여 투자 수익 관리자에 관심 영역을 추가 | 도구 | 투자 수익 관리자 | 추가, 또는 키보드에서 T 를 눌러.
- 스택 창에서 탭을 이동 하 여 또는 shift 키를 누르면 다음 프레임으로 진행 + < 키보드, 다음 이전 작업을 반복. 동영상의 과정을 통해 관심의 셀 따라 고 각 프레임의 ROI ROI 관리자 추가. 최종 투자 수익 더 를 클릭 하 여 추적 된 각 셀에 대 한 설정 저장 | 저장...에 ROI 관리자.
참고: 셀 분할 후 두 딸 셀을 개별적으로 추적 하 고 배경 빼기 후 그들의 통합된 강렬을 합계 (단계 4.6 4.9 참조) 세포 분열 동안 단백질 희석에 대 한 계정. 두 딸 세포에 대 한 투자 수익 집합을 저장 합니다. - 일단 관심의 셀은 영화 내내 추적, 측정, 평균 강도와 ROIs의 영역에 대 한 값을 클릭 합니다. 전자 스프레드시트 프로그램으로 얻은 값을 복사 하 고 각 시간 지점에 대 한 셀의 원시 통합된 강도 다음과 같이 계산.

어디 c의미평균 강도 및지역C해당 투자 수익의 영역. - 로컬 백그라운드를 예상 하려면 각 시간 프레임에 대 한 관심의 셀에 가까운 수익을 추가 합니다. 어떤 세포 형광 신호를 포함 하지 마십시오. 셀의 크기는 대략 원형 ROI를 사용 하 여 이동 하거나 필요한 경우, 예를 들어 인접 셀 간섭 하는 경우 적절 하 게 축소 합니다. 투자 수익 배경 투자 수익을 얻으려면 설정 설정 휴대와 앞에서 설명한 대로 진행 하 고 스프레드시트에 측정된 강도 값을 복사 합니다.
- 셀의 통합 된 강도 대 한 배경 수정 값을 가져오려면 먼저 각 시간 지점에 대 한 백그라운드의 통합된 강도 계산:

배경 신호 및 지역C 의 평균 강도 BG 의미 셀 포위 망 투자 수익의 영역입니다. 두 영역 모두 동일한 크기를 가지 않는 한 ROI, 배경 영역을 사용 하지 마십시오. - 각 시간 지점에 대 한 셀의 마지막 배경 빼고 통합된 강도 계산:

- 정규화 단일 셀 감퇴 곡선, 첫째 시간 점의 강도 값으로 각 시간 점의 강도 값을 나눕니다.
참고: 커브 피팅 (단계 4.11 참조)도 수행할 수 있습니다 각 단일 셀에서 또는 인구 평균에. 기반 인구 평균 셀 사이의 다른 형광 강렬에서 편견을 피하기 위해 계산 하는 경우에 정규화가 필요 합니다. 정규화는 따라서 각 셀 마지막 감퇴 곡선을 같은 무게로 공헌 한다 다는 것을 보장 합니다. 또한, 정상화 ( 그림 3A-3 C참조) 그들의 절대 형광 강렬은 별도로 단일 셀 부패 한다를 시각화 하기 위해 유용할 수 있습니다. 그러나, 경우에 단일 셀 반감기를 결정 하 고 데이터 평균 하지, 정규화 단계를 생략할 수 있다 고 커브 피팅 원시 데이터에서 직접 수행할 수 있습니다. - 단백질 반감기 예측에 대 한 커브 피팅 도구를 사용 합니다. 이 연구의 MATLAB 사용자 인터페이스 애플 리 케이 션 섹션에서 기본적으로 위치한 MATLAB 커브 피팅 도구 상자 3.4.1가 사용 되었다. 형광 강도 가져오기 및 가져오기 데이터 탭에서 클릭 하 여 MATLAB에 전자 스프레드시트에서 시간 값 커브 피팅 도구 상자를 열고 선택한 시간 포인트 및 형광 붕괴 데이터 X 데이터 및 Y 데이터 탭 선택 사용자 정의 방정식 곡선 맞춤 탭을 입력 하는 지 수 감퇴의 공식:
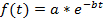
f (t) 주어진 시간 포인트, 형광 강도가 는 초기 강도 및 b 감퇴 속도. 맞는 옵션탭, 0 모두 a 와 b의 낮은 한도 대 한 선택. A 와 b 에 대 한 예상된 값 결과 창에 나타납니다. 다음과 같이 반감기를 계산.
Access restricted. Please log in or start a trial to view this content.
결과
설명된 프로토콜 스냅인 태그 융합 어떤 주어진된 단백질에 대 한 하프 라이프 셀 가변성의 견적을 제공 합니다. 이미징 판의 코팅에 대 한 재조합 형 전자 cadherin Fc를 사용 하 여 단일 셀 해상도 ES 세포는 식민지에서 성장에 대 한 수 있습니다. 단일 셀 영화 ( 그림 1A)의 과정을 통해 개별적으로 추적할 수 있습니다.
각 단일 셀에 대 한 단백질 반감기를 ?...
Access restricted. Please log in or start a trial to view this content.
토론
가장 중요 한 단계는 아니 잔여 언바운드 염료 남아 매체 또는 셀으로 세척 후 그것을 보장 하기 위해 수도 바인딩할 새로 단백질 부패 모니터링 스냅인 태그를 사용 하 여 생산 실험 및 거기 동안 나중 스냅인 태그 분자 eby 타협 감퇴 곡선입니다. 이것은 한 손으로 모든 설명된 세척 단계를 신중 하 게 수행 하 여 달성 에입니다. 다른 한편으로, 염료 농도 유지 되어야 한다, 가능한 한 낮은 여전히 ?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 없다.
감사의 말
시간 경과 현미경 실험에는 바이오 검사 시설 (BSF), EPFL을 수행 했다. 우리는 videography 및 영화 편집을 위한 마크 Delachaux (서비스 Audiovisuel, EPFL) 감사 합니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
참고문헌
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745(2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156(2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15(2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유