È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cella singola quantificazione dei tassi di degradazione delle proteine mediante microscopia a fluorescenza time-lapse in coltura delle cellule aderenti
In questo articolo
Riepilogo
Questo protocollo descrive un metodo per determinare la proteina emivite in singole cellule aderenti, utilizzando impulsi etichettatura e time-lapse imaging di fluorescenza di proteine di fusione SNAP-tag.
Abstract
Le proteine sono in uno stato dinamico di sintesi e degradazione e la loro emivita può essere regolata in varie circostanze. Tuttavia, la maggior parte comunemente utilizzati approcci per determinare l'emivita della proteina o sono limitate a medie della popolazione dalla cellule lisate o richiedono l'uso di inibitori della sintesi di proteina. Questo protocollo descrive un metodo per misurare la proteina emivite in singole cellule aderenti, con proteine di fusione SNAP-tag in combinazione con Time-lapse microscopia di fluorescenza. Qualsiasi proteina di interesse fusa ad un SNAP-tag possa legarsi covalentemente da una tintura di permeabili delle cellule fluorescente, che è accoppiata ad un derivato di benzylguanine, e il decadimento della popolazione proteina marcata può essere monitorato dopo interruzione della tintura residua. Successiva delle cellule di rilevamento e la quantificazione dell'intensità di fluorescenza integrata rispetto ai risultati di tempo in una curva di decadimento esponenziale per ogni cella cingolato, permettendo per determinare i tassi di degradazione delle proteine in cellule singole di adattamento della curva. Questo metodo fornisce una stima per l'eterogeneità delle emivite in una popolazione di cellule in coltura, che non può essere valutato facilmente con altri metodi. L'approccio qui presentato è applicabile a qualsiasi tipo di cellule aderenti coltivate che esprime una proteina di interesse fusa a un tag di SNAP. Qui usiamo cellule staminali embrionali (ES) topo coltivate su piastre per colture cellulari E-cadherin-rivestito per illustrare come singola cella degradazione tassi di proteine con una vasta gamma di dimezzamento possono essere determinati.
Introduzione
È noto che proteine cellulari subiscono ampio turnover, con tassi di sintesi e degradazione essendo specifico per ogni proteina e soggette a regolazione fisiologica. Tradizionalmente, tassi di degradazione delle proteine sono stati misurati utilizzando metodi di massa, quali l'analisi di chase impulso radioattivo, o che coinvolgono gli inibitori della sintesi di proteine quali cicloesimmide1. Più recentemente, isotopo stabile etichettatura con gli amminoacidi nella coltura delle cellule (SILAC) in combinazione con la spettrometria di massa è stato istituito per quantificare il turnover delle proteine su una scala globale2. Tuttavia, questi metodi sono limitati da una media di popolazione e informazioni sulla variabilità della cellula--cellula sono quindi persi. Inoltre, i cambiamenti transitori nella degradazione delle proteine che non sono sincronizzati in tutta la popolazione delle cellule non possono essere identificati.
In alternativa, emivite di proteina possono essere determinate anche da approcci basati sulla fluorescenza, che spesso hanno il vantaggio di fornire la risoluzione di singole cellule. Ad esempio, una proteina fluorescente verde fuse (paGFP) è stata utilizzata per determinare Oct4 emivita in embrione dei mammiferi primi3. Un altro metodo per monitorare il decadimento della proteina in cellule viventi è l'uso di un tag di SNAP in combinazione con Time-lapse imaging di fluorescenza. Il tag di SNAP è una versione mutante del DNA riparazione enzima O6- alchilguanina DNA-alchiltransferase (AGT) che reagisce specificamente con benzylguanine derivati (BG), che possono essere accoppiati a sonde molecolari4,5, 6. di conseguenza, qualsiasi proteina di fusione SNAP-tag può essere irreversibilmente etichettata con un colorante fluorescente, di permeabili delle cellule. Impulso di etichettatura di una proteina di interesse fusa per il SNAP-tag, seguito da interruzione della tintura residua, permette per il monitoraggio del degrado della popolazione proteina marcata e così per determinare il tempo di dimezzamento della proteina. SNAP-tag sono stati utilizzati con successo per impulso-insegua etichettatura delle proteine e per la determinazione della proteina emivite in aderente delle cellule di cultura ed in vivo5,7,8,9. Una grande varietà di substrati SNAP-tag che coprono comunemente usato fluorescente spectra è disponibili in commercio, consentendo la selezione della tintura ottima per ogni specifica applicazione. Così, SNAP-tag possono essere utilizzati anche per l'imaging multicolore in combinazione con altre proteine di fusione fluorescenti o coloranti. Cella-impermeabile coloranti sono adatti per l'etichettatura delle proteine di membrana-legato, mentre cellula-permeabile coloranti sono applicabili per il monitoraggio di proteine intracellulari e di membrana-limitano. Inoltre, alcune di queste sonde non presentano quasi nessuna fluorescenza basale e solo avviare l'emissione di un segnale fluorescente forte legandosi ad un SNAP-tag10.
Questo protocollo viene descritto come misurare i tassi di degradazione delle proteine differenti di interesse in singole cellule utilizzando un tag di SNAP. Qui abbiamo applicare questo metodo a cellule staminali embrionali (ES) topo coltivate su E-caderina, ma dovrebbe essere possibile usarlo con qualsiasi tipo di cellule in coltura aderente. Indichiamo che impulso etichettatura delle proteine di fusione SNAP-tag seguite da formazione immagine di fluorescenza time-lapse consente di determinare le emivite unicellulare di varie proteine di interesse e fornisce una stima della variabilità della cellula--cellula di emivite in un popolazione di cellule coltivate.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Nota: In questo studio, è stata utilizzata la linea cellulare E14 ES. Tuttavia, questo protocollo è direttamente applicabile a qualsiasi altra linea di mouse ES cellula che esprime una proteina di interesse fusa a un tag di SNAP, codifica la proteina endogena oppure mediante sovraespressione. Per gli esempi riportati nella sezione risultati, linee cellulari di doxiciclina-inducible SNAP-tag fusione sono stati usati (SNAP-tag fusa per le seguenti proteine: Nanog, Oct4, Srsf11, o per le proteine fluorescenti mOrange2 e sfGFP e messo sotto il controllo di un doxiciclina-inducible promotore. Vedi11 per ulteriori informazioni sui plasmidi utilizzati per la generazione di doxiciclina-inducible linee cellulari fusione SNAP-tag). Il sistema doxiciclina-viscoelastico può essere particolarmente utile, in quanto consente di controllare strettamente la tempistica e l'intensità dell'espressione della proteina di interesse. C-terminale di posizionamento del tag SNAP è consigliato, come cambiare il N-terminale sequenza dell'amminoacido è più probabile che altera l'emivita della proteina (N-fine regola12).
1. E-caderina rivestimento e semina delle cellule
- Generare una linea di cellule ES che esprime una proteina di interesse fusa per il SNAP-tag4.
- Cappotto 100 mm piastre di coltura cellulare con 4 mL di 0,1% di gelatina (diluito in tampone fosfato salino (PBS) senza Ca2 +/Mg2 +) per 1 h. rimuovere gelatina e lasciare asciugare per 1 h. uso immediatamente o conservare i piatti rivestiti per fino a 2 mesi.
- Crescere la linea cellulare di interesse nel mezzo di coltura cellulare ES (Glasgow minimo essenziale Medium, supplementato con 10% ES cella-qualificato siero bovino fetale, piruvato di sodio di 2 mM, gli aminoacidi non essenziali di 1%, 1% di penicillina/streptomicina, 2mm L-glutamina, 100 µM 2- mercaptoetanolo, fattore inibitorio di leucemia (LIF), 3 µM CHIR99021 e 0,8 µM PD184352) rivestite con gelatina piatti a 37 ° C e 5% di CO2 per 2 giorni fino a raggiungere una confluenza delle cellule Mio 10-20 per ogni piatto.
Nota: Qui, LIF è stato prodotto da transitori di transfezione delle cellule HEK 293T, seguita da surnatante raccolta e filtraggio. Ogni lotto di LIF è stato testato per il suo potenziale mantenere la pluripotenza, ma non sono state determinate concentrazioni. Tuttavia, ricombinante LIF è anche disponibile in commercio e la concentrazione comunemente usata per la coltura delle cellule di topo ES è 1000 unità/mL13. - Ricoprire una piastra a 96 pozzetti adatta per imaging con 30 µ l di mouse recombinant della proteina chimera di E-caderina Fc (E-cad-Fc) per pozzetto (5 ng / µ l, diluito in PBS con Ca2 +/Mg2 +. Utilizzare concentrazioni stock di 100 ng / µ l, conservato a-80 ° C). Evitare di pipettaggio estesa, come E-cad-Fc è molto fragile. Incubare a 37 ° C per 1,5 ore.
Nota: A seconda del microscopio, altri formati potrebbero essere utilizzati. - Aspirare E-cad-Fc, lavare una volta con 100 µ l di PBS con Ca2 +/Mg2 +e aggiungere 100 µ l di terreno di coltura delle cellule ES pre-riscaldato.
- Lavare le cellule di interesse con 5 mL di PBS senza Ca2 +/Mg2 +. Aspirare e aggiungere 2 mL di tripsina-EDTA 0,25%. Incubare per 4 min a 37 ° C. Aggiungere 4 mL di terreno di coltura delle cellule di ES, risospendere e spin giù a 1000 x g per 4 min. aspirare il supernatante e risospendere il pellet in 2 mL di terreno di coltura di cellule ES nuovo.
Nota: PBS senza Ca2 +/Mg2 + dovrebbe essere usato per lavare le cellule prima di trypsinization al fine di rimuovere ioni Ca2 + , che sono necessari per l'adesione delle cellule. Al contrario, per la diluizione E-cad-Fc e rivestimento (punto 1.4), è possibile utilizzare PBS con Ca2 +/Mg2 +, poiché l'adesione delle cellule di ES per E-cad-Fc piatti rivestiti è Ca2 +-dipendente14. - Diluire il sedimento celle 01:10 in 1 mL di terreno di coltura delle cellule di ES e conta, utilizzando una camera di conteggio (carico 10 µ l per una camera di 0,1 mm di profondità).
- Seme 30.000 cellule/cm2 a E-cad-Fc rivestite in lamiera di imaging. Riempire fino a 200 µ l per pozzetto con mezzo di coltura cellulare ES. Per linee cellulari doxiciclina-inducibile, indurre con una dose adeguata di doxiciclina (ottimizzare la dose e la tempistica dell'induzione in anticipo. Le linee cellulari utilizzate qui sono state trattate con doxiciclina di 500 ng/mL 24 h prima di imaging). Incubare a 37 ° C e 5% CO2 e procedere con l'impulso di etichettatura e di imaging 24 h dopo la semina delle cellule.
2. etichettatura del SNAP-tag di impulso
Nota: Per gli esperimenti di decadimento di proteina è fondamentale utilizzare un'adeguata concentrazione di tintura SNAP. La concentrazione deve essere sufficientemente elevata per produrre un segnale luminoso all'inizio di time-lapse, come la fluorescenza si riduce nel tempo. Tuttavia, utilizzando concentrazioni di tintura troppo elevato potrebbe causare tintura residua lasciata nel terreno o nelle cellule anche dopo il lavaggio. Il colorante libero potrebbe associare successivamente recentemente prodotti SNAP-tag molecole nel corso del film, che distorcono la curva di decadimento. Il segnale di fluorescenza osservata dipenderanno le proprietà della tintura, la linea cellulare utilizzata, nonché il livello di espressione della proteina corrispondente. Di conseguenza, è fondamentale ottimizzare la concentrazione di colorante testando diluizioni diverse, a partire dalla diluizione suggerita per l'imaging cellulare diretta dal produttore. Per questo studio, è stato utilizzato un substrato fluorescente da'. Una concentrazione ottimale di 12 nM è stato determinato per linee cellulari sovraespressione doxiciclina-inducibile.
- Diluire la tintura SNAP alla concentrazione appropriata nel terreno di coltura delle cellule ES pre-riscaldato. Usare una pipetta per rimuovere delicatamente il mezzo dalle cellule precedentemente seminate nella piastra 96 pozzetti e aggiungere 50 µ l di colorante diluito di SNAP per pozzetto. Incubare per 30 min a 37 ° C.
- Il colorante diluito con una pipetta di aspirare e lavare 3 volte con 200 µ l di PBS preriscaldata (senza Ca2 +/Mg2 +). Aggiungere 200 µ l di terreno di coltura delle cellule di ES ed incubare per 15 min a 37 ° C.
- Ripetere i passaggi precedenti di lavaggio due volte di più.
Nota: Vasto lavaggio è importante al fine di rimuovere qualsiasi colorante non associato. I passaggi ripetuti lavaggi con PBS rimuovere il residuo dalla tintura extracellulare nel mezzo, considerando che le incubazioni di 15 min garantiscono etichettatura robusto delle proteine di fusione SNAP-tag prima di iniziare l'esperimento di formazione immagine. Tuttavia, eseguire la procedura di lavaggio pipettando delicato e non utilizzare un aspiratore automatico, in quanto le cellule seminate su E-cad-Fc tendono a staccarsi facilmente. - Aggiungere 200 µ l di medie di imaging. Per ridurre la fluorescenza di fondo, usare il mezzo libero rosso fenolo (supplementato con 10% ES cella-qualificato siero bovino fetale, piruvato di sodio di 2 mM, gli aminoacidi non essenziali di 1%, 1% di penicillina/streptomicina, 2mm L-glutamina, 100 µM 2-mercaptoetanolo, LIF, 3 µM CHIR99021 e 0,8 µM PD184352).
3. Time-lapse microscopia
- Utilizzare un microscopio per formazione immagine live, che permette di CO2e temperatura controllata. Impostare la temperatura a 37 ° C e la CO2 al 5% prima dell'uso e permettono di equilibrare per 1-2 h.
- Inserire la piastra al microscopio e trovare i punti adatti per l'imaging. Selezionare i punti dove le cellule sono distribuite uniformemente, ma non troppo densa, come questo faciliterà l'analisi. Regolare il numero di punti selezionati per il numero di cellule necessarie per l'analisi. In questo studio, sono state registrate 3-5 punti per ogni linea cellulare.
- Selezionare l'obiettivo. Un obiettivo X 20 è adatto per le dimensioni delle cellule di ES.
- Selezionare le impostazioni di illuminazione per i canali fluorescente per essere registrato. Regolare la potenza del laser e l'esposizione secondo l'intensità del segnale di fluorescenza da celle di interesse. Assicurati di ottenere un forte segnale iniziale, poiché sarà diminuire nel corso del film, ma evitare laser poteri superiori al 30% al fine di ridurre al minimo la fototossicità e photobleaching. Per questo studio, un filtro di eccitazione di 632/22 nm (lunghezza d'onda/passa-banda), filtro di emissione di 679/34 nm (lunghezza d'onda/passa-banda), potere del laser di 10% e tempo di esposizione di 100 ms sono stati usati.
- Selezionare gli intervalli di imaging e la durata dell'esperimento time-lapse. Per le proteine con emivite previste di 2-20 h, scegliere tempi di acquisizione di 12-24 h con intervalli di tempo di 15 min. Per le proteine studiano, ridurre gli intervalli e tempi di acquisizione, per aumento di proteine più longevo di conseguenza.
- Avviare imaging.
4. elaborazione ed analisi di immagini
- Raccogliere le immagini acquisite come una pila in formato tiff. Per l'ulteriore elaborazione e analisi, utilizzare il software di FIJI15. Se i dati di time-lapse non viene raccolta come una pila dal software microscopio, aprire tutti i fotogrammi dell'esperimento time-lapse del software (sia facendo clic su File | Apri... o trascinare e far cadere i file corrispondenti sulla barra degli strumenti) e fare clic su immagine | Stacks | Immagini di Stack.
- Utilizzare la funzione di sfondo Sottrai per rimuovere lo sfondo da tutte le foto nello stack. A tale scopo, fare clic su processo | Sottrarre il Background. Selezionare il raggio di rotolamento sfere nella finestra a comparsa. Utilizzare un raggio che è almeno la dimensione delle celle imaged. Per applicare la sottrazione di sfondo a tutte le celle dello stack, selezionare Sì nella stack processo? finestra pop-up.
- Selezionare una cella di interesse e disegnare un'area di interesse (ROI) intorno ad esso utilizzando la barra degli strumenti. Per segnali nucleari, è possibile utilizzare la selezione ovale primo facendo clic sulla scheda corrispondente nella barra degli strumenti e quindi facendo clic sul nucleo di interesse e modificando l'ovale intorno ad esso. Per segnali citoplasmatici o regioni molto dense utilizzare la selezione a mano libera per essere in grado di seguire da vicino i contorni delle cellule. Evitare di includere troppo ampie regioni dello sfondo, anche se non è necessario seguire i contorni delle cellule proprio a causa di successiva sottrazione di tutti i pixel dello sfondo (passo 4.8-4.9).
- Aggiungere la regione di interesse per il manager di ROI facendo Analyze | Strumenti | Manager di ROI | Aggiungi, o premendo T sulla tastiera.
- Procedere alla cornice successiva spostando la scheda nella finestra stack o premendo MAIUSC + < sulla tastiera, quindi ripetere le operazioni precedenti. Seguire la cella di interesse durante tutto il corso del film e aggiungere il ROI di ogni fotogramma al manager di ROI. Salvare il ROI finale impostata per ogni cella cingolato cliccando più | Salvare... nel manager di ROI.
Nota: Dopo le divisioni delle cellule, monitorare separatamente due cellule della figlia e sommare le relative intensità integrate dopo la sottrazione di sfondo (vedere i passaggi 4.6-4.9) per tenere conto per la diluizione della proteina durante la divisione cellulare. Salvare i set di ROI per due cellule della figlia. - Una volta che la cella di interesse viene tenuta traccia per tutto il film, fare clic su misura, per ottenere i valori di intensità media e l'area della ROIs. Copiare i valori ottenuti in un programma di foglio elettronico e calcolare l'intensità integrato prima della cella per ogni punto di tempo come segue:

dove direcè l'intensità media ezonaCla zona del ROI corrispondente. - Per stimare lo sfondo locale, aggiungere un ROI vicino alla cella di interesse per ogni intervallo di tempo. Evitare di includere qualsiasi segnale di fluorescenza cellulare. Utilizzare un ROI circolare che ha circa le dimensioni di una cella e spostare o ridurre di conseguenza se necessario, ad esempio se le cellule vicine interferiscano. Procedere come descritto in precedenza con il cellulare impostato al fine di ottenere uno sfondo ROI ROI impostare e copiare i valori di intensità misurata al foglio di calcolo.
- Per ottenere un valore corretto di sfondo per l'intensità integrata della cella, prima di calcolare l'intensità integrata dello sfondo per ogni punto di tempo:

dove direBG è l'intensità media del segnale di fondo e AreaC è l'area del ROI che circondano la cella. Non utilizzare l'area dello sfondo ROI, a meno che entrambe le aree hanno la stessa dimensione. - Calcolare l'intensità integrata sottratto sfondo finale della cella per ogni punto di tempo:

- Per normalizzare le curve di decadimento di singola cellula, dividere il valore di intensità di ogni tempo-punto per il valore di intensità del primo punto di tempo.
Nota: L'adattamento della curva (vedere il punto 4.11) possa essere eseguito su ogni singola cella o sulla media della popolazione. Una normalizzazione è richiesta se viene calcolata una media basati sulla popolazione al fine di evitare pregiudizi da intensità di fluorescenza diverse fra le cellule. La normalizzazione assicura quindi che ogni cella contribuisce con lo stesso peso alla curva di decadimento finale. Inoltre, la normalizzazione può essere utile per visualizzare i decadimenti di singola cellula indipendentemente dalla loro intensità di fluorescenza assoluta (Vedi Figure 3A-3C). Tuttavia, se solo la cella singola emivite sono determinate e i dati non sono una media, il passo di normalizzazione può essere omesso e l'adattamento delle curve può essere eseguita direttamente sui dati raw. - Per la stima della emivita della proteina, è necessario utilizzare uno strumento di montaggio di curva. In questo studio, la curva MATLAB toolbox 3.4.1 di montaggio è stata utilizzata, che si trova per impostazione predefinita nella sezione applicazioni dell'interfaccia utente MATLAB. Importare l'intensità di fluorescenza e i valori di ora da foglio di calcolo elettronico in MATLAB facendo clic sulla scheda Importa dati aprire la curva di raccordo della casella degli strumenti e selezionare i punti di tempo e dati di decadimento fluorescenza nei dati X e Y di dati Tab. Scegli Custom equazione nel raccordo curva scheda e immettere l'equazione per un decadimento esponenziale:
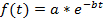
dove f (t) è l'intensità di fluorescenza in un dato punto di tempo, un l'intensità iniziale e b il tasso di decadimento. In Opzioni di adattamento..., selezionare 0 per il limite inferiore di un e b. I valori stimati per un e b apparirà nella finestra dei risultati. Calcolare il tempo di dimezzamento come segue:
Access restricted. Please log in or start a trial to view this content.
Risultati
Il protocollo descritto fornisce una stima della variabilità cellula--cellula in Half-Life per qualsiasi data proteina fusa a un tag di SNAP. L'uso di ricombinanti E-cadherin-Fc per il rivestimento della piastra imaging consente risoluzione unicellulare in cellule ES, che altrimenti crescere nelle colonie. Cellule singole possono essere monitorate separatamente durante tutto il corso del film ( Figura 1A).
Al fine di determinare il tempo di dimezzamento di protei...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il passaggio più importante quando si utilizza un tag di SNAP per monitorare il decadimento della proteina è per garantire che nessun residuo colorante non legato è lasciato nel terreno o nelle cellule dopo il lavaggio, altrimenti si potrebbe associare a recentemente prodotte molecole SNAP-tag più tardi nel corso dell'esperimento e ther Eby compromesso la curva di decadimento. Questo è un lato ottenuta eseguendo attentamente tutti i passaggi di lavaggio descritta. D'altra parte, la concentrazione di colorante dovreb...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Esperimenti di microscopia time-lapse sono stati eseguiti presso biomolecolari Screening Facility (BSF), EPFL. Ringraziamo Marc Delachaux (servizio Audiovisuel, EPFL) per la videografia e montaggio del film.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| ES cell line expressing a SNAP-tag fusion protein of interest | - | - | |
| Falcon 100 mm TC-Treated Cell Culture Dish | Corning | 353003 | |
| 96 Well, Black/Clear, Tissue Culture Treated Plate | Corning | 353219 | |
| Neubauer-improved counting chamber, 0.1 mm | Marienfeld-superior | 640030 | |
| CO2 Incubator | Panasonic | MCO-170AICUV-PE | |
| Centrifuge 5804 R | Eppendorf | 5804000528 | |
| InCell Analyzer 2200 Cell Imaging System | GE Healthcare Life Sciences | 29027886 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Glasgow Minimum Essential Medium | Sigma-Aldrich | G5154 | |
| Fetal Bovine Serum, embyonic stem cell-qualified | ThermoFisher | 16141-079 | |
| Sodium pyruvate solution | Sigma-Aldrich | 113-24-6 | |
| Minimum Essential Medium Non-Essential Amino Acids | ThermoFisher | 11140-035 | |
| Penicillin-Streptomycin | BioConcept | 4-01F00H | |
| L-Glutamine 200mM | ThermoFisher | 25030-024 | |
| 2-Mercaptoethanol | Sigma-Aldrich | 63689-25ML-F | |
| Leukemia Inhibitory factor | - | - | Produced in the lab by transient transfection of HEK-293T cells, followed by collection and filtering of the supernatant. |
| CHIR99021 (GSK-3 Inhibitor XVI) | Merck Millipore | 361559 | |
| PD 0325901 | Sigma-Aldrich | 391210-10-9 | |
| Gelatin from bovine skin | Sigma-Aldrich | 9000-70-8 | |
| Dulbecco's PBS 10x concentrated | BioConcept | 3-05K00-I | |
| Dulbecco's PBS Without Ca++/Mg++ | BioConcept | 3-05F29-I | |
| Trypsin-EDTA-Solution 0.25% | Sigma-Aldrich | T4049 | |
| Recombinant Mouse E-Cadherin Fc Chimera protein | R&D systems | 748-EC-050 | |
| Doxycycline hyclate | Sigma-Aldrich | D9891 | |
| SNAP-Cell 647-SiR | New England BioLabs | S9102S | |
| FluoroBrite DMEM | ThermoFisher | A18967-01 | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| FIJI | - | - | Open-source image analysis software |
| MATLAB R2014a | Mathworks | - | |
| Microsoft Excel | Microsoft | - |
Riferimenti
- Zhou, P. Determining Protein Half-Lives. Signal Transduct Protoc. , 67-77 (2004).
- Schwanhäusser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Plachta, N., Bollenbach, T., Pease, S., Fraser, S. E., Pantazis, P. Oct4 kinetics predict cell lineage patterning in the early mammalian embryo. Nature Cell Biol. 13 (2), 117-123 (2011).
- Keppler, A., Gendreizig, S., Gronemeyer, T., Pick, H., Vogel, H., Johnsson, K. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nature Biotechnol. 21 (1), 86-89 (2002).
- Keppler, A., Pick, H., Arrivoli, C., Vogel, H., Johnsson, K. Labeling of fusion proteins with synthetic fluorophores in live cells. Proc Nat Acad Sci U S A. 101 (27), 9955-9959 (2004).
- Gronemeyer, T., Chidley, C., Juillerat, A., Heinis, C., Johnsson, K. Directed evolution of O6-alkylguanine-DNA alkyltransferase for applications in protein labeling. Protein Eng Design Select. 19 (7), 309-316 (2006).
- Jansen, L. E. T., Black, B. E., Foltz, D. R., Cleveland, D. W. Propagation of centromeric chromatin requires exit from mitosis. J Cell Biol. 176 (6), 795-805 (2007).
- Bojkowska, K., et al. Measuring In Vivo Protein Half-Life. Chem Biol. 18 (6), 805-815 (2011).
- Mandic, A., Strebinger, D., Regali, C., Phillips, N. E., Suter, D. M. A novel method for quantitative measurements of gene expression in single living cells. Methods. 120, 65-75 (2017).
- Komatsu, T., et al. Real-Time Measurements of Protein Dynamics Using Fluorescence Activation-Coupled Protein Labeling Method. J Am Chem Soc. 133 (17), 6745(2011).
- Deluz, C., et al. A role for mitotic bookmarking of SOX2 in pluripotency and differentiation. Genes Dev. 30 (22), 2538-2550 (2016).
- Varshavsky, A. The N-end rule pathway and regulation by proteolysis. Protein Sci. 20 (8), 1298-1345 (2011).
- Tamm, C., Pijuan Galitó, S., Annerén, C. A Comparative Study of Protocols for Mouse Embryonic Stem Cell Culturing. PLoS ONE. 8 (12), e81156(2013).
- Nagaoka, M., et al. E-Cadherin-Coated Plates Maintain Pluripotent ES Cells without Colony Formation. PLoS ONE. 1 (1), e15(2006).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Meth. 9 (7), 676-682 (2012).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotech. 34 (7), 703-706 (2016).
- Blanchoud, S., Nicolas, D., Zoller, B., Tidin, O., Naef, F. CAST: An automated segmentation and tracking tool for the analysis of transcriptional kinetics from single-cell time-lapse recordings. Methods. 85, 3-11 (2015).
- Peng, T., et al. A BaSiC tool for background and shading correction of optical microscopy images. Nature Comm. 8, (2017).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nature Methods. 12 (5), 404-406 (2015).
- Chae, H. D., Lee, M. R., Broxmeyer, H. E. 5-Aminoimidazole-4-carboxyamide Ribonucleoside Induces G1/S Arrest and Nanog Downregulation via p53 and Enhances Erythroid Differentiation. Stem Cells. 30 (2), 140-149 (2012).
- Lin, Y., et al. Reciprocal Regulation of Akt and Oct4 Promotes the Self-Renewal and Survival of Embryonal Carcinoma Cells. Mol Cell. 48 (4), 627-640 (2012).
- Wei, F., Scholer, H. R., Atchison, M. L. Sumoylation of Oct4 Enhances Its Stability, DNA Binding, and Transactivation. J Biol Chem. 282 (29), 21551-21560 (2007).
- Gautier, A., et al. An Engineered Protein Tag for Multiprotein Labeling in Living Cells. Chem Biol. 15 (2), 128-136 (2008).
- Los, G. V., et al. HaloTag: A Novel Protein Labeling Technology for Cell Imaging and Protein Analysis. ACS Chem Biol. 3 (6), 373-382 (2008).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon