Method Article
أداة دراسة رابطة المسار لتحليل GWAS لمعلومات المسار الأيضي
In This Article
Erratum Notice
Summary
من خلال تشغيل أداة دراسة جمعية المسار (PAST) ، إما من خلال التطبيق اللامع أو من خلال وحدة التحكم R ، يمكن للباحثين اكتساب فهم أعمق للمعنى البيولوجي لنتائج دراسة الارتباط على نطاق الجينوم (GWAS) من خلال التحقيق في المسارات الأيضية المعنية.
Abstract
في الآونة الأخيرة ، تم تطوير وتنفيذ جديد لطريقة موصوفة سابقا لتفسير بيانات دراسة الارتباط على نطاق الجينوم (GWAS) باستخدام تحليل المسار الأيضي والإفراج عنه. وقد وضعت أداة دراسة رابطة المسارات لمعالجة الشواغل المتعلقة بسهولة الاستخدام والتحليلات البطيئة. وقد تم الافراج عن هذه الأداة الجديدة سهلة الاستخدام على الموصل الحيوي وGithub. في الاختبار، أجرت PAST تحليلات في أقل من ساعة واحدة كانت تتطلب في السابق أربع وعشرين ساعة أو أكثر. في هذه المقالة، نقدم البروتوكول لاستخدام التطبيق Shiny أو وحدة التحكم R لتشغيل الماضي.
Introduction
دراسات الارتباط على نطاق الجينوم (GWAS) هي طريقة شائعة لدراسة الصفات المعقدة والمناطق الجينومية المرتبطة بها1و2و3. في هذا النوع من الدراسة، يتم اختبار مئات الآلاف من علامات تعدد الأشكال النيوكليوتيدات واحدة (SNP) لارتباطها مع سمة، ويتم تقييم أهمية الجمعيات. يتم الاحتفاظ بارتباطات العلامات التي تفي بخطة معدل الاكتشاف الزائف (أو نوع آخر من عتبة الأهمية) للدراسة ، ولكن قد يتم تصفية الاقترانات الحقيقية. بالنسبة للسمات المعقدة المتعددة الجينات ، قد يكون تأثير كل جين صغيرا (وبالتالي يتم تصفيته) ، ويتم التعبير عن بعض الأليل فقط في ظروف محددة قد لا تكون موجودة فيالدراسة 3. وهكذا، في حين أنه قد يتم الاحتفاظ بالعديد من الشركات غير المناقلة على أنها مرتبطة بالسمة، فإن كل منها قد يكون له تأثير صغير جدا. وسوف يكون هناك الكثير من المكالمات SNP في عداد المفقودين، وتفسير المعنى البيولوجي والهندسة الوراثية للسمة قد تكون غير مكتملة ومربكة. تحليل المسار الأيضي يمكن أن يساعد على معالجة بعض هذه القضايا من خلال التركيز على الآثار المشتركة للجينات مجمعة وفقا لوظيفتها البيولوجية4،5،6.
تم الانتهاء من العديد من الدراسات باستخدام تطبيق سابق للطريقة الموصوفة في هذه المقالة. Aflatoxin تراكم7، الذرة earworm المقاومة8، والنفط biosynthesis9 ودرست جميع مع التنفيذ السابق. وفي حين كانت هذه التحليلات ناجحة، إلا أن عملية التحليل كانت معقدة وتستغرق وقتا طويلا ومرهقة، لأن أدوات التحليل كتبت في مزيج من R و Perl و Bash، ولم يكن خط الأنابيب آليا. وبسبب المعرفة المتخصصة المطلوبة لتعديل هذه الطريقة لكل تحليل، تم الآن تطوير طريقة جديدة يمكن مشاركتها مع باحثين آخرين.
وقد صممت أداة دراسة رابطة المسارات (PAST)10 لمعالجة أوجه القصور في الطريقة السابقة من خلال اشتراط معرفة أقل بلغات البرمجة وإجراء التحليلات في فترة أقصر. في حين تم اختبار هذه الطريقة مع الذرة، لا تقدم PAST أي افتراضات خاصة بالأنواع. يمكن تشغيل PAST من خلال وحدة التحكم R ، كطبيق لامع ، ومن المتوقع أن يتوفر إصدار عبر الإنترنت قريبا على MaizeGDB.
Protocol
1. الإعداد
- تثبيت R، إذا لم يكن مثبتا مسبقا.
ملاحظة: PAST هو مكتوب في R ولذلك، يتطلب أن يكون المستخدمين الخاص به R مثبتة. في وقت كتابة هذه السطور، يتطلب تثبيت PAST مباشرة من الموصل الحيوي R4.0. يمكن تثبيت الإصدارات القديمة من PAST من الموصل الحيوي ل R3.6 ، ويمكن تثبيت PAST من Github للمستخدمين الذين لديهم R3.5. يمكن تنزيل إرشادات التثبيت R من الرابط التالي: https://www.r-project.org/. - تثبيت أحدث إصدار من RStudio سطح المكتب أو تحديث RStudio (اختياري).
ملاحظة: RStudio بيئة مفيدة للعمل مع لغة R. ينصح به التثبيت، وخاصة بالنسبة لأولئك الذين يختارون لتشغيل الماضي في سطر الأوامر بدلا من خلال تطبيق واجهة المستخدم الرسومية لامعة. RStudio وإرشادات التثبيت الخاصة به يمكن العثور على الرابط التالي: https://rstudio.com/products/rstudio/. - تثبيت الماضي من الموصل الحيوي11 باتباع الإرشادات على الموصل الحيوي.
ملاحظة: التثبيت من خلال الموصل الحيوي يجب معالجة تثبيت تبعيات PAST. بالإضافة إلى ذلك، يمكن تثبيت PAST من Github12،ولكن التثبيت من Github لن يقوم بتثبيت التبعيات تلقائيا. - تثبيت الماضي لامعة (اختياري). تحميل الملف "التطبيق. R" من صفحة الإصدارات من مستودع Github: https://github.com/IGBB/PAST/releases/، وتذكر أين يقع الملف الذي تم تنزيله.
ملاحظة: يمكن استخدام PAST عن طريق استدعاء أساليبه مباشرة مع R ولكن يمكن للمستخدمين الذين هم أقل دراية R تشغيل التطبيق PAST Shiny الذي يوفر واجهة مستخدم موجهة. PAST Shiny هو برنامج نصي R متوفر في الفرع shiny_app من مستودع PAST Github. سيحاول PAST Shiny تثبيت التبعيات الخاصة به أثناء التشغيل الأول. - ابدأ التحليل من خلال بدء التطبيق بإحدى الطرق الثلاث الموضحة أدناه.
- الماضي لامعة مع RStudio
- باستخدام RStudio، إنشاء مشروع جديد في المجلد حيث التطبيق. يقع R. انقر فوق | الملف مشروع جديد وحدد هذا المجلد.
- بمجرد إنشاء مشروع جديد، افتح التطبيق. R تحميلها في وقت سابق. RStudio يتعرف على هذا التطبيق. R هو تطبيق لامع ويخلق زر تشغيل التطبيق على الشريط فوق التعليمات البرمجية المصدر المعروضة. انقر فوق تشغيل التطبيق. RStudio ثم سيتم تشغيل إطار يعرض تطبيق PAST لامعة.
- الماضي لامعة مع وحدة التحكم R
- تشغيل R وتشغيل التعليمات البرمجية التالية لبدء تطبيق PAST لامعة: لامعة::runApp('path/to/folder/with/shiny/app. R'. استبدل النص في علامات الاقتباس بالمجلد الذي التطبيق. تم تحميل R، والحفاظ على علامات الاقتباس.
- الماضي دون R لامعة
- تشغيل مكتبة (PAST) في وحدة تحكم R لتحميل الماضي.
- الماضي لامعة مع RStudio
2. تخصيص تحليل لامعة (اختياري)
- تغيير عنوان التحليل من "تحليل جديد" إلى شيء يعكس بشكل أفضل نوع التحليل الذي يتم تشغيله مما يساعد على تتبع التحليلات المتعددة (انظر الشكل 1).
الشكل 1. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
- تعديل عدد النوى ووضع. تعيين عدد النوى إلى أي رقم بين 1 والعدد الإجمالي على الجهاز ولكن كن على علم بأن تكريس المزيد من الموارد إلى الماضي قد تبطئ العمليات الأخرى على الجهاز. تعيين الوضع استنادا إلى الوصف في المقطع 6.
3. تحميل بيانات GWAS
ملاحظة: تحقق من أن يتم تحديد علامة التبويب بيانات GWAS. تأكد من أن ملف الاقتران يحتوي على الأعمدة التالية: سمة، اسم علامة، الموضع أو الكروموسوم، الموقف على الكروموسوم، قيمة p، و R2 قيمة لعلامة. تأكد من أن ملف التأثيرات يحتوي على الأعمدة التالية: سمة، اسم علامة، مكان أو كروموسوم، موقف على الكروموسوم، وتأثير. ترتيب هذه الأعمدة غير مهم، حيث يمكن للمستخدم تحديد أسماء الأعمدة عند تحميل البيانات. يتم تجاهل أي أعمدة إضافية. يمكن استخدام TASSEL13 لإنتاج هذه الملفات.
- تحميل بيانات GWAS مع لامعة الماضي.
- حدد ملف اقتران وملف تأثيرات باستخدام مربعات التحديد ملف اقتران و ملف تأثيرات. تغيير أسماء الأعمدة في المربعات إدخال اسم العمود اقتران و تأثيرات اسم الأعمدة أسفل مربعات تحديد الملف لعكس أسماء الأعمدة في البيانات.
الشكل 2. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
- تحميل بيانات GWAS مع PAST في وحدة التحكم R.
- تعديل وتشغيل التعليمات البرمجية التالية:
gwas_data = load_GWAS_data("المسار/إلى/association_file.tsv"، "المسار/إلى/effects_file.tsv"، association_columns = c("Trait"، "marker"، "Locus"، "الموقع"، "p"، "marker_R2")، effects_columns = c("trait"، "marker"، "Locus"، "الموقع"، "التأثير")
- تعديل وتشغيل التعليمات البرمجية التالية:
- ملاحظة: تغيير المسارات إلى الموقع الفعلي لملفات GWAS. القيم المتوفرة association_columns و effects_columns هي القيم الافتراضية. إذا لم تتطابق الأسماء مع القيم الافتراضية، حدد أسماء الأعمدة. وإلا، يمكن حذف هذه.
4. بيانات اختلال موازنة الربط التحميل (LD)
ملاحظة: تحقق من أن بيانات عدم التوازن (LD) الارتباط هو علامة التبويب محدد ويحتوي على الأنواع التالية من البيانات: الموضع، Position1، Site1 Position2، Site2، المسافة في أزواج أساسية بين Position1 و Position2، وقيمة R2.
- تحميل بيانات LD مع PAST لامعة.
- حدد الملف الذي يحتوي على بيانات LD. تغيير أسماء الأعمدة في مربعات الإدخال أسماء الأعمدة LD أسفل مربع تحديد الملف لمطابقة أسماء الأعمدة في بيانات LD إذا لزم الأمر.
الشكل 3. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
- تحميل بيانات LD مع PAST في وحدة التحكم R.
- تعديل وتشغيل التعليمات البرمجية التالية لتحميل بيانات LD:
LD = load_LD("المسار/إلى/LD.tsv", LD_columns = c("Locus1", "Position1", "Site1", "Position2", "Site2", "Dist_bp", "R.2")
ملاحظة: تغيير المسار إلى الموقع الفعلي لملف LD. القيم المتوفرة LD_columns هي القيم الافتراضية. إذا لم تتطابق الأسماء مع هذه الإعدادات الافتراضية، حدد الأسماء الصحيحة للأعمدة؛ وإلا، يمكن حذف هذه.
- تعديل وتشغيل التعليمات البرمجية التالية لتحميل بيانات LD:
5. تعيين SNPs للجينات
ملاحظة: تحميل أو تحديد موقع التعليقات التوضيحية في تنسيق GFF. ويمكن العثور على هذه الشروح في كثير من الأحيان في قواعد البيانات على الانترنت لكائنات محددة. كن حذرا بشأن التعليقات التوضيحية منخفضة الجودة ، حيث ستؤثر جودة بيانات التعليقات التوضيحية على جودة تحليل المسار. تأكد من أن العمود الأول من هذه التعليقات التوضيحية (الكروموسوم) يطابق شكل الموضع / الكروموسوم في الارتباط والآثار وبيانات LD. على سبيل المثال، يجب أن لا تسمى التعليقات التوضيحية الكروموسوم الأول "chr1" إذا كانت ملفات بيانات GWAS و LD تسمي الكروموسوم الأول "1".
- تعيين SNPs إلى الجينات مع الماضي لامعة.
ملاحظة: يمكن العثور على مزيد من المعلومات حول تحديد قطع R2 المناسبة في تانغ وآخرون6، في القسم المسمى "SNP إلى خوارزمية الجينات لتحليل المسار".- حدد الملف الذي يحتوي على تعليقات توضيحية ل GFF. النظر في حجم النافذة وقطع R2 هي الأكثر ملاءمة للأنواع التي يجري النظر فيها وتعديل إذا كانت الافتراضات لا تناسب البيانات التي تم تحميلها.
ملاحظة: القيم الافتراضية في PAST تعكس بشكل أساسي القيم المناسبة للذرة. يتم استخدام عدد النوى التي تم تعيينها في بداية التحليل PAST Shiny (الخطوة 2.2) في هذه الخطوة.
- حدد الملف الذي يحتوي على تعليقات توضيحية ل GFF. النظر في حجم النافذة وقطع R2 هي الأكثر ملاءمة للأنواع التي يجري النظر فيها وتعديل إذا كانت الافتراضات لا تناسب البيانات التي تم تحميلها.
الشكل 4. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
- تعيين SNPs إلى الجينات مع الماضي في وحدة التحكم R.
- تعديل وتشغيل التعليمات البرمجية التالية لتعيين SNPs إلى الجينات:
الجينات = assign_SNPs_to_genes (gwas_data، LD، "المسار/إلى/annotations.gff"، ج("جين")، 1000، 0.8، 2)
ملاحظة: في هذا النموذج التعليمات البرمجية، يتم توفير العديد من الاقتراحات الافتراضية: 1000 هو حجم الإطار حول SNP للبحث عن الجينات; 0.8 هي قيمة القطع ل R2؛ 2 هو عدد النوى المستخدمة للمعالجة المتوازية. يجب أيضا تغيير المسار إلى التعليقات التوضيحية إلى الموقع الفعلي لملف التعليقات التوضيحية.
- تعديل وتشغيل التعليمات البرمجية التالية لتعيين SNPs إلى الجينات:
6. اكتشاف مسارات هامة
ملاحظة: تحقق من أن ملف المسارات يحتوي على البيانات التالية في تنسيق محدد علامة التبويب مع سطر واحد لكل جين في كل مسار: معرف المسار - معرف مثل "PWY-6475-1"; وصف المسار - وصف أطول لما تفعله المسارات مثل "التمثيل الحيوي عبر الليكوبين"؛ جين - جين في المسار، والتي ينبغي أن تتطابق مع الأسماء الواردة في الشروح. يمكن العثور على معلومات المسار على الأرجح في قواعد البيانات عبر الإنترنت لكائنات حية محددة، مثل MaizeGDB. الخيار الثاني المحدد من قبل المستخدم هو الوضع. تشير "الزيادة" إلى الأنماط الظاهرية التي تعكس عندما تكون القيمة المتزايدة للسمة المقاسة مرغوبة ، مثل العائد ، في حين يشير "التناقص" إلى سمة يكون فيها انخفاض القيم المقاسة مفيدا ، مثل تقييمات تلف الحشرات. يتم اختبار أهمية المسارات باستخدام الطرق الموصوفة سابقا4و6و14.
- اكتشف مسارات هامة مع PAST Shiny.
- حدد الملف الذي يحتوي على بيانات المسارات وتأكد من تحديد الوضع في خيارات التحليل. إذا لزم الأمر، تغيير عدد الجينات التي يجب أن تكون في مسار للاحتفاظ بها للتحليل وعدد التباديل المستخدمة لإنشاء التوزيع الخالي لاختبار أهمية التأثير.
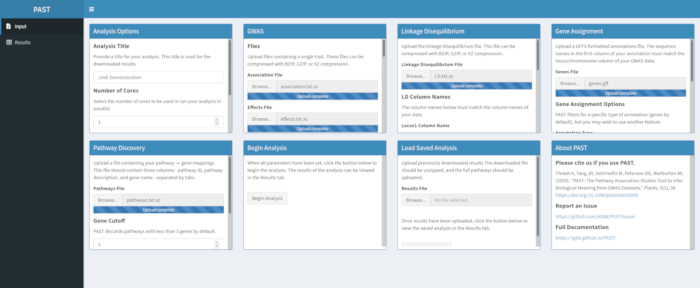
الشكل 5. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
ملاحظة: يتم استخدام عدد النوى ووضع تعيين في بداية التحليل PAST Shiny (الخطوة 2.2) في هذه الخطوة. يتم تعيين العدد الافتراضي للجينات حاليا في 5 جينات، لذلك سيتم إزالة المسارات مع عدد أقل من الجينات المعروفة. يمكن للمستخدم خفض هذه القيمة إلى 4 أو 3 ، لتشمل مسارات أقصر ، ولكن القيام بذلك سيخاطر بنتائج إيجابية خاطئة. زيادة هذه القيمة يمكن أن تزيد من قوة التحليل ولكن سوف يزيل المزيد من المسارات من التحليل. تغيير عدد التباديل المستخدمة يزيد ويقلل من قوة الاختبار.
- اكتشف مسارات هامة مع PAST في وحدة التحكم R.
- تعديل وتشغيل التعليمات البرمجية التالية لاكتشاف مسارات هامة:
rugplots_data <- find_pathway_significance (الجينات، "المسار/إلى/pathways.tsv"، 5، "زيادة"، 1000، 2)
ملاحظة: في هذا النموذج التعليمات البرمجية، يتم توفير العديد من الافتراضات المقترحة. 5 هو الحد الأدنى لعدد الجينات التي يجب أن تكون في مسار من أجل الحفاظ على المسار في التحليل ، وتشير الزيادة إلى كمية متزايدة من السمة المقاسة (يوصى بأن يقوم المستخدم بتشغيل كل من الزيادة والتناقص ، بغض النظر عن السمة ؛ يختلف تفسير البيانات عن الاثنين ، ومع ذلك) ، فإن 1000 هو عدد المرات التي يتم فيها أخذ عينات من الآثار لتحديد التوزيع الخالي ، و 2 هو عدد النوى المستخدمة للمعالجة المتوازية. تغيير المسار إلى الموقع الفعلي لملف المسارات.
- تعديل وتشغيل التعليمات البرمجية التالية لاكتشاف مسارات هامة:
7. عرض Rugplots
- عرض Rugplots مع الماضي لامعة.
- بمجرد تحميل كافة المدخلات وتعيينها، انقر فوق بدء التحليل. سيظهر شريط تقدم ويشير إلى الخطوة التي اكتملت آخر مرة من التحليل. عند اكتمال التحليل، سوف ينتقل PAST Shiny إلى علامة التبويب النتائج. سيتم عرض جدول النتائج في العمود الأيسر (المسمى "المسارات") وسيتم عرض Rugplots في العمود الأيمن (المسمى "المؤامرات").
- استخدم شريط التمرير للتحكم في معلمات التصفية. عندما يكون مستوى التصفية مرضيا، انقر فوق الزر تنزيل النتائج في أسفل اليسار لتنزيل كافة الصور والجداول بشكل فردي إلى ملف ZIP الذي تم تسميته بعنوان التحليل. يحتوي هذا الملف ZIP على الجدول المصفاة وجدول غير المصفاة وصورة واحدة لكل مسار في الجدول المصفاة.
الشكل 6. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
الشكل 7. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الرقم.
- عرض Rugplots مع الماضي في وحدة التحكم R
- تعديل وتشغيل التعليمات البرمجية التالية لحفظ النتائج:
plot_pathways(rugplots_data، "pvalue"، 0.02، "زيادة"، "output_folder")
ملاحظة: في هذا النموذج التعليمات البرمجية، يتم توفير العديد من الافتراضات المقترحة. pvalue يوفر البيانات التي يمكن استخدامها لتصفية مسارات تافهة بعد اختيار عتبة أهمية من قبل المستخدم; 0.02 هي القيمة الافتراضية المستخدمة في التصفية، وتشير الزيادة إلى كمية متزايدة من السمة المقاسة (من المستحسن أن يقوم المستخدم بتشغيل كل من الزيادة والتناقص، بغض النظر عن السمة؛ ومع ذلك، فإن تفسير البيانات يختلف بالنسبة للاثنين)؛ output_folder هو المجلد حيث سيتم كتابة الصور والجداول (يجب أن يكون هذا المجلد موجودا قبل تشغيل الدالة). تتم كتابة جدول النتائج المصفاة والنتائج غير المصفاة والصور الفردية لكل مسار في النتائج التي تمت تصفيتها إلى هذا المجلد.
- تعديل وتشغيل التعليمات البرمجية التالية لحفظ النتائج:
النتائج
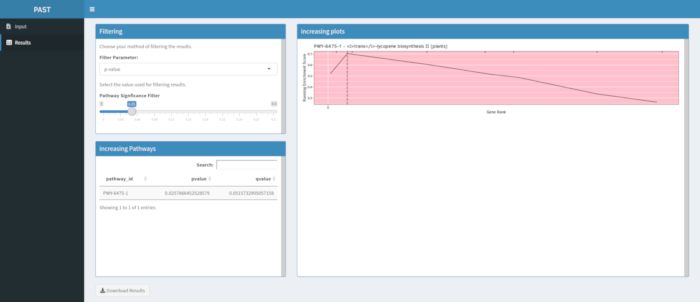
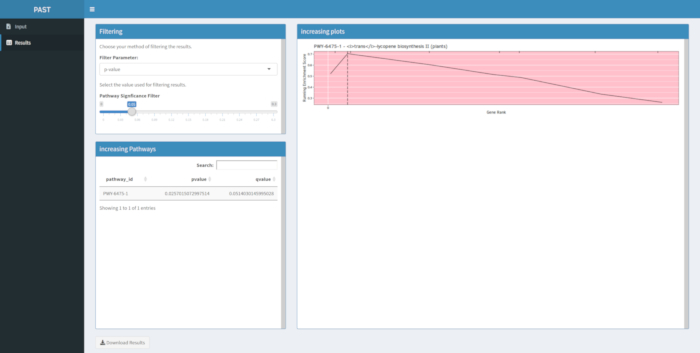
إذا لم يتم إنتاج النتائج بعد تشغيل أداة البرامج PAST تحقق للتأكد من أن كافة ملفات الإدخال منسقة بشكل صحيح. يظهر في الشكل 8تشغيل ناجح باستخدام بيانات المثال في حزمة PAST، والتي تستند إلى GWAS الذرة من لون الحبوب. يمكن تنزيل هذا الجدول والصورة الناتجة باستخدام الزر تنزيل النتائج. يظهر مثال للصورة التي تم تنزيلها في الشكل 210. قد تؤدي الإعدادات غير الصحيحة إلى نتائج لا معنى لها بيولوجيا ، ولكن تحديد عدم الصحة يجب أن يكون متروكا للباحث ، الذي يجب أن يتحقق مرتين من صحة الإعدادات المختارة والنظر في جميع الأدلة المعروفة المتعلقة بسمة الاهتمام.
ويبين الشكل 910 السجادة الناتجة عن تحليل المسار لنتائج ال GWAS التي تم إنشاؤها باستخدام لوحة ذرة مكونة من 288 خطا أصيلا تم كتابتها بشكل الظاهري للون الحبوب. هذا المثال التبسيطي، حيث كانت الأنماط الظاهرية إما "بيضاء" أو "صفراء"، تم استخدامه لأن المسار المسؤول عن إنشاء أصباغ الكاروتينويد الصفراء الزاهية معروف ويجب أن يكون مسؤولا عن معظم النمط الظاهري. وهكذا، كنا نتوقع أن نرى مسار التمثيل الحيوي عبر الليكوبين (التي تنتج الكاروتينات) أن تكون مرتبطة بشكل كبير مع لون الحبوب، وهو عليه. يتم سرد معرف المسار والاسم في أعلى الرسم البياني. يصنف المحور الأفقي للرسم البياني جميع الجينات التي تم تضمينها في التحليل ، مرتبة من اليسار إلى اليمين من أجل التأثير الأكبر على السمة إلى الأصغر. ومع ذلك ، يتم وضع علامة فقط على الجينات في مسار التمثيل الحيوي عبر الليكوبين (في الجزء العلوي من الرسم البياني ، كعلامات فقس ، تظهر في رتبة الجينات لتأثيرها مقارنة بجميع الجينات الأخرى في التحليل). هناك 7 جينات في هذا المسار. يتم رسم درجة الإثراء قيد التشغيل (ES) على طول المحور الرأسي. يتم إضافة ES لكل جين في المجموع الجاري حسب التأثير ويتم تعديل المجموع إلى عدد الجينات التي تم تحليلها. وهكذا، تتغير النتيجة مع تحرك المرء مباشرة على طول المحور الأفقي ويميل إلى الزيادة مع تضمين جينات التأثير الأكبر، ولكن في مرحلة ما، تكون الزيادة في التأثير أصغر من التعديل ستضاف جين آخر، وتبدأ النتيجة بأكملها في الانخفاض. يتم وضع علامة على قمة خط ES قيد التشغيل بخط عمودي منقط؛ هذا هو ES للمسار بأكمله ويستخدم من قبل البرنامج لتحديد ما إذا كان يتم اختيار المسار وتقديمه على أنه سجادة.
الشكل 8: الانتهاء من تشغيل الماضي لامعة. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
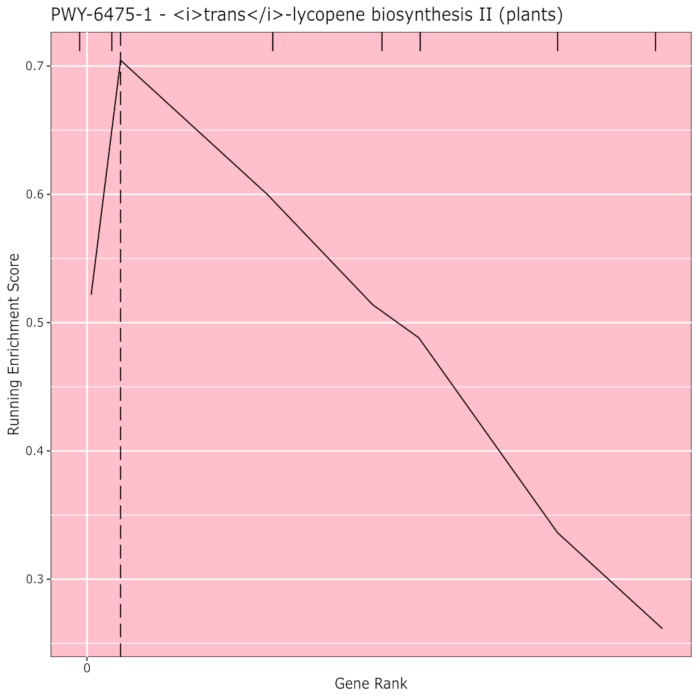
الشكل 9: صورة المسار من تشغيل الانتهاء من الماضي (أو تحميلها من لامعة). وقد استشهد بهذا الرقم من Thrashوآخرون. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Discussion
ويتمثل الهدف الرئيسي من "PAST" في تقديم تحليلات المسار الأيضي لبيانات GWAS إلى جمهور أوسع، وخاصة بالنسبة للكائنات غير البشرية وغير الحيوانية. غالبا ما تكون الطرق البديلة ل PAST هي برامج سطر الأوامر التي تركز على البشر أو الحيوانات. وكان سهولة الاستخدام هدفا أساسيا في تطوير الماضي، سواء في اختيار لتطوير تطبيق لامعة وفي اختيار استخدام R و Bioconduductor للافراج عن التطبيق. المستخدمين لا تحتاج إلى معرفة كيفية ترجمة البرامج من أجل استخدام الماضي.
وكما هو الحال مع معظم أنواع برامج التحليل، فإن نتائج برنامج PAST لا تكون جيدة إلا بقدر ما تكون بيانات الإدخال؛ إذا كان لدى بيانات الإدخال أخطاء أو تنسيقها بشكل غير صحيح، سوف تفشل PAST في تشغيل أو إنتاج نتائج غير مفيدة. التأكد من أن يتم تنسيق البيانات GWAS والبيانات LD والتعليقات التوضيحية وملفات المسارات بشكل صحيح أمر بالغ الأهمية لتلقي الإخراج الصحيح من الماضي. الماضي يحلل علامات ثنائية allelic فقط ، ويمكن تشغيل سمة واحدة فقط لكل مجموعة من البيانات الإدخال. وبالإضافة إلى ذلك، فإن بيانات GWAS الناتجة عن سوء الكتابة الجينية أو الفينوتيبينج غير الصحيح أو غير الدقيق من غير المرجح أن تسفر عن نتائج واضحة أو قابلة للتكرار أيضا. يمكن أن تساعد PAST في التفسير البيولوجي لنتائج GWAS ولكن من غير المرجح أن توضح مجموعات البيانات الفوضوية إذا لم يتم حساب الاختلاف البيئي أو الخطأ التجريبي أو الهيكل السكاني بشكل صحيح.
يمكن للمستخدمين اختيار تغيير بعض معلمات التحليل، سواء في التطبيق لامعة وتمرير تلك المعلمات إلى وظائف الماضي في وحدة التحكم R. يمكن تغيير هذه المعلمات النتائج التي تم الإبلاغ عنها بواسطة PAST و يجب على المستخدمين توخي الحذر عند تعديل هذه من الإعدادات الافتراضية. لأن LD يتم قياسها بواسطة المستخدمين، عادة باستخدام نفس مجموعة بيانات العلامة التي تم استخدامها أيضا في GWAS، قياسات LD محددة للسكان. وبالنسبة لجميع الدراسات، وخاصة بالنسبة لأنواع أخرى غير الذرة ، (وخاصة التلقيح الذاتي ، أو الأنواع متعددة الأضلاع ، أو غير المتجانسة للغاية) ، قد يكون هناك ما يبرر حدوث تغييرات في حالات التخلف عن السداد.
Disclosures
وليس لدى صاحبي البلاغ ما يكشفان عنه.
Acknowledgements
اي.
Materials
| Name | Company | Catalog Number | Comments |
| Computer | NA | NA | Any computer with 8GB RAM should be sufficient |
| R | R Project | NA | R 4.0 or greater is required to install from Bioconductor 3.11 |
References
- Rafalski, J. Association genetics in crop improvement. Current Opinion in Plant Biology. 13 (2), 174-180 (2010).
- Yan, J., Warburton, M., Crouch, J. Association Mapping for Enhancing Maize (Zea mays L.) Genetic Improvement. Crop Science. 51 (2), 433-449 (2011).
- Xiao, Y., Liu, H., Wu, L., Warburton, M., Yan, J. Genome-wide Association Studies in Maize: Praise and Stargaze. Molecular Plant. 10 (3), 359-374 (2017).
- Wang, K., Li, M., Bucan, M. Pathway-Based Approaches for Analysis of Genomewide Association Studies. The American Journal of Human Genetics. 81 (6), 1278-1283 (2007).
- Weng, L., et al. SNP-based pathway enrichment analysis for genome-wide association studies. BMC Bioinformatics. 12 (1), 99(2011).
- Tang, J., Perkins, A., Williams, W., Warburton, M. Using genome-wide associations to identify metabolic pathways involved in maize aflatoxin accumulation resistance. BMC Genomics. 16 (1), 673(2015).
- Warburton, M., et al. Genome-Wide Association Mapping of Aspergillus flavus and Aflatoxin Accumulation Resistance in Maize. Crop Science. 55 (5), 1857-1867 (2015).
- Warburton, M., et al. Genome-Wide Association and Metabolic Pathway Analysis of Corn Earworm Resistance in Maize. The Plant Genome. 11 (1), 170069(2018).
- Li, H., Thrash, A., Tang, J., He, L., Yan, J., Warburton, M. Leveraging GWAS data to identify metabolic pathways and networks involved in maize lipid biosynthesis. The Plant Journal. 98 (5), 853-863 (2019).
- Thrash, A., Tang, J., DeOrnellis, M., Peterson, D., Warburton, M. PAST: The Pathway Association Studies Tool to Infer Biological Meaning from GWAS Datasets. Plants. 9 (1), 58(2020).
- Adam, T., Mason, D. PAST: Pathway Association Study Tool (PAST). Bioconductor version: Release (3.10). , (2020).
- Thrash, A., DeOrnellis, M. IGBB/PAST. , at https://github.com/IGBB/PAST (2019).
- Bradbury, P., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Subramanian, A., et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences U.S.A. 102, 15545-15550 (2005).
Erratum
Formal Correction: Erratum: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information
Posted by JoVE Editors on 10/08/2021. Citeable Link.
An erratum was issued for: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information. One of the affiliations was updated.
The second affiliation was updated from:
USDA-ARS Corn Host Plant Resistance Research Unit, Mississippi State University
to:
Corn Host Plant Resistance Research Unit, USDA-ARS
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved