Method Article
代謝経路情報のGWAS解析のための経路関連研究ツール
Erratum Notice
要約
シャイニーアプリケーションまたはRコンソールを通じて経路協会研究ツール(PAST)を実行することにより、研究者は関連する代謝経路を調査することによって、ゲノム全体の関連研究(GWAS)の結果の生物学的意味をより深く理解することができます。
要約
近年、代謝経路解析を用いたゲノムワイド関連解析(GWAS)データの解釈に関する新たな実装が開発され、発表された。経路協会スタディツール(PAST)は、ユーザーの使いやすさと遅い分析の懸念に対処するために開発されました。この新しいユーザーフレンドリーなツールは、バイオコンダクターとGithubでリリースされました。テストでは、PASTは以前は24時間以上必要な分析を1時間以内に実行しました。この記事では、書き込みアプリケーションまたは R コンソールを使用して PAST を実行するためのプロトコルを紹介します。
概要
ゲノムワイド関連研究(GWAS)は、複雑な形質とそれらに関連するゲノム領域を1、2、3に関連するゲノム領域を研究する一般的な方法である。このタイプの研究では、数十万個の一塩基多型(SNP)マーカーが形質との関連についてテストされ、関連の有意性が評価される。誤検出率 (FDR) のしきい値 (またはその他の種類の有意性しきい値) を満たすマーカー特性アソシエーションは、調査のために保持されますが、真の関連付けは除外される場合があります。複雑なポリジェニック形質の場合、各遺伝子の効果は小さく(したがって除外される)可能性があり、一部の対立遺伝子は研究3に存在しないかもしれない特定の条件でのみ発現される。したがって、多くのSNPsは形質に関連付けられているように保持されるかもしれませんが、それぞれが非常に小さな効果を有する可能性があります。あまりにも多くのSNP呼び出しが欠落し、形質の生物学的意味と遺伝的アーキテクチャの解釈が不完全で混乱する可能性があります。代謝経路解析は、生物学的機能4、5、6に従ってグループ化された遺伝子の複合効果に焦点を当てることで、これらの問題のいくつかに対処するのに役立ちます。
この記事で説明したメソッドの以前の実装を使用して、いくつかの調査が完了しました。アフラトキシン蓄積7、コーンミミズ耐性8、及び油生合成9 はいずれも、以前の実施で検討した。これらの解析は成功しましたが、分析ツールは R、Perl、Bash の組み合わせで作成され、パイプラインは自動化されていないため、分析プロセスは複雑で時間がかかり、面倒でした。解析ごとにこの方法を変更するために必要な専門的な知識のため、他の研究者と共有できる新しい方法が開発されました。
経路関連スタディツール(PAST)10 は、プログラミング言語の知識を減らし、より短い期間で分析を実行することで、以前の方法の欠点に対処するように設計されました。この方法はトウモロコシでテストされましたが、PASTは種固有の仮定をしません。PASTは、シャイニーアプリとしてRコンソールを介して実行することができ、オンラインバージョンはまもなくMaizeGDBで利用可能になると予想されます。
プロトコル
1. セットアップ
- R がインストールされていない場合は、R をインストールします。
注: PAST は R で記述されているため、ユーザーには R がインストールされている必要があります。この書き込みの時点で、生物伝導体から直接PASTをインストールするにはR4.0が必要です。 PAST の古いバージョンは、R3.6 のバイオコンダクタからインストールでき、また、R3.5 を使用するユーザーは Github からインストールできます。R のインストール手順は、次のリンクからダウンロードできます https://www.r-project.org/。 - RStudio デスクトップの最新バージョンをインストールするか、RStudio を更新します (省略可能)。
注: RStudio は、R 言語を使用する場合に役立つ環境です。特に、シンニー GUI アプリケーションを使用するのではなく、コマンド ラインで PAST を実行することを選択したユーザーには、インストールすることをお勧めします。RStudio とそのインストール手順は、次のリンク https://rstudio.com/products/rstudio/にあります。 - 生体伝導体11 から、生体伝導体の指示に従ってPASTをインストールします。
注: 生体伝導体によるインストールは、PAST の依存関係のインストールを処理する必要があります。さらに、PAST は Github12からインストールできますが、Github からインストールすると依存関係は自動的にインストールされません。 - 過去のシャイニー(オプション)をインストールします。ファイル"アプリ"をダウンロードします。Github リポジトリの [リリース] ページの R" https://github.com/IGBB/PAST/releases/、ダウンロードしたファイルの場所を覚えておいてください。
注: PAST は R でメソッドを直接呼び出すことによって使用できますが、R にあまり慣れていないユーザーは、ガイド付きユーザー インターフェイスを提供する PAST シャイニー アプリケーションを実行できます。PAST シャイニーは、過去の Github リポジトリのshiny_appブランチで利用できる R スクリプトです。PAST シャイニーは、最初の実行時にその依存関係をインストールしようとします。 - 次に説明する 3 つの方法のいずれかで、アプリケーションを開始して分析を開始します。
- RStudioと過去の光沢
- RStudio を使用して、アプリのフォルダーに新しいプロジェクトを作成します。R が配置されます。[ ファイル] |新しいプロジェクト をクリックし、そのフォルダを選択します。
- 新しいプロジェクトが作成されたら、アプリを開きます。R ファイルは以前にダウンロードされました。RStudio はそのアプリを認識します。R は、シャイニー アプリで、表示されたソース コードの上のバーに [アプリの実行 ] ボタンを作成します。[ アプリケーションの実行] をクリックします。その後、RStudio は PAST のシャイニー アプリケーションを表示するウィンドウを起動します。
- Rコンソールで過去の光沢
- Rを起動し、PASTシャイニーアプリケーションを起動するために次のコードを実行します: 光沢::runApp('パス/フォルダ/ウィズ/シャイニー/アプリ。R'。引用符で囲まれたテキストを、アプリのフォルダーに置き換えます。Rがダウンロードされ、引用符を保持します。
- Rシャイニーなしの過去
- ライブラリ (PAST)を Rコンソールで実行して、PASTをロードします。
- RStudioと過去の光沢
2. シャイニー分析のカスタマイズ (オプション)
- 解析のタイトルを「新規分析」から、複数の分析を追跡するのに役立つ、実行されている分析のタイプをよりよく反映したものに変更します ( 図 1を参照)。
図1. ここをクリックして、この図の大きなバージョンを表示してください。
- コアの数とモードを変更します。コアの数を 1 からコンピューターの合計数の間の任意の数に設定しますが、PAST にリソースを追加すると、マシン上の他の操作が遅くなる可能性があることに注意してください。セクション 6 の説明に基づいてモードを設定します。
3. GWAS データの読み込み
注: GWAS データがタブ区切りであることを確認します。関連ファイルに、特性、マーカー名、軌跡または染色体、染色体上の位置、p値、およびマーカーの R2 値が含まれていることを確認します。エフェクトファイルに、特性、マーカー名、軌跡または染色体、染色体上の位置、および効果の列が含まれていることを確認します。ユーザーはデータを読み込むときに列の名前を指定できるため、これらの列の順序は重要ではありません。追加の列は無視されます。TASSEL13 は、これらのファイルを生成するために使用できます。
- 過去の光沢のあるGWASデータをロードします。
- [関連付けファイル] および [エフェクト ファイル] 選択ボックスを使用して、 関連付けファイル と エフェクト ファイル を選択します。ファイル選択ボックスの下にある [関連列名 ]および[ エフェクト列名] 入力ボックスの列名を変更して、データ内の列名を反映します。
図2. この図の大きなバージョンを表示するには、ここをクリックしてください。
- R コンソールで PAST を使用して GWAS データをロードします。
- 次のコードを変更して実行します。
gwas_data =load_GWAS_data("path/to/association_file.tsv"、"パス effects_file/association_file.tsv"、association_columns=c(「形質」、「マーカー」、「ローカス」、「サイト」、「サイト」、「p」、「marker_R2」)、effects_columns=c(「形質」、「マーカー」、「ローカ」、「サイト」、「効果」)
- 次のコードを変更して実行します。
- 注: GWAS ファイルの実際の場所へのパスを変更します。association_columnsとeffects_columnsに指定された値は、既定値です。名前がデフォルト値と一致しない場合は、列名を指定します。それ以外の場合は省略できます。
4. リンケージの不一定衡 (LD) データの読み込み
注: リンケージの不衡 (LD) データがタブ区切りで、次の種類のデータが含まれていることを確認します: 軌跡、位置 1、サイト 1、位置 2、サイト 2、位置 1 と位置 2 の間のベース ペアの距離、および R2 値。
- 過去のシャイニーで LD データをロードします。
- LD データを含むファイルを選択します。必要に応じて 、LD データの列名 と一致するように、ファイル選択ボックスの下にある [LD 列名] 入力ボックスの列名を変更します。
図 3. この図の大きなバージョンを表示するには、ここをクリックしてください。
- LD データを PAST を使用して R コンソールにロードします。
- LD データを読み込むには、次のコードを変更して実行します。
LD = load_LD("パス/先/LD.tsv"、LD_columns=c(「ローカス1」、「位置1」、「サイト1」、「位置2」、「サイト2」、「Dist_bp」、「R.2」)
メモ: LD ファイルの実際の場所へのパスを変更します。LD_columnsに指定された値は、デフォルト値です。名前がこれらのデフォルト値と一致しない場合は、列の正しい名前を指定します。それ以外の場合は省略できます。
- LD データを読み込むには、次のコードを変更して実行します。
5. 遺伝子へのスナップの割り当て
注: GFF 形式の注釈をダウンロードまたは検索します。これらの注釈は、多くの場合、特定の生物のためのオンラインデータベースで見つけることができます。注釈データの品質が経路解析の品質に影響を与えるため、低品質の注釈については注意が必要です。これらの注釈の最初の列(染色体)が、関連付け、効果、LD データの軌跡/染色体の形式と一致することを確認します。たとえば、GWAS と LD データ ファイルが最初の染色体 "1" を呼び出す場合、注釈は最初の染色体 "chr1" を呼び出す必要があります。
- PASTシャイニーを使用して遺伝子にスナップを割り当てます。
注: 適切な R2 カットオフの決定に関する詳細については、Tang et al.6の「経路解析のための SNP to 遺伝子アルゴリズム」のセクションで見つけることができます。- GFF 注釈を含むファイルを選択します。考慮する種に最適なウィンドウ サイズと R2 カットオフを検討し、デフォルトがアップロードされたデータに適合しない場合は変更します。
注: PAST のデフォルト値は、主にトウモロコシに適した値を反映します。このステップでは、PAST シャイニー解析の最初に設定されたコアの数 (ステップ 2.2) を使用します。
- GFF 注釈を含むファイルを選択します。考慮する種に最適なウィンドウ サイズと R2 カットオフを検討し、デフォルトがアップロードされたデータに適合しない場合は変更します。
図 4. この図の大きなバージョンを表示するには、ここをクリックしてください。
- R コンソールで PAST を使用して、SNPs を遺伝子に割り当てます。
- 次のコードを変更して実行し、SNPs を遺伝子に割り当てます。
遺伝子 = assign_SNPs_to_genes(gwas_data、LD、「パス/パス/注釈.gff」、c(「遺伝子」)、1000、0.8、2)
注: このサンプル コードでは、いくつかの既定の提案が提供されています: 1000 は、遺伝子を検索する SNP の周囲のウィンドウのサイズです。0.8 は R2のカットオフ値です。2 は、並列処理に使用されるコアの数です。注釈へのパスも、注釈ファイルの実際の場所に変更する必要があります。
- 次のコードを変更して実行し、SNPs を遺伝子に割り当てます。
6. 重要な経路を発見する
注: 経路ファイルに、各経路の各遺伝子に対して 1 行のデータがタブ区切り形式で含まれていることを確認します。経路記述 - 経路が「トランスリコピン生合成」などの何をするかのより長い説明;gene - アノテーションで提供される名前と一致する経路の遺伝子。経路情報は、MaizeGDB などの特定の生物のオンライン データベースで見つかる可能性があります。2 番目のユーザー指定オプションはモードです。「増加」とは、歩留まりなど測定された形質の増加値が望ましい場合に反射する表現型を指し、「減少」とは、昆虫の損傷評価などの測定値の減少が有益である形質を指す。経路の重要性は、前に説明した方法4,6,14を使用してテストされます。
- PASTシャイニーで重要な経路を発見してください。
- パス データを含むファイルを選択し、解析オプションでモードが選択されていることを確認します。必要に応じて、分析のためにそれを保持するために経路に必要な遺伝子の数と、効果の有意性をテストするためにヌル分布を作成するために使用される順列の数を変更します。
図 5. この図の大きなバージョンを表示するには、ここをクリックしてください。
注: このステップでは、PAST シャイニー解析の開始時に設定されたコア数とモードセットを使用します。遺伝子のデフォルト数は現在5遺伝子に設定されているので、既知の遺伝子が少ない経路は除去されます。ユーザーはこの値を 4 または 3 に下げて短い経路を含めることができますが、これを行うと誤検出の結果が生じる危険性があります。この値を大きくすると、解析の力が増えますが、解析からより多くの経路が除去されます。使用される順列の数を変更すると、テストの能力が増加し、減少します。
- RコンソールでPASTを使用して重要な経路を発見してください。
- 重要な経路を検出するには、次のコードを変更して実行します。
rugplots_data <-find_pathway_significance(遺伝子、「パス/パス/パス/パス.tsv」、5、「増加」、1000、2)
注: このサンプル コードでは、推奨される既定値がいくつか用意されています。5は、分析中の経路を維持するために経路内になければならない遺伝子の最小数であり、増加は測定された形質の増加量を指し(ユーザーは形質に関係なく増加と減少の両方を実行することが推奨され、データ解釈は2つの場合は異なるが、1000はヌル分布を決定する効果をサンプリングする回数である。 2 は並列処理に使用されるコアの数です。パスをパスパスファイルの実際の場所に変更します。
- 重要な経路を検出するには、次のコードを変更して実行します。
7. ラグプロットを表示する
- 過去のシャイニーでラグプロットを表示します。
- すべての入力がアップロードされ、設定されたら、[ 分析の開始] をクリックします。進行状況バーが表示され、解析のどのステップが最後に完了したかを示します。解析が完了すると、PAST シャイニーが [結果] タブに切り替わります。結果の表が左側の列(「経路」とラベル付け)に表示され、ラグプロットが右側の列(「プロット」とラベル付け)に表示されます。
- スライダーを使用して、フィルター処理パラメーターを制御します。フィルタリングレベルが十分な場合は、左下の [結果のダウンロード ]ボタンをクリックして、すべての画像とテーブルを、解析タイトルで指定されたZIPファイルに個別にダウンロードします。この ZIP ファイルには、フィルター処理されたテーブル、フィルタ処理されていないテーブル、およびフィルター選択されたテーブルのパスごとに 1 つのイメージが含まれています。
図 6. この図の大きなバージョンを表示するには、ここをクリックしてください。
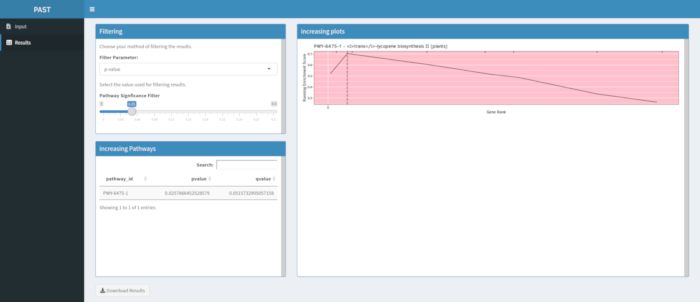
図 7. この図の大きなバージョンを表示するには、ここをクリックしてください。
- R コンソールで PAST を使用してラグプロットを表示する
- 次のコードを変更して実行し、結果を保存します。
plot_pathways(rugplots_data、"pvalue"、0.02、「増加」、「output_folder」)
注: このサンプル コードでは、推奨される既定値がいくつか用意されています。pvalue は、有意性しきい値がユーザーによって選択された後に、重要でない経路をフィルタリングするために使用できるデータを提供します。0.02 はフィルター処理で使用される既定値で、増加とは、測定された特性の増加量を指します (ユーザーは、特性に関係なく、増加と減少の両方を実行することをお勧めします。output_folderは、イメージとテーブルが書き込まれるフォルダです (このフォルダは、関数を実行する前に存在している必要があります)。フィルター処理された結果、フィルター処理されていない結果、およびフィルター処理された結果内のすべての経路の個々のイメージの表がこのフォルダーに書き込まれます。
- 次のコードを変更して実行し、結果を保存します。
結果
PAST ソフトウェアツールの実行後に結果が生成されない場合は、すべての入力ファイルが正しくフォーマットされていることを確認してください。図 8に、グレイン カラーのトウモロコシ GWAS に基づく PAST パッケージのサンプル データを使用した正常な実行を示します。この表と結果の画像は、[結果のダウンロード] ボタンを使用してダウンロードできます。ダウンロードした画像の例を 図 210に示します。誤った設定は生物学的に意味をなさない結果につながる可能性がありますが、誤りを判断することは研究者次第であり、選択した設定の妥当性を再確認し、関心のある特性に関するすべての既知の証拠を考慮する必要があります。
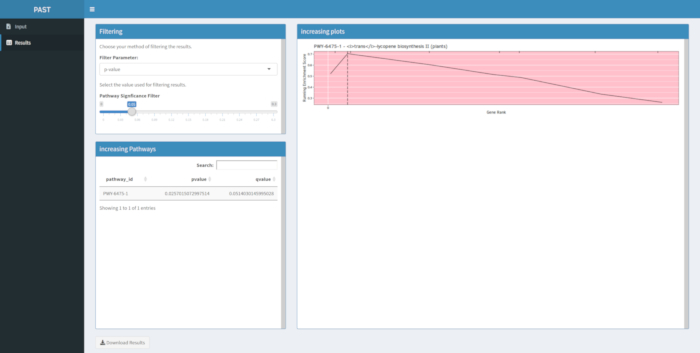
図910 は、粒色に対してフェノタイプされた288本の近交系ラインのトウモロコシパネルで作成されたGWAS結果の経路解析から生成されたラグプロットを示す。この単純な例は、表現型が「白」または「黄色」であったが、明るい黄色のカロテノイド顔料を作成する経路が知られており、表現型の大部分を担うべきであるため、使用された。したがって、トランスリコピン生合成経路(カロテノイドを産生する)が穀物色と有意に関連することを期待した。パス ID と名前は、グラフの上部に表示されます。グラフの横軸は、分析に含まれていたすべての遺伝子を、形質に対する最大の効果から最小の順に左から右に並べ替えます。しかし、トランスリコピン生合成経路の遺伝子のみがマークされています(グラフの上部には、ハッチマークとして、分析中の他のすべての遺伝子と比較してその効果の遺伝子ランクに現れます)。この経路には7つの遺伝子があります。ランニングエンリッチメントスコア(ES)は、垂直軸に沿ってプロットされます。各遺伝子のESは効果の順序で走行合計に加え、合計は分析された遺伝子の数に合わせて調整されます。したがって、スコアは水平軸に沿って右に移動するにつれて変化し、より大きな効果遺伝子が含まれるにつれて増加する傾向がありますが、ある時点で、効果の増加は別の遺伝子を追加した場合の調整よりも小さく、スコア全体が減少し始めます。実行中の ES 線の頂点は、垂直点線で示されます。これは経路全体のESであり、経路が選択され、ラグプロットとして提示されているかどうかを判断するためにプログラムによって使用されます。
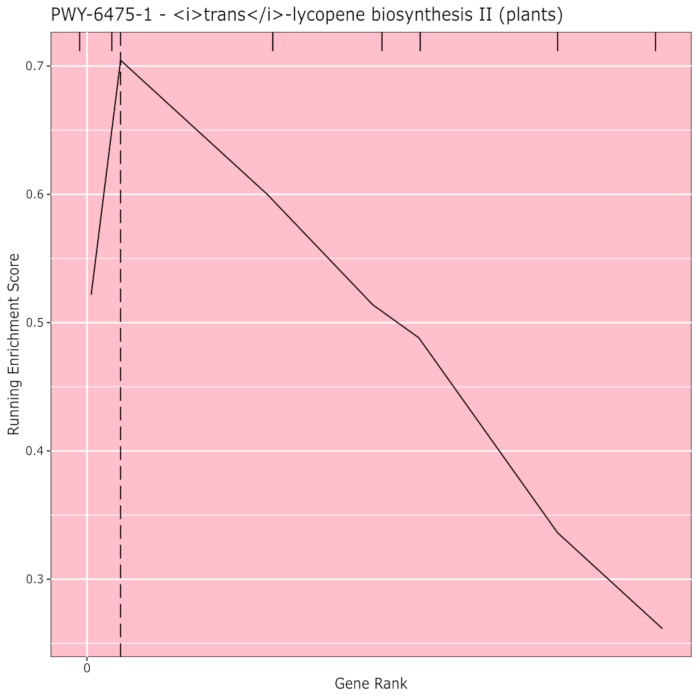
図9:PASTの実行が完了した(またはシャイニーからダウンロードされた)からの経路画像。 この図は、スラッシュら10から引用されています。 この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
PASTの主な目標は、GWASデータの代謝経路分析を、特に非ヒトおよび非動物生物に対してより多くの聴衆に持ち込むことである。PAST の代替方法は、多くの場合、人間や動物に焦点を当てたコマンドラインプログラムです。ユーザーフレンドリさは、光沢のあるアプリケーションを開発することを選択することと、Rとバイオコンダクターを使用してアプリケーションをリリースするという点の両方で、PASTの開発における主要な目標でした。ユーザーは PAST を使用するためにプログラムをコンパイルする方法を学ぶ必要はありません。
ほとんどのタイプの解析ソフトウェアと同様に、PAST の結果は入力データと同じ結果にすぎません。入力データにエラーがある場合や、形式が誤っている場合、PAST は実行に失敗するか、または情報が得られない結果を生成します。GWAS データ、LD データ、アノテーション、およびパス・ファイルが正しくフォーマットされていることを確認することは、PAST から正しい出力を受け取るうえで重要です。PAST は、バイアラリック マーカーのみを分析し、入力データのセットごとに 1 つの特性のみを実行できます。さらに、遺伝子型入力が不十分、誤った、または不正確な型定義によって生成されたGWASデータは、明確または再現可能な結果を生み出す可能性は低い。PASTはGWASの結果の生物学的解釈に役立ちますが、環境変動、実験的エラー、または人口構造が適切に考慮されなかった場合、混沌としたデータセットを明確にすることはほとんどありません。
ユーザーは、シャイニーアプリケーションと、それらのパラメータをRコンソールのPASTの関数に渡すことによって、分析の一部のパラメータを変更することができます。これらのパラメータは PAST によって報告される結果を変更する可能性があり、ユーザーはデフォルトからこれらの結果を変更する際に注意する必要があります。LDはユーザによって測定されるので、通常はGWASでも使用されていたのと同じマーカーデータセットを使用するため、LD測定値は母集団に固有である。すべての研究、特にトウモロコシ以外の種(特に自己受粉、多発性、または非常に異種種)については、デフォルトの変化が保証される可能性があります。
開示事項
著者らは開示するものは何もない。
謝辞
何一つ。
資料
| Name | Company | Catalog Number | Comments |
| Computer | NA | NA | Any computer with 8GB RAM should be sufficient |
| R | R Project | NA | R 4.0 or greater is required to install from Bioconductor 3.11 |
参考文献
- Rafalski, J. Association genetics in crop improvement. Current Opinion in Plant Biology. 13 (2), 174-180 (2010).
- Yan, J., Warburton, M., Crouch, J. Association Mapping for Enhancing Maize (Zea mays L.) Genetic Improvement. Crop Science. 51 (2), 433-449 (2011).
- Xiao, Y., Liu, H., Wu, L., Warburton, M., Yan, J. Genome-wide Association Studies in Maize: Praise and Stargaze. Molecular Plant. 10 (3), 359-374 (2017).
- Wang, K., Li, M., Bucan, M. Pathway-Based Approaches for Analysis of Genomewide Association Studies. The American Journal of Human Genetics. 81 (6), 1278-1283 (2007).
- Weng, L., et al. SNP-based pathway enrichment analysis for genome-wide association studies. BMC Bioinformatics. 12 (1), 99(2011).
- Tang, J., Perkins, A., Williams, W., Warburton, M. Using genome-wide associations to identify metabolic pathways involved in maize aflatoxin accumulation resistance. BMC Genomics. 16 (1), 673(2015).
- Warburton, M., et al. Genome-Wide Association Mapping of Aspergillus flavus and Aflatoxin Accumulation Resistance in Maize. Crop Science. 55 (5), 1857-1867 (2015).
- Warburton, M., et al. Genome-Wide Association and Metabolic Pathway Analysis of Corn Earworm Resistance in Maize. The Plant Genome. 11 (1), 170069(2018).
- Li, H., Thrash, A., Tang, J., He, L., Yan, J., Warburton, M. Leveraging GWAS data to identify metabolic pathways and networks involved in maize lipid biosynthesis. The Plant Journal. 98 (5), 853-863 (2019).
- Thrash, A., Tang, J., DeOrnellis, M., Peterson, D., Warburton, M. PAST: The Pathway Association Studies Tool to Infer Biological Meaning from GWAS Datasets. Plants. 9 (1), 58(2020).
- Adam, T., Mason, D. PAST: Pathway Association Study Tool (PAST). Bioconductor version: Release (3.10). , (2020).
- Thrash, A., DeOrnellis, M. IGBB/PAST. , at https://github.com/IGBB/PAST (2019).
- Bradbury, P., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Subramanian, A., et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences U.S.A. 102, 15545-15550 (2005).
Erratum
Formal Correction: Erratum: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information
Posted by JoVE Editors on 10/08/2021. Citeable Link.
An erratum was issued for: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information. One of the affiliations was updated.
The second affiliation was updated from:
USDA-ARS Corn Host Plant Resistance Research Unit, Mississippi State University
to:
Corn Host Plant Resistance Research Unit, USDA-ARS
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved