Method Article
כלי מחקר של איגוד הנתיבים עבור ניתוחי GWAS של מידע על מסלול מטבולי
In This Article
Erratum Notice
Summary
על ידי הפעלת כלי המחקר של איגוד הנתיבים (PAST), באמצעות היישום הנוצץ או באמצעות קונסולת R, החוקרים יכולים לקבל הבנה עמוקה יותר של המשמעות הביולוגית של תוצאות מחקר הקשר הגנום שלהם (GWAS) על ידי חקירת המסלולים המטבוליים המעורבים.
Abstract
לאחרונה, יישום חדש של שיטה שתוארה בעבר לפענוח נתוני מחקר איגוד גנום רחב (GWAS) באמצעות ניתוח מסלול מטבולי פותח ושוחרר. כלי המחקר של איגוד הנתיבים (PAST) פותח כדי לטפל בדאגות עם ידידותיות למשתמש ניתוחים איטיים. כלי חדש וידידותי למשתמש זה שוחרר על Bioconductor ו Github. בבדיקות, PAST ביצעה ניתוחים בפחות משעה שדרשו בעבר עשרים וארבע שעות או יותר. במאמר זה, אנו מציגים את הפרוטוקול לשימוש ביישום מבריק או במסוף R להפעלת PAST.
Introduction
מחקרי אסוציאציה כלל-גנומית (GWAS) הם שיטה פופולרית לחקר תכונות מורכבות והאזורים הגנומיים הקשורים אליהם1,2,3. בסוג זה של מחקר, מאות אלפי סמני פולימורפיזם נוקלאוטיד יחיד (SNP) נבדקים על הקשר שלהם עם התכונה, ואת המשמעות של האסוציאציות מוערך. שיוכים בעלי תכונות סמן העונים על סף שיעור גילוי כוזב (FDR) (או סוג אחר של סף משמעות) נשמרים עבור המחקר, אך ניתן לסנן שיוכים אמיתיים. עבור תכונות מורכבות, פוליגניות, ההשפעה של כל גן עשויה להיות קטנה (ובכך מסוננת החוצה), וכמה אללים באים לידי ביטוי רק בתנאים ספציפיים שעשויים שלא להיות נוכחים במחקר3. לכן, בעוד SNPs רבים עשויים להישמר כמזוהה עם התכונה, כל אחד עשוי להיות השפעה קטנה מאוד. יותר מדי שיחות SNP יחסרו, ופרשנות של המשמעות הביולוגית והארכיטקטורה הגנטית של התכונה עשויה להיות לא שלמה ומבלבלת. ניתוח מסלול מטבולי יכול לעזור לטפל בכמה מבעיות אלה על ידי התמקדות בהשפעות המשולבות שלגניםהמקובצים לפי הפונקציה הביולוגית שלהם 4,5,6.
מספר מחקרים הושלמו באמצעות יישום קודם של השיטה המתוארת במאמר זה. הצטברות Aflatoxin7, התנגדות תולעי אוזני תירס8, וביוסינטזה שמן9 נחקרו כולם עם היישום הקודם. בעוד שניתוחים אלה היו מוצלחים, תהליך הניתוח היה מסובך, גוזל זמן ומסורבל, מכיוון שכלי הניתוח נכתבו בשילוב של R, פרל ובאש, והצינור לא היה אוטומטי. בגלל הידע המיוחד הנדרש לשינוי שיטה זו עבור כל ניתוח, פותחה כעת שיטה חדשה שניתן לשתף עם חוקרים אחרים.
כלי הלימוד של איגוד הנתיבים (PAST)10 נועד לטפל בחסרונות של השיטה הקודמת על ידי דרישת פחות ידע בשפות תכנות ועל ידי הפעלת ניתוחים בתקופה קצרה יותר. בעוד שהשיטה נבדקה בתירס, PAST אינה מניחה הנחות ספציפיות למינים. העבר יכול להיות מופעל באמצעות קונסולת R, כאפליקציה מבריקה, וגרסה מקוונת צפויה להיות זמינה בקרוב ב- MaizeGDB.
Protocol
1. התקנה
- התקן את R, אם הוא עדיין לא מותקן.
הערה: PAST כתוב ב- R, ולכן דורש שהמשתמשים שלו יתקנו R. בעת כתיבת שורות אלה, התקנת PAST ישירות מ- Bioconductor דורשת R4.0. ניתן להתקין גרסאות ישנות יותר של PAST מ- Bioconductor עבור R3.6, וניתן להתקין את PAST מגיתוב עבור משתמשים עם R3.5. ניתן להוריד הוראות התקנה R מהקישור הבא: https://www.r-project.org/. - התקן את הגירסה העדכנית ביותר של RStudio שולחן העבודה או עדכן RStudio (אופציונלי).
הערה: RStudio היא סביבה מועילה לעבודה עם שפת ה- R. ההתקנה שלו מומלצת, במיוחד עבור אלה שבוחרים להפעיל את PAST בשורת הפקודה ולא באמצעות יישום GUI מבריק. RStudio והוראות ההתקנה שלה ניתן למצוא בקישור הבא: https://rstudio.com/products/rstudio/. - התקן את PAST מ- Bioconductor11 על-ידי ביצוע ההוראות בנושא מוליך ביולוגי.
הערה: התקנה באמצעות מוליך ביולוגי אמורה לטפל בהתקנת יחסי התלות של PAST. בנוסף, ניתן להתקין את PAST מגיתוב12, אך התקנה מ- Github לא תתקין יחסי תלות באופן אוטומטי. - התקן את PAST מבריק (אופציונלי). הורד את הקובץ "אפליקציה. R" מהדף מהדורות של מאגר Github: https://github.com/IGBB/PAST/releases/, וזכרו היכן ממוקם הקובץ שהורד.
הערה: בעבר ניתן להשתמש על ידי קורא השיטות שלה ישירות עם R, אבל משתמשים שמכירים פחות R יכול להפעיל את היישום בעבר מבריק, המספק ממשק משתמש מודרך. PAST Shiny הוא סקריפט R הזמין בסניף shiny_app של מאגר גיטוב בעבר. בעבר מבריק ינסה להתקין את יחסי התלות שלה במהלך ההפעלה הראשונה. - התחל בניתוח על-ידי הפעלת היישום באחת משלוש הדרכים המתוארות להלן.
- עבר מבריק עם RStudio
- באמצעות RStudio, צור פרוייקט חדש בתיקיה שבה היישום. R ממוקם. לחץ על קובץ | פרוייקט חדש ובחר תיקיה זו.
- לאחר יצירת פרוייקט חדש, פתח את היישום. קובץ R שהורד קודם לכן. RStudio מזהה את האפליקציה הזו. R היא אפליקציה מבריקה ויוצרת לחצן הפעל יישום בסרגל שמעל קוד המקור המוצג. לחץ על הפעל יישום. לאחר מכן RStudio תשיק חלון המציג את היישום PAST Shiny.
- מעבר מבריק עם מסוף R
- הפעל R והפעל את הקוד הבא כדי להפעיל את היישום המבריק של העבר: מבריק::runApp('נתיב/אל/תיקיה/עם/מבריק/אפליקציה. ר'. החלף את הטקסט במרכאות בתיקיה שאליה האפליקציה. R הורד, ולשמור את הציטוטים.
- עבר ללא R מבריק
- הפעל ספריה(PAST) במסוף R כדי לטעון את PAST.
- עבר מבריק עם RStudio
2. התאמה אישית של ניתוח מבריק (אופציונלי)
- שנה את כותרת הניתוח מ"ניתוח חדש" למשהו שמשקף טוב יותר את סוג הניתוח המנוהל, המסייע לעקוב אחר ניתוחים מרובים (ראו איור 1).
איור 1. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- שנה את מספר הליבות ואת המצב. הגדר את מספר הליבות למספר כלשהו בין 1 למספר הכולל במחשב, אך שים לב לכך שהעברת משאבים נוספים ל- PAST עשויה להאט פעולות אחרות במחשב. הגדר את המצב בהתבסס על התיאור בסעיף 6.
3. טען נתוני GWAS
הערה: ודא כי נתוני GWAS מופרדים באמצעות כרטיסיות. ודא שקובץ השיוך מכיל את העמודות הבאות: תכונה, שם סמן, לוקוס או כרומוזום, מיקום על ערך הכרומוזום, p-value ו- R2 עבור הסמן. ודא שקובץ האפקטים מכיל את העמודות הבאות: תכונה, שם סמן, לוקוס או כרומוזום, מיקום על הכרומוזום ואפקט. סדר עמודות אלה אינו חשוב, שכן המשתמש יכול לציין את שמות העמודות בעת טעינת הנתונים. המערכת מתעלמת מעמודות נוספות. ניתן להשתמש ב- TASSEL13 כדי להפיק קבצים אלה.
- טען נתוני GWAS באמצעות מעבר מבריק.
- בחר קובץ שיוך וקובץ אפקטים באמצעות תיבות הבחירה קובץ שיוך וקובץ אפקטים. שנה את שמות העמודות בתיבות הקלט שם עמודת שיוך ועמודות אפקטים מתחת לתיבות בחירת הקבצים כדי לשקף את שמות העמודות בנתונים.
איור 2. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- טען נתוני GWAS עם PAST במסוף R.
- שנה והפעל את הקוד הבא:
gwas_data = load_GWAS_data("path/to/association_file.tsv", "path/to/effects_file.tsv", association_columns = c("תכונה", "Marker", "לוקוס", "אתר", "p", "marker_R2"), effects_columns = c("תכונה", "מרקר", "לוקוס", "אתר", "אפקט")
- שנה והפעל את הקוד הבא:
- הערה: שנה את הנתיבים למיקום הממשי של קבצי GWAS. הערכים המסופקים עבור association_columns effects_columns הם ערכי ברירת המחדל. אם השמות אינם תואמים לערכי ברירת המחדל, ציין את שמות העמודות. אחרת, ניתן להשמיט אותם.
4. נתוני אי-שוויון הצמדה לטעינה (LD)
הערה: ודא כי נתוני אי-שוויון ההצמדה (LD) מופרדים באמצעות כרטיסיות ומכילה את סוגי הנתונים הבאים: לוקוס, מיקום1, Site1, Position2, Site2, מרחק בזוגות בסיס בין מיקום1 ל- Position2 וערך R2.
- טען נתוני LD באמצעות מעבר מבריק.
- בחר את הקובץ המכיל נתוני LD. שנה את שמות העמודות בתיבות הקלט שמות עמודות LD מתחת לתיבת בחירת הקבצים כך שיתאימו לשמות העמודות בנתוני LD במידת הצורך.
איור 3. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- טען נתוני LD עם PAST במסוף R.
- שנה והפעל את הקוד הבא כדי לטעון נתוני LD:
LD = load_LD("path/to/LD.tsv", LD_columns = c("Locus1", "Position1", "Site1", "Position2", "Site2", "Dist_bp", "R.2")
הערה: שנה את הנתיב למיקום הממשי של קובץ LD. הערכים המסופקים עבור LD_columns הם ערכי ברירת המחדל. אם השמות אינם תואמים לברירות מחדל אלה, ציין את השמות הנכונים של העמודות; אחרת, ניתן להשמיט אותם.
- שנה והפעל את הקוד הבא כדי לטעון נתוני LD:
5. הקצה SNPs לגנים
הערה: הורד או אתר ביאורים באופן אחר בתבנית GFF. ביאורים אלה ניתן למצוא לעתים קרובות במסדי נתונים מקוונים עבור אורגניזמים ספציפיים. היזהרו לגבי ביאורים באיכות נמוכה, שכן איכות נתוני הביאורים תשפיע על איכות ניתוח המסלול. ודא שהעמודה הראשונה של ביאורים אלה (הכרומוזום) תואמת לתבנית הלוקוס/כרומוזום בנתוני השיוך, האפקטים וה- LD. לדוגמה, הביאורים לא צריכים לקרוא לכרומוזום הראשון "chr1" אם קבצי הנתונים GWAS ו- LD מכנים את הכרומוזום הראשון "1".
- הקצה SNPs לגנים עם בעבר מבריק.
הערה: מידע נוסף על קביעת חתך R2 מתאים ניתן למצוא טאנג ואח'6, בסעיף שנקרא "SNP לאלגוריתם גנים לניתוח המסלול".- בחר את הקובץ המכיל ביאורים GFF. שקול איזה גודלחלון וחיתוך R 2 מתאימים ביותר למינים הנלקחים בחשבון ולשנות אם ברירות המחדל אינן מתאימות לנתונים שהועלו.
הערה: ערכי ברירת המחדל ב- PAST משקפים בעיקר ערכים המתאימים לתירס. מספר הליבות שנקבעו בתחילת ניתוח ה- PAST Shiny (שלב 2.2) משמש בשלב זה.
- בחר את הקובץ המכיל ביאורים GFF. שקול איזה גודלחלון וחיתוך R 2 מתאימים ביותר למינים הנלקחים בחשבון ולשנות אם ברירות המחדל אינן מתאימות לנתונים שהועלו.
איור 4. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- הקצה SNPs לגנים עם PAST במסוף R.
- שנה והפעל את הקוד הבא כדי להקצות SNPs לגנים:
גנים = assign_SNPs_to_genes(gwas_data, LD, "path/to/anotations.gff", c("גן"), 1000, 0.8, 2)
הערה: בקוד לדוגמה זה, מסופקות מספר הצעות ברירת מחדל: 1000 הוא גודל החלון סביב SNP כדי לחפש גנים; 0.8 הוא ערך הקיצוץ עבור R2; 2 הוא מספר הליבות המשמשות לעיבוד מקבילי. יש לשנות את הנתיב לביאורים גם למיקום הממשי של קובץ הביאורים.
- שנה והפעל את הקוד הבא כדי להקצות SNPs לגנים:
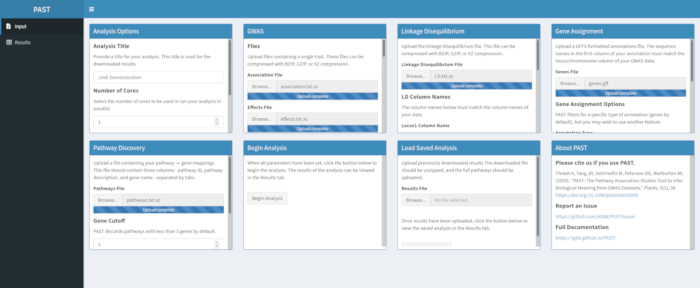
6. גלה מסלולים משמעותיים
הערה: ודא שקובץ הנתיבים מכיל את הנתונים הבאים בתבנית מופרדת באמצעות טאבים, עם שורה אחת עבור כל גן בכל מסלול: מזהה מסלול - מזהה כגון "PWY-6475-1"; תיאור מסלול - תיאור ארוך יותר של מה המסלולים לעשות כגון "טרנס-ליקופן biosynthesis"; גן - גן במסלול, אשר צריך להתאים את השמות המסופקים ביאורים. מידע מסלול ניתן למצוא ככל הנראה במסדי נתונים מקוונים עבור אורגניזמים ספציפיים, כגון MaizeGDB. האפשרות השנייה שצוינה על-ידי המשתמש היא המצב. "הגדלת" מתייחס פנוטיפים המשקפים כאשר ערך עולה של התכונה הנמדדת רצוי, כגון תשואה, בעוד "ירידה" מתייחס תכונה שבה ירידה בערכים הנמדדים מועילה, כגון דירוגי נזק חרקים. המשמעות של מסלולים נבדקת בשיטות שתוארו בעבר4,6,14.
- גלה מסלולים משמעותיים עם עבר מבריק.
- בחר את הקובץ המכיל נתוני מסלולים וודא שהמצב נבחר באפשרויות הניתוח. במידת הצורך, שנה את מספר הגנים שחייבים להיות במסלול כדי לשמור אותו לניתוח ואת מספר התמורות המשמשות ליצירת התפלגות ה- Null כדי לבדוק את משמעות ההשפעה.
איור 5. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
הערה: מספר הליבות והמצב שהוגדר בתחילת ניתוח ה- PAST Shiny (שלב 2.2) משמש בשלב זה. מספר ברירת המחדל של הגנים מוגדר כיום על 5 גנים, כך שמסלולים עם פחות גנים ידועים יוסרו. המשתמש יכול להוריד ערך זה ל- 4 או 3, כדי לכלול מסלולים קצרים יותר, אך פעולה זו תסתכן בתוצאות חיוביות שגויות. הגדלת ערך זה יכולה להגדיל את כוח הניתוח אך תסיר מסלולים נוספים מהניתוח. שינוי מספר התמורות המשמשות מגדיל ומקטין את כוח הבדיקה.
- גלה מסלולים משמעותיים עם PAST במסוף R.
- שנה והפעל את הקוד הבא כדי לגלות מסלולים משמעותיים:
rugplots_data <- find_pathway_significance (גנים, "path/to/paths.tsv", 5, "גדל", 1000, 2)
הערה: בקוד לדוגמה זה, מסופקות מספר ברירות מחדל מוצעות. 5 הוא המספר המינימלי של גנים שחייבים להיות במסלול כדי לשמור על המסלול בניתוח, הגדלת מתייחסת לכמות הולכת וגדלה של התכונה הנמדדת (מומלץ שהמשתמש יפעל הן גדל והן יורד, ללא קשר לתכונה; פרשנות נתונים תהיה שונה עבור השניים, עם זאת), 1000 הוא מספר הפעמים לדגום את ההשפעות כדי לקבוע את התפלגות האפס, ו- 2 הוא מספר הליבות המשמשות לעיבוד מקבילי. שנה את הנתיב למיקום הממשי של קובץ הנתיבים.
- שנה והפעל את הקוד הבא כדי לגלות מסלולים משמעותיים:
7. צפו ברוגפלוטס
- צפה ברוגפלוטס עם עבר מבריק.
- לאחר העלאת כל הקלטים והגדרתם,לחץ על התחל ניתוח . מד התקדמות יופיע ויציין איזה שלב של הניתוח הושלם לאחרונה. לאחר השלמת הניתוח, PAST Shiny יעבור לכרטיסיה תוצאות. בעמודה השמאלית (שכותרתה "מסלולים") תוצג טבלת תוצאות (הנקראת "נתיבים") ו-Rugplots יוצגו בעמודה הימנית (שכותרתה "התוויות").
- השתמש במחוון כדי לשלוט בפרמטרים של הסינון. כאשר רמת הסינון משביעת רצון, לחץ על לחצן הורד תוצאות בפינה הימנית התחתונה כדי להוריד את כל התמונות והטבלאות בנפרד לקובץ ZIP ששמו מופיע עם כותרת הניתוח. קובץ ZIP זה מכיל את הטבלה המסוננת, הטבלה הלא מסוננת ותמונה אחת לכל מסלול בטבלה המסוננת.
איור 6. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
איור 7. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- הצגת רוגלוטים עם עבר בקונסולת R
- שנה והפעל את הקוד הבא כדי לשמור את התוצאות:
plot_pathways(rugplots_data, "pvalue", 0.02, "גדל", "output_folder")
הערה: בקוד לדוגמה זה, מסופקות מספר ברירות מחדל מוצעות. pvalue מספק את הנתונים שניתן להשתמש בהם לסינון מסלולים לא משמעותיים לאחר בחירת סף משמעות על-ידי המשתמש; 0.02 הוא ערך ברירת המחדל המשמש לסינון, והגדלה מתייחסת לכמות הולכת וגדלה של התכונה הנמדדת (מומלץ שהמשתמש יפעל הן גדל והן יורד, ללא קשר לתכונה; פרשנות הנתונים תהיה שונה עבור השניים, עם זאת); output_folder היא התיקיה שבה יכתבו התמונות והטבלאות (תיקיה זו חייבת להתקיים לפני הפעלת הפונקציה). טבלה של תוצאות מסוננות, התוצאות הלא מסוננות ותמונות בודדות עבור כל מסלול בתוצאות המסוננות נכתבות לתיקיה זו.
- שנה והפעל את הקוד הבא כדי לשמור את התוצאות:
תוצאות
אם התוצאות אינן מיוצרות לאחר הפעלה של כלי התוכנה PAST, ודא שכל קבצי הקלט מעוצבים כראוי. ריצה מוצלחת באמצעות הנתונים לדוגמה בחבילת PAST, המבוססים על תירס GWAS בצבע תבואה, מוצגת באיור 8. ניתן להוריד טבלה זו ואת התמונה המתקבלת באמצעות לחצן הורד תוצאות. דוגמה לתמונה שהורדה מוצגת באיור 210. הגדרות שגויות עשויות להוביל לתוצאות שאינן הגיוניות ביולוגית, אך קביעת אי-תקינות חייבת להיות תלויה אצל החוקר, שצריך לבדוק שוב את תוקפן של ההגדרות שנבחרו ולשקול את כל הראיות הידועות לגבי תכונת העניין.
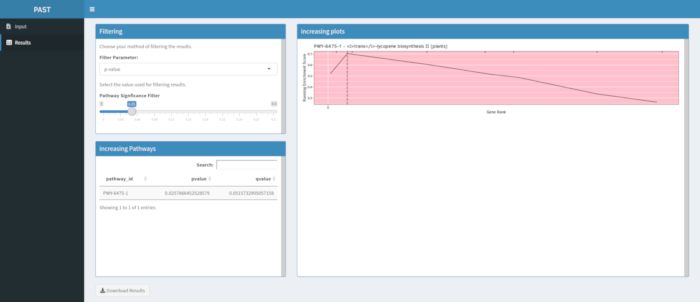
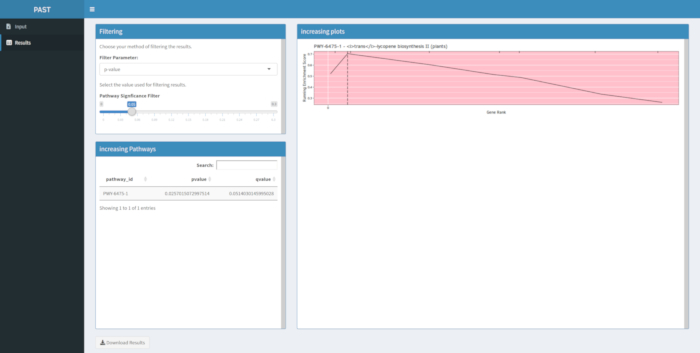
איור910 מציג את הרוגלוט שהופק מניתוח המסלול של תוצאות GWAS שנוצרו עם לוח תירס של 288 קווים מלידה שהופקו לצבע התבואה. דוגמה פשטנית זו, שבה הפנוטיפים היו "לבנים" או "צהובים", שימשה מכיוון שהמסלול האחראי ליצירת פיגמנטים קרוטנואידים צהובים בהירים ידוע וצריך להיות אחראי לרוב הפנוטיפ. לכן, ציפינו לראות את מסלול הביוסינתזה טרנס-ליקופן (המייצר קרוטנואידים) להיות קשור באופן משמעותי עם צבע התבואה, וזה. מזהה הנתיב והשם מפורטים בחלק העליון של הגרף. הציר האופקי של הגרף מדרג את כל הגנים שנכללו בניתוח, המסודרים משמאל לימין לפי סדר ההשפעה הגדולה ביותר על התכונה לקטנה ביותר. עם זאת, רק הגנים במסלול הביוסינתזה הטרנס-ליקופן מסומנים (בחלק העליון של הגרף, כסימני צוהר, המופיעים בדירוג הגנים של השפעתם בהשוואה לכל הגנים האחרים בניתוח). יש 7 גנים במסלול הזה. ציון העשרת הריצה (ES) מתוות לאורך הציר האנכי. ה- ES עבור כל גן מתווסף לסך הכל הפועל לפי סדר ההשפעה והסכום הכולל מותאם למספר הגנים שנותחו. לכן, הציון משתנה ככל שאדם נע ממש לאורך הציר האופקי ונוטה לגדול ככל שהגנים בעלי ההשפעה הגדולה יותר כלולים, אך בשלב מסוים, הגידול בהשפעה קטן יותר מההתאמה להוספת גן אחר, והציון כולו מתחיל לרדת. פסגת קו ה- ES הפועל מסומנת בקו אנכי מנוקד; זהו ES עבור המסלול כולו והוא משמש את התוכנית כדי לקבוע אם המסלול נבחר ומוצג כמו רוגלוט.
איור 8: הריצה המוגמרת של PAST Shin. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
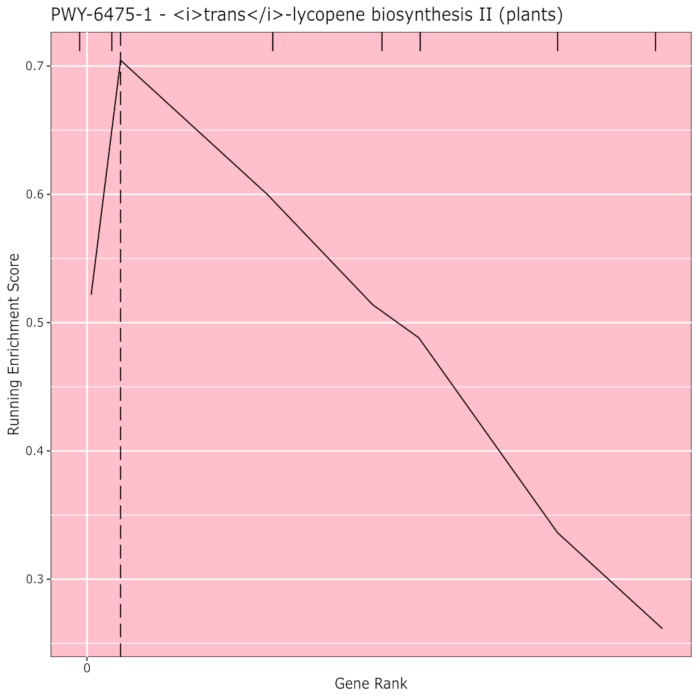
איור 9: תמונת מסלול מהפעלה שהושלמה של PAST (או הורדה מ-Shin). נתון זה צוטט מתרש ואח '10. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
המטרה העיקרית של העבר היא להביא ניתוחי מסלול מטבוליים של נתוני GWAS לקהל רחב יותר, במיוחד עבור אורגניזמים שאינם אנושיים ולא בעלי חיים. שיטות חלופיות ל-PAST הן לעתים קרובות תוכניות שורת פקודה המתמקדות בבני אדם או בבעלי חיים. ידידותיות למשתמש הייתה מטרה עיקרית בפיתוח של PAST, הן בבחירה לפתח יישום מבריק והן בבחירה להשתמש R ו Bioconductor לשחרר את היישום. משתמשים אינם צריכים ללמוד כיצד לבצע הידור תוכניות כדי להשתמש ב- PAST.
כמו ברוב סוגי תוכנות הניתוח, התוצאות של PAST טובות רק כמו נתוני הקלט; אם נתוני הקלט כוללים שגיאות או מעוצבים באופן שגוי, הפונקציה PAST לא תפעל או תפיק תוצאות לא חשובות. הקפדה על עיצוב נכון של נתוני GWAS, נתוני LD, ביאורים ומסלולים היא קריטית לקבלת פלט נכון מ- PAST. PAST מנתחת סמנים דו-אלליים בלבד ויכולה להפעיל תכונה אחת בלבד עבור כל קבוצה של נתוני קלט. בנוסף, נתוני GWAS המיוצרים על ידי genotyping המסכן או פנוטיפינג שגוי או לא מדויק אינו צפוי לייצר תוצאות ברורות או חוזרות על עצמן. העבר יכול לסייע בפרשנות הביולוגית של תוצאות GWAS, אך לא סביר שיבהיר ערכות נתונים כאוטיות אם וריאציה סביבתית, שגיאה ניסיונית או מבנה האוכלוסייה לא נלקחו בחשבון כראוי.
משתמשים יכולים לבחור לשנות פרמטרים מסוימים של הניתוח, הן ביישום Shiny והן על-ידי העברת פרמטרים אלה לפונקציות של PAST במסוף R. פרמטרים אלה יכולים לשנות את התוצאות שדווחו על-ידי PAST, ועל המשתמשים לדאוג בעת שינוי אלה מברירות המחדל. מאחר ש- LD נמדד על-ידי המשתמשים, בדרך כלל באמצעות אותה ערכת נתוני סמן ששימשה גם ב- GWAS, מדידות LD ספציפיות לאוכלוסייה. עבור כל המחקרים, במיוחד עבור מינים שאינם תירס, (במיוחד האבקה עצמית, פוליפלואיד, או מינים הטרוגניים מאוד), שינויים בברירות המחדל עשויים להיות מוצדקים.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
ללא.
Materials
| Name | Company | Catalog Number | Comments |
| Computer | NA | NA | Any computer with 8GB RAM should be sufficient |
| R | R Project | NA | R 4.0 or greater is required to install from Bioconductor 3.11 |
References
- Rafalski, J. Association genetics in crop improvement. Current Opinion in Plant Biology. 13 (2), 174-180 (2010).
- Yan, J., Warburton, M., Crouch, J. Association Mapping for Enhancing Maize (Zea mays L.) Genetic Improvement. Crop Science. 51 (2), 433-449 (2011).
- Xiao, Y., Liu, H., Wu, L., Warburton, M., Yan, J. Genome-wide Association Studies in Maize: Praise and Stargaze. Molecular Plant. 10 (3), 359-374 (2017).
- Wang, K., Li, M., Bucan, M. Pathway-Based Approaches for Analysis of Genomewide Association Studies. The American Journal of Human Genetics. 81 (6), 1278-1283 (2007).
- Weng, L., et al. SNP-based pathway enrichment analysis for genome-wide association studies. BMC Bioinformatics. 12 (1), 99(2011).
- Tang, J., Perkins, A., Williams, W., Warburton, M. Using genome-wide associations to identify metabolic pathways involved in maize aflatoxin accumulation resistance. BMC Genomics. 16 (1), 673(2015).
- Warburton, M., et al. Genome-Wide Association Mapping of Aspergillus flavus and Aflatoxin Accumulation Resistance in Maize. Crop Science. 55 (5), 1857-1867 (2015).
- Warburton, M., et al. Genome-Wide Association and Metabolic Pathway Analysis of Corn Earworm Resistance in Maize. The Plant Genome. 11 (1), 170069(2018).
- Li, H., Thrash, A., Tang, J., He, L., Yan, J., Warburton, M. Leveraging GWAS data to identify metabolic pathways and networks involved in maize lipid biosynthesis. The Plant Journal. 98 (5), 853-863 (2019).
- Thrash, A., Tang, J., DeOrnellis, M., Peterson, D., Warburton, M. PAST: The Pathway Association Studies Tool to Infer Biological Meaning from GWAS Datasets. Plants. 9 (1), 58(2020).
- Adam, T., Mason, D. PAST: Pathway Association Study Tool (PAST). Bioconductor version: Release (3.10). , (2020).
- Thrash, A., DeOrnellis, M. IGBB/PAST. , at https://github.com/IGBB/PAST (2019).
- Bradbury, P., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Subramanian, A., et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences U.S.A. 102, 15545-15550 (2005).
Erratum
Formal Correction: Erratum: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information
Posted by JoVE Editors on 10/08/2021. Citeable Link.
An erratum was issued for: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information. One of the affiliations was updated.
The second affiliation was updated from:
USDA-ARS Corn Host Plant Resistance Research Unit, Mississippi State University
to:
Corn Host Plant Resistance Research Unit, USDA-ARS
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved