Method Article
Инструмент исследования ассоциаций путей для анализа GWAS информации о метаболических путях
В этой статье
Erratum Notice
Резюме
Запустив Pathway Association Study Tool (PAST), либо через приложение Shiny, либо через консоль R, исследователи могут получить более глубокое понимание биологического значения результатов своего общегеномного ассоциативного исследования (GWAS), исследуя вовлеченные метаболические пути.
Аннотация
Недавно была разработана и выпущена новая реализация ранее описанного метода интерпретации данных общегеномного ассоциативного исследования (GWAS) с использованием анализа метаболических путей. Инструмент исследования Ассоциации Pathway (PAST) был разработан для решения проблем, связанных с удобством для пользователя и медленным анализом. Этот новый удобный инструмент был выпущен на Bioconductor и Github. В тестировании PAST проводил анализы менее чем за один час, что ранее требовало двадцати четырех или более часов. В этой статье мы представляем протокол для использования приложения Shiny или консоли R для запуска PAST.
Введение
Общегеномные ассоциативные исследования (GWAS) являются популярным методом изучения сложных признаков и связанных с ними геномныхобластей 1,2,3. В этом типе исследования сотни тысяч маркеров однонуклеотидного полиморфизма (SNP) проверяются на их связь с признаком, и оценивается значимость ассоциаций. Ассоциации маркеров-признаков, которые соответствуют порогу ложного уровня обнаружения (FDR) (или какому-либо другому типу порога значимости), сохраняются для исследования, но истинные ассоциации могут быть отфильтрованы. Для сложных полигенных признаков эффект каждого гена может быть небольшим (и, следовательно, отфильтрованным), а некоторые аллели экспрессируются только в определенных условиях, которые могут отсутствовать в исследовании3. Таким образом, хотя многие SMP могут быть сохранены как связанные с признаком, каждый из них может иметь очень небольшой эффект. Слишком много вызовов SNP будет отсутствовать, а интерпретация биологического значения и генетической архитектуры признака может быть неполной и запутанной. Анализ метаболических путей может помочь решить некоторые из этих проблем, сосредоточив внимание на комбинированных эффектах генов, сгруппированных в соответствии с их биологической функцией4,5,6.
Несколько исследований были завершены с использованием предыдущей реализации метода, описанного в этой статье. Накопление афлатоксина7,резистентность к кукурузному ушному червю8и биосинтез масла9 были изучены с предыдущей реализацией. Хотя эти анализы были успешными, процесс анализа был сложным, трудоемким и громоздким, потому что инструменты анализа были написаны в комбинации R, Perl и Bash, а конвейер не был автоматизирован. Из-за специализированных знаний, необходимых для модификации этого метода для каждого анализа, в настоящее время разработан новый метод, которым можно поделиться с другими исследователями.
Инструмент изучения Pathway Association (PAST)10 был разработан для устранения недостатков предыдущего метода, требуя меньшего знания языков программирования и проводя анализ за более короткий период. Хотя метод был протестирован на кукурузе, PAST не делает никаких видовых предположений. PAST можно запускать через консоль R, как приложение Shiny, и ожидается, что онлайн-версия скоро будет доступна на MaizeGDB.
протокол
1. Настройка
- Установите R, если он еще не установлен.
ПРИМЕЧАНИЕ: PAST написан на R и, следовательно, требует, чтобы у его пользователей был установлен R. На момент написания этой статьи для установки PAST непосредственно из Bioconductor требуется R4.0. Старые версии PAST могут быть установлены из Bioconductor для R3.6, а PAST могут быть установлены с Github для пользователей с R3.5. Инструкцию по установке R можно скачать по следующей ссылке: https://www.r-project.org/. - Установите последнюю версию RStudio Desktop или обновите RStudio (необязательно).
ПРИМЕЧАНИЕ: RStudio является полезной средой для работы с языком R. Рекомендуется его установка, особенно для тех, кто предпочитает запускать PAST в командной строке, а не через приложение Shiny GUI. RStudio и инструкции по его установке можно найти по следующей ссылке: https://rstudio.com/products/rstudio/. - Установите PAST из Биопроводника11, следуя инструкциям на Биопроводнике.
ПРИМЕЧАНИЕ: Установка через Bioconductor должна обрабатывать установку зависимостей PAST. Кроме того, PAST можно установить с Github12,но установка с Github не будет устанавливать зависимости автоматически. - Установите PAST Shiny (необязательно). Скачайте файл "app. R" со страницы Релизы репозитория Github: https://github.com/IGBB/PAST/releases/ и запомнить, где находится загруженный файл.
ПРИМЕЧАНИЕ: PAST можно использовать, вызывая его методы непосредственно с помощью R, но пользователи, которые менее знакомы с R, могут запускать приложение PAST Shiny, которое предоставляет управляемый пользовательский интерфейс. PAST Shiny — это скрипт R, доступный в ветви shiny_app репозитория PAST Github. PAST Shiny попытается установить свои зависимости во время первого запуска. - Начните анализ с запуска приложения одним из трех способов, описанных ниже.
- PAST Shiny с RStudio
- С помощью RStudio создайте новый проект в папке, в которой находится приложение. R находится. Нажмите файл | Создать проект и выбрать эту папку.
- После создания нового проекта откройте приложение. R файл, загруженный ранее. RStudio распознает это приложение. R является блестящим приложением и создает кнопку «Запустить приложение» на панели над отображаемым исходным кодом. Щелкните Запустить приложение. Затем RStudio запустит окно, в котором отображается приложение PAST Shiny.
- PAST Блестящий с консолью R
- Запустите R и выполните следующий код, чтобы запустить приложение PAST Shiny: shiny::runApp('path/to/folder/with/shiny/app. Р'. Замените текст в кавычках папкой, в которую вложено приложение. R был загружен, и сохраните котировки.
- PAST без R Shiny
- Запустите библиотеку (PAST) в консоли R для загрузки PAST.
- PAST Shiny с RStudio
2. Настройка блестящего анализа (опционально)
- Измените название анализа с «Новый анализ» на что-то, что лучше отражает тип запускаемого анализа, что помогает отслеживать несколько анализов (см. Рисунок 1).
Рисунок 1. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Измените количество ядер и режим. Установите для числа ядер любое число от 1 до общего числа на компьютере, но имейте в виду, что выделение большего количества ресурсов на PAST может замедлить другие операции на компьютере. Установите режим на основе описания в разделе 6.
3. Загрузка данных GWAS
ПРИМЕЧАНИЕ: Убедитесь, что данные GWAS разделены табуляцией. Убедитесь, что файл ассоциации содержит следующие столбцы: признак, имя маркера, локус или хромосома, положение на хромосоме, значение p и значениеR2 для маркера. Убедитесь, что файл эффектов содержит следующие столбцы: признак, имя маркера, локус или хромосома, положение на хромосоме и эффект. Порядок этих столбцов не важен, так как пользователь может указать имена столбцов при загрузке данных. Все дополнительные столбцы игнорируются. TASSEL13 может быть использован для создания этих файлов.
- Загрузите данные GWAS с помощью PAST Shiny.
- Выберите ассоциативный файл и файл эффектов с помощью полей выбора Файл ассоциации и Файл эффектов. Измените имена столбцов в полях ввода Имя столбца ассоциации и Имя столбцов эффектов под полями выбора файла, чтобы отразить имена столбцов в данных.
Рисунок 2. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Загрузите данные GWAS с помощью PAST в консоли R.
- Измените и запустите следующий код:
gwas_data = load_GWAS_data("path/to/association_file.tsv", "path/to/effects_file.tsv", association_columns = c("Признак", "Маркер", "Локус", "Сайт", "p", "marker_R2"), effects_columns = c("Признак", "Маркер", "Локус", "Сайт", "Эффект")
- Измените и запустите следующий код:
- ПРИМЕЧАНИЕ: Измените пути к фактическому расположению файлов GWAS. Значения, предоставленные для association_columns и effects_columns, являются значениями по умолчанию. Если имена не совпадают со значениями по умолчанию, укажите имена столбцов. В противном случае они могут быть опущены.
4. Данные о неравновесии связей нагрузки (LD)
ПРИМЕЧАНИЕ: Убедитесь, что данные неравновесия связи (LD) разделены табуляцией и содержат следующие типы данных: Locus, Position1, Site1, Position2, Site2, Distance in base pairs between Position1 и Position2 и значение R2.
- Загрузите данные LD с помощью PAST Shiny.
- Выберите файл, содержащий данные LD. При необходимости измените имена столбцов в полях ввода ИМЕНА СТОЛБЦОВ LD под полем выбора файла, чтобы они соответствовали именам столбцов в данных LD.
Рисунок 3. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Загрузите данные LD с помощью PAST в консоли R.
- Измените и запустите следующий код для загрузки данных LD:
LD = load_LD("path/to/LD.tsv", LD_columns = c("Locus1", "Position1", "Site1", "Position2", "Site2", "Dist_bp", "R.2")
ПРИМЕЧАНИЕ: Измените путь к фактическому расположению файла LD. Значения, предоставляемые для LD_columns, являются значениями по умолчанию. Если имена не совпадают с именами по умолчанию, укажите правильные имена столбцов; в противном случае они могут быть опущены.
- Измените и запустите следующий код для загрузки данных LD:
5. Назначение SFP генам
ПРИМЕЧАНИЕ: Загрузите или найдите аннотации в формате GFF. Эти аннотации часто можно найти в онлайн-базах данных для конкретных организмов. Будьте осторожны с аннотациями низкого качества, так как качество данных аннотаций повлияет на качество анализа пути. Подтвердите, что первый столбец этих аннотаций (хромосома) соответствует формату локуса/хромосомы в данных ассоциации, эффектов и LD. Например, аннотации не должны называть первую хромосому «chr1», если файлы данных GWAS и LD называют первую хромосому «1».
- Назначайте SFP генам с PAST Shiny.
ПРИМЕЧАНИЕ: Более подробную информацию об определении соответствующего отсеченияR2 можно найти в Tang et al.6,в разделе под названием «SNP к генному алгоритму для анализа путей».- Выберите файл, содержащий аннотации GFF. Рассмотрим, какой размер окна и отсечку R2 наиболее подходят для рассматриваемого вида, и измените, если значения по умолчанию не подходят для загруженных данных.
ПРИМЕЧАНИЕ: Значения по умолчанию в PAST в основном отражают значения, подходящие для кукурузы. На этом шаге используется количество ядер, заданных в начале анализа PAST Shiny (шаг 2.2).
- Выберите файл, содержащий аннотации GFF. Рассмотрим, какой размер окна и отсечку R2 наиболее подходят для рассматриваемого вида, и измените, если значения по умолчанию не подходят для загруженных данных.
Рисунок 4. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Назначьте SFP генам с PAST в консоли R.
- Измените и запустите следующий код, чтобы назначить SFP генам:
гены = assign_SNPs_to_genes(gwas_data, LD, "path/to/annotations.gff", c("gene"), 1000, 0.8, 2)
ПРИМЕЧАНИЕ: В этом примере кода приведено несколько предложений по умолчанию: 1000 - размер окна вокруг SNP для поиска генов; 0,8 - величина отсечения дляR2; 2 — количество ядер, используемых для параллельной обработки. Путь к аннотациям также должен быть изменен на фактическое расположение файла аннотаций.
- Измените и запустите следующий код, чтобы назначить SFP генам:
6. Откройте для себя важные пути
ПРИМЕЧАНИЕ: Убедитесь, что файл pathways содержит следующие данные в формате с разделителями табуляции, с одной строкой для каждого гена в каждом пути: pathway ID - идентификатор, такой как "PWY-6475-1"; описание пути - более длинное описание того, что делают пути, такие как «биосинтез транс-ликопина»; gene - ген в пути, который должен соответствовать названиям, приведенным в аннотациях. Информацию о путях распространения, вероятно, можно найти в онлайновых базах данных для конкретных организмов, таких как MaizeGDB. Второй параметр, заданный пользователем, — это режим. «Увеличение» относится к фенотипам, которые отражают, когда желательно увеличение значения измеряемого признака, например, урожайность, в то время как «уменьшение» относится к признаку, где снижение измеренных значений полезно, например, рейтинги повреждения насекомыми. Значимость путей проверяется с помощьюранееописанных методов4,6,14.
- Откройте для себя важные пути с PAST Shiny.
- Выберите файл, содержащий данные путей, и убедитесь, что режим выбран в параметрах анализа. При необходимости измените количество генов, которые должны быть в пути, чтобы сохранить его для анализа, и количество перестановок, используемых для создания нулевого распределения, чтобы проверить значимость эффекта.
Рисунок 5. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
ПРИМЕЧАНИЕ: На этом шаге используется количество ядер и режим, установленный в начале анализа PAST Shiny (шаг 2.2). Стандартное количество генов в настоящее время установлено на уровне 5 генов, поэтому пути с меньшим количеством известных генов будут удалены. Пользователь может понизить это значение до 4 или 3, включив в него более короткие пути, но это может привести к ложноположительным результатам. Увеличение этого значения может увеличить мощность анализа, но удалит больше путей из анализа. Изменение количества используемых перестановок увеличивает и уменьшает мощность теста.
- Откройте для себя важные пути с помощью PAST в консоли R.
- Измените и запустите следующий код, чтобы обнаружить важные пути:
rugplots_data <- find_pathway_significance (гены, "путь/к/пути.цв", 5, "увеличение", 1000, 2)
ПРИМЕЧАНИЕ: В этом примере кода приведено несколько предлагаемых параметров по умолчанию. 5 - минимальное количество генов, которые должны быть в пути, чтобы сохранить путь в анализе, увеличение относится к увеличению количества измеренного признака (рекомендуется, чтобы пользователь запускал как увеличивающийся, так и уменьшающийся, независимо от признака; интерпретация данных будет отличаться для двух, однако), 1000 - это количество раз, чтобы выборка эффектов для определения нулевого распределения, и 2 — количество ядер, используемых для параллельной обработки. Измените путь к фактическому расположению файла pathways.
- Измените и запустите следующий код, чтобы обнаружить важные пути:
7. Просмотр Rugplots
- Посмотрите Rugplots с PAST Shiny.
- После загрузки и настройки всех входных данных нажмите кнопку Начать анализ. Появится индикатор выполнения, который покажет, какой шаг анализа был завершен последним. После завершения анализа PAST Shiny переключится на вкладку Результаты. Таблица результатов будет отображаться в левом столбце (помечена как «пути»), а Rugplots будет отображаться в правом столбце (помечено как «графики»).
- Ползунок используется для управления параметрами фильтрации. Когда уровень фильтрации будет удовлетворительным, нажмите кнопку Загрузить результаты в левом нижнем углу, чтобы загрузить все изображения и таблицы по отдельности в ZIP-файл, названный в соответствии с заголовком анализа. Этот ZIP-файл содержит отфильтрованную таблицу, неотфильтрованную таблицу и одно изображение на путь в отфильтроированной таблице.
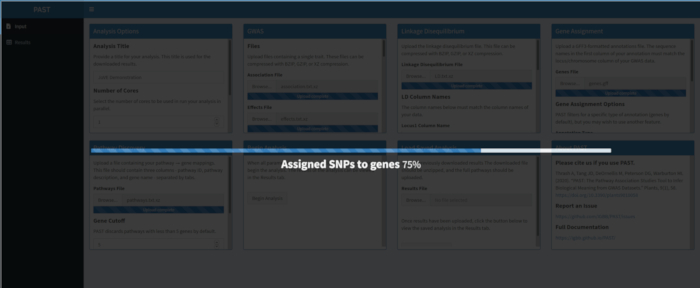
Рисунок 6. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
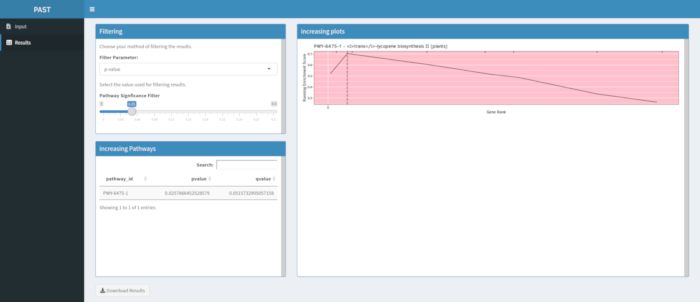
Рисунок 7. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Просмотр rugplots с помощью PAST в консоли R
- Измените и запустите следующий код, чтобы сохранить результаты:
plot_pathways (rugplots_data, "pvalue", 0.02, "увеличение", "output_folder")
ПРИМЕЧАНИЕ: В этом примере кода приведено несколько предлагаемых параметров по умолчанию. pvalue предоставляет данные, которые могут быть использованы для фильтрации незначительных путей после выбора пользователем порога значимости; 0,02 — значение по умолчанию, используемое при фильтрации, а увеличение относится к увеличению количества измеряемого признака (рекомендуется, чтобы пользователь запускал как увеличивающийся, так и уменьшающийся, независимо от признака; интерпретация данных будет отличаться для них, однако); output_folder — это папка, в которую будут записаны изображения и таблицы (эта папка должна существовать до запуска функции). В эту папку записывается таблица отфильтрованных результатов, нефильтрованных результатов и отдельных изображений для каждого пути в отфильтрованных результатах.
- Измените и запустите следующий код, чтобы сохранить результаты:
Результаты
Если результаты не получены после запуска программного средства PAST, убедитесь, что все входные файлы правильно отформатированы. Успешный запуск с использованием примера данных в пакете PAST, которые основаны на GWAS кукурузы зернового цвета, показан на рисунке 8. Эту таблицу и полученное изображение можно загрузить с помощью кнопки Загрузить результаты. Пример загруженного изображения показан на рисунке 210. Неправильные настройки могут привести к результатам, которые не имеют биологического смысла, но определение неправильности должно быть на стороне исследователя, который должен дважды проверить достоверность выбранных настроек и рассмотреть все известные доказательства относительно интересующей черты.
На рисунке 910 показан рудглот, полученный в результате анализа путей результатов GWAS, созданных с помощью кукурузной панели из 288 инбредных линий, которые были фенотипированы для цвета зерна. Этот упрощенный пример, где фенотипы были либо «белыми», либо «желтыми», был использован, потому что путь, ответственный за создание ярко-желтых каротиноидных пигментов, известен и должен отвечать за большую часть фенотипа. Таким образом, мы ожидали увидеть, что путь биосинтеза транс-ликопина (который производит каротиноиды) будет значительно связан с цветом зерна, которым он является. Идентификатор и имя пути перечислены в верхней части графика. Горизонтальная ось графа ранжирует все гены, которые были включены в анализ, расположенные слева направо в порядке наибольшего влияния на признак к наименьшему. Однако отмечены только гены в пути биосинтеза транс-ликопина (в верхней части графика, как метки хэтча, появляющиеся в генном ранге их эффекта по сравнению со всеми другими генами в анализе). В этом пути есть 7 генов. Показатель бегущего обогащения (ES) строится вдоль вертикальной оси. ES для каждого гена добавляется в ходовую сумму в порядке эффекта, а общая сумма корректируется на количество проанализированных генов. Таким образом, оценка изменяется по мере движения вправо вдоль горизонтальной оси и имеет тенденцию увеличиваться по мере включения генов большего эффекта, но в какой-то момент увеличение эффекта меньше, чем корректировка на добавление другого гена, и вся оценка начинает уменьшаться. Вершина бегущей линии ES отмечена пунктирной вертикальной линией; это ES для всего пути и используется программой для определения того, выбран ли путь и представлен ли он как rugplot.
Рисунок 8:Завершенный запуск PAST Shiny. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
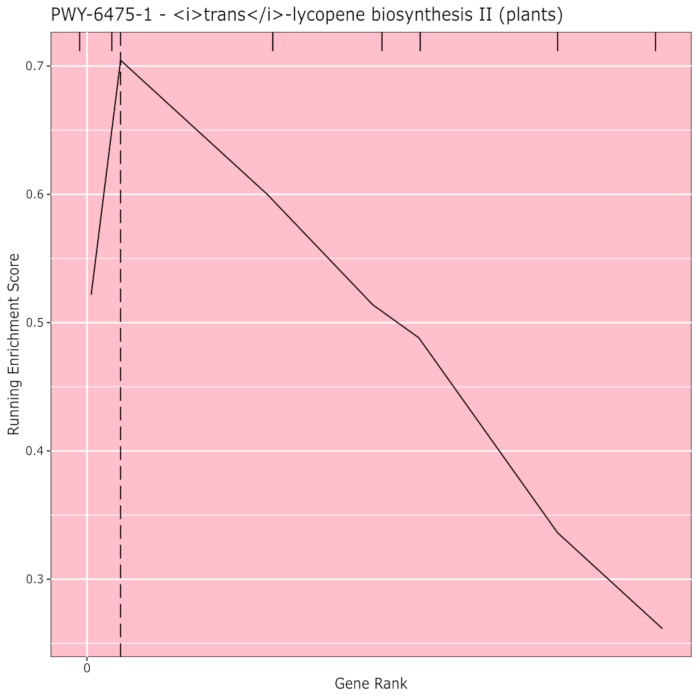
Рисунок 9:Изображение пути из завершенного запуска PAST (или загруженное из Shiny). Эта цифра была приведена из Thrash et al.10. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Обсуждение
Основной целью PAST является доведения анализа метаболических путей данных GWAS до более широкой аудитории, особенно для нечеловеческих и неживотных организмов. Альтернативными методами PAST часто являются программы командной строки, которые фокусируются на людях или животных. Удобство использования было основной целью в разработке PAST, как при выборе разработки приложения Shiny, так и при выборе использования R и Bioconductor для выпуска приложения. Пользователям не нужно учиться компилировать программы, чтобы использовать PAST.
Как и в большинстве типов аналитического программного обеспечения, результаты PAST хороши настолько, насколько хороши входные данные; если входные данные имеют ошибки или неправильно отформатированы, PAST не будет запущен или выдать неинформативные результаты. Обеспечение правильного форматирования данных GWAS, данных LD, аннотаций и путей имеет решающее значение для получения правильных выходных данных от PAST. PAST анализирует только двухаллельные маркеры и может выполнять только один признак для каждого набора входных данных. Кроме того, данные GWAS, полученные в результате плохого генотипирования или неправильного или неточного фенотипирования, также вряд ли приведут к четким или повторяемым результатам. PAST может помочь в биологической интерпретации результатов GWAS, но вряд ли прояснит хаотические наборы данных, если изменения окружающей среды, экспериментальные ошибки или структура популяции не были должным образом учтены.
Пользователи могут изменить некоторые параметры анализа, как в приложении Shiny, так и путем передачи этих параметров функциям PAST в консоли R. Эти параметры могут изменять результаты, сообщаемые PAST, и пользователи должны проявлять осторожность при изменении их по умолчанию. Поскольку LD измеряется пользователями, как правило, с использованием того же набора маркерных данных, который также использовался в GWAS, измерения LD специфичны для населения. Для всех исследований, особенно для других видов, кроме кукурузы (особенно самоопыляющихся, полиплоидных или сильно гетерогенных видов), могут быть оправданы изменения в значениях по умолчанию.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Никакой.
Материалы
| Name | Company | Catalog Number | Comments |
| Computer | NA | NA | Any computer with 8GB RAM should be sufficient |
| R | R Project | NA | R 4.0 or greater is required to install from Bioconductor 3.11 |
Ссылки
- Rafalski, J. Association genetics in crop improvement. Current Opinion in Plant Biology. 13 (2), 174-180 (2010).
- Yan, J., Warburton, M., Crouch, J. Association Mapping for Enhancing Maize (Zea mays L.) Genetic Improvement. Crop Science. 51 (2), 433-449 (2011).
- Xiao, Y., Liu, H., Wu, L., Warburton, M., Yan, J. Genome-wide Association Studies in Maize: Praise and Stargaze. Molecular Plant. 10 (3), 359-374 (2017).
- Wang, K., Li, M., Bucan, M. Pathway-Based Approaches for Analysis of Genomewide Association Studies. The American Journal of Human Genetics. 81 (6), 1278-1283 (2007).
- Weng, L., et al. SNP-based pathway enrichment analysis for genome-wide association studies. BMC Bioinformatics. 12 (1), 99(2011).
- Tang, J., Perkins, A., Williams, W., Warburton, M. Using genome-wide associations to identify metabolic pathways involved in maize aflatoxin accumulation resistance. BMC Genomics. 16 (1), 673(2015).
- Warburton, M., et al. Genome-Wide Association Mapping of Aspergillus flavus and Aflatoxin Accumulation Resistance in Maize. Crop Science. 55 (5), 1857-1867 (2015).
- Warburton, M., et al. Genome-Wide Association and Metabolic Pathway Analysis of Corn Earworm Resistance in Maize. The Plant Genome. 11 (1), 170069(2018).
- Li, H., Thrash, A., Tang, J., He, L., Yan, J., Warburton, M. Leveraging GWAS data to identify metabolic pathways and networks involved in maize lipid biosynthesis. The Plant Journal. 98 (5), 853-863 (2019).
- Thrash, A., Tang, J., DeOrnellis, M., Peterson, D., Warburton, M. PAST: The Pathway Association Studies Tool to Infer Biological Meaning from GWAS Datasets. Plants. 9 (1), 58(2020).
- Adam, T., Mason, D. PAST: Pathway Association Study Tool (PAST). Bioconductor version: Release (3.10). , (2020).
- Thrash, A., DeOrnellis, M. IGBB/PAST. , at https://github.com/IGBB/PAST (2019).
- Bradbury, P., et al. TASSEL: software for association mapping of complex traits in diverse samples. Bioinformatics. 23 (19), 2633-2635 (2007).
- Subramanian, A., et al. Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences U.S.A. 102, 15545-15550 (2005).
Erratum
Formal Correction: Erratum: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information
Posted by JoVE Editors on 10/08/2021. Citeable Link.
An erratum was issued for: A Pathway Association Study Tool for GWAS Analyses of Metabolic Pathway Information. One of the affiliations was updated.
The second affiliation was updated from:
USDA-ARS Corn Host Plant Resistance Research Unit, Mississippi State University
to:
Corn Host Plant Resistance Research Unit, USDA-ARS
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены