Method Article
تحليل حلقة R بواسطة Dot-Blot
In This Article
Summary
يوضح هذا البروتوكول بالتفصيل طريقة بسيطة تحدد الحلقة R ، وهي بنية حمض نووي ثلاثية الشريطة تتكون من هجين من الحمض النووي الريبي والحمض النووي وخيط DNA نازح.
Abstract
يتم التعرف بشكل متزايد على هيكل الحمض النووي ثلاثي الشريطة ، R-loop ، لدوره في تنظيم الجينات. في البداية ، كان يعتقد أن حلقات R هي المنتجات الثانوية للنسخ. لكن النتائج الحديثة لعدد أقل من حلقات R في الخلايا المريضة أوضحت أن حلقات R لها أدوار وظيفية في مجموعة متنوعة من الخلايا البشرية. بعد ذلك ، من الأهمية بمكان فهم أدوار حلقات R وكيف توازن الخلايا وفرتها. يتمثل التحدي في هذا المجال في القياس الكمي للحلقات R لأن الكثير من العمل يعتمد على الجسم المضاد أحادي النسيلة S9.6 الذي تم التشكيك في خصوصيته لهجينة الحمض النووي الريبي. هنا ، نستخدم البقع النقطية مع الجسم المضاد S9.6 لتحديد حلقات R وإظهار حساسية وخصوصية هذا الاختبار مع RNase H و RNase T1 و RNase III التي تشق هجينة الحمض النووي الريبي والحمض النووي الريبي أحادي الشريطة والحمض النووي الريبي المزدوج الشريطة ، على التوالي. هذه الطريقة قابلة للتكرار بدرجة كبيرة ، وتستخدم معدات المختبرات العامة والكواشف ، وتوفر النتائج في غضون يومين. يمكن استخدام هذا الاختبار في البحث والإعدادات السريرية لتحديد الحلقات R وتقييم تأثير الطفرات في الجينات مثل سيناتاكسين على وفرة الحلقة R.
Introduction
يوفر هذا البروتوكول دليلا تفصيليا لمقايسة اللطخة النقطية التي تسمح بتقييم مقارن سريع لوفرة الحلقة R ، وهي بنية حمض نووي ثلاثية الشريط. تتشكل حلقة R عندما يغزو الحمض النووي الريبي الحمض النووي المزدوج الشريطة لتوليد هجين من الحمض النووي الريبي والحمض النووي ويزيح خيط الحمض النووي الآخر. تم العثور على حلقات R في مراحل مختلفة من دورة حياة الحمض النووي الريبي. في مجمع النسخ ، يتم تصنيع الحمض النووي الريبي الناشئ مكملا للحمض النووي للقالب ، ويتم إزاحة الخيط غير القالب. يتم حل هجين الحمض النووي الريبي القصير (<10 نقطة أساس) لتحرير الحمض النووي الريبي الناشئ حتى يتمكن من مغادرة مركب بوليميراز الحمض النووي الريبي من خلال قناة الخروج1،2. خارج مجمع النسخ ، يكون الحمض النووي الريبي الناشئ قريبا من قالب الحمض النووي الخاص به ، والذي لا يزال غير ملفوف قليلا من النسخ ، وبالتالي يمكن للحمض النووي الريبي إعادة التهجين مع قالب الحمض النووي الخاص به الذي يشكل حلقات R3. بالإضافة إلى ذلك ، يمكن أن تتشكل حلقات R عندما تصطدم مجمعات النسخ المتماثل والنسخ4 ، وفي النسخ المضادللمعنى 5. نظرا للفرص العديدة لتكوينها ، فإن حلقات R ليست نادرة ، ويمكن العثور عليها في 3-5٪ من الجينوم البشري6 ، اعتمادا على حالة نسخ الخلية. تم العثور على حلقات R في محفزات الجينات7 ومواقع الإنهاء5 في mRNA ، وعلى طول الحمض النووي الريبيالريبوسومي 8 بالإضافة إلى نقل الحمض النوويالريبي 9. توجد حلقات R أيضا في المناطق التيلوميرية من الكروموسومات.
تلعب حلقات R دورا تنظيميا. إنهم ينظمون التعبير الجيني من خلال التأثير على النسخ في المروجين10،11 ، والتوسط في إعادة تركيب تبديل الفصل12 ، وتسهيل تحرير الجينوم المستند إلى كريسبر13،14،15. مثل العديد من الأحداث الخلوية ، يتم معايرة وفرة حلقة R بإحكام. تؤثر حلقات R كثيرة جدا أو قليلة جدا على وظيفة الخلية الطبيعية16،17. يتم تنظيم حلقات R بواسطة مجموعة متنوعة من البروتينات بما في ذلك RNase H و senataxin والهليكازات الأخرى التي تفكك الهجينة RNA-DNA18،19،20،21،22.
لمراقبة وفرة حلقات R ، تثري الطرق على مستوى الجينوم أولا حلقات R بالجسم المضاد S9.68،23،24 أو مع نوكليازات أخرى25 بما في ذلك RNase H10،26،27 ، ثم قم بتقييم عدد حلقات R المخصبة عن طريق التسلسل. لم تحقق الإصدارات المبكرة من هذه الأساليب القائمة على التسلسل تغطية تسلسل كافية للسماح بالقياس الكمي الدقيق ، لكن التحسين السريع في تقنيات التسلسل يسمح الآن بتحليل حلقة R موضع تلو الآخر. كما تم استخدام تقنيات التألق المناعي لتحديد وتوطين حلقات R10،17. هذه الأساليب شاملة ، لكنها ليست عملية في العديد من البيئات السريرية أو كتقييمات أولية لأنها تتطلب معدات باهظة الثمن وتحليلات متخصصة.
هناك حاجة إلى إجراء يمكن إجراؤه بشكل موحد عبر المختبرات في البيئات السريرية. توفر البقع النقطية مثل هذا الخيار حيث يمكن إجراؤها دون أي معدات محددة أو تحليل حسابي. كخطوة أولية أو في الإعدادات السريرية لتقييم آثار الطفرات على حلقات R ، يجب أن توفر هذه البقع النقطية نتائج حساسة ومحددة. هنا ، نصف فحصنا الذي يحدد حلقات R على وجه التحديد ؛ يستبعد الإشارات من الحمض النووي المزدوج الشريطة (DS) ، والحمض النووي الريبي المزدوج الشريطة ، والحمض النووي الريبي أحادي الشريطة. يستخدم بروتوكولنا الجسم المضاد S9.627 لتحديد الهجينة من الحمض النووي الريبي والحمض النووي في حلقات R ويتضمن RNase H ، وهو نوكلياز داخلي ينشق وبالتالي يؤدي إلى تدهور الحمض النووي الريبي في هجين الحمض النوويالريبي-الحمض النووي 20،28 ، للتأكد من أن الإشارات المكتشفة هي تلك الخاصة بالهجينة. قمنا أيضا بدمج RNase T1 الذي يشق الحمض النووي الريبي أحادي الشريطة عند الجوانين29،30 ، و RNase III الذي يشق الحمض النووي الريبي المزدوج الشريطة بما في ذلك الحلقات الجذعية31،32 للتحقق من وجود إشارات غير محددة. يتعرف الجسم المضاد S9.6 على الهجينة من الحمض النووي الريبي والحمض النووي بأطوال متفاوتة ، حتى تلك التي يبلغ طولها 8 نيوكليوتيداتفقط 33.
هنا ، نقدم البروتوكول الذي يبدأ بعزل الحمض النووي متبوعا بإعداد اللطخة النقطية ، واكتشاف الحلقة R بالجسم المضاد S9.6. يتضمن بروتوكولنا خطوات لضمان تحميل كميات متساوية من العينات ، وأن الإشارات محددة. يوفر قليل النوكليوتيدات لتكون بمثابة ضوابط إيجابية وسلبية. هذه طريقة سريعة وسهلة وسهلة الاستخدام لتقييم وفرة حلقة R.
Protocol
1. تحلل الخلايا للتجزئة النووية
- اغسل الخلايا بمحلول ملحي مخزن بالفوسفات 1x (PBS) مرتين. قم بإزالة الخلايا من أطباق زراعة الأنسجة باستخدام تقنيات تفكك الخلايا القياسية مثل التربسين. عد الخلايا باستخدام مقياس كثافة الدم.
ملاحظة: تم استخدام الخطوات الموضحة أدناه لتحليل الخلايا الليفية الأولية لجلد الإنسان ، على الرغم من أنه يمكن فحص مجموعة من أنواع الخلايا. نمت الخلايا الليفية في وسط قاعدي يحتوي على 10٪ مصل بقري جنيني. بدلا من ذلك ، يمكن إضافة المخزن المؤقت لتحلل الخلية (الجدول 1) مباشرة إلى مزرعة الخلية بعد الغسيل. - انقل تعليق الخلية إلى أنبوب سعة 1.5 مل لتكسير الخلايا.
- جهاز الطرد المركزي للعينة عند 300 × جم لمدة 5 دقائق عند 4 درجات مئوية. استنشق الوسائط.
- اغسل مرتين باستخدام 1x PBS المثلج باستخدام إعدادات الطرد المركزي في الخطوة 1.3.
- أضف المخزن المؤقت لتحلل الخلايا الباردة (الجدول 1) إلى حبيبات الخلية (300 ميكرولتر لكل 2 × 106 خلايا). الماصة لأعلى ولأسفل لإعادة تعليق الحبيبات.
- احتضن على الثلج لمدة 10 دقائق.
- تدور عند 500 × جم لمدة 5 دقائق لتكوير النوى.
- تخلص من المادة الطافية وأعد تعليق الحبيبات النووية في 400 ميكرولتر من محلول التحلل النووي البارد (الجدول 1).
- احتضن على الثلج لمدة 10 دقائق.
ملاحظة: يضمن تجزئة المقصورات النووية والسيتوبلازمية للخلايا خصوصية الإشارة. يمكن تقييم جودة الفصل النووي والسيتوبلازمي قبل المتابعة (الجدول 2). - أضف 3 ميكرولتر من 20 مجم / مل من بروتيناز K واحتضنه لمدة 3-5 ساعات عند 55 درجة مئوية.
ملاحظة: وحدات التخزين المشار إليها مخصصة ل 2 × 106 خلايا ، قم بتوسيع نطاقها أو تصغيرها حسب الضرورة.
2. تنقية الحمض النووي الجيني (الذي يتضمن هجينة الحمض النووي الريبي)
- إذا كان الحمض النووي لزجا ، فقم بإجراء صوتنة لتقليل اللزوجة (على سبيل المثال ، صوتنة عند خرج طاقة عالية ، 30 ثانية / 30 ثانية إيقاف التشغيل ، لمدة دقيقتين باستخدام حمام مائي 4 °C).
- أضف 400 ميكرولتر من محلول الشطف (الجدول 1) و 400 ميكرولتر من الفينول: الكلوروفورم: كحول الأيزواميل (25: 24: 1 درجة الحموضة 8.0).
- دوامة لمدة 10 ثوان.
- قم بالدوران عند 12,000 × جم لمدة 5 دقائق عند 4 درجات مئوية.
- نقل الطور المائي (حوالي 350 ميكرولتر) إلى أنبوب جديد.
- استخرج مرة واحدة باستخدام حجم واحد من الكلوروفورم ، دوامة لمدة 10 ثوان ، ثم قم بالدوران عند 12,000 × جم لمدة 5 دقائق عند 4 درجات مئوية. نقل الطور المائي إلى أنبوب جديد (حوالي 300 ميكرولتر).
- أضف 35 ميكرولتر من أسيتات الصوديوم 3 M (درجة الحموضة 5.2) و 1 ميكرولتر من الجليكوجين و 700 ميكرولتر من الإيثانول المثلج البارد بنسبة 100٪.
- دوامة لمدة 10 ثوان وتدور لأسفل عند 12,000 × جم لمدة 30 دقيقة عند 4 درجات مئوية.
- اغسل الحبيبات ب 1 مل من 70٪ إيثانول.
- دوامة لمدة 10 ثوان وتدور لأسفل عند 12,000 × جم لمدة 15 دقيقة عند 4 درجات مئوية.
- تخلص من المادة الطافية واترك الحبيبات تجف في الهواء.
- أضف 12 ميكرولتر من المخزن المؤقت للشطف والدوامة لمدة 10 ثوان لإعادة التعليق. احتضان العينة لمدة 30 دقيقة عند 37 درجة مئوية مع التحريك أو عند 4 درجات مئوية طوال الليل لإعادة تعليق الحبيبات.
- قياس تركيز الحمض النووي باستخدام قياس الطيف الضوئي القياسي.
ملاحظة: وحدات التخزين المشار إليها مخصصة ل 2 × 106 خلايا ، قم بتوسيع نطاقها أو تصغيرها حسب الضرورة. يمكن تخزين الحمض النووي (مع هجينة الحمض النووي الريبي) عند -20 درجة مئوية ، إذا لزم الأمر.
3. نشاف عينات الحمض النووي (والتي تشمل هجينة الحمض النووي الريبي-الحمض النووي) على أغشية النايلون
- تحضير تخفيفات الأحماض النووية إلى التركيزات المرغوبة في المخزن المؤقت للشطف (أي 50 نانوغرام / ميكرولتر ، أو 25 نانوغرام / ميكرولتر ، أو 12.5 نانوغرام / ميكرولتر). تضمن هذه العينات ذات التركيزات (200 ، 100 ، 50 ، 25 ، 12.5 نانوغرام) وجود إشارات ضمن النطاق الخطي.
ملاحظة: تأكد من إعداد عينة كافية للتكرارات التقنية والبيولوجية ، وبالنسبة لعلاجات RNase المختلفة ، انظر الخطوة 5. - قم بإعداد غشاء نايلون موجب الشحنة بحيث يكون هناك مساحة لكل عينة 2 ميكرولتر لشغل مساحة 0.5 سم 2.
- ضع 2 ميكرولتر من كل عينة على غشاءين: أحدهما للجسم المضاد S9.6 والآخر للجسم المضاد dsDNA. بدلا من ذلك ، يمكن استخدام جهاز اللطخة النقطية أو اللطخة النقطية الذي يسمح بتحميل العينات ذات الأحجام الأكبر.
- اسمح للعينات بالتشبع في الغشاء. انتظر 2 دقيقة على الأقل قبل ربط الغشاء بضوء الأشعة فوق البنفسجية.
- ضع الغشاء في وسط جهاز الأشعة فوق البنفسجية وقم بربط الغشاء باستخدام الوصلة الشابكة للأشعة فوق البنفسجية باستخدام إعداد "الارتباط المتقاطع التلقائي" (1,200 ميكروجول × 100).
4. الكشف الهجين عن الحمض النووي الريبي والحمض النووي باستخدام الجسم المضاد S9.6
- احتضان الغشاء في محلول مانع (5٪ حليب في محلول ملحي مخزن في تريس مع 0.05٪ توين -20 (TBST) لمدة ساعة واحدة في درجة حرارة الغرفة على شاكر.
ملاحظة: يجب أن يكون هناك ما يكفي من محلول الحجب لتغطية الغشاء. - احتضان الأغشية طوال الليل في الجسم المضاد الأولي (في 5٪ حليب في TBST) عند 4 درجات مئوية مع الرج. أضف الجسم المضاد ل dsDNA (تخفيف 1: 10,000) إلى غشاء واحد. أضف 1 ميكروغرام / مل الجسم المضاد S9.6 إلى الغشاء الثاني (تخفيف 1: 1,000).
ملاحظة: الجسم المضاد S9.6 متاح تجاريا أو من الدكتور S. Leppla ، NIAID ، المعاهد الوطنية للصحة. - قم بإزالة الجسم المضاد الأساسي واغسله 3 مرات باستخدام TBST. قم بإجراء كل غسلة لمدة 5-10 دقائق مع الرج في درجة حرارة الغرفة.
- احتضان الجسم المضاد الثانوي المترافق مع بيروكسيداز الفجل (HRP) (مضاد للفأر ، تخفيف 1: 5,000) في حليب 5٪ في TBST مع رج في درجة حرارة الغرفة.
ملاحظة: مضاد dsDNA وهجين مضاد للحمض النووي الريبي والحمض النووي كلاهما من الأجسام المضادة للفأر. - قم بإزالة الجسم المضاد الثانوي واغسله 3 مرات باستخدام TBST لمدة 5-10 دقائق مع رج في درجة حرارة الغرفة.
- التطوير باستخدام كواشف التلألؤ الكيميائي المعزز (ECL) للحصول على إشارات للتصوير.
- تحديد كثافة الإشارة باستخدام أدوات معالجة الصور القياسية مثل ImageJ.
ملاحظة: يتم تفصيل استكشاف الأخطاء وإصلاحها في الجدول 2.
5. علاجات الريبونوكلياز لتقييم خصوصية الإشارة
ملاحظة: يجب إجراء معالجة RNase على عينات الحمض النووي لإثبات خصوصية ارتباط S9.6. يجب أن يؤدي العلاج ب RNase H ، ولكن ليس RNase T1 أو RNase III إلى انخفاض في التلوين المناعي S9.6.
- هضم العينات التي تحتوي على هجينة الحمض النووي الريبي والحمض النووي عن طريق تحضيرها في أربعة أنابيب منفصلة. عالج كل من العينات الأربعة إما ب 5 U RNase H أو 1000 U RNase T1 أو 0.5 U RNase III أو وهمية. احتضان العينات عند 37 درجة مئوية لمدة 15 دقيقة بأحجام 20 ميكرولتر.
- قم بتحميل 2 ميكرولتر من كل عينة على غشاء كما هو موضح في القسم 3.
6. إعداد أدوات التحكم في قليل النوكليوتيدات لتقييم خصوصية الإشارة
ملاحظة: يمكن استخدام أدوات التحكم في قليل النوكليوتيدات لإثبات خصوصية ربط S9.6. يتعرف S9.6 على هجينة الحمض النووي الريبي ، ولكن ليس عناصر تحكم dsDNA أو dsRNA ، كما تم الإبلاغ عنهسابقا 34.
- قم بإذابة قليل النوكليوتيدات (الجدول 3) في محلول التلدين (10 ملي مولار تريس ، درجة الحموضة 8.0 ؛ 50 ملي كلوريد الصوديوم ، 1 ملي مولار EDTA) إلى 100 ميكرومتر.
- تحضير 4 أنابيب تفاعل ل
- هجين الحمض النووي الريبي - الحمض النووي الريبي # 1: امزج 10 ميكرولتر من الخيط العلوي ssRNA مع 10 ميكرولتر من الخيط السفلي ssDNA و 80 ميكرولتر من المخزن المؤقت للتلدين.
- هجين الحمض النووي الريبي والحمض النووي # 2: امزج 10 ميكرولتر من الخيط العلوي ssDNA مع 10 ميكرولتر من الخيط السفلي ssRNA و 80 ميكرولتر من المخزن المؤقت للتلدين.
- dsRNA: امزج 10 ميكرولتر من الخيط العلوي ssRNA مع 10 ميكرولتر من الخيط السفلي ssRNA و 80 ميكرولتر من المخزن المؤقت للتلدين.
- dsDNA: امزج 10 ميكرولتر من الخيط العلوي ssDNA مع 10 ميكرولتر من الخيط السفلي ssDNA و 80 ميكرولتر من المخزن المؤقت للتلدين.
- سخني 4 مخاليط من الخطوة 6.2 عند 95 درجة مئوية لمدة 10 دقائق.
- اترك الأنابيب تبرد ببطء إلى درجة حرارة الغرفة للسماح بإعادة تلدين الخيوط. يمكن تخزين المعايير الملدنة عند -20 درجة مئوية لاستخدامها لاحقا.
ملاحظة: يجب التحقق من كفاءة التلدين عن طريق الرحلان الكهربائي للهلام غير المتغير. تهاجر الازدواج بشكل أبطأ من قليل النوكليوتيدات غير الملدنة (الجدول 2). - قم بتحميل 2 ميكرولتر من كل عينة على غشاءين ، أحدهما للجسم المضاد S9.6 والآخر للجسم المضاد dsDNA ، كما هو موضح في القسم 3.
- تنفيذ الخطوات الموضحة في القسم 4.
7. القياس الكمي وتطبيع شدة إشارة S9.6 R-loop باستخدام ImageJ.
- احفظ صور S9.6 ، تلطيخ dsDNA بتنسيق TIFF ، وقم بتحليلها باستخدام برنامج ImageJ (https://imagej.nih.gov/ij/).
- حدد خيار عكس الصورة (تحرير | انعكاس). بعد الانعكاس ، ستكون كل نقطة مرئية باللون الأبيض على خلفية داكنة.
- استخدم أداة تحديد الصورة البيضاوية لتحديد شكل بيضاوي كبير بما يكفي لإحاطة أكبر نقطة على الصورة.
- استخدم مدير عائد الاستثمار لإضافة المنطقة المحددة للقياس الكمي. تأكد من تحديد خياري "إظهار الكل" و "التسميات" بحيث يمكن تصور مناطق الاهتمام.
- استخدم نفس منطقة التحديد البيضاوية المستخدمة خلال الخطوة 7.3 لإضافة مناطق إضافية ذات أهمية حول كل نقطة ليتم قياسها كميا. استخدم اختصار Command + Shift + E لنسخ المنطقة المحددة من الخطوة 7.3 إلى كل نقطة من النقاط اللاحقة.
- قياس الكثافة المتكاملة لكل منطقة من مناطق الاهتمام.
- اقسم شدة إشارة S9.6 لكل عينة على قياس dsDNA للحصول على نسبة إشارة S9.6 / dsDNA. تحقق من النتائج عن طريق تكرار التجارب (ثلاث نسخ على الأقل لكل من الحصول على إشارة S9.6 و dsDNA). يمكن حساب الخطأ المعياري للمتوسط من نسب إشارة S9.6 / dsDNA.
النتائج
العلاج الأنزيمي لتقييم خصوصية الجسم المضاد S9.6 (RNA-DNA).
نمت الخلايا الليفية الأولية لجلد الإنسان17. تم عزل الحمض النووي مع هجينة الحمض النووي الريبي والحمض النووي وقياسه كميا. تم هضم ميكروغرام من العينات باستخدام RNase T1 أو RNase H أو RNase III لمدة 15 دقيقة عند 37 درجة مئوية. كما تم تحليل عينة وهمية للمقارنة بالعينات المعالجة ب RNase. تم مسح العينات (200 أو 100 أو 50 أو 25 أو 12.5 أو 6.25 نانوغرام) على غشاءين مختلفين كما هو موضح في القسم 3. تم ربط الأغشية وحظرها وتم فحص أحدها بجسم مضاد S9.6 (الشكل 1 أ).
أظهرت النتائج أن إشارة S9.6 ترتبط بوفرة العينة المحملة. يؤدي العلاج باستخدام RNase H ، ولكن ليس RNase T1 أو RNase III إلى تقليل تلطيخ S9.6.
تم فحص غشاء ثان بجسم مضاد dsDNA (الشكل 1 ب) للتطبيع. تم استخدام الصورة J لتحليل شدة الإشارة. تم اختيار عينات 50 نانوغرام للقياس الكمي حيث كانت شدة الإشارة من الأجسام المضادة S9.6 و dsDNA ضمن النطاق الديناميكي. تم تطبيع شدة الإشارة لتلك الموجودة في عينات وهمية. البيانات موضحة في الشكل 2.
S9.6 لطخة نقطية للجسم المضاد باستخدام أدوات التحكم في النيوكليوتيدات الاصطناعية.
لتقييم خصوصية الجسم المضاد S9.6 ، استخدمنا قليل النوكليوتيدات المقابلة ل dsRNA و dsDNA و RNA-DNA كما هو موضح في القسم 6. تم تحضير سلسلة تخفيف من نيوكليوتيدات الحمض النووي الريبي وdsRNA و dsDNA ومسحها على غشاء النايلون كما هو موضح في القسم 3. تم فحص الغشاء بالجسم المضاد S9.6 (الشكل 3). أظهرت النتائج أن الجسم المضاد S9.6 يرتبط على وجه التحديد بهجينة الحمض النووي الريبي والحمض النووي بطريقة تعتمد على الجرعة وأظهر الحد الأدنى من التفاعل المتبادل مع dsRNAs و dsDNAs.
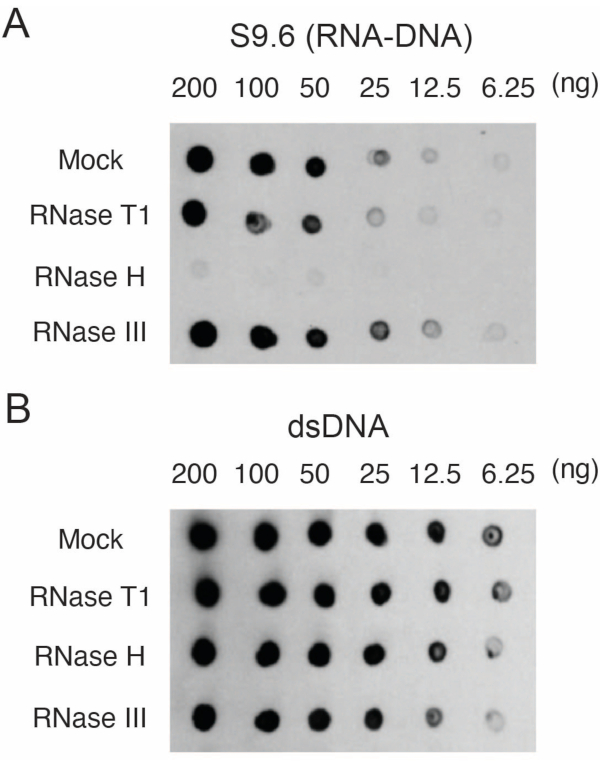
الشكل 1: خصوصية S9.6 كما هو موضح في اللطخة النقطية المحملة بالأحماض النووية من الخلايا الليفية البشرية. تمت معالجة عينات الحمض النووي من الخلايا الليفية البشرية إما بشكل وهمي أو معالجتها باستخدام RNase T1 أو RNase H أو RNase III ثم تم تحميلها على أغشية النايلون في سلسلة تخفيف من 200 و 100 و 50 و 25 و 12.5 و 6.25 نانوغرام لكل 2 ميكرولتر نقطة. ثم تم فحص الأغشية باستخدام الجسم المضاد S9.6 (A) ، أو الجسم المضاد dsDNA (B). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
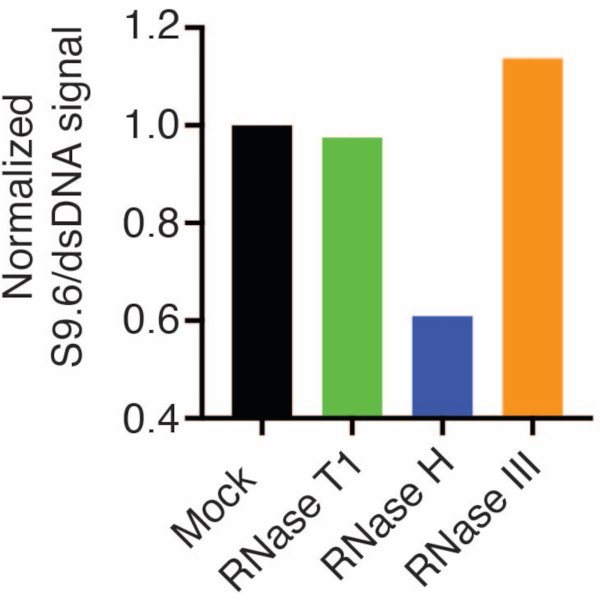
الشكل 2: القياس الكمي لتلوين حلقة S9.6 R. تم اختيار 50 نانوغرام من العينات من الشكل 1 للقياس الكمي باستخدام ImageJ. تم تقسيم إشارة S9.6 على شدة إشارة dsDNA ، ثم تم تطبيعها إلى العينة الوهمية باتباع الخطوات الموضحة في القسم 7. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
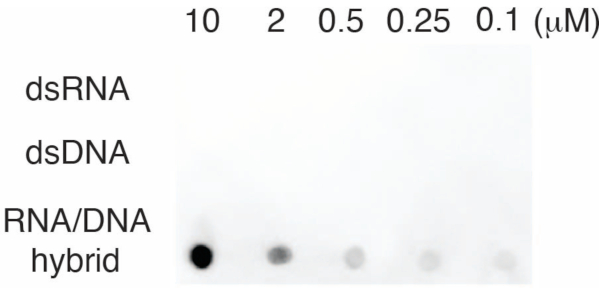
الشكل 3: S9.6 نقطية لطخة باستخدام عناصر تحكم قليل النوكليوتيدات. S9.6 لطخة نقطية للجسم المضاد ضد سلسلة تخفيف من قليل النوكليوتيدات الاصطناعية مثل dsRNA أو dsDNA أو RNA-DNA الهجين. يرتبط S9.6 على وجه التحديد بهجينة الحمض النووي الريبي والحمض النووي بطريقة تعتمد على الجرعة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
| عازلة تحلل الخلايا | لمدة 10 مل | ل 200 مل | النهائي Conc. |
| ماء خال من النوكلياز | 9 مل | 180 مل | - |
| 10٪ NP-40 | 0.5 مل | 10 مل | 0.5% |
| 2 مليون كيلو سي إل | 0.4 مل | 8 مل | 80 ملي متر |
| أنابيب 0.5 متر (درجة الحموضة 8.0) | 100 ميكرولتر | 2 مل | 5 مللي متر |
| المخزن المؤقت للتحلل النووي | لمدة 10 مل | ل 200 مل | النهائي Conc. |
| ماء خال من النوكلياز | 8.65 مل | 173 مل | - |
| 10٪ SDS | 1 مل | 20 مل | 1% |
| 1 متر تريس حمض الهيدروكلوريك (درجة الحموضة 8.0) | 0.25 مل | 5 مل | 25 ملم |
| 0.5 مليون EDTA | 100 ميكرولتر | 2 مل | 5 مللي متر |
| المخزن المؤقت للشطف | لمدة 10 مل | ل 200 مل | النهائي Conc. |
| 1 متر Tris-Cl ، درجة الحموضة 8.5 | 0.1 مل | 2 مل | 10 ملم |
| ماء خال من النوكلياز | 9.9 مل | 198 مل | - |
الجدول 1. تحضير المخازن المؤقتة
| درج | مشكلة | السبب المحتمل | حل |
| 1.9 | غير متاح | التحقق من تجزئة الخلايا | يمكن تقييم جودة الفصل النووي والسيتوبلازمي عن طريق إضافة كوكتيلات مثبطة البروتياز القياسية إلى الخلية والمخازن المؤقتة للتحلل النووي (الجدول 1). يمكن تقييم الكسور السيتوبلازمية والنووية عن طريق تحليل النشاف الغربي لتأكيد التقييد الكافي لوضع العلامات باستخدام العلامات السيتوبلازمية (على سبيل المثال ، GAPDH أو HSP90) في الكسور السيتوبلازمية ووضع العلامات بعلامات نووية (على سبيل المثال ، HDAC1 أو Histone H3) في الكسور النووية. يمكن تقييم مساهمة تلوث الميتوكوندريا في الجزء النووي عن طريق تحليل qPCR باستخدام مجسات خاصة بالحمض النووي للميتوكوندريا. |
| 2.1 | العينة لزجة جدا | رقم الخلية مرتفع للغاية. | قلل الحمض النووي بمقدار النصف واستمر في خطوة الصوتنة 2.1 |
| 2.12 | لا توجد بيليه مرئي | مادة أولية غير كافية أو خسارة أثناء الاستخراج. | ابدأ من البداية باستخدام المزيد من الخلايا. |
| 2.13 | تركيز منخفض للحمض النووي | ||
| 3.1 | لا يوجد ما يكفي من الحمض النووي للتخفيفات | ||
| 3.4 | لن تشبع العينة في الغشاء | نقع الغشاء في 1x TBST. اترك المخزن المؤقت الزائد حتى يجف وابدأ في الخطوة 3.4 | |
| 4.6 | تم الكشف عن نمط غير مكتمل أو مرقط | أضف 0.1٪ من كبريتات دوديسيل الصوديوم (SDS) إلى العينة واستمر في الخطوة 3.1 | |
| 4.6 | النقاط لها مظهر "حلقة القهوة" | أضف 0.01٪ ساركوسيل إلى العينة واستمر في الخطوة 3.1 | |
| 5.2 | لا يظهر عنصر تحكم RNaseH أي انخفاض في الإشارة | هضم الريبونوكلاز غير مكتمل. | زيادة الحضانة أو زيادة تركيز الإنزيم. |
| 6.5 | لا توجد إشارة لعناصر التحكم oligo | لم يتم تشكيل دوبلكس. لم يتم تلدين قليل النوكليوتيدات بشكل صحيح. | تحقق من نسب قليل النوكليوتيدات والمخزن المؤقت للتلدين. |
| 6.5 | إشارة S9.6 ليست خاصة بالهجينة | دفعة الأجسام المضادة S9.6 لها ارتباط غير محدد | التحقق من صحة حساسية وخصوصية الدفعات الجديدة من الجسم المضاد S9.6 باستخدام علاجات إنزيم RNase أو تحليل قليل النوكليوتيدات الاصطناعية. |
الجدول 2: استكشاف الأخطاء وإصلاحها
| ssRNA ، أعلى حبلا | 5'-UGGGGGCUCGUCCGGGAUAUGGGAACCACUGAUCCC-3' |
| ssDNA ، حبلا علوي | 5'-TGGGGGCTCGTCCGGGATATGGGAACCACTGATCCC-3' |
| ssRNA ، حبلا سفلي | 5'-GGGAUCAGUGGUUCCCAUAUCCCGGACGCCCCCA-3' |
| ssDNA ، حبلا سفلي | 5'-GGGATCAGTGGTTCCCATATCCCCCGGACGAGCCCCCA-3 ' |
الجدول 3. التحكم في تسلسل قليل النوكليوتيدات
Discussion
تتشكل الأحماض النووية المكونة من 3 تقطعت بهم السبل ، حلقات R ، في مراحل مختلفة خلال دورة حياة الحمض النووي الريبي ووجد بشكل متزايد أنها تنظم العمليات الخلوية. لفهم حلقات R بشكل كامل ، من الضروري استخدام تقنيات موثوقة لاكتشاف حلقة R. هنا ، نصف نهجا للاستجواب في وفرة الحلقات R باستخدام الجسم المضاد S9.68،23،24. تسمح هذه الطريقة بإجراء تقييم سريع لوفرة حلقة R من الخلايا وعينات زراعة الأنسجة. لا يتطلب معدات خاصة أو كمية كبيرة من مواد البداية. يضمن نتائج محددة وقابلة للتكرار باستخدام مزيج من علاجات RNase.
أبلغ البعض عن مخاوف بشأن خصوصية الجسم المضاد S9.6. كما هو الحال مع أي كاشف ، قد يكون هناك تباين من دفعة إلى دفعة مع الجسم المضاد S9.6. يتضمن بروتوكولنا RNase H و RNase T1 و RNase III للتحقق من خصوصية الإشارة. بالإضافة إلى ذلك ، نستخدم قليل النوكليوتيدات الاصطناعية لضمان خصوصية كل دفعة من الجسم المضاد S9.6.
بيولوجيا R-loop هو مجال متنامي. سيؤدي تطوير طرق الكشف والقياس الكمي الموثوقة ، مثل تلك المعروضة هنا ، إلى تسهيل الدراسات الميكانيكية لتوضيح متى تتشكل حلقات R ، وكيف يتم تنظيمها ، وما تنظمه. مع عناصر التحكم المناسبة ، يعد اختبار اللطخة النقطية طريقة بسيطة للكشف عن وفرة الحلقة R في الإعدادات السريرية والبحثية.
Disclosures
المؤلفون ليس لديهم ما يكشفون عنه.
Acknowledgements
تم دعم هذا العمل من قبل معهد هوارد هيوز الطبي والبحوث الداخلية في المعهد الوطني للاضطرابات العصبية والسكتة الدماغية.
نشكر الدكتور ستيفن ليبلا على تقديم دفعات من الجسم المضاد S9.6 للتحليل. نشكر أيضا الدكتور Dongjun Li على مساعدته في علاجات الريبونوكلياز.
Materials
| Name | Company | Catalog Number | Comments |
| Anti-dsDNA antibody | Abcam | ab27156 | |
| Anti-RNA-DNA hybrid antibody (S9.6) | Kerafast | ENH001 | |
| Biorupter sonicator | Diagenode | UCD-200 | |
| EB Buffer | Qiagen | 19086 | |
| EDTA (0.5M) | Invitrogen | AM9261 | |
| Hybond N+ nylon membrane | GE healthcare Life Sciences | RPN203B | |
| KCl (2M) | Invitrogen | AM9640G | |
| NP-40 (Igepal CA-630) | Sigma | I8896 | |
| PBS | Invitrogen | 10010-023 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593031 | |
| PIPES (0.5M, pH 8.0) | VWR | AAJ61406-AE | |
| Proteinase K | Qiagen | 19131 | |
| RNase III | Invitrogen | AM2290 | |
| RNase H | New England Biolabs | M0297 | |
| RNase T1 | ThermoFisher Sci. | EN0541 | |
| SDS (10%) | Invitrogen | 15553027 | |
| sodium acetate (3M, pH 5.2) | Invitrogen | AM9740 | |
| Tris-buffered saline (10X) | Corning | 46-012-CM | |
| Tris-HCl (1M, pH 8.0) | KD Medical | RGF-3360 | |
| TrypLE | Invitrogen | 12605010 | |
| Tween-20 | Sigma | P9416 | |
| UV Stratalinker 2400 | Stratagene | Stratalinker 2400 | |
| Whatman marking pen | Sigma | WHA10499001 |
References
- Daube, S. S., von Hippel, P. H. RNA displacement pathways during transcription from synthetic RNA-DNA bubble duplexes. Biochemistry. 33 (1), 340-347 (1994).
- Westover, K. D., Buschnell, D. A., Kornberg, R. D. Structural basis of transcription: separation of RNA from DNA by RNA polymerase II. Science. 303 (5660), 1014-1016 (2004).
- Itoh, T., Tomizawa, J. Formation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proceedings of the National Academy of Science USA. 77 (5), 2450-2454 (1980).
- Hamperl, S., Bocek, M. J., Saldivar, J. C., Swigut, T., Cimprich, K. A. Transcription-replication conflict orientation modulates R-loop levels and activates distinct DNA damage responses. Cell. 170 (4), 774-786 (2017).
- Skourti-Stathaki, K., Kamieniarz-Gdula, K., Proudfoot, N. J. R-loops induce repressive chromatin marks over mammalian gene terminators. Nature. 516 (7531), 436-439 (2014).
- Sanz, L. A., et al. conserved R-loop structures associate with specific epigenomic signatures in mammals. Molecular Cell. 63 (1), 167-178 (2016).
- Chen, L., et al. R-ChIP using inactive RNase H reveals dynamic coupling of R-loops with transcriptional pausing at gene promoters. Molecular Cell. 68 (4), 745-757 (2017).
- El Hage, A., French, S. L., Beyer, A. L., Tollervey, D. Loss of topoisomerase I leads to R-loop mediated transcriptional blocks during ribosomal RNA synthesis. Genes and Development. 24 (14), 1546-1558 (2010).
- Tran, P., et al. PIF1 family DNA helicases suppress R-loop mediated genome instability at tRNA genes. Nature Communication. 8, 15025(2017).
- Ginno, P. A., Lott, P. L., Christensen, H. C., Korf, I., Chedin, F. R-loop formation is a distinctive characteristic of unmethylated human CpG island promoters. Molecular Cell. 45 (6), 814-825 (2012).
- Colak, D., et al. Promoter-bound trinucleotide repeat mRNA drives epigenetic silencing in fragile X syndrome. Science. 343 (6174), 1002-1005 (2014).
- Yu, K., Chedin, F., Hsieh, C., Wilson, T. E., Lieber, M. R. R-loops at immunoglobulin class switch regions in the chromosomes of stimulated B cells. Nature Immunology. 4 (5), 442-451 (2003).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleotide complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Science USA. 109 (39), 2579-2586 (2012).
- Westra, E. R., et al. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3. Molecular Cell. 46 (5), 595-605 (2012).
- Jiang, F., et al. Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science. 351 (6275), 867-871 (2016).
- Huertas, P., Aguilera, A. Cotranscriptionally formed DNA:RNA hybrids mediate transcription elongation impairment and transcription-associated recombination. Molecular Cell. 12 (3), 711-721 (2003).
- Grunseich, C., et al. Senataxin mutation reveals how R-loops promote transcription by blocking DNA methylation at gene promoters. Molecular Cell. 69 (3), 426-437 (2018).
- Kim, H. D., Choe, J., Seo, Y. S. The sen1(+) gene of Schizosaccharomyces pombe, a homologue of budding yeast SEN1, encodes an RNA and DNA helicase. Biochemistry. 38 (44), 14697-14710 (1999).
- Stein, H., Hausen, P. Enzyme from calf thymus degrading the RNA moiety of DNA-RNA hybrids: effect on DNA-dependent RNA polymerase. Science. 166 (3903), 393-395 (1969).
- Cerritelli, S. M., Crouch, R. J. Ribonuclease H: the enzymes in eukaryotes. FEBS Journal. 276 (6), 1494-1505 (2009).
- Hyjek, M., Figiel, M., Nowotny, M. RNases H: Structure and mechanism. DNA Repair. 84, 102672(2019).
- Pohl, T. J., Zakian, V. A. Pif1 family DNA helicases: A helpmate to RNase H. DNA Repair. 84, 102633(2019).
- Pohjoismaki, J. L., et al. Mammalian mitochondrial DNA replication intermediates are essentially duplex but contain extensive tracts of RNA/DNA hybrid. Journal of Molecular Biology. 397 (5), 1144-1155 (2010).
- Sanz, L. A., Chedin, F. High-resolution, strand-specific R-loop mapping via S9.6-based DNA-RNA immunoprecipitation and high-throughput sequencing. Nature Protocols. 14 (6), 1734-1755 (2019).
- Wahba, L., Costantino, L., Tan, F. J., Zimmer, A., Koshland, D. S1-DRIP-seq identifies high expression and polyA tracts as major contributors to R-loop formation. Genes and Development. 30 (11), 1327-1338 (2016).
- Yan, Q., Shields, E. J., Bonasio, R., Sarma, K. Mapping native r-loops genome-wide using a targeted nuclease approach. Cell Reports. 29 (5), 1369-1380 (2019).
- Hu, Z., Zhang, A., Storz, G., Gottesman, S., Leppla, S. H. An antibody-based microarray assay for small RNA detection. Nucleic Acids Research. 34 (7), 52(2006).
- Nowotny, M., Gaidamakov, S. A., Crouch, R. J., Yang, W. Crystal structures of RNase H bound to an RNA/DNA hybrid: substrate specificity and metal-dependent catalysis. Cell. 121 (7), 1005-1016 (2005).
- Sato, K., Egami, F. Studies on ribonucleases in takadiastase. Journal of Biochemistry. 44 (11), Tokyo. 753-767 (1957).
- Takahashi, K., Moore, S. Ribonuclease T1. Enzymes. 15, 435-468 (1982).
- Dunn, J. J. RNase III cleavage of single-stranded RNA: Effect of ionic strength in the fidelity of cleavage. Journal of Biological Chemistry. 251 (12), 3807-3814 (1976).
- Pertzev, A., Nicholson, A. W. Characterization of RNA sequence determinants and antideterminants of processing reactivity for a minimal substrate of Escherichia coli ribonuclease III. Nucleic Acids Research. 34 (13), 3708-3721 (2006).
- Phillips, D. D., et al. The sub-nanomolar binding of DNA-RNA hybrids by the single chain Fv fragment of antibody S9.6. Journal of Molecular Recognition. 26 (8), 376-381 (2013).
- Haruki, M., Noguchi, E., Kanaya, S., Crouch, R. J. Kinetic and stoichiometric analysis for the binding of Escherichia coli ribonuclease H1 to RNA-DNA hybrids using surface plasmon resonance. Journal of Biological Chemistry. 272 (35), 22015-22022 (1997).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved