Method Article
Dot-Blot ile R-Loop Analizi
Bu Makalede
Özet
Bu protokol, bir RNA-DNA melezi ve yer değiştirmiş bir DNA zincirinden oluşan üç sarmallı bir nükleik asit yapısı olan R-döngüsünü ölçen basit bir yöntemi detaylandırır.
Özet
Üç sarmallı nükleik asit yapısı, R-döngüsü, gen düzenlemesindeki rolü nedeniyle giderek daha fazla tanınmaktadır. Başlangıçta, R-döngülerinin transkripsiyonun yan ürünleri olduğu düşünülüyordu; ancak hastalıklı hücrelerde daha az R-döngüsünün son bulguları, R-döngülerinin çeşitli insan hücrelerinde işlevsel rollere sahip olduğunu açıkça ortaya koydu. Daha sonra, R-döngülerinin rollerini ve hücrelerin bolluklarını nasıl dengelediğini anlamak çok önemlidir. Alandaki bir zorluk, R-döngülerinin nicelikçiliğidir, çünkü çalışmanın çoğu, RNA-DNA hibritleri için özgüllüğü sorgulanan S9.6 monoklonal antikoruna dayanmaktadır. Burada, R-döngülerini ölçmek için S9.6 antikoru ile nokta lekeleri kullanıyoruz ve bu testin RNA-DNA hibritlerini, tek sarmallı RNA'yı ve çift sarmallı RNA'yı parçalayan RNase H, RNase T1 ve RNase III ile duyarlılığını ve özgüllüğünü gösteriyoruz. Bu yöntem yüksek oranda tekrarlanabilir, genel laboratuvar ekipmanı ve reaktifleri kullanır ve iki gün içinde sonuç verir. Bu test, R-döngülerini ölçmek ve senataksin gibi genlerdeki mutasyonların R-döngüsü bolluğu üzerindeki etkisini değerlendirmek için araştırma ve klinik ortamlarda kullanılabilir.
Giriş
Bu protokol, üç sarmallı bir nükleik asit yapısı olan R-döngüsünün bolluğunun hızlı bir karşılaştırmalı değerlendirmesine izin veren bir nokta-leke tahlili için adım adım bir kılavuz sağlar. R-döngüsü, RNA, bir RNA-DNA melezi oluşturmak için çift sarmallı bir DNA'yı istila ettiğinde ve diğer DNA zincirinin yerini aldığında oluşur. R-döngüleri, RNA'nın yaşam döngüsünün farklı aşamalarında bulunur. Transkripsiyonel komplekste, yeni ortaya çıkan RNA, şablon DNA'ya tamamlayıcı olarak sentezlenir ve şablon olmayan iplikçik yer değiştirir. Kısa RNA-DNA hibriti (<10 bp), yeni ortaya çıkan RNA'yı serbest bırakmak için çözülür, böylece RNA polimeraz kompleksini çıkış kanalı 1,2'den terk edebilir. Transkripsiyonel kompleksin dışında, yeni ortaya çıkan RNA, kopyalanmaktan hala biraz çözülmüş olan DNA şablonuna yakındır, bu nedenle RNA, R-döngüleri3 oluşturan şablon DNA'sı ile yeniden hibritleşebilir. Ek olarak, replikasyon ve transkripsiyon kompleksleriçarpıştığında R-döngüleri oluşabilir 4 ve antisens transkripsiyonda5. Oluşumları için birçok fırsat göz önüne alındığında, R-döngüleri nadir değildir ve hücrenin transkripsiyon durumuna bağlı olarak insan genomunun% 3-5'inde6 bulunabilir. R-döngüleri, mRNA'daki gen promotörleri7 ve sonlandırma5 bölgelerinde ve ribozomal RNA8'in yanı sıra transfer RNA9 boyunca bulunur. R-döngüleri ayrıca kromozomların telomerik bölgelerinde bulunur.
R-döngüleri düzenleyici bir rol oynar. Promotörlerde10,11 transkripsiyonu etkileyerek, sınıf anahtarı rekombinasyonuna12 aracılık ederek ve CRISPR tabanlı genom düzenlemesini kolaylaştırarak gen ekspresyonunu düzenlerler 13,14,15. Birçok hücresel olay gibi, R-döngü bolluğu da sıkı bir şekilde titre edilir; çok fazla veya çok az R-döngüsü normal hücre fonksiyonunu etkiler 16,17. R-döngüleri, RNase H, senataxin ve RNA-DNA hibritlerini 18,19,20,21,22 çözen diğer helikazlar dahil olmak üzere çeşitli proteinler tarafından düzenlenir.
R-döngülerinin bolluğunu izlemek için, genom çapında yöntemler önce R-döngüleri için antikor S9.6 8,23,24 veya RNase H10,26,27 dahil olmak üzere diğer nükleazlar 25 ile zenginleştirir ve daha sonra dizileme ile zenginleştirilmiş R-döngülerinin sayısını değerlendirir. Bu dizileme tabanlı yöntemlerin ilk sürümleri, kesin nicelemeye izin vermek için yeterli dizi kapsamı elde etmedi, ancak dizileme teknolojilerindeki hızlı gelişme artık lokus lokus R-döngü analizine izin veriyor. R-döngülerini ölçmek ve lokalize etmek için immünofloresan teknikleri de kullanılmıştır10,17. Bu yöntemler kapsamlıdır, ancak pahalı ekipman ve özel analiz gerektirdiğinden birçok klinik ortamda veya ilk değerlendirmeler olarak pratik değildirler.
Klinik ortamlarda laboratuvarlar arasında aynı şekilde yapılabilen bir prosedür gereklidir. Nokta lekeleri, herhangi bir özel ekipman veya hesaplama analizi olmadan gerçekleştirilebildikleri için böyle bir seçenek sunar. Mutasyonların R-döngüleri üzerindeki etkilerini değerlendirmek için bir ön adım olarak veya klinik ortamlarda, bu nokta lekeleri hassas ve spesifik sonuçlar sağlamalıdır. Burada, R-döngülerini özel olarak tanımlayan tahlilimizi açıklıyoruz; çift sarmallı (ds) DNA, çift sarmallı RNA ve tek sarmallı RNA'dan gelen sinyalleri hariç tutar. Protokolümüz, R-döngülerindeki RNA-DNA hibritlerini tanımlamak için S9.6 antikoru27'yi kullanır ve tespit edilen sinyallerin hibritlere ait olduğundan emin olmak için bir RNA-DNA hibridinde20,28 parçalanan ve dolayısıyla RNA'nın bozulmasına yol açan bir endoribonükleaz olan RNase H'yi içerir. Ayrıca, guanin 29,30'da tek sarmallı RNA'yı parçalayan RNase T1'i ve spesifik olmayan sinyalleri kontrol etmek için kök halkalar 31,32 dahil olmak üzere çift sarmallı RNA'yı parçalayan RNase III'ü de dahil ettik. S9.6 antikoru, sadece 8 nükleotid uzunluğunda olanlar da dahil olmak üzere, değişen uzunluklardaki RNA-DNA hibritlerini tanır33.
Burada, nükleik asit izolasyonu ile başlayan, ardından nokta-blot hazırlığı ve S9.6 antikoru ile R-loop tespiti ile devam eden protokolü sunuyoruz. Protokolümüz, eşit miktarda numunenin yüklenmesini ve sinyallerin spesifik olmasını sağlamak için adımlar içerir. Oligonükleotidlerin pozitif ve negatif kontroller olarak hizmet etmesini sağlar. Bu, R-döngü bolluğunu değerlendirmek için hızlı, kolay ve kullanıcı dostu bir yöntemdir.
Protokol
1. Nükleer fraksiyonasyon için hücre lizisi
- Hücreleri 1x fosfat tamponlu salin (PBS) ile iki kez yıkayın. Tripsin gibi standart hücre ayrışma tekniklerini kullanarak hücreleri doku kültürü kaplarından çıkarın. Bir hemositometre kullanarak hücreleri sayın.
NOT: Aşağıda açıklanan adımlar, birincil insan derisi fibroblastlarının analizi için kullanılmıştır, ancak bir dizi hücre tipi test edilebilir. Fibroblastlar %10 fetal sığır serumu içeren bazal besiyerinde büyütüldü. Alternatif olarak, hücre lizis tamponu (Tablo 1) yıkamadan sonra doğrudan hücre kültürüne eklenebilir. - Hücreleri peletlemek için hücre süspansiyonunu 1.5 mL'lik bir tüpe aktarın.
- Numuneyi 300 x g'da 4 °C'de 5 dakika santrifüjleyin. Medyayı aspire edin.
- Adım 1.3'teki santrifüj ayarlarını kullanarak buz gibi soğuk 1x PBS ile iki kez yıkayın.
- Hücre peletine (2 x 106 hücre başına 300 μL) soğuk hücre lizis tamponu (Tablo 1) ekleyin. Peleti yeniden süspanse etmek için yukarı ve aşağı pipetleyin.
- Buz üzerinde 10 dakika inkübe edin.
- Çekirdekleri peletlemek için 5 dakika boyunca 500 x g'da döndürün.
- Süpernatanı atın ve nükleer peleti 400 μL soğuk nükleer lizis tamponunda yeniden süspanse edin (Tablo 1).
- Buz üzerinde 10 dakika inkübe edin.
NOT: Hücrelerin nükleer ve sitoplazmik bölmelerinin fraksiyonlanması, sinyal özgüllüğünü sağlar. Devam etmeden önce nükleer ve sitoplazmik ayırmanın kalitesi değerlendirilebilir (Tablo 2). - 3 μL 20 mg/mL proteinaz K ekleyin ve 55 °C'de 3-5 saat inkübe edin.
NOT: Belirtilen hacimler 2 x 106 hücre içindir, gerektiği gibi büyütün veya küçültün.
2. Genomik DNA'nın saflaştırılması (RNA-DNA hibritlerini içerir)
- DNA viskoz ise, viskoziteyi azaltmak için sonikasyon yapın (örneğin, yüksek güç çıkışında sonikasyon, 30 s AÇIK / 30 s KAPALI, 4 °C su banyosu kullanarak 2 dakika boyunca).
- 400 μL elüsyon tamponu (Tablo 1) ve 400 μL fenol:kloroform:izoamil alkol (25:24:1 pH 8.0) ekleyin.
- 10 saniye boyunca girdap.
- 4 ° C'de 5 dakika boyunca 12.000 x g'da döndürün.
- Sulu fazı (yaklaşık 350 μL) yeni bir tüpe aktarın.
- 1 hacim kloroform kullanarak bir kez ekstrakte edin, 10 saniye boyunca girdaplayın, ardından 4 ° C'de 5 dakika boyunca 12.000 x g'da döndürün. Sulu fazı yeni bir tüpe (yaklaşık 300 μL) aktarın.
- 35 μL 3 M sodyum asetat (pH 5.2), 1 μL glikojen ve 700 μL buz gibi %100 etanol ekleyin.
- 10 saniye boyunca vorteksleyin ve 4 ° C'de 30 dakika boyunca 12.000 x g'da döndürün.
- Peleti 1 mL% 70 etanol ile yıkayın.
- 10 saniye boyunca vorteksleyin ve 4 ° C'de 15 dakika boyunca 12.000 x g'da döndürün.
- Süpernatanı atın ve pelet havasının kurumasını bekleyin.
- 12 μL elüsyon tamponu ekleyin ve yeniden askıya almak için 10 saniye boyunca girdap yapın. Peleti yeniden süspanse etmek için numuneyi 37 °C'de 37 °C'de 30 dakika veya gece boyunca 4 °C'de inkübe edin.
- Standart spektrofotometri kullanarak DNA konsantrasyonunu ölçün.
NOT: Belirtilen hacimler 2 x 106 hücre içindir, gerektiği gibi büyütün veya küçültün. Gerekirse DNA (RNA-DNA hibritleri ile) -20 °C'de saklanabilir.
3. DNA örneklerinin (RNA-DNA hibritlerini içeren) naylon zarlar üzerine lekelenmesi
- Nükleik asitlerin seyreltmelerini elüsyon tamponunda istenen konsantrasyonlara hazırlayın (ör., 50 ng/μL, 25 ng/μL veya 12.5 ng/μL). Bir konsantrasyon aralığına (200, 100, 50, 25, 12.5 ng) sahip bu numuneler, doğrusal aralık içinde sinyallerin olmasını sağlar.
NOT: Teknik ve biyolojik kopyalar için yeterli numune hazırladığınızdan emin olun ve çeşitli RNaz tedavileri için Adım 5'e bakın. - Her 2 μL'lik numune için 0,5 cm2'lik bir alanı kaplayacak şekilde pozitif yüklü bir naylon membran hazırlayın.
- Her numunenin 2 μL'sini 2 zar üzerine yerleştirin: biri S9.6 antikoru ve diğeri dsDNA antikoru için. Alternatif olarak, daha büyük hacimli numunelerin yüklenmesine izin veren bir nokta-leke veya yarık-leke aparatı kullanılabilir.
- Numunelerin zara doymasına izin verin. Membranı UV ışığı ile çapraz bağlamadan önce en az 2 dakika bekleyin.
- Membranı UV cihazının merkezine yerleştirin ve "Otomatik Çapraz Bağlama" ayarını (1.200 μJ x 100) kullanarak bir UV çapraz bağlayıcı kullanarak membranı çapraz bağlayın.
4. S9.6 antikoru ile RNA-DNA hibrid tespiti
- Membranı bir çalkalayıcı üzerinde oda sıcaklığında 1 saat boyunca bloke edici çözelti (% 0.05 Tween-20 (TBST) ile Tris tamponlu salin içinde% 5 süt inkübe edin.
NOT: Membranı örtmek için yeterli blokaj solüsyonu bulunmalıdır. - Membranları gece boyunca primer antikorda (TBST'de% 5 sütte) 4 ° C'de çalkalayarak inkübe edin. Bir zara anti-dsDNA antikoru (1: 10.000 seyreltme) ekleyin. İkinci zara 1μg / mL S9.6 antikoru ekleyin (1: 1.000 seyreltme).
NOT: S9.6 antikoru ticari olarak veya Dr. S. Leppla, NIAID, Ulusal Sağlık Enstitüleri'nden temin edilebilir. - Birincil antikoru çıkarın ve 3x'i TBST ile yıkayın. Her yıkamayı oda sıcaklığında çalkalanarak 5-10 dakika boyunca gerçekleştirin.
- Yaban turpu peroksidaz (HRP) konjuge ikincil antikoru (anti-fare, 1: 5.000 seyreltme) ile TBST'de% 5 süt içinde oda sıcaklığında çalkalanarak inkübe edin.
NOT: Anti-dsDNA ve anti-RNA-DNA hibridinin her ikisi de fare antikorlarıdır. - İkincil antikoru çıkarın ve oda sıcaklığında çalkalanarak 5-10 dakika boyunca TBST ile 3x'i yıkayın.
- Görüntüleme için sinyaller elde etmek için gelişmiş kemilüminesans (ECL) reaktifleri ile geliştirin.
- ImageJ gibi standart görüntü işleme araçlarını kullanarak sinyal yoğunluğunu ölçün.
NOT: Sorun giderme Tablo 2'de ayrıntılı olarak açıklanmıştır.
5. Sinyal özgüllüğünü değerlendirmek için ribonükleaz tedavileri
NOT: S9.6 bağlanmasının özgüllüğünü göstermek için nükleik asit numuneleri üzerinde RNaz tedavisi yapılmalıdır. RNase H ile tedavi, ancak RNase T1 veya RNase III ile tedavi, S9.6 immün boyamada bir azalmaya neden olmalıdır.
- RNA-DNA hibritleri içeren örnekleri dört ayrı tüpte hazırlayarak sindirin. 4 numunenin her birine 5 U RNase H, 1000 U RNase T1, 0.5 U RNase III veya sahte ile muamele edin. Numuneleri 37 °C'de 20 μL'lik hacimlerde 15 dakika inkübe edin.
- Her bir numuneden 2 μL'yi bölüm 3'te açıklandığı gibi bir membran üzerine yükleyin.
6. Sinyal özgüllüğünü değerlendirmek için oligonükleotid kontrollerinin hazırlanması
NOT: Oligonükleotid kontrolleri, S9.6 bağlanmasının özgüllüğünü göstermek için kullanılabilir. S9.6, RNA-DNA hibritlerini tanır, ancak daha önce bildirildiği gibi dsDNA veya dsRNA kontrollerini tanımaz34.
- Oligonükleotidleri (Tablo 3) tavlama tamponunda (10 mM Tris, pH 8.0; 50 mM NaCl, 1 mM EDTA) 100 μM'ye çözün.
- İçin 4 reaksiyon tüpü hazırlayın
- RNA-DNA hibriti #1: 10 μL ssRNA üst zincirini 10 μL ssDNA alt ipliği ve 80 μL tavlama tamponu ile karıştırın.
- RNA-DNA hibriti #2: 10 μL ssDNA üst zincirini 10 μL ssRNA alt ipliği ve 80 μL tavlama tamponu ile karıştırın.
- dsRNA: 10 μL ssRNA üst ipliği ile 10 μL ssRNA alt ipliği ve 80 μL tavlama tamponu karıştırın.
- dsDNA: 10 μL ssDNA üst ipliğini, 10 μL ssDNA alt ipliği ve 80 μL tavlama tamponu ile karıştırın.
- Adım 6.2'deki 4 karışımı 95 °C'de 10 dakika ısıtın.
- Tellerin yeniden tavlanmasına izin vermek için tüplerin yavaşça oda sıcaklığına soğumasına izin verin. Tavlanmış standartlar daha sonra kullanılmak üzere -20 °C'de saklanabilir.
NOT: Tavlama verimliliği, denatüre olmayan jel elektroforezi ile kontrol edilmelidir. Dupleksler, tavlanmamış oligonükleotidlerden daha yavaş göç eder (Tablo 2). - Bölüm 3'te açıklandığı gibi, biri S9.6 antikoru ve diğeri dsDNA antikoru için olmak üzere 2 membran üzerine her numuneden 2 μL yükleyin.
- Bölüm 4'te açıklanan adımları gerçekleştirin.
7. ImageJ kullanılarak S9.6 R-döngü sinyal yoğunluğunun ölçülmesi ve normalleştirilmesi.
- S9.6, dsDNA boyama görüntülerini TIFF formatında kaydedin ve bunları ImageJ yazılımını (https://imagej.nih.gov/ij/) kullanarak analiz edin.
- Görüntüyü ters çevir seçeneğini seçin (Düzenle | Ters Çevir). Ters çevirme işleminden sonra, her nokta koyu bir arka plana karşı beyaz olarak görünecektir.
- Görüntüdeki en büyük noktayı çevreleyecek kadar büyük bir oval seçmek için oval görüntü seçim aracını kullanın.
- Niceleme için seçilen alanı eklemek için ROI yöneticisini kullanın. İlgilenilen bölgelerin görselleştirilebilmesi için "Tümünü Göster" ve "Etiketler" seçeneklerinin seçili olduğundan emin olun.
- Ölçülecek her noktanın etrafına ek ilgi alanları eklemek için adım 7.3'te kullanılan aynı oval seçim alanını kullanın. Seçili alanı adım 7.3'ten sonraki noktaların her birine kopyalamak için Command + Shift + E kısayolunu kullanın.
- İlgilenilen bölgelerin her birinin entegre yoğunluğunu ölçün.
- S9.6/dsDNA sinyal oranını elde etmek için her numune için S9.6 sinyal yoğunluğunu dsDNA ölçümüne bölün. Deneyleri tekrarlayarak sonuçları doğrulayın (hem S9.6 hem de dsDNA sinyal alımı için en az üç katına çıkar). Ortalamanın standart hatası, S9.6 / dsDNA sinyal oranlarından hesaplanabilir.
Sonuçlar
S9.6 (RNA-DNA) antikorunun özgüllüğünü değerlendirmek için enzimatik tedavi.
Birincil insan derisi fibroblastları büyütüldü17. RNA-DNA hibritleri olan DNA izole edildi ve niceliği belirlendi. Numunelerin iki μg'ı 37 ° C'de 15 dakika boyunca RNase T1, RNase H veya RNase III ile sindirildi. RNaz ile muamele edilen numunelerle karşılaştırma için sahte bir numune de analiz edildi. Numuneler (200, 100, 50, 25, 12.5 veya 6.25 ng), bölüm 3'te tarif edildiği gibi iki farklı zar üzerine lekelendi. Membranlar çapraz bağlandı, bloke edildi ve bunlardan biri S9.6 antikoru ile problandı (Şekil 1A).
Sonuçlar, S9.6 sinyalinin yüklenen numunenin bolluğu ile ilişkili olduğunu gösterdi. RNase H ile tedavi, ancak RNase T1 veya RNase III ile tedavi edilmemesi, S9.6 boyamasında bir azalmaya neden olur.
Normalizasyon için ikinci bir membran bir dsDNA antikoru (Şekil 1B) ile incelendi. Sinyal yoğunluklarını analiz etmek için resim J kullanıldı. S9.6 ve dsDNA antikorlarından gelen sinyal yoğunlukları dinamik aralık içinde olduğu için 50 ng numuneler miktar tayini için seçildi. Sinyal yoğunlukları, sahte örneklerdekilere göre normalleştirildi. Veriler Şekil 2'de gösterilmiştir.
Sentetik nükleotid kontrolleri kullanan S9.6 antikor nokta lekesi.
S9.6 antikorunun özgüllüğünü değerlendirmek için, bölüm 6'da açıklandığı gibi dsRNA, dsDNA ve RNA-DNA'ya karşılık gelen oligonükleotidleri kullandık. RNA-DNA, dsRNA ve dsDNA nükleotidlerinden oluşan bir seyreltme serisi hazırlandı ve bölüm 3'te tarif edildiği gibi naylon zar üzerine lekelendi. Membran S9.6 antikoru ile incelendi (Şekil 3). Sonuçlar, S9.6 antikorunun doza bağlı bir şekilde spesifik olarak RNA-DNA hibritlerine bağlandığını ve dsRNA'lara ve dsDNA'lara minimum çapraz reaktivite gösterdiğini gösterdi.
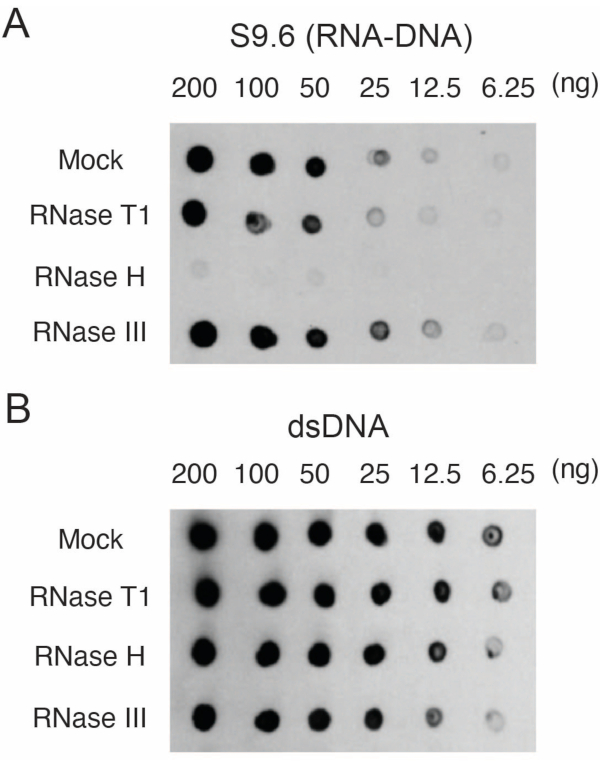
Şekil 1: İnsan fibroblastlarından nükleik asitlerle yüklü nokta-leke ile gösterildiği gibi S9.6'nın özgüllüğü. İnsan fibroblastlarından alınan nükleik asit örnekleri ya sahte muamele edildi ya da RNase T1, RNase H veya RNase III ile muamele edildi ve daha sonra 2 μL nokta başına 200, 100, 50, 25, 12.5 ve 6.25 ng'lik bir seyreltme serisinde naylon membranlara yüklendi. Membranlar daha sonra S9.6 antikoru (A) veya dsDNA antikoru (B) ile incelendi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
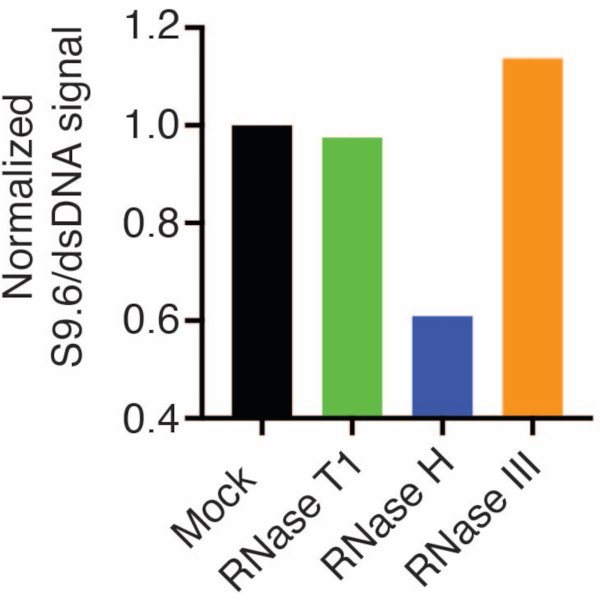
Şekil 2: S9.6 R-loop boyamanın miktar tayini. Şekil 1'den 50 ng numune, ImageJ ile miktar tayini için seçildi. S9.6 sinyali, dsDNA sinyal yoğunluğuna bölündü, daha sonra bölüm 7'de belirtilen adımlar izlenerek sahte numuneye normalize edildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
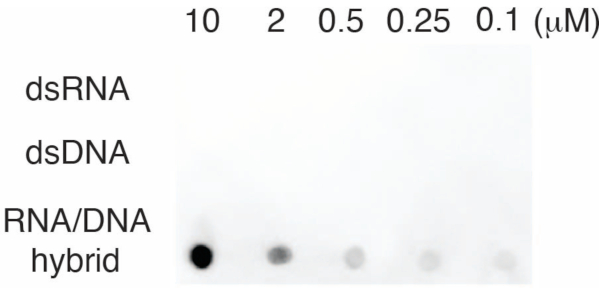
Şekil 3: Oligonükleotid kontrollerini kullanan S9.6 nokta lekesi. dsRNA, dsDNA veya RNA-DNA hibriti olarak bir dizi sentetik oligonükleotidin seyreltilmesine karşı S9.6 antikoru nokta-lekesi. S9.6, doza bağlı bir şekilde spesifik olarak RNA-DNA hibritlerine bağlanır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
| Hücre lizis tamponu | 10 mL için | 200 mL için | Son Danışmanlık. |
| Su, nükleaz içermez | 9 milyon | 180 milyon | - |
| % 10 NP-40 | 0,5 mL | 10 milyon | 0.5% |
| 2 milyon KCl | 0,4 mL | 8 milyon | 80 milyon |
| 0,5 M BORULAR (PH 8,0) | 100 uL | 2 milyon | 5 milyon |
| Nükleer lizis tamponu | 10 mL için | 200 mL için | Son Danışmanlık. |
| Su, nükleaz içermez | 8,65 milyon | 173 milyon | - |
| %10 SDS | 1 mL | 20 milyon | 1% |
| 1M Tris-HCl (pH 8.0) | 0,25 mL | 5 milyon | 25 milyon |
| 0,5 milyon EDTA | 100 uL | 2 milyon | 5 milyon |
| Elüsyon Tamponu | 10 mL için | 200 mL için | Son Danışmanlık. |
| 1M Tris-Cl, pH 8.5 | 0.1 mL | 2 mL | 10 milyon |
| Su, nükleaz içermez | 9.9 mL | 198 mL | - |
Tablo 1. Tamponların hazırlanması
| Adım | Sorun | Olası neden: | Çözüm |
| 1.9 | YOK | Hücre fraksiyonunu doğrulayın | Nükleer ve sitoplazmik ayırmanın kalitesi, hücreye standart proteaz inhibitör kokteylleri ve nükleer lizis tamponları eklenerek değerlendirilebilir (Tablo 1). Sitoplazmik ve nükleer fraksiyonlar, sitoplazmik fraksiyonlarda sitoplazmik belirteçlerle (örneğin, GAPDH veya HSP90) etiketlemenin ve nükleer fraksiyonlarda nükleer belirteçlerle (örneğin, HDAC1 veya Histon H3) etiketlemenin yeterli kısıtlamasını doğrulamak için batı lekeleme analizi ile değerlendirilebilir. Mitokondriyal kontaminasyonun nükleer fraksiyona katkısı, mitokondriyal DNA'ya özgü problarla qPCR analizi ile değerlendirilebilir. |
| 2.1 | Örnek çok viskoz | Hücre sayısı çok yüksek. | DNA'yı yarı yarıya azaltın ve sonikasyon adımı 2.1 ile devam edin |
| 2.12 | Görünür saçma yok | Yetersiz başlangıç materyali veya ekstraksiyon sırasında kayıp. | Daha fazla hücre kullanarak baştan başlayın. |
| 2.13 | Düşük DNA konsantrasyonu | ||
| 3.1 | Seyreltmeler için yeterli DNA yok | ||
| 3.4 | Numune zara doymaz | Membranı 1x TBST'ye batırın. Fazla tamponun kurumasını bekleyin ve adım 3.4'ten başlayın | |
| 4.6 | Yamalı veya benekli desen algılandı | Numuneye %0,1 Sodyum dodesil sülfat (SDS) ekleyin ve 3.1. adımdan devam edin | |
| 4.6 | Noktalar "kahve halkası" görünümündedir | Numuneye %0,01 Sarkosyl ekleyin ve adım 3.1'de devam edin | |
| 5.2 | RNaseH kontrolü sinyalde azalma göstermez | Ribonükleaz sindirimi eksiktir. | İnkübasyonu artırın veya enzim konsantrasyonunu artırın. |
| 6.5 | Oligo kontrolleri için sinyal yok | Dubleks oluşturulmadı. Oligonükleotidler uygun şekilde tavlanmadı. | Oligonükleotidlerin ve tavlama tamponunun oranlarını doğrulayın. |
| 6.5 | S9.6 sinyali hibritlere özgü değildir | S9.6 antikor partisi spesifik olmayan bağlanmaya sahiptir | RNaz enzim tedavileri veya sentetik oligonükleotid analizi kullanarak yeni S9.6 antikor partilerinin duyarlılığını ve özgüllüğünü doğrulayın. |
Tablo 2: Sorun Giderme
| ssRNA, üst iplikçik | 5'-UGGGGGCUCGUCCGGGAUAUGGGAACCACUGAUCCC-3' |
| ssDNA, üst iplikçik | 5'-TGGGGGCTCGTCCGGGATATGGGAACCACTGATCCC-3' |
| ssRNA, alt iplikçik | 5'-GGGAUCAGUGGUUCCCAUAUCCCCCACGAGCCCCCA-3' |
| ssDNA, alt iplikçik | 5'-GGGATCAGTGGTTCCCATATCCCGGACGAGCCCCCA-3' |
Tablo 3. Oligonükleotid dizilerini kontrol edin
Tartışmalar
3 sarmallı nükleik asitler, R-döngüleri, RNA'nın yaşam döngüsü boyunca farklı aşamalarda oluşur ve hücresel süreçleri düzenlediği giderek daha fazla bulunur. R-döngülerini tam olarak anlamak için, R-döngüsü tespiti için güvenilir teknikler gereklidir. Burada, S9.6 antikoru 8,23,24 kullanarak R-döngülerinin bolluğunu sorgulamak için bir yaklaşım açıklıyoruz. Bu yöntem, hücrelerden ve doku kültürü örneklerinden R-döngü bolluğunun hızlı bir şekilde değerlendirilmesine izin verir. Özel ekipman veya büyük miktarda başlangıç malzemesi gerektirmez. RNase tedavilerinin bir kombinasyonunu kullanarak spesifik ve tekrarlanabilir sonuçlar sağlar.
Bazıları S9.6 antikorunun özgüllüğü hakkında endişeler bildirmiştir. Herhangi bir reaktifte olduğu gibi, S9.6 antikoru ile partiden partiye değişkenlik olabilir. Protokolümüz, sinyal özgüllüğünü kontrol etmek için RNase H, RNase T1 ve RNase III'ü içerir. Ek olarak, her bir S9.6 antikor partisinin özgüllüğünü sağlamak için sentetik oligonükleotidler kullanıyoruz.
R-döngü biyolojisi büyüyen bir alandır; Burada sunulanlar gibi güvenilir algılama ve niceleme yöntemlerinin geliştirilmesi, R-döngülerinin ne zaman oluştuğunu, nasıl düzenlendiklerini ve neyi düzenlediklerini aydınlatmak için mekanik çalışmaları kolaylaştıracaktır. Uygun kontrollerle, bu nokta-leke testi, klinik ve araştırma ortamlarında R-döngü bolluğunu taramak için basit bir yöntemdir.
Açıklamalar
Yazarların ifşa edecek hiçbir şeyi yok.
Teşekkürler
Bu çalışma Howard Hughes Tıp Enstitüsü ve Ulusal Nörolojik Bozukluklar ve İnme Enstitüsü'ndeki Intramural Araştırma tarafından desteklenmiştir.
Analiz için S9.6 antikoru partilerini sağladığı için Dr. Stephen Leppla'ya teşekkür ederiz. Ayrıca ribonükleaz tedavilerindeki yardımı için Dr. Dongjun Li'ye teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Anti-dsDNA antibody | Abcam | ab27156 | |
| Anti-RNA-DNA hybrid antibody (S9.6) | Kerafast | ENH001 | |
| Biorupter sonicator | Diagenode | UCD-200 | |
| EB Buffer | Qiagen | 19086 | |
| EDTA (0.5M) | Invitrogen | AM9261 | |
| Hybond N+ nylon membrane | GE healthcare Life Sciences | RPN203B | |
| KCl (2M) | Invitrogen | AM9640G | |
| NP-40 (Igepal CA-630) | Sigma | I8896 | |
| PBS | Invitrogen | 10010-023 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593031 | |
| PIPES (0.5M, pH 8.0) | VWR | AAJ61406-AE | |
| Proteinase K | Qiagen | 19131 | |
| RNase III | Invitrogen | AM2290 | |
| RNase H | New England Biolabs | M0297 | |
| RNase T1 | ThermoFisher Sci. | EN0541 | |
| SDS (10%) | Invitrogen | 15553027 | |
| sodium acetate (3M, pH 5.2) | Invitrogen | AM9740 | |
| Tris-buffered saline (10X) | Corning | 46-012-CM | |
| Tris-HCl (1M, pH 8.0) | KD Medical | RGF-3360 | |
| TrypLE | Invitrogen | 12605010 | |
| Tween-20 | Sigma | P9416 | |
| UV Stratalinker 2400 | Stratagene | Stratalinker 2400 | |
| Whatman marking pen | Sigma | WHA10499001 |
Referanslar
- Daube, S. S., von Hippel, P. H. RNA displacement pathways during transcription from synthetic RNA-DNA bubble duplexes. Biochemistry. 33 (1), 340-347 (1994).
- Westover, K. D., Buschnell, D. A., Kornberg, R. D. Structural basis of transcription: separation of RNA from DNA by RNA polymerase II. Science. 303 (5660), 1014-1016 (2004).
- Itoh, T., Tomizawa, J. Formation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. Proceedings of the National Academy of Science USA. 77 (5), 2450-2454 (1980).
- Hamperl, S., Bocek, M. J., Saldivar, J. C., Swigut, T., Cimprich, K. A. Transcription-replication conflict orientation modulates R-loop levels and activates distinct DNA damage responses. Cell. 170 (4), 774-786 (2017).
- Skourti-Stathaki, K., Kamieniarz-Gdula, K., Proudfoot, N. J. R-loops induce repressive chromatin marks over mammalian gene terminators. Nature. 516 (7531), 436-439 (2014).
- Sanz, L. A., et al. conserved R-loop structures associate with specific epigenomic signatures in mammals. Molecular Cell. 63 (1), 167-178 (2016).
- Chen, L., et al. R-ChIP using inactive RNase H reveals dynamic coupling of R-loops with transcriptional pausing at gene promoters. Molecular Cell. 68 (4), 745-757 (2017).
- El Hage, A., French, S. L., Beyer, A. L., Tollervey, D. Loss of topoisomerase I leads to R-loop mediated transcriptional blocks during ribosomal RNA synthesis. Genes and Development. 24 (14), 1546-1558 (2010).
- Tran, P., et al. PIF1 family DNA helicases suppress R-loop mediated genome instability at tRNA genes. Nature Communication. 8, 15025(2017).
- Ginno, P. A., Lott, P. L., Christensen, H. C., Korf, I., Chedin, F. R-loop formation is a distinctive characteristic of unmethylated human CpG island promoters. Molecular Cell. 45 (6), 814-825 (2012).
- Colak, D., et al. Promoter-bound trinucleotide repeat mRNA drives epigenetic silencing in fragile X syndrome. Science. 343 (6174), 1002-1005 (2014).
- Yu, K., Chedin, F., Hsieh, C., Wilson, T. E., Lieber, M. R. R-loops at immunoglobulin class switch regions in the chromosomes of stimulated B cells. Nature Immunology. 4 (5), 442-451 (2003).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleotide complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Science USA. 109 (39), 2579-2586 (2012).
- Westra, E. R., et al. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3. Molecular Cell. 46 (5), 595-605 (2012).
- Jiang, F., et al. Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. Science. 351 (6275), 867-871 (2016).
- Huertas, P., Aguilera, A. Cotranscriptionally formed DNA:RNA hybrids mediate transcription elongation impairment and transcription-associated recombination. Molecular Cell. 12 (3), 711-721 (2003).
- Grunseich, C., et al. Senataxin mutation reveals how R-loops promote transcription by blocking DNA methylation at gene promoters. Molecular Cell. 69 (3), 426-437 (2018).
- Kim, H. D., Choe, J., Seo, Y. S. The sen1(+) gene of Schizosaccharomyces pombe, a homologue of budding yeast SEN1, encodes an RNA and DNA helicase. Biochemistry. 38 (44), 14697-14710 (1999).
- Stein, H., Hausen, P. Enzyme from calf thymus degrading the RNA moiety of DNA-RNA hybrids: effect on DNA-dependent RNA polymerase. Science. 166 (3903), 393-395 (1969).
- Cerritelli, S. M., Crouch, R. J. Ribonuclease H: the enzymes in eukaryotes. FEBS Journal. 276 (6), 1494-1505 (2009).
- Hyjek, M., Figiel, M., Nowotny, M. RNases H: Structure and mechanism. DNA Repair. 84, 102672(2019).
- Pohl, T. J., Zakian, V. A. Pif1 family DNA helicases: A helpmate to RNase H. DNA Repair. 84, 102633(2019).
- Pohjoismaki, J. L., et al. Mammalian mitochondrial DNA replication intermediates are essentially duplex but contain extensive tracts of RNA/DNA hybrid. Journal of Molecular Biology. 397 (5), 1144-1155 (2010).
- Sanz, L. A., Chedin, F. High-resolution, strand-specific R-loop mapping via S9.6-based DNA-RNA immunoprecipitation and high-throughput sequencing. Nature Protocols. 14 (6), 1734-1755 (2019).
- Wahba, L., Costantino, L., Tan, F. J., Zimmer, A., Koshland, D. S1-DRIP-seq identifies high expression and polyA tracts as major contributors to R-loop formation. Genes and Development. 30 (11), 1327-1338 (2016).
- Yan, Q., Shields, E. J., Bonasio, R., Sarma, K. Mapping native r-loops genome-wide using a targeted nuclease approach. Cell Reports. 29 (5), 1369-1380 (2019).
- Hu, Z., Zhang, A., Storz, G., Gottesman, S., Leppla, S. H. An antibody-based microarray assay for small RNA detection. Nucleic Acids Research. 34 (7), 52(2006).
- Nowotny, M., Gaidamakov, S. A., Crouch, R. J., Yang, W. Crystal structures of RNase H bound to an RNA/DNA hybrid: substrate specificity and metal-dependent catalysis. Cell. 121 (7), 1005-1016 (2005).
- Sato, K., Egami, F. Studies on ribonucleases in takadiastase. Journal of Biochemistry. 44 (11), Tokyo. 753-767 (1957).
- Takahashi, K., Moore, S. Ribonuclease T1. Enzymes. 15, 435-468 (1982).
- Dunn, J. J. RNase III cleavage of single-stranded RNA: Effect of ionic strength in the fidelity of cleavage. Journal of Biological Chemistry. 251 (12), 3807-3814 (1976).
- Pertzev, A., Nicholson, A. W. Characterization of RNA sequence determinants and antideterminants of processing reactivity for a minimal substrate of Escherichia coli ribonuclease III. Nucleic Acids Research. 34 (13), 3708-3721 (2006).
- Phillips, D. D., et al. The sub-nanomolar binding of DNA-RNA hybrids by the single chain Fv fragment of antibody S9.6. Journal of Molecular Recognition. 26 (8), 376-381 (2013).
- Haruki, M., Noguchi, E., Kanaya, S., Crouch, R. J. Kinetic and stoichiometric analysis for the binding of Escherichia coli ribonuclease H1 to RNA-DNA hybrids using surface plasmon resonance. Journal of Biological Chemistry. 272 (35), 22015-22022 (1997).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır