A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تحديد البروتينات الملزمة للليجاندات الصغيرة مع العمل الشعيرات الدموية الشعاعية التفاضلية من المقايسة ليغاند (DRaCALA)
In This Article
Summary
يمكن استخدام العمل الشعيرات الدموية الشعاعية التفاضلية من المقايسة ليغاند (DRaCALA) لتحديد البروتينات ملزمة ليغاند الصغيرة من كائن حي باستخدام مكتبة ORFeome.
Abstract
شهد العقد الماضي تقدما هائلا في فهم جزيئات الإشارات الصغيرة في فسيولوجيا البكتيريا. وعلى وجه الخصوص، تم تحديد البروتينات المستهدفة للعديد من الرسل الثانويين المشتقة من النيوكليوتيدات (NSMs) بشكل منهجي ودراسة في الكائنات الحية النموذجية. ويعزى هذا الإنجاز أساسا إلى تطوير العديد من التقنيات الجديدة بما في ذلك تقنية مركب التقاط والعمل الشعيرات الدموية الشعاعية التفاضلية من المقايسة ليغاند (DRaCALA)، والتي استخدمت لتحديد البروتينات المستهدفة بشكل منهجي من هذه الجزيئات الصغيرة. تصف هذه الورقة استخدام NSMs، وبانتوسين بنتا- و tetraphosphates (p)ppGpp، كمثال ومظاهرة فيديو لتقنية DRaCALA. باستخدام DRaCALA، تم تحديد 9 من أصل 20 البروتينات المستهدفة المعروفة و 12 من (ع) ppGpp في الكائن الحي النموذجي، Escherichia القولونية K-12، مما يدل على قوة هذا المقايسة. من حيث المبدأ ، يمكن استخدام DRaCALA لدراسة الليغندات الصغيرة التي يمكن تصنيفها بالنظائر المشعة أو الأصباغ الفلورية. تتم مناقشة الخطوات الحاسمة والايجابيات والسلبيات من DRaCALA هنا لمزيد من التطبيق لهذه التقنية.
Introduction
البكتيريا استخدام عدة جزيئات الإشارات الصغيرة للتكيف مع بيئات المتغيرة باستمرار1،2. على سبيل المثال، فإن الحثات الذاتية، N-acylhomoserine lactones وoligopeptides المعدلة، والتوسط في الاتصالات بين الخلايا بين البكتيريا لتنسيق السلوك السكاني، وهي ظاهرة تعرف باسم استشعار النصابالقانوني 2. مجموعة أخرى من جزيئات الإشارات الصغيرة هي NSMs ، بما في ذلك الأدينوسين الدوري الذي تمت دراسته على نطاق واسع أحادي الفوسفات (cAMP) ، ودي أمبير الدوري ، والفوسفات الأحادي الدوري دي جوانوسين (الدوري دي GMP) ، وغوانوزين بنتا - وتترا فوسفات (p)ppGpp1. البكتيريا تنتج هذه NSMs كاستجابة لمجموعة متنوعة من ظروف الإجهاد المختلفة. بمجرد إنتاجها ، ترتبط هذه الجزيئات بالبروتينات المستهدفة وتنظم العديد من المسارات الفسيولوجية والأيضية المختلفة للتعامل مع الضغوط التي تواجهها وتعزيز البقاء البكتيري. لذلك ، فإن تحديد البروتينات المستهدفة هو شرط أساسي لا مفر منه لفك الرموز الوظائف الجزيئية لهذه الجزيئات الصغيرة.
وقد شهد العقد الماضي طفرة في المعرفة بهذه الجزيئات الصغيرة التي تشير إلى ذلك، ويرجع ذلك أساسا إلى العديد من الابتكارات التقنية التي كشفت عن البروتينات المستهدفة لهذه الجزيئات الصغيرة. وتشمل هذه تقنية مركب التقاط3،4،5، والعمل الشعيرات الدموية الشعاعية التفاضلية من المقايسة ليغاند (DRaCALA)6 لمناقشتها في هذه الورقة.
اخترعها فنسنت لي وزملاء العمل في 20116, DRaCALA تنشر قدرة غشاء النيتروسليلوز لعزل التفاضلية الحرة والبروتين ملزمة ligands. جزيئات مثل البروتينات لا يمكن أن تنتشر على غشاء النيتروسليلوز، في حين أن الليغنديات الصغيرة، مثل NSMs، قادرة على. عن طريق خلط NSM(على سبيل المثال، ppGpp) مع البروتين لاختبارها واكتشافها على الغشاء ، يمكن توقع سيناريوهين(الشكل 1):إذا (ع) ppGpp يربط البروتين ، سيتم الاحتفاظ بالعلامات المشعة (p) ppGpp في وسط البقعة بواسطة البروتين ولن ينتشر إلى الخارج ، مما يعطي نقطة صغيرة مكثفة (أي، إشارة مشعة قوية) تحت الفوسفوريماجر. ومع ذلك، إذا (ع)ppGpp لا يرتبط البروتين، وسوف تنتشر بحرية إلى الخارج لإنتاج بقعة كبيرة مع إشارة مشعة خلفية موحدة.
وعلاوة على ذلك، يمكن الكشف عن DRaCALA التفاعل بين جزيء صغير وبروتين غير مبور في lysate خلية كاملة إذا كان البروتين موجود في كمية كافية. هذه البساطة تسمح باستخدام DRaCALA في تحديد أهداف البروتين بسرعة باستخدام مكتبة التعبير ORFeome. في الواقع، البروتينات المستهدفة من cAMP7، دوري دي AMP8، دوري دي GMP9،10، و (ع) ppGpp11،12،13 وقد تم تحديد منهجي باستخدام DRaCALA. تستخدم هذه المقالة الفيديو (p)ppGpp كمثال على إظهار ووصف الخطوات والاعتبارات الهامة في إجراء فحص DRaCALA ناجحة. ملاحظة، ينصح بشدة وصف أكثر شمولا من DRaCALA14 لقراءة بالاشتراك مع هذه المقالة قبل تنفيذ DRaCALA.
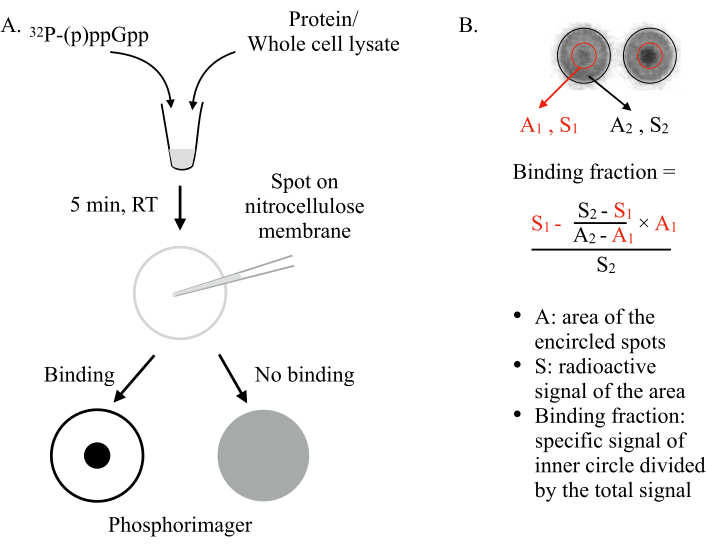
الشكل 1: مبدأ DRaCALA. (أ) التخطيطي للدراكالا المقايسة. راجع النص للحصول على التفاصيل. (ب)تحديد وحساب الكسر الملزم. راجع النص للحصول على التفاصيل. باختصار، سيتم تحليل البقع DRaCALA عن طريق رسم دائرتين التي تحد من بقعة كاملة والنقطة المظلمة الداخلية(أي.،وتحتفظ (ع) ppGpp بسبب ربط البروتين اختبارها). إشارة الربط المحددة هي الإشارة المشعة للدائرة الداخلية (S1) بعد طرح إشارة الخلفية غير المحددة (محسوبة ب A1 × ((S2-S1)/(A2-A1)). الكسر الملزم هو إشارة الربط المحددة مقسومة على إجمالي الإشارة المشعة (S2). المختصرات: DRaCALA = العمل الشعيرات الشعرية الشعاعية التفاضلية من المقايسة ليغاند; (ع) ppGpp = جوانوسين بنتا - وتيترافوسفات؛ RT = درجة حرارة الغرفة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. إعداد lysates الخلية بأكملها
- تلقيح E. coli K-12 ASKA ORFeome جمعسلالات 15 إلى 1.5 مل مرق ليسوجيني (LB) التي تحتوي على 25 ميكروغرام / مل الكلورامفينيكول في 96 جيدا لوحات الآبار العميقة. تنمو بين عشية وضحاها (O / N) لمدة 18 ساعة في 30 درجة مئوية مع اهتزاز في 160 دورة في الدقيقة. في اليوم التالي، أضف الأيزوبروبيل β-د-1-ثيوغالاكتوبترانوزيد (IPTG) (0.5 مليون متر نهائي) إلى ثقافات O/N للحث على التعبير عن البروتين عند 30 درجة مئوية لمدة 6 ساعة.
- الكريات الخلايا في 500 × ز لمدة 10 دقيقة. تجميد الكريات في -80 درجة مئوية حتى الاستخدام. تحلل الخلايا، إضافة 150 ميكرولتر من تحلل العازلة L1 (40 mM تريس درجة الحموضة 7.5، 100 م م كلوريد كلوريد، 10 مللي متر ملغم2،تكملها 2 mM فلوريد الفينيلميل سلفونيل (PMSF)، 40 ميكروغرام/ مل DNase 1، و 0.5 ملغم/ مل ليسوزيم) لإعادة إنفاق بيليه.
- تجميد الخلايا في -80 درجة مئوية لمدة 30 دقيقة، ثم إذابة في 37 درجة مئوية لمدة 20 دقيقة. كرر هذه الدورة ثلاث مرات لليس الخلايا. تخزين lysates في -80 درجة مئوية قبل الاستخدام.
2. تنقية ريلseq و GppA
ملاحظة: تستخدم البروتينات المؤتلفة Relseq من العقدية equisimilis وGppA من E. coli K-12 لتجميع pppGpp وppGpp ذات العلامات المشعة، على التوالي.
- تنمو وجمع الخلايا التعبير عن كل بروتين.
- تنمو سلالة E. coli BL21 DE3 تصل إلى مرحلة الأسي (الكثافة البصرية (OD) ~ 0.3-0.4) في مرق LB، وتدور أسفل 1 مل من الثقافة في 6000 × ز لمدة 5 دقائق. Decant supernatant، وإعادة إنفاق الخلايا مع 100 ميكرولتر من مرق TSB الجليد الباردة (LB مرق تكملها مع 0.1 غرام/مل PEG3350، 0.05 مل / مل ديميثيل سلف أكسيد، 20 مللي متر ملغكل2).
- مزيج البلازميدات التي تحمل الهستيدين الموسومة relseq و gppA، كل 100 نانوغرام ، مع تعليق الخلية أعلاه في TSB ، واحتضان على الجليد لمدة 30 دقيقة. الحرارة صدمة الخلايا في 42 درجة مئوية لمدة 40 s. ضع الخليط على الثلج لمدة دقيقتين، وأضف 1 مل من مرق LB في درجة حرارة الغرفة للسماح للخلايا بالتعافي لمدة ساعة واحدة عند 37 درجة مئوية مع الهياج عند 160 دورة في الدقيقة.
- لوحة الخلايا المستردة على لوحات أجار LB تكملهاالمضادات الحيوية المقابلة (ريل seq:100 ميكروغرام / مل أمبيسلين; GppA: 30 ميكروغرام / مل كاناميسين). في اليوم التالي، تلقيح المستعمرات في مرق LB لبدء O / N الثقافات المسبقة لكلا النوعين في 37 درجة مئوية.
- بعد 18 ساعة، تلقيح 500 مل من LB المتوسطة مع 10 مل من الثقافات O / N والمضادات الحيوية المقابلة. تنمو الثقافات عن طريق اهتزاز في 160 دورة في الدقيقة في 37 درجة مئوية. عندما يصل OD600nm 0.5-0.7، حث التعبير البروتين بإضافة 0.5 M IPTG وتنمو لمدة 3 ساعة في 30 درجة مئوية مع اهتزاز في 160 دورة في الدقيقة.
- جمع الخلايا عن طريق الغزل في 6084 × ز لمدة 10 دقيقة في 4 درجة مئوية. Resuspend بيليه في 20 مل من الجليد الباردة 1x الفوسفات العازلة المالحة (PBS)، وإعادة الطرد المركزي في 1912 × ز لمدة 20 دقيقة في 4 درجة مئوية. decant supernatant، وتجميد الكريات في -20 درجة مئوية قبل الاستخدام.
- تنقية حمض النيكل والنتريلوترياستيك (Ni-NTA)
ملاحظة: من هذه النقطة فصاعدا، تأكد من أن العينات الباردة.- إضافة 40 مل من الجليد الباردة تحلل العازلة L2 (50 mM تريس pH 7.5, 150 mM NaCl, 5٪ الجلسرين, 10 mM imidazole, 10 mM β ميركابتوثانول تكملها مثبطات البروتيز (قرص خالية من EDTA; انظر جدول المواد) لإعادة إنفاق بيليه. Lyse الخلايا عن طريق سونيكيشن (60٪ السعة، 2 ق ON / 4 ق OFF لمدة 8 دقائق ON). مسح lysate عن طريق الغزل في 23426 × ز لمدة 40 دقيقة في 4 درجة مئوية ، والاستمرار مع supernatant لتنقية.
- أثناء الطرد المركزي أعلاه، وإعداد راتنج ني-NTA.
- نقل 500 ميكرولتر من راتنج ني-NTA المتجانس إلى عمود كروماتوغرافيا البولي بروبلين الدائمة، والسماح لها تسوية لمدة 15 دقيقة واستنزاف حل التخزين من خلال. غسل الراتنج مع 15 مل من المياه فائقة الشراء مرتين، ومن ثم غسل العمود مع 15 مل من تحلل العازلة L2.
- تحميل supernatant مسح الخلية lysate من الخطوة 1 على العمود، والسماح لها بالتدفق من خلال. غسل العمود مع 30 مل من العازلة الغسيل (50 mM تريس pH 7.5, 150 mM NaCl, 5٪ الجلسرين, 20 mM imidazole).
- Elute البروتينات مع 400 ميكرولتر من العازلة elution (50 mM تريس درجة الحموضة 7.5، 150 م م NaCl، 5٪ الجلسرين، 500 mM imidazole) ثلاث مرات. ثم كرر elution مع آخر 300 μL من المخزن المؤقت elution. الجمع بين البروتينات الملوتة إلى حجم نهائي من 700 ميكرولتر.
- الترشيح هلام
- إعداد هلام الترشيح العازلة (50 mM تريس, pH 7.5; 200 mM NaCl; 5٪ الجلسرين). اغسل عمود استبعاد الحجم بحجم عمود واحد (25 مل) من المخزن المؤقت لترشيح الجل.
- قم بتحميل العينة أعلاه 700 ميكرولتر باستخدام حلقة 500 ميكرولتر، وتشغيلها بمعدل 0.5 مل/دقيقة، وجمع 2-3 كسور، كل منها بحجم 0.5 مل، تحتوي على البروتينات المعنية.
- الجمع بين وتركيز الكسور التي تحتوي على كل من البروتينات باستخدام عمود تدور، وقياس تركيز البروتين باستخدام المقايسة برادفورد.
3. توليف 32P-المسمى pppGpp وppGpp
- تجميع رد فعل على نطاق صغير Relseq في أنبوب غطاء المسمار (انظر الجدول 1).
ملاحظة: العمل مع الكواشف المشعة فقط في مكان مرخص ومع معدات الحماية الشخصية.
| حجم الصوت (ميكرولتر) | ||
| نطاق صغير | نطاق واسع | |
| المياه فائقة البور | ||
| 10x ريلseq العازلة * | 2 | 50 |
| ATP (نهائي 8 mM) | ||
| ريلسيك (4 μم النهائي) | ||
| 32 P-α-جوانوسين ثلاثي الفوسفات (GTP) (النهائي 120 nM) (تحذير) | 0.2 | 5 |
| مجموع | 20 | 500 |
الجدول 1: تجميع المعلومات لتفاعلات توليف صغيرة وكبيرة النطاق من 32P-المسمى pppGpp. * 10x Relseq العازلة يحتوي على 250 mM تريس-HCl، درجة الحموضة 8.6؛ 1M NaCl; 80 م م2. اختصار: pppGpp = جوانوسين بنتافوسفات.
- احتضان أنبوب في 37 درجة مئوية في thermomixer لمدة 1 ساعة، ثم في 95 درجة مئوية لمدة 5 دقائق، ووضعها على الجليد لمدة 5 دقائق. تدور أسفل البروتين عجلت في 15700 س ز لمدة 5 دقائق، ونقل supernatant (توليفها 32P-pppGpp) إلى أنبوب غطاء المسمار الجديد.
- لتجميع 32P-ppGpp من 32P-pppGpp، نقل نصف المنتج 32ف pppGpp إلى أنبوب غطاء المسمار الجديد، وإضافة 1 ميكرومتر GppA. احتضان الأنبوب في 37 درجة مئوية لمدة 10 دقائق، في 95 درجة مئوية لمدة 5 دقائق، ومن ثم وضعها على الجليد لمدة 5 دقائق.
- تدور أسفل البروتين عجلت في 15700 × ز لمدة 5 دقائق، ونقل supernatant (توليفها 32P-ppGpp) إلى أنبوب غطاء المسمار الجديد.
- تحليل 32P-pppGpp و 32P-ppGpp عن طريق تشغيل 1 ميكرولتر من العينات على طبقة رقيقة الكروماتوغرافيا (TLC) لوحة (البولي ايثيلينين تعديل السليلوز TLC لوحات) باستخدام 1.5 M KH2PO4, pH 3.4, كمرحلة متنقلة.
ملاحظة: استخدامα-32P-المسمى جوانوسين 5'-ثلاثي الفوسفات(32ف-α-GTP) كعنصر تحكم. - جفف لوحة TLC تماما، ضعها بين مجلد بلاستيكي شفاف، وعرضها لشاشة تخزين فوسفور لمدة 5 دقائق. تصور وقياس الإشارات باستخدام الفوسفوريماجر.
ملاحظة: عندما نسب 32P-pppGpp و 32P-ppGpp أعلى من 85٪, رد فعل واسع النطاق (500 ميكرولتر, كافية لفحص 20 لوحات 96-جيدا) يمكن تجميعها وتوليفها باستخدام الجدول 1.
4. فحص DRaCALA من البروتينات المستهدفة من (ع) ppGpp
- ذوبان ونقل 20 ميكرولتر من lysates الخلية بأكملها إلى لوحة 96 جيدا V-أسفل microtiter. إضافة 2.5 U/ بئر من الإندونوكليا من Serratia marcescens، واحتضان في 37 درجة مئوية لمدة 15 دقيقة للحد من لزوجة ليسات. ضع الlysates على الجليد لمدة 20 دقيقة.
- امزج 32P-pppGpp و 32P-ppGpp بنسبة 1:1، وأضف 1x تحلل العازل L1 لجعل التركيز النهائي ل (p)ppGpp يساوي 4 nM.
ملاحظة: نظرا للتشابه الكيميائي بين pppGpp وppGpp، فإن مزيجا من المادتين الكيميائيتين سيبسط عملية الفحص. - استخدام ماصة متعددة القنوات ونصائح ماصة المصفاة لإضافة وخلط 10 ميكرولتر من خليط (ع) ppGpp مع lysate الخلية. احتضان في درجة حرارة الغرفة (RT) لمدة 5 دقائق.
- اغسل أداة 96 × دبوس عن طريق وضع محلول 0.01٪ من المنظفات غير الأيونية لمدة 30 s ، وجفف على ورق الأنسجة لمدة 30 s. كرر غسل أداة دبوس 3x.
- ضع أداة الدبوس في لوحات العينة أعلاه 96-well، وانتظر لمدة 30 s. رفع أداة دبوس على التوالي، ووضعه مباشرة إلى أسفل على غشاء النيتروسليلوز لمدة 30 s.
ملاحظة: إذا كان هناك بقعة مفقودة، بقعة 2 ميكرولتر من العينات المقابلة مع ماصة ونصائح تصفيتها. من المستحسن أن تجعل بقعة مكررة من نفس العينة كما هو مبين أدناه. - جفف الغشاء لمدة 5 دقائق في RT. ضع الغشاء بين مجلد بلاستيكي شفاف، وعرضه لشاشة الفوسفور التخزين لمدة 5 دقائق. تصور باستخدام الفوسفوريماجر.
5. تحديد وتحديد البروتينات المستهدفة المحتملة
- استخدم برنامج التحليل المرتبط بفوسفوريماجر لفتح ملف .gel للوحات المرئية. استخدم دالة تحليل Array لتعريف البقع 96 بإعداد شبكة من 12 عمودا × 8 صفوف.
- حدد دوائر كبيرة لتحديد الحافة الخارجية للبقع بأكملها (انظر الشكل 1B). تصدير Volumn + الخلفية والمنطقة من الدوائر الكبيرة المحددة ، وحفظ في جدول البيانات.
ملاحظة: إذا لزم الأمر، قم بتغيير موضع كل دائرة على حدة لتتداخل تماما مع البقع، ثم قم بتغيير حجم كل دائرة على حدة لجعلها أكبر قليلا من البقعة الفعلية. - الحجم أسفل الدوائر المحددة للحد من النقاط الداخلية الصغيرة. تصدير Volumn + الخلفية، ومساحة الدوائر الصغيرة المعرفة ، وحفظ في جدول بيانات.
- حساب الكسور الربط في جدول البيانات باستخدام المعادلة في الشكل 1B، ورسم البيانات. تحديد البروتينات الملزمة المحتملة في الآبار التي تظهر كسورا عالية الربط بالمقارنة مع غالبية الآبار الأخرى.

الشكل 2:سير العمل العام لعملية فحص DRaCALA. يتم حث إنتاج البروتين من مجموعة إسكشيا القولونية، ويتم التخلص من الخلايا. وفي الوقت نفسه، يتم تنقية البروتينات المؤتلفة Relseq-His و GppA-His واستخدامها لتجميع 32pppGpp وppGpp المسمى P من 32P-α-GTP. ثم يتم خلط جزيئات ppGpp المسماة إشعاعيا (p) مع الليسات ، ويتم استخدام أداة دبوس 96 لاكتشاف الخلائط على غشاء النيتروسليلوز للتعرض اللاحق لشاشة تخزين الفوسفور والتصوير وتحديد كمي للإشارات المشعة. المختصرات: DRaCALA = العمل الشعيرات الشعرية الشعاعية التفاضلية من المقايسة ليغاند; (ع) ppGpp = جوانوسين بنتا - وتيترافوسفات؛ RT = درجة حرارة الغرفة. IPTG = ايزوبروبيل β-د-1-ثيوغالاتوبيرانوزيد; GTP = جوانوسين 5'-ثلاثي الفوسفات; SDS-PAGE = دودسيلسلفيت الصوديوم-البولي أكريلاميد هلام الكهربائي; TLC = طبقة رقيقة الكروماتوغرافيا. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Access restricted. Please log in or start a trial to view this content.
النتائج
باتباع البروتوكول المذكور أعلاه سوف تسفر عادة نوعين من النتائج (الشكل 3).
ويبين الشكل 3 ألف لوحة ذات إشارات ربط خلفية منخفضة نسبيا (كسور ملزمة < 0.025) من غالبية الآبار. إشارة الربط الإيجابية من بئر H3 يعطي جزء ملزم من ~ 0.35 وهذا هو أعلى بكثير من تلك ا?...
Access restricted. Please log in or start a trial to view this content.
Discussion
واحدة من الخطوات الحاسمة في أداء فحص DRaCALA هو الحصول على lysates خلية كاملة جيدة. أولا، ينبغي إنتاج البروتينات المختبرة بكميات كبيرة وفي أشكال قابلة للذوبان. ثانيا، يجب أن يكون تحلل الخلايا كاملا، ويجب أن تكون لزوجة التحلل ضئيلة. إدراج lysozyme واستخدام ثلاث دورات من تجميد ذوبان غالبا ما تكون كافي?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ولا يوجد لدى صاحبي البلاغ تضارب في المصالح يكشفان عنه.
Acknowledgements
ويدعم هذا العمل منحة مشروع NNF (NNF19OC0058331) إلى YEZ، وبرنامج أفق الاتحاد الأوروبي للبحث والابتكار لعام 2020 بموجب اتفاقية منحة ماري سكلودوسكا كوري (Nº 801199) إلى MLS.

Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
References
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404(2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232(2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188(2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388(2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved