É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação das Proteínas De Ligação de Pequenos Ligantes com a Ação Capilar Radial Diferencial do Ensaio de Ligand (DRaCALA)
Neste Artigo
Resumo
A Ação Capilar De Ligania Diferencial do Ensaio de Ligamento (DRaCALA) pode ser usada para identificar pequenas proteínas de ligação de ligantes de um organismo usando uma biblioteca ORFeome.
Resumo
A última década teve um tremendo progresso na compreensão de pequenas moléculas de sinalização na fisiologia bacteriana. Em particular, as proteínas-alvo de vários mensageiros secundários derivados de nucleotídeos (NSMs) têm sido sistematicamente identificadas e estudadas em organismos modelo. Essas conquistas se devem principalmente ao desenvolvimento de várias novas técnicas, incluindo a técnica do composto de captura e a ação capilar radial diferencial do ensaio ligante (DRaCALA), que foram utilizadas para identificar sistematicamente proteínas-alvo dessas pequenas moléculas. Este artigo descreve o uso dos NSMs, guanosine penta e tetrafosfatos (p)ppGpp, como exemplo e demonstração de vídeo da técnica DRaCALA. Utilizando DRaCALA, 9 das 20 proteínas-alvo conhecidas e 12 novas proteínas-alvo de (p)ppGpp foram identificadas no organismo modelo, Escherichia coli K-12, demonstrando o poder deste ensaio. Em princípio, o DRaCALA poderia ser usado para estudar pequenos ligantes que podem ser rotulados por isótopos radioativos ou corantes fluorescentes. Os passos críticos, prós e contras do DRaCALA são discutidos aqui para posterior aplicação desta técnica.
Introdução
As bactérias usam várias pequenas moléculas de sinalização para se adaptar em ambientes em constante mudança1,2. Por exemplo, os autoindutores, N-acylhomoserine lactones e seus oligopeptídeos modificados, mediam a comunicação intercelular entre bactérias para coordenar o comportamento populacional, um fenômeno conhecido como quórum sensoriando2. Outro grupo de pequenas moléculas de sinalização são os NSMs, incluindo o monofosfato de adenosina cíclica amplamente estudado (cAMP), di-AMP cíclico, monofosfato de di-guanosina cíclico (di-GMP cíclico), e penta-e tetra fosfatos (p)ppGpp1. As bactérias produzem esses NSMs como resposta a uma variedade de diferentes condições de estresse. Uma vez produzidas, essas moléculas se ligam às suas proteínas-alvo e regulam várias vias fisiológicas e metabólicas diferentes para lidar com as tensões encontradas e aumentar a sobrevivência bacteriana. Portanto, a identificação das proteínas alvo é um pré-requisito inevitável para decifrar as funções moleculares dessas pequenas moléculas.
A última década testemunhou um boom de conhecimento dessas pequenas moléculas de sinalização, principalmente devido a várias inovações técnicas que revelaram as proteínas-alvo dessas pequenas moléculas. Estes incluem a técnica do composto de captura3,4,5, e a ação capilar radial diferencial do ensaio de ligantes (DRaCALA)6 a ser discutido neste artigo.
Inventado por Vincent Lee e colegas de trabalho em 20116,o DRaCALA implanta a capacidade de uma membrana nitrocelulose para sequestrar diferencialmente ligantes livres e ligados à proteína. Moléculas como proteínas não podem se difundir em uma membrana de nitrocelulose, enquanto pequenos ligantes, como os NSMs, são capazes de. Ao misturar o NSM (por exemplo,ppGpp) com a proteína a ser testada e localizá-las na membrana, dois cenários podem ser esperados (Figura 1): Se (p)ppGpp se ligar à proteína, o radiolabeled (p)ppGpp será mantido no centro do local pela proteína e não se difundirá para fora, dando um pequeno ponto intenso (i.e., forte sinal radioativo) sob um fosforrimager. No entanto, se (p)ppGpp não se ligar à proteína, ele irá difundir livremente para fora para produzir um grande ponto com sinal radioativo de fundo uniforme.
Além disso, o DRaCALA pode detectar a interação entre uma pequena molécula e uma proteína não purificada em uma célula inteira lysate se a proteína estiver presente em uma quantidade suficiente. Essa simplicidade permite o uso de DRaCALA na identificação rápida de alvos proteicos usando uma biblioteca de expressão ORFeome. De fato, as proteínas-alvo de cAMP7,cíclico di-AMP8,di-GMPciclic 9,10e (p)ppGpp11,12,13 foram sistematicamente identificadas pelo uso de DRaCALA. Este artigo de vídeo usa (p)ppGpp como um exemplo para demonstrar e descrever as etapas e considerações críticas na realização de uma triagem DRaCALA bem sucedida. Note-se que uma descrição mais completa do DRaCALA14 é altamente recomendada para ler em combinação com este artigo antes de realizar o DRaCALA.
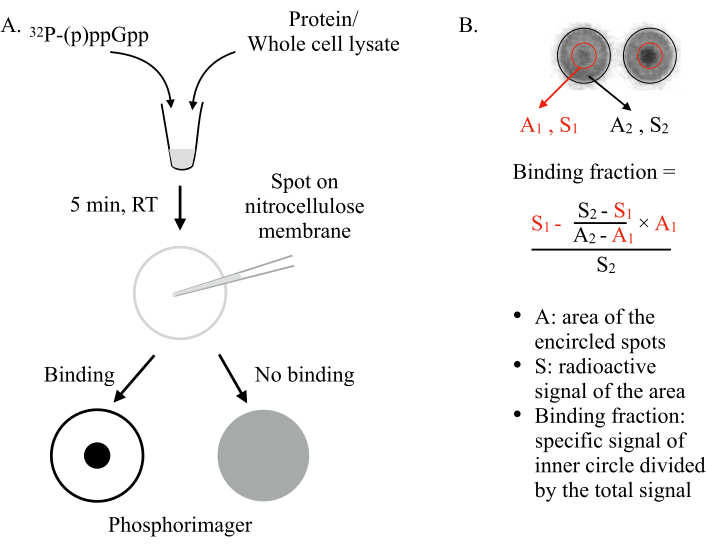
Figura 1: O princípio da DRaCALA. (A) Esquema do ensaio DRaCALA. Consulte o texto para obter detalhes. (B) Quantificação e cálculo da fração vinculante. Consulte o texto para obter detalhes. Resumidamente, as manchas DRaCALA serão analisadas desenhando dois círculos que circunscrevem todo o local e o ponto escuro interno (ou seja,o retido (p)ppGpp devido à ligação da proteína testada). O sinal de ligação específico é o sinal radioativo do círculo interno (S1) após subtrair o sinal de fundo não específico (calculado por A1 × ((S2-S1)/(A2-A1)). A fração de ligação é o sinal de ligação específico dividido pelo sinal radioativo total (S2). Abreviaturas: DRaCALA = Ação Capilar Radial Diferencial do Ensaio de Ligante; p)ppGpp = penta e tetrafosfatos de guanosina; RT = temperatura ambiente. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação de lises celulares inteiras
- Inocular a coleção E. coli K-12 ASKA ORFeome estica15 em 1,5 mL Caldo de Lysogeny (LB) contendo 25 μg/mL chloramphenicol em placas de poços profundos de 96. Cresça durante a noite (O/N) por 18 h a 30 °C com agitação a 160 rpm. No dia seguinte, adicione isopropílico β-d-1-thiogalactopyranoside (IPTG) (final de 0,5 mM) às culturas O/N para induzir expressão proteica a 30 °C por 6 h.
- Células de pelota a 500 x g por 10 min. Congele as pelotas a -80 °C até usar. Para lise as células, adicione 150 μL de tampão de lise L1 (40 mM Tris pH 7.5, 100 mM NaCl, 10 mM MgCl2, suplementado com fluoreto de fenilmetilsulfonyl de 2 mM (PMSF), 40 μg/mL DNase 1 e 0,5 mg/mL de lysozime) para ressususpensar a pelota.
- Congele as células a -80 °C por 30 min e descongele a 37 °C por 20 min. Repita este ciclo três vezes para lise as células. Armazene as lises a -80 °C antes de usar.
2. Purificação de Relseq e GppA
NOTA: As proteínas recombinantes Relseq de Streptococcus equisimilis e GppA de E. coli K-12 são usadas para sintetizar o pppGpp radiolabeled e ppGpp, respectivamente.
- Crescer e coletar células superexpressando cada proteína.
- Cresça a cepa E. coli BL21 DE3 até a fase exponencial (densidade óptica (OD) ~0,3-0,4) em caldo LB, e gire 1 mL de cultura a 6000 x g por 5 min. Decante o supernascimento, e resuspenja as células com 100 μL de caldo TSB gelado (caldo LB suplementado com 0,1 g/mL PEG3350, 0,05 mL/meL dimetilsulfoxida, 20 mM MgCl2).
- Misture os plasmídeos com o relseq e gppAmarcados por histidina, cada 100 ng, com as suspensões celulares acima em TSB, e incubar no gelo por 30 minutos. Choque térmico das células a 42 °C para 40 s. Coloque a mistura no gelo por 2 min, e adicione 1 mL de caldo LB à temperatura ambiente para permitir que as células se recuperem por 1h a 37 °C com agitação a 160 rpm.
- Placa as células recuperadas em placas de ágar LB suplementadas com os antibióticos correspondentes (Relseq:100 μg/mL ampicillin; GppA: 30 μg/mL kanamycin). No dia seguinte, inocular as colônias em caldo LB para iniciar as pré-culturas O/N de ambas as cepas a 37 °C.
- Após 18 h, inocular 500 mL de meio LB com 10 mL das culturas O/N e os antibióticos correspondentes. Cresça as culturas agitando a 160 rpm a 37 °C. Quando o OD600nm atingir 0,5-0,7, induzir expressão proteica adicionando 0,5 mM IPTG e crescendo por 3h a 30 °C com agitação a 160 rpm.
- Colete as células girando a 6084 x g por 10 min a 4 °C. Resuspense a pelota em 20 mL de salina tamponada de fosfato de 1x (PBS) e re-centrífuga a 1912 x g por 20 min a 4 °C. Decante o supernatante e congele as pelotas a -20 °C antes de usar.
- Purificação de afinidade do ácido nitrilotriactico de níquel (Ni-NTA)
NOTA: A partir deste ponto, certifique-se de que as amostras estão frias.- Adicione 40 mL de tampão de lise gelada L2 (50 mM Tris pH 7.5, 150 mM NaCl, 5% glicerol, 10 mM imidazol, 10 mM β-mercaptoetanol complementado com inibidores de protease (comprimido livre de EDTA; ver a Tabela de Materiais) para resuspensar a pelota. Lise as células através de sônica (60% de amplitude, 2 s ON/ 4 s OFF por 8 min ON). Limpe o lise girando a 23.426 x g por 40 min a 4 °C, e continue com o sobrenatante para a purificação.
- Durante a centrifugação acima, prepare a resina Ni-NTA.
- Transfira 500 μL de resina Ni-NTA homogeneizada para uma coluna de cromatografia de polipropileno em pé, e deixe-a se contentar com 15 minutos e a solução de armazenamento escorra. Lave a resina com 15 mL de água ultrauso duas vezes e depois lave a coluna com 15 mL do tampão de lise L2.
- Carregue o supernanato limpo de lise celular do passo 1 para a coluna, e deixe-o fluir. Lave a coluna com 30 mL de tampão de lavagem (50 mM Tris pH 7,5, 150 mM NaCl, 5% glicerol, 20 mM imidazole).
- Elute as proteínas com 400 μL do tampão de eluição (50 mM Tris pH 7.5, 150 mM NaCl, 5% glicerol, 500 mM imidazol) três vezes. Em seguida, repita a elução com outros 300 μL do buffer de eluição. Combine as proteínas elucidas a um volume final de 700 μL.
- Filtragem de gel
- Prepare o tampão de filtragem de gel (50 mM Tris, pH 7,5; 200 mM NaCl; 5% glicerol). Lave a coluna de exclusão de tamanho com um volume de coluna (25 mL) do tampão de filtragem de gel.
- Carregue a amostra acima de 700 μL usando um loop de 500 μL, execute a 0,5 mL/min e colete 2-3 frações, cada uma de 0,5 mL de volume, contendo as respectivas proteínas.
- Combine e concentre as frações que contêm cada uma das proteínas usando uma coluna de spin, e meça a concentração de proteínas usando o ensaio de Bradford.
3. Síntese de PPPGpp e ppGpp com etiqueta em P de 32P
- Monte uma reação relseq em pequena escala em um tubo de tampa de parafuso (ver Tabela 1).
NOTA: Trabalhe com reagentes radioativos apenas em local licenciado e com equipamento de proteção individual.
| Volume (μL) | ||
| Pequena escala | Grande escala | |
| Água ultrapura | ||
| 10x Relseq buffer* | 2 | 50 |
| ATP (final de 8 mM) | ||
| Relseq (4 μM final) | ||
| 32 Triptofato P-α-Guanosine (GTP) (final 120 nM) (ATENÇÃO) | 0.2 | 5 |
| total | 20 | 500 |
Tabela 1: A montagem de informações para as reações de síntese de pequena e grande escala de pppGpp. *10x Relseq buffer contém 250 mM Tris-HCl, pH 8.6; 1M NaCl; 80 mM MgCl2. Abreviação: pppGpp = pentafosfato de guanosina.
- Incubar o tubo a 37 °C em um termomixer por 1h, depois a 95 °C por 5 min, e coloque no gelo por 5 min. Gire a proteína precipitada a 15.700 x g por 5 min, e transfira o supernante (sintetizado 32P-pppGpp) para um novo tubo de tampa de parafuso.
- Para sintetizar 32P-ppGpp de 32P-pppGpp, transfira metade do produto 32p-pppGpp para um novo tubo de tampa de parafuso e adicione 1 μM GppA. Incubar o tubo a 37 °C por 10 min, a 95 °C por 5 min, e depois coloque no gelo por 5 minutos.
- Gire a proteína precipitada a 15.700 x g por 5 min, e transfira o supernante (sintetizado 32P-ppGpp) para um novo tubo de tampa de parafuso.
- Analise a placa de 32P-pppGpp e 32P-ppGpp executando 1 μL das amostras em uma placa de cromatografia de camada fina (TLC) (placas TLC modificadas por polietileneimina) utilizando as placas TLC 1,5 M KH2PO4, pH 3.4, como fase móvel.
NOTA: Use o α-32P-rotulado guanosina 5'-triphosphate (32P-α-GTP) como controle. - Seque completamente a placa TLC, coloque-a entre uma pasta de plástico transparente e exponha-a a uma tela de fósforo de armazenamento por 5 minutos. Visualize e quantifique os sinais usando um fósforo.
NOTA: Quando as proporções de 32P-pppGpp e 32P-ppGpp forem superiores a 85%, uma reação em larga escala (500 μL, suficiente para triagem de 20 placas de 96 poços) pode ser montada e sintetizada usando a Tabela 1.
4. Triagem DRaCALA das proteínas-alvo de (p)ppGpp
- Descongele e transfira 20 μL de lises celulares inteiras para uma placa de microtiter fundo V de 96 poços. Adicione 2,5 U/poço de endonuclease de Serratia marcescens, e incubar a 37 °C por 15 min para reduzir a viscosidade lysate. Coloque os lises no gelo por 20 minutos.
- Misture o tampão de 32P-pppGpp e 32P-ppGpp em uma proporção de 1:1, e adicione 1x tampão de lise L1 para tornar a concentração final de (p)ppGpp igual a 4 nM.
NOTA: Dada a similaridade química entre o pppGpp e o ppGpp, uma mistura de ambos os produtos químicos simplificará o processo de triagem. - Use uma pipeta multicanal e pontas de pipeta filtradas para adicionar e misturar 10 μL da mistura (p)ppGpp com o lise celular. Incubar em temperatura ambiente (RT) por 5 min.
- Lave a ferramenta de 96 x pino colocando em 0,01% a solução de detergente não iônico para 30 s e seque em um papel de tecido para 30 s. Repita a lavagem da ferramenta do pino 3x.
- Coloque a ferramenta de pinos nas placas de amostra acima de 96 poços e espere por 30 s. Levante a ferramenta do pino para cima e coloque-a em linha reta para baixo em uma membrana de nitrocelulose por 30 s.
NOTA: Se faltar uma mancha, local 2 μL das amostras correspondentes com uma pipeta e pontas filtradas. É aconselhável fazer um ponto duplicado da mesma amostra que indicado abaixo. - Seque a membrana por 5 minutos no RT. Coloque a membrana entre uma pasta de plástico transparente e exponha-a a uma tela de fósforo de armazenamento por 5 minutos. Visualize usando um fósforo.
5. Quantificação e identificação de potenciais proteínas-alvo
- Use o software de análise associado ao fosforrimager para abrir o arquivo .gel das placas visualizadas. Use a função de análise Array para definir os 96 pontos configurando uma grade de 12 colunas x 8 linhas.
- Defina círculos grandes para circunscrever a borda externa de todos os pontos (ver Figura 1B). Exporte o Volumn+Background e Área dos grandes círculos definidos e economize em uma planilha.
NOTA: Se necessário, reposicione cada círculo individual para se sobrepor perfeitamente com as manchas e redimensione cada círculo individual para torná-lo ligeiramente maior do que o local real. - Dimensione os círculos definidos para circunscrever os pequenos pontos internos. Exporte o Volumn+Backgrounde Área dos pequenos círculos definidos e economize em uma planilha.
- Calcule as frações de vinculação na planilha usando a equação na Figura 1Be plote os dados. Identifique as proteínas de ligação potenciais nos poços que apresentam altas frações de ligação em comparação com a maioria dos outros poços.

Figura 2: Fluxo de trabalho global do processo de triagem DRaCALA. A produção de proteínas de uma coleção Escherichia coli ASKA é induzida, e as células são lístidas. Enquanto isso, as proteínas recombinantes Relseq-His e GppA-His são purificadas e usadas para sintetizar pppGpp e ppGpp de 32P-α-GTP. As moléculas de ppGpp radioativamente rotuladas (p)ppGpp são então misturadas com os lises, e uma ferramenta de 96 pinos é usada para detectar as misturas em uma membrana nitrocelulose para posterior exposição a uma tela de armazenamento de fósforo, imagem e quantificação dos sinais radioativos. Abreviaturas: DRaCALA = Ação Capilar Radial Diferencial do Ensaio de Ligante; p)ppGpp = penta e tetrafosfatos de guanosina; RT = temperatura ambiente; IPTG = isopropílico β-d-1-thiogalactopyranoside; GTP = guanosina 5'-triphosfato; SDS-PAGE = eletroforese de gel de dodecylsulfato-poliacrilamida de sódio ; TLC = cromatografia de camada fina. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Seguindo o protocolo acima descrito normalmente produzirá dois tipos de resultados (Figura 3).
A Figura 3A mostra uma placa com sinais de ligação de fundo relativamente baixos (frações de ligação < 0,025) da maioria dos poços. O sinal de ligação positivo do poço H3 dá uma fração de ligação de ~0,35 que é muito maior do que a observada para os outros poços. Mesmo sem quantificação, bem H3 é notável, sugerindo que ...
Access restricted. Please log in or start a trial to view this content.
Discussão
Uma das etapas críticas na realização da triagem DRaCALA é obter bons lises celulares inteiras. Em primeiro lugar, as proteínas testadas devem ser produzidas em grandes quantidades e em formas solúveis. Em segundo lugar, a lise das células deve estar completa, e a viscosidade do lysate deve ser mínima. A inclusão da lise e o uso de três ciclos de congelamento são muitas vezes suficientes para lise completamente as células. No entanto, o DNA cromossômico liberado torna o viscoso lísato e gera alto sinal de l...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflito de interesses para divulgar.
Agradecimentos
O trabalho é apoiado por um NNF Project Grant (NNF19OC0058331) para a YEZ, e o programa de pesquisa e inovação Horizon 2020 da União Europeia sob o acordo de subvenção Marie Skłodowska-Curie (Nº 801199) à MLS.

Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
Referências
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404(2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232(2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188(2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388(2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados