JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
리간드 분석의 차동 방사형 모세관 작용으로 작은 리간드의 결합 단백질 식별 (DRaCALA)
요약
리간드 분석(DRaCALA)의 차동 적개 모세관 작용은 ORFeome 라이브러리를 사용하여 유기체의 작은 리간 결합 단백질을 식별하는 데 사용될 수 있다.
초록
지난 10 년 세균성 생리학에 있는 작은 신호 분자의 이해에 있는 엄청난 진전을 보았습니다. 특히, 여러 뉴클레오티드 유래 이차 메신저(NSM)의 표적 단백질은 모델 유기체에서 체계적으로 확인되고 연구되었다. 이러한 성과는 주로 이러한 소분자의 표적 단백질을 체계적으로 식별하는 데 사용되었던 리간드 분석시(DRaCALA)의 포획 화합물 기술과 차동 적대모세 혈관 작용을 포함한 몇 가지 새로운 기술의 개발 때문입니다. 이 논문은 DRaCALA 기술의 예 및 비디오 데모로 NSM, 과노신 펜타 및 테트라포산염(p)ppGpp의 사용을 설명합니다. DRaCALA를 사용하여, 20명 중 9개 및 12개의 새로운 표적 단백질(p)ppGpp가 모델 유기체, 대장균 K-12에서 확인되었으며, 이 분석의 힘을 입증하였다. 원칙적으로, DRaCALA는 방사성 동위원소 또는 형광 염료에 의해 표시될 수 있는 작은 리간드를 공부하기 위하여 이용될 수 있었습니다. DRaCALA의 중요한 단계, 장단점 및 단점은 이 기술을 추가로 적용하기 위해 여기에서 논의됩니다.
서문
박테리아는 끊임없이 변화하는 환경에 적응하기 위해 여러 개의 작은 신호 분자를 사용1,2. 예를 들어, 자가유도기, N-아실모케린 락톤 및 수정된 올리고펩티드, 박테리아 간의 세포간 통신을 중재하여 인구 거동, 쿼럼 센싱2로알려진 현상을 조정한다. 작은 신호 분자의 또 다른 그룹은 널리 연구 된 순환 아데노신 모노 포스 페이트 (cAMP), 순환 디-AMP, 순환 디 구아노신 모노 인산염 (순환 디-GMP), 및 구아노신 펜타 및 테트라 인산염(ppp)을 포함하여 NSM입니다. 박테리아는 다양한 스트레스 조건에 대한 응답으로 이러한 NSM을 생산합니다. 일단 생성되면, 이 분자는 그들의 표적 단백질에 결합하고 만나는 응고에 대처하고 세균성 생존을 강화하기 위하여 몇몇 다른 생리적이고 신진 대사 통로를 조절합니다. 따라서, 표적 단백질의 식별은 이 작은 분자의 분자 기능을 해독하기 위한 피할 수 없는 전제 조건이다.
지난 10 년은 이 작은 분자의 표적 단백질을 공개한 몇몇 기술 혁신 때문에 주로 이 작은 신호 분자의 지식의 붐을 목격했습니다. 이들 로는 이 논문에서 논의될 리간 분석(DRaCALA)6의 포획 화합물 기술3,4,5,및 차동 방사형 모세관 작용을 포함한다.
2011년6년빈센트 리와 동료들이 발명한 DRaCALA는 니트로셀룰로오스 막의 능력을 차별화적으로 분리하여 무료 및 단백질 바운드 리간드에 배치합니다. 단백질과 같은 분자는 니트로셀룰로오스 막에서 확산될 수 없으며, NSM과 같은 작은 리간드는 확산될 수 있습니다. NSM(예를들어,ppGpp)을 시험할 단백질과 혼합하여 멤브레인에서 이를 발견하면 2가지 시나리오를 기대할 수있습니다(그림 1):(p)ppGpp가 단백질에 결합하면, 방사성 라벨(p)ppGpp는 단백질에 의해 그 자리의 중앙에 유지되고, 강렬한 작은 점(i.i.) 강한 방사성 신호) 인산화증하에서. 그러나(p)ppGpp가 단백질에 결합하지 않으면, 균일한 배경 방사성 신호로 큰 지점을 생성하기 위해 자유롭게 바깥쪽으로 확산된다.
더욱이, DRaCALA는 단백질이 충분한 양으로 존재하는 경우에 전체 세포 용액에 있는 작은 분자와 불정제 단백질 사이 상호 작용을 검출할 수 있습니다. 이러한 단순성으로 ORFeome 발현 라이브러리를 사용하여 단백질 표적을 빠르게 식별할 수 있는 DRaCALA를 사용할 수 있습니다. 실제로, cAMP7,순환 디-암8,순환 디-GMP9,10,및 (p)ppGpp11,12,13의 표적 단백질은 DRaCALA를 사용하여 체계적으로 확인되었다. 이 비디오 문서에서는 (p) ppGpp를 예로 사용하여 성공적인 DRaCALA 스크리닝을 수행하는 데 중요한 단계와 고려 사항을 설명하고 설명합니다. 참고, DRaCALA의 더 철저한 설명14 는 DRaCALA를 수행 하기 전에이 문서와 함께 읽는 것이 좋습니다.
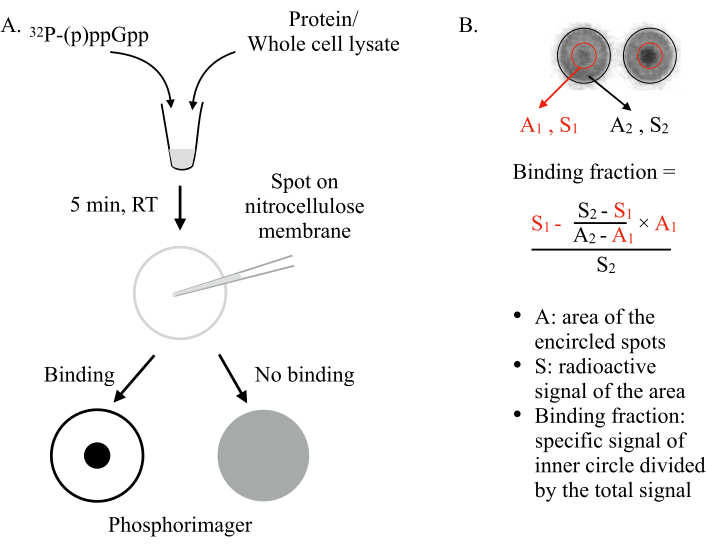
그림 1: DRaCALA의 원리. (A)DRaCALA 분석의 회로도. 자세한 내용은 텍스트를 참조하십시오. (B)바인딩 분획의 정량화 및 계산. 자세한 내용은 텍스트를 참조하십시오. 간단히, DRaCALA 반점은 전체 반점과 내부 다크닷(즉,테스트된 단백질의 결합으로 인해 유지된(p)ppGpp를 둘레에 두 개의 원을 그려 분석될 것이다. 특이적 결합 신호는 비특이적 배경 신호(A1 ×(S 2-S 1)/(A2-A1)에의해계산된)를 빼낸 후 내부 원(S1)의 방사성 신호이다. 결합 분획은 총 방사성 신호(S2)로 나눈 특정 결합 신호이다. 약어: DRaCALA = 리간드 분석의 차동 방사형 모세관 작용; (p)ppGpp = 구아노신 펜타 및 테트라포산염; RT = 실온. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 전체 세포 리스의 준비
- 대장균 K-12 ASKA ORFeome 컬렉션 균주15 에 1.5 mL 리소제니 국물 (LB) 포함 25 μg/mL 클로람페니콜 96 잘 깊은 우물 플레이트. 160 rpm에서 흔들리면 30 °C에서 18 시간 동안 하룻밤 (O / N)으로 성장하십시오. 다음 날, O/N 배양에 이소프로필 β-d-1-티오갈라크토피라노사이드(최종 0.5mMMM)를 첨가하여 단백질 발현을 6시간 동안 30°C로 유도한다.
- 펠릿 셀은 500 x g에서 10 분 동안. 사용 전까지 -80°C에서 펠릿을 얼리면 됩니다. 세포를 용해하려면, 용해 버퍼 L1 (40 mM Tris pH 7.5, 100 mM NaCl, 10 mM MgCl2,2 mM 페닐메틸술포닐 불소 (PMSF), 40 μg /mL DNase 1, 및 0.5 mgmel을 추가하십시오.
- 세포를 -80°C에서 30분 동안 동결한 다음 37°C에서 20분 동안 해동합니다. 이 주기를 세 번 반복하여 셀을 lyse합니다. 사용하기 전에 -80 °C에 lysates를 저장합니다.
2. 렐세크와 GppA의 정화
참고: 대장균 K-12의 연쇄상구균 과키시닐리스및 GppA의 재조합 단백질 Relseq는 각각 방사성 표지ppp와 ppGpp를 합성하는 데 사용됩니다.
- 성장하고 각 단백질을 과발 현현하는 세포를 수집합니다.
- LB 국물에 대장균 BL21 DE3 균주를 최대 기하급수적 단계(광학 밀도(OD)~0.3-0.4)까지 늘리고, 5분 동안 6000 x g에서 1mL의 배양을 회전시한다. 상체를 데수제, 얼음-차가운 TSB 국물의 100 μL로 세포를 다시 중단 (LB 국물은 0.1 g / mL PEG3350, 0.05 mL / mL 디메틸술프리산화물, 20 mM MgCl2로보충).
- 히스티딘 태그가 달린 릴세크와 gppA를베어링 플라스미드를 각각 100 ng, TSB의 위의 세포 현탁액과 혼합하고 30 분 동안 얼음에 배양하십시오. 40s에 대해 42°C에서 세포를 열 충격. 혼합물을 2분 동안 얼음위에 놓고, 실온에 1mL의 LB 국물을 추가하여 세포가 160 rpm에서 교반하여 37°C에서 1시간 동안 회복될 수 있도록 합니다.
- 해당 항생제로 보충된 LB 한천 플레이트상에서 회수된 세포를 플레이트(Relseq:100 μg/mL 암피실린; GppA: 30 μg/mL 카나마이신). 다음 날, LB 국물에 있는 식민지를 접종하여 37°C에서 두 균주의 O/N 프리컬쳐를 시작합니다.
- 18h 후, O/N 배양 및 해당 항생제의 10mL를 가진 LB 배지의 500mL를 접종하였다. 37 °C에서 160 rpm에서 흔들어 배양을 성장시다. OD600nm이 0.5-0.7에 도달하면 0.5mM IPTG를 추가하고 160 rpm에서 흔들림으로 30°C에서 3시간 동안 성장하여 단백질 발현을 유도한다.
- 4°C에서 10분 동안 6084 x g로 회전하여 세포를 수집합니다. 4°C에서 20분 동안 20mL의 얼음-감기 1x 인산염 완충식식염(PBS)으로 펠릿을 재중단하고, 1912x g에서 재원심분리기에서 4°C에서 20분 동안 재원심분리기를 재차중단한다. 상류제를 데칭하고, 사용하기 전에 -20°C에서 펠릿을 동결한다.
- 니켈-니트리로트리아세산(Ni-NTA) 친화성 정화
참고: 이 시점부터 샘플이 차가운지 확인합니다.- 얼음 냉기 용해 버퍼 L2 (50 mM Tris pH 7.5, 150 mM NaCl, 5 % 글리세롤, 10 mM 이미다졸레, β 프로테아제 억제제 (EDTA 프리 태블릿; 재료의 표 참조)를추가하여 펠릿을 다시 일시 중단합니다. 초음파 처리를 통해 세포를 lyse (60% 진폭, 2 s ON / 4 S OFF 8 분 ON). 4°C에서 40분 동안 23,426 x g에서 회전하여 용액을 지우고, 정화를 위한 상퍼로 계속한다.
- 위의 원심 분리 중에 Ni-NTA 수지를 준비하십시오.
- 균질화된 Ni-NTA 수지 500μL을 서있는 폴리프로필렌 크로마토그래피 컬럼으로 옮기고 15분 동안 정착하고 저장 용액이 배출됩니다. 수지 15mL의 초순수를 두 번 세척한 다음, 리시스 버퍼 L2의 15mL로 컬럼을 세척한다.
- 클리어된 셀 리셀러트(supersate)를 1단계에서 열에 로드하고 통과하게 합니다. 30mL의 세탁 버퍼(50m Tris pH 7.5, 150m NaCl, 5% 글리세롤, 20m imidazole)로 컬럼을 세척합니다.
- 용출 완충제400μL(50m Tris pH 7.5, 150mM NaCl, 5% 글리세롤, 500mM imidazole)로 단백질을 3회 엘ute하였다. 그런 다음 용출 버퍼의 또 다른 300 μL으로 용출을 반복합니다. 용출된 단백질을 700 μL의 최종 부피에 결합합니다.
- 젤 여과
- 젤 여과 버퍼 (50 mM Tris, pH 7.5; 200 mM NaCl; 5 % 글리세롤)를 준비하십시오. 크기 배제 열을 젤 여과 버퍼의 하나의 열 부피(25 mL)로 세척합니다.
- 위의 700 μL 샘플을 500 μL 루프를 사용하여 로드하고 0.5 mL/min에서 실행하고 각각 0.5 mL 부피의 2-3 분획을 수집하여 각각 단백질을 함유합니다.
- 스핀 컬럼을 사용하여 각 단백질을 함유하는 분획을 결합하고 농축하고, 브래드포드 분석시를 사용하여 단백질 농도를 측정합니다.
3. 32P 라벨 pppGpp 및 ppGpp의 합성
- 나사 캡 튜브에 작은 규모의 Relseq 반응을 조립합니다(표 1참조).
참고: 허가된 장소와 개인 보호 장비에서만 방사성 시약으로 작업하십시오.
| 부피(μL) | ||
| 작은 규모 | 대규모 | |
| 초순수 | ||
| 10x Relseq 버퍼* | 2 | 50 |
| ATP (8mM 결승) | ||
| 릴세크 (4 μ M 결승) | ||
| 32 P-α-구아노신 트리호스페이트 (GTP) (최종 120 nM) (주의) | 0.2 | 5 |
| 합계 | 20 | 500 |
표 1: 32P-labeled pppGpp의 소규모 및 대규모 합성 반응에 대한 정보를 조립합니다. * 10x Relseq 버퍼에는 250mM Tris-HCl, pH 8.6이 포함되어 있습니다. 1M NaCl; 80 mM MgCl2. 약어: pppGpp = 구아노신 펜타포스페이트.
- 1 시간 동안 열믹서에서 37 °C에서 튜브를 배양 한 다음 95 °C에서 5 분 동안 얼음위에 놓고 5 분 동안 얼음위에 놓습니다. 침전된 단백질을 15,700 x g에서 5분 동안 스핀다운하고, 새로운 스크류 캡 튜브로 수퍼내트(syntheed 32P-pppGpp)를 전달합니다.
- 32P-pppGpp에서 32P-ppGpp를 합성하려면 32p-pppGpp 제품의 절반을 새로운 스크류 캡 튜브로 옮기고 1 μM GppA를 추가합니다. 튜브를 37°C에서 10분 동안, 95°C에서 5분 동안 배양한 다음 얼음 위에 5분 동안 놓습니다.
- 침전 된 단백질을 15,700 x g에서 5 분 동안 회전시키고 수퍼나티 (합성 32P-ppGpp)를 새로운 스크류 캡 튜브로 옮긴다.
- 1.5M KH 2 PO4,pH 3.4를 사용하여 얇은 층 크로마토그래피(TLC) 플레이트(polyethyleneimine-변형 셀룰로오스 TLC 플레이트)에서 샘플의 1μl을 실행하여 32P-pppGpp 및 32P-ppGpp를 분석한다.
참고:α-32P-라벨 구아노신 5'-triphosphate(32P-α-GTP)를 제어로 사용하십시오. - TLC 플레이트를 완전히 건조하고 투명 플라스틱 폴더 사이에 놓고 5 분 동안 저장 인광판 화면에 노출하십시오. 인산화증을 사용하여 신호를 시각화하고 정량화합니다.
참고: 32P-pppGpp 및 32P-ppGpp의 비율이 85% 이상이면, 대형 반응(500 μL, 20 96웰 플레이트 스크리닝에 충분함)을 표 1을사용하여 조립및 합성할 수 있다.
4. DRaCALA (p)ppGpp의 표적 단백질의 검열
- 전체 셀의 20 μL을 96웰 V-바닥 마이크로티터 플레이트로 해동 및 이송합니다. 세라티아 마르케센에서2.5 U/웰의 엔도누테스를 추가하고 37°C에서 15분 동안 배양하여 용액 점도를 줄입니다. 20분 동안 얼음 위에 용사를 놓습니다.
- 32P-pppGpp 및 32P-ppGpp를 1:1 비율로 혼합하고 1x 용해 버퍼 L1을 추가하여 (p)ppGpp의 최종 농도를 4 nM에 동일하게 만듭니다.
참고: pppGpp와 ppGpp 사이의 화학적 유사성을 감안할 때, 두 화학 물질의 혼합은 선별 과정을 단순화합니다. - 멀티채널 파이펫과 필터링된 파이펫 팁을 사용하여 (p) ppGpp 혼합물의 10 μL을 셀 리세테와 추가하여 혼합합니다. 실온(RT)에서 5분 동안 배양합니다.
- 96 x 핀 공구를 30s용 비이온 세제용 0.01% 용액으로 넣고 30s의 티슈 페이퍼에 건조시하십시오. 핀 공구3배의 세척을 반복합니다.
- 위의 96웰 샘플 플레이트에 핀 도구를 놓고 30s를 기다립니다. 핀 도구를 똑바로 들어 올리고 30s의 니트로셀룰로오스 멤브레인에 똑바로 놓습니다.
참고: 스팟이 없는 경우 파이펫과 필터링된 팁이 있는 해당 샘플의 2 μL을 발견합니다. 아래에 표시된 것과 동일한 샘플의 중복 지점을 만드는 것이 좋습니다. - RT에서 멤브레인을 5분 동안 건조시킵니다. 인산화증을 사용하여 시각화합니다.
5. 잠재적인 표적 단백질의 정량화 및 식별
- 인산화기와 관련된 분석 소프트웨어를 사용하여 시각화된 플레이트의 .gel 파일을 엽니다. 배열 분석 함수를 사용하여 12개의 열 x 8열의 그리드를 설정하여 96개의 스팟을 정의합니다.
- 큰 원을 정의하여 전체 반점의 바깥쪽 가장자리를 돌례합니다(그림 1B참조). 정의된 큰 원의 볼륨 +배경 및 영역을 내보내고 스프레드시트에 저장합니다.
참고: 필요한 경우 각 개별 원을 재배치하여 반점과 완벽하게 겹치고 각 개별 원을 조정하여 실제 스팟보다 약간 더 크게 만듭니다. - 정의된 원을 아래로 크기로 조정하여 작은 내부 점을 돌례합니다. Volumn+Background및 정의된 작은 원의 영역을 내보내고 스프레드시트에 저장합니다.
- 도 1B의방정식을 사용하여 스프레드시트의 바인딩 분수를 계산하고 데이터를 플롯합니다. 다른 우물의 대다수에 비해 높은 결합 분수를 보여주는 우물에서 잠재적인 결합 단백질을 확인합니다.

그림 2: DRaCALA 심사 프로세스의 전체 워크플로우. 에체리치아 대장균 ASKA 수집으로부터단백질 생산이 유도되고, 세포가 리세이드된다. 한편, 재조합 단백질 Relseq-His와 GppA-His는 32P-α-GTP에서 32P 라벨 pppGpp 및 ppGpp를 합성하는 데 사용됩니다. 방사성 표지(p)ppGpp 분자는 용액과 혼합된 다음, 96핀 툴이 질소셀룰로오스 막에 혼합물을 발견하여 인광저장 스크린, 이미징 및 방사성 신호의 정량화를 위해 사용된다. 약어: DRaCALA = 리간드 분석의 차동 방사형 모세관 작용; (p)ppGpp = 구아노신 펜타 및 테트라포산염; RT = 실온; IPTG = 이소프로필 β-d-1-티오갈라크토피라노사이드; GTP = 구아노신 5'-triphosphate; SDS-PAGE = 나트륨 도데킬설페이트-폴리아크라이알라미드 젤 전기포고증; TLC = 얇은 층 크로마토그래피. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
전술한 프로토콜에 따라 일반적으로 두 가지 유형의 결과를 산출한다(그림3).
도 3A는 대부분의 우물에서 상대적으로 낮은 배경 바인딩 신호(바인딩 분획 < 0.025)를 가진 플레이트를 나타낸다. 웰 H3의 양수 결합 신호는 다른 우물에서 관찰된 것보다 훨씬 높은 ~0.35의 결합 분획을 제공합니다. 정량화없이도, 잘 H3는 놀라운, 잘 H3에서 ?...
토론
DRaCALA 스크리닝을 수행하는 중요한 단계 중 하나는 좋은 전체 세포 리스를 얻는 것입니다. 첫째, 테스트된 단백질은 다량과 수용성 형태로 생산되어야 합니다. 둘째, 세포의 용해가 완료되어야하며, 용물의 점도는 최소화해야합니다. 리소지메의 포함과 동결 해동의 세 주기의 사용은 종종 완전히 세포를 lyse 충분하다. 그러나, 방출된 염색체 DNA는 용액점성 및 높은 배경 결합 신호를 생성하여
공개
저자는 공개 할 이해상충이 없습니다.
감사의 말
이 작품은 NNF 프로젝트 보조금(NNF19OC00058331)이 YEZ에 지원하고, MLS에 마리 Skłodowska-Curie 보조금 계약 (Nº 801199)에 따라 유럽 연합의 호라이즌 2020 연구 및 혁신 프로그램에 의해 지원됩니다.

자료
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
참고문헌
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404 (2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232 (2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188 (2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388 (2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유