Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification des protéines de liaison des petits ligands avec l’action capillaire radiale différentielle du test de ligand (DRaCALA)
Dans cet article
Résumé
L’action capillaire radiale différentielle du test de ligand (DRaCALA) peut être utilisée pour identifier les petites protéines de liaison au ligand d’un organisme à l’aide d’une bibliothèque ORFeome.
Résumé
La dernière décennie a vu d’énormes progrès dans la compréhension des petites molécules de signalisation dans la physiologie bactérienne. En particulier, les protéines cibles de plusieurs messagers secondaires dérivés de nucléotides (NSM) ont été systématiquement identifiées et étudiées dans des organismes modèles. Ces réalisations sont principalement dues au développement de plusieurs nouvelles techniques dont la technique du composé de capture et l’action capillaire radiale différentielle du dosage des ligands (DRaCALA), qui ont été utilisées pour identifier systématiquement les protéines cibles de ces petites molécules. Cet article décrit l’utilisation des NSM, de la guanosine penta- et des tétraphosphates (p)ppGpp, à titre d’exemple et de démonstration vidéo de la technique DRaCALA. En utilisant DRaCALA, 9 des 20 protéines cibles connues et 12 nouvelles de (p)ppGpp ont été identifiées dans l’organisme modèle, Escherichia coli K-12, démontrant la puissance de ce test. En principe, DRaCALA pourrait être utilisé pour étudier de petits ligands qui peuvent être marqués par des isotopes radioactifs ou des colorants fluorescents. Les étapes critiques, les avantages et les inconvénients de DRaCALA sont discutés ici pour une application ultérieure de cette technique.
Introduction
Les bactéries utilisent plusieurs petites molécules de signalisation pour s’adapter à des environnements en constante évolution1,2. Par exemple, les auto-inducteurs, les lactones N-acylhomosérine et leurs oligopeptides modifiés, médient la communication intercellulaire entre les bactéries pour coordonner le comportement de la population, un phénomène connu sous le nom de quorum sensing2. Un autre groupe de petites molécules de signalisation est les NSM, y compris l’adénosine monophosphate cyclique (AMPc), le di-AMP cyclique, la di-guanosine monophosphate cyclique (di-GMP cyclique) et les penta- et tétraphosphates de guanosine (p)ppGpp1. Les bactéries produisent ces NSM en réponse à une variété de conditions de stress différentes. Une fois produites, ces molécules se lient à leurs protéines cibles et régulent plusieurs voies physiologiques et métaboliques différentes pour faire face aux stress rencontrés et améliorer la survie bactérienne. Par conséquent, l’identification des protéines cibles est une condition préalable inévitable pour déchiffrer les fonctions moléculaires de ces petites molécules.
La dernière décennie a été témoin d’un boom des connaissances sur ces petites molécules de signalisation, principalement en raison de plusieurs innovations techniques qui ont dévoilé les protéines cibles de ces petites molécules. Il s’agit notamment de la technique du composé de capture3,4,5et de l’action capillaire radiale différentielle du test de ligand (DRaCALA)6 qui sera discutée dans cet article.
Inventé par Vincent Lee et ses collègues en 20116,DRaCALA déploie la capacité d’une membrane de nitrocellulose à séquestrer différemment des ligands libres et liés aux protéines. Les molécules telles que les protéines ne peuvent pas diffuser sur une membrane de nitrocellulose, tandis que de petits ligands, tels que les NSM, sont capables de le faire. En mélangeant le NSM(par exemple,ppGpp) avec la protéine à tester et en les repérant sur la membrane, on peut s’attendre à deux scénarios (Figure 1): Si (p)ppGpp se lie à la protéine, le (p)ppGpp radiomarqué sera retenu au centre de la tache par la protéine et ne diffusera pas vers l’extérieur, donnant un petit point intense(c’est-à-dire, signal radioactif fort) sous un imageur phosphoré. Cependant, si (p)ppGpp ne se lie pas à la protéine, elle diffusera librement vers l’extérieur pour produire une grande tache avec un signal radioactif de fond uniforme.
De plus, DRaCALA peut détecter l’interaction entre une petite molécule et une protéine non purifiée dans un lysate cellulaire entier si la protéine est présente en quantité suffisante. Cette simplicité permet l’utilisation de DRaCALA dans l’identification rapide de cibles protéiques à l’aide d’une bibliothèque d’expression ORFeome. En effet, les protéines cibles de l’AMPc7,du di-AMP8cyclique,du di-GMPcyclique 9,10et du (p)ppGpp11,12,13 ont été systématiquement identifiées à l’aide de DRaCALA. Cet article vidéo utilise (p)ppGpp comme exemple pour démontrer et décrire les étapes et les considérations critiques dans la réalisation d’un dépistage DRaCALA réussi. Il est à noter qu’il est fortement recommandé de lire une description plus complète de DRaCALA14 en combinaison avec cet article avant d’effectuer DRaCALA.
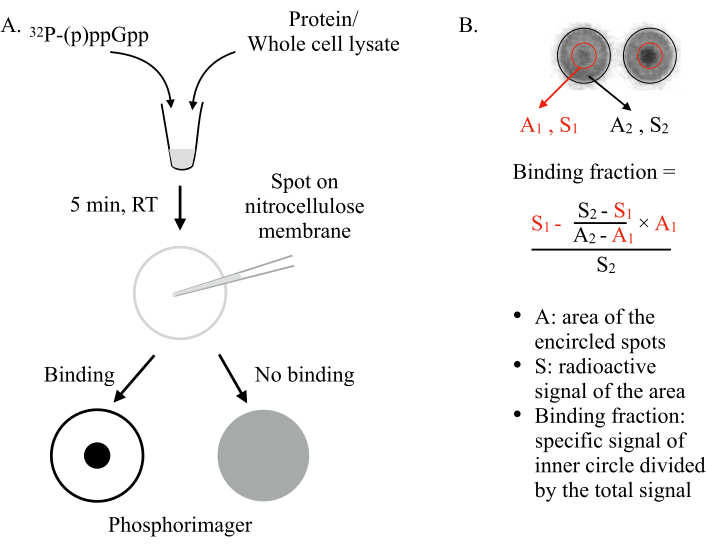
Figure 1: Le principe de DRaCALA. (A) Schéma du test DRaCALA. Voir le texte pour plus de détails. (B) Quantification et calcul de la fraction liante. Voir le texte pour plus de détails. En bref, les taches DRaCALA seront analysées en dessinant deux cercles qui circonscrivent la tache entière et le point sombre interne(c’est-à-direle (p)ppGpp retenu en raison de la liaison de la protéine testée). Le signal de liaison spécifique est le signal radioactif du cercle intérieur (S1) après soustraction du signal de fond non spécifique (calculé par A1 × ((S2-S1)/(A2-A1))). La fraction de liaison est le signal de liaison spécifique divisé par le signal radioactif total (S2). Abréviations: DRaCALA = Action capillaire radiale différentielle du test de ligand; (p)ppGpp = guanosine penta- et tétraphosphates; RT = température ambiante. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation de lysates de cellules entières
- Inoculer les souches de collecte E. coli K-12 ASKA ORFeome15 dans un bouillon de lysogénie (LB) de 1,5 mL contenant 25 μg/mL de chloramphénicol dans des plaques de puits profondes de 96 puits. Cultiver pendant la nuit (O/N) pendant 18 h à 30 °C en agitant à 160 tr/min. Le lendemain, ajouter l’isopropyle β-d-1-thiogalactopyranoside (IPTG) (final 0,5 mM) aux cultures O/N pour induire l’expression des protéines à 30 °C pendant 6 h.
- Cellules à granulés à 500 x g pendant 10 min. Congeler les granulés à -80 °C jusqu’à leur utilisation. Pour lyser les cellules, ajouter 150 μL de tampon de lyse L1 (40 mM Tris pH 7,5, 100 mM NaCl, 10 mM MgCl2, complété par 2 mM de fluorure de phénylméthylsulfonyle (PMSF), 40 μg/mL de DNase 1 et 0,5 mg/mL de lysozyme) pour ressusposer la pastille.
- Congeler les cellules à -80 °C pendant 30 min, puis décongeler à 37 °C pendant 20 min. Répétez ce cycle trois fois pour lyser les cellules. Conserver les lysates à -80 °C avant utilisation.
2. Purification de Relseq et GppA
REMARQUE: Les protéines recombinantes Relseq de Streptococcus equisimilis et GppA de E. coli K-12 sont utilisées pour synthétiser les pppGpp et ppGpp radiomarqués, respectivement.
- Cultiver et collecter des cellules surexprimant chaque protéine.
- Cultiver la souche E. coli BL21 DE3 jusqu’à la phase exponentielle (densité optique (OD) ~ 0,3-0,4) dans le bouillon LB et faire tourner 1 mL de culture à 6000 x g pendant 5 min. Décanter le surnageant et ressuspender les cellules avec 100 μL de bouillon de BST glacé (bouillon LB complété par 0,1 g/mL de PEG3350, 0,05 mL/mL de diméthylsulfoxyde, 20 mMMgCl2).
- Mélanger les plasmides portant le rel seq et le gppAmarqués à l’histidine, chacun de 100 ng, avec les suspensions cellulaires ci-dessus dans le TSB, et incuber sur de la glace pendant 30 min. Choc thermique des cellules à 42 °C pendant 40 s. Placer le mélange sur de la glace pendant 2 min, et ajouter 1 mL de bouillon LB à température ambiante pour permettre aux cellules de récupérer pendant 1 h à 37 °C avec agitation à 160 tr/min.
- Plaquer les cellules récupérées sur des plaques de gélose LB complétées par les antibiotiques correspondants (Relseq:100 μg/mL d’ampicilline; GppA : 30 μg/mL de kanamycine). Le lendemain, inoculez les colonies dans du bouillon LB pour commencer les précultures O/N des deux souches à 37 °C.
- Après 18 h, inoculer 500 mL de milieu LB avec 10 mL des cultures O/N et des antibiotiques correspondants. Cultivez les cultures en les agitant à 160 tr/min à 37 °C. Lorsque la DO600nm atteint 0,5-0,7, induire l’expression des protéines en ajoutant 0,5 mM d’IPTG et en augmentant pendant 3 h à 30 °C en agitant à 160 tr/min.
- Prélever les cellules en les tournant à 6084 x g pendant 10 min à 4 °C. Resuspendez la pastille dans 20 mL de solution saline glacée 1x tamponnée au phosphate (PBS) et recentrifugez à 1912 x g pendant 20 min à 4 °C. Décantez le surnageant et congelez les granulés à -20 °C avant utilisation.
- Purification par affinité de l’acide nickel-nitrilotriacétique (Ni-NTA)
REMARQUE: À partir de ce moment, assurez-vous que les échantillons sont froids.- Ajouter 40 mL de tampon de lyse glacé L2 (50 mM Tris pH 7,5, 150 mM naCl, 5 % de glycérol, 10 mM d’imidazole, 10 mM de β-mercaptoéthanol complété par des inhibiteurs de la protéase (comprimé sans EDTA; voir la table des matériaux)pour ressuspender la pastille. Lyser les cellules par sonication (amplitude de 60%, 2 s ON / 4 s OFF pendant 8 min ON). Nettoyer le lysate en filant à 23 426 x g pendant 40 min à 4 °C, et continuer avec le surnageant pour la purification.
- Pendant la centrifugation ci-dessus, préparez la résine Ni-NTA.
- Transférer 500 μL de résine Ni-NTA homogénéisée dans une colonne de chromatographie en polypropylène sur pied et laisser se déposer pendant 15 min et la solution de stockage s’écouler à travers. Lavez la résine avec 15 mL d’eau ultrapure deux fois, puis lavez la colonne avec 15 mL du tampon de lyse L2.
- Chargez le surnageant dégagé du lysate de cellule de l’étape 1 sur la colonne et laissez-le s’écouler. Laver la colonne avec 30 mL de tampon de lavage (50 mM Tris pH 7,5, 150 mM NaCl, 5% glycérol, 20 mM d’imidazole).
- Éluez les protéines avec 400 μL du tampon d’élution (50 mM Tris pH 7,5, 150 mM NaCl, 5% glycérol, 500 mM imidazole) trois fois. Ensuite, répétez l’élution avec 300 μL de plus du tampon d’élution. Combiner les protéines éluées pour un volume final de 700 μL.
- Filtration sur gel
- Préparer un tampon de filtration en gel (50 mM Tris, pH 7,5; 200 mM NaCl; 5% de glycérol). Lavez la colonne d’exclusion de taille avec un volume de colonne (25 mL) du tampon de filtration en gel.
- Chargez l’échantillon de 700 μL ci-dessus à l’aide d’une boucle de 500 μL, exécutez à 0,5 mL / min et collectez 2 à 3 fractions, chacune de volume de 0,5 mL, contenant les protéines respectives.
- Combinez et concentrez les fractions contenant chacune des protéines à l’aide d’une colonne de spin et mesurez la concentration en protéines à l’aide du test de Bradford.
3. Synthèse de 32pppGpp et ppGpp marqués P
- Assembler une réaction Relseq à petite échelle dans un tube à bouchon à vis (voir tableau 1).
REMARQUE : Travailler avec des réactifs radioactifs uniquement dans un endroit autorisé et avec de l’équipement de protection individuelle.
| Volume (μL) | ||
| Petite échelle | À grande échelle | |
| Eau ultrapure | ||
| 10x Relseq buffer* | 2 | 50 |
| ATP (finale 8 mM) | ||
| Relseq (4 μM final) | ||
| 32 P-α-Guanosine triphosphate (GTP) (final 120 nM) (ATTENTION) | 0.2 | 5 |
| total | 20 | 500 |
Tableau 1 : Informations d’assemblage pour les réactions de synthèse à petite et à grande échelle de 32pppGpp marqués P. *10x Le tampon Relseq contient 250 mM de Tris-HCl, pH 8,6 ; 1M NaCl; 80 mM MgCl2. Abréviation : pppGpp = guanosine pentaphosphate.
- Incuber le tube à 37 °C dans un thermomélangeur pendant 1 h, puis à 95 °C pendant 5 min, et placer sur de la glace pendant 5 min. Faites tourner la protéine précipitée à 15 700 x g pendant 5 min et transférez le surnageant (synthétisé 32P-pppGpp) dans un nouveau tube à bouchon à vis.
- Pour synthétiser 32P-ppGpp à partir de 32P-pppGpp, transférez la moitié du produit 32p-pppGpp dans un nouveau tube à bouchon à vis et ajoutez 1 μM GppA. Incuber le tube à 37 °C pendant 10 min, à 95 °C pendant 5 min, puis placer sur de la glace pendant 5 min.
- Faites tourner la protéine précipitée à 15 700 x g pendant 5 min et transférez le surnageant (synthétisé 32P-ppGpp) dans un nouveau tube à bouchon à vis.
- Analyser les 32P-pppGpp et 32P-ppGpp en faisant fonctionner 1 μL des échantillons sur une plaque de chromatographie sur couche mince (TLC) (plaques TLC de cellulose modifiée à la polyéthylèneimine) en utilisant le 1,5 M KH2PO4, pH 3,4, comme phase mobile.
REMARQUE: Utilisez leα-32P-marqué guanosine 5'-triphosphate(32P-α-GTP) comme témoin. - Séchez complètement la plaque TLC, placez-la entre un dossier en plastique transparent et exposez-la à un écran de stockage au phosphore pendant 5 minutes. Visualisez et quantifiez les signaux à l’aide d’un phosphorimager.
REMARQUE: Lorsque les rapports de 32P-pppGpp et 32P-ppGpp sont supérieurs à 85%, une réaction à grande échelle (500 μL, suffisant pour le criblage de 20 plaques de 96 puits) peut être assemblée et synthétisée à l’aide du tableau 1.
4. Dépistage DRaCALA des protéines cibles de (p)ppGpp
- Décongeler et transférer 20 μL de lysates de cellules entières vers une plaque de microtitrage à fond en V de 96 puits. Ajouter 2,5 U/puits d’endonucléase de Serratia marcescenset incuber à 37 °C pendant 15 min pour réduire la viscosité du lysate. Placez les lysates sur de la glace pendant 20 min.
- Mélanger les 32P-pppGpp et 32P-ppGpp dans un rapport de 1:1, et ajouter 1x tampon de lyse L1 pour que la concentration finale de (p)ppGpp soit égale à 4 nM.
REMARQUE: Compte tenu de la similitude chimique entre pppGpp et ppGpp, un mélange des deux produits chimiques simplifiera le processus de criblage. - Utilisez une pipette multicanal et des embouts de pipette filtrés pour ajouter et mélanger 10 μL du mélange (p)ppGpp avec le lysate cellulaire. Incuber à température ambiante (RT) pendant 5 min.
- Lavez l’outil à 96 broches en le plaçant dans une solution à 0,01% de détergent non ionique pendant 30 s et séchez-le sur un papier de soie pendant 30 s. Répétez le lavage de l’outil de broche 3x.
- Placez l’outil à broches dans les plaques d’échantillonnage de 96 puits ci-dessus et attendez 30 s. Soulevez l’outil à goupille vers le haut et placez-le directement sur une membrane de nitrocellulose pendant 30 s.
REMARQUE: S’il manque un point, repérez 2 μL des échantillons correspondants avec une pipette et des pointes filtrées. Il est conseillé de faire une tache dupliquée du même échantillon comme indiqué ci-dessous. - Sécher la membrane pendant 5 min à TA. Placez la membrane entre un dossier en plastique transparent et exposez-la à un écran de stockage au phosphore pendant 5 min. Visualisez à l’aide d’un phosphorimageur.
5. Quantification et identification des protéines cibles potentielles
- Utilisez le logiciel d’analyse associé au phosphorimager pour ouvrir le fichier .gel des plaques visualisées. Utilisez la fonction d’analyse Array pour définir les 96 points en configurant une grille de 12 colonnes x 8 lignes.
- Définissez de grands cercles pour circonscrire le bord extérieur de l’ensemble des taches (voir Figure 1B). Exportez volumn+background et area des grands cercles définis et enregistrez-les dans une feuille de calcul.
REMARQUE: Si nécessaire, repositionnez chaque cercle individuel pour qu’il se chevauche parfaitement avec les taches et redimensionnez chaque cercle individuel pour le rendre légèrement plus grand que le point réel. - Dimensionnez les cercles définis pour circonscrire les petits points intérieurs. Exportez Volumn+Backgroundet Area des petits cercles définis, puis enregistrez-les dans une feuille de calcul.
- Calculez les fractions de liaison dans la feuille de calcul à l’aide de l’équation de la figure 1Bet tracez les données. Identifier les protéines de liaison potentielles dans les puits qui présentent des fractions de liaison élevées par rapport à la majorité des autres puits.

Figure 2: Flux de travail global du processus de sélection DRaCALA. La production de protéines à partir d’une collection Escherichia coli ASKA est induite et les cellules sont lysées. Pendant ce temps, les protéines recombinantes Relseq-Hiset GppA-His sont purifiées et utilisées pour synthétiser 32pppGpp et ppGpp marqués P à partir de 32P-α-GTP. Les molécules (p)ppGpp radioactivement étiquetées sont ensuite mélangées avec les lysates, et un outil à 96 broches est utilisé pour repérer les mélanges sur une membrane de nitrocellulose pour une exposition ultérieure à un écran de stockage du phosphore, l’imagerie et la quantification des signaux radioactifs. Abréviations: DRaCALA = Action capillaire radiale différentielle du test de ligand; (p)ppGpp = guanosine penta- et tétraphosphates; RT = température ambiante; IPTG = isopropyle β-d-1-thiogalactopyranoside; GTP = guanosine 5'-triphosphate; SDS-PAGE = électrophorèse sur gel de dodécylsulfate-polyacrylamide de sodium ; TLC = chromatographie sur couche mince. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
Suivre le protocole décrit ci-dessus donnera généralement deux types de résultats (Figure 3).
La figure 3A montre une plaque avec des signaux de liaison de fond relativement faibles (fractions de liaison < 0,025) provenant de la majorité des puits. Le signal de liaison positif du puits H3 donne une fraction de liaison d’environ 0,35 qui est beaucoup plus élevée que celle observée pour les autres puits. Même sans quantificat...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’une des étapes critiques dans la réalisation du dépistage DRaCALA est d’obtenir de bons lysates de cellules entières. Tout d’abord, les protéines testées doivent être produites en grande quantité et sous des formes solubles. Deuxièmement, la lyse des cellules doit être complète et la viscosité du lysate doit être minimale. L’inclusion de lysozyme et l’utilisation de trois cycles de gel-dégel sont souvent suffisantes pour lyser complètement les cellules. Cependant, l’ADN chromosomique libéré...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Le travail est soutenu par une subvention de projet NNF (NNF19OC0058331) à YEZ, et le programme de recherche et d’innovation Horizon 2020 de l’Union européenne dans le cadre de la convention de subvention Marie Skłodowska-Curie (Nº 801199) à MLS.

Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
Références
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404(2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232(2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188(2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388(2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon