このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リガンドアッセイの微小径毛管作用による小リガンドの結合タンパク質の同定(DRaCALA)
要約
リガンドアッセイの微小径径毛管作用(DRaCALA)は、ORFeomeライブラリを使用して生物の小さなリガンド結合タンパク質を同定するために使用できます。
要約
過去10年間、細菌生理学における小信号分子の理解は大きく進歩しています。特に、いくつかのヌクレオチド由来二次メッセンジャー(NSM)の標的タンパク質を体系的に同定し、モデル生物で研究されている。これらの成果は、主に、これらの小分子の標的タンパク質を体系的に同定するために使用されたリガンドアッセイ(DRaCALA)の捕獲化合物技術および微分径状毛細管作用を含むいくつかの新しい技術の開発によるものです。本論文では、DRaCALA技術の例及びビデオデモとして、NSM、グアノシンペンタおよびテトラリン酸(p)ppGppの使用について説明する。DRaCALAを用いて、(p)ppGppの20個の既知および12個の新しい標的タンパク質のうち9個がモデル生物 のエシェリヒア・コリ K-12で同定され、このアッセイの力を実証した。DRaCALAは、原理的に、放射性同位体や蛍光色素によって標識できる小さなリガンドの研究に使用できます。この手法のさらなる応用のために、DRaCALA の重要なステップ、長所、および短所について説明します。
概要
細菌は、絶えず変化する環境1、2に適応するために、いくつかの小さなシグナル伝達分子を使用しています。例えば、オートインデューサーは、N-アシルホモセリンラクトンおよびそれらの改変オリゴペプチドと、集団行動を調整するために細菌間の細胞間通信を仲介し、クォーラムセンシング2として知られる現象である。小シグナル伝達分子の別のグループは、NSMであり、広く研究された環状アデノシン一リン酸(cAMP)、環状ジAMP、環状ジグアノシン一リン酸(環状ジGMP)、およびグアノシンペンタおよびテトラリン酸(p)ppGpp1を含む。細菌は、さまざまな異なるストレス状態に対する応答としてこれらのNSMを産生する。いったん産生されると、これらの分子は標的タンパク質に結合し、遭遇したストレスに対処し、細菌の生存を高めるためにいくつかの異なる生理学的および代謝経路を調節する。したがって、標的タンパク質の同定は、これらの小分子の分子機能を解読するための避けられない前提条件である。
過去10年間、これらの小さなシグナル伝達分子に関する知識のブームを目の当たりにしてきました, 主にこれらの小分子の標的タンパク質を発表したいくつかの技術革新.これらには、この論文で議論されるリガンドアッセイ(DRaCALA)6の捕捉化合物技術3、4、5、および微分径状毛細管作用が含まれる。
2011年6年にヴィンセント・リーと同僚によって発明されたDRaCALAは、ニトロセルロース膜の能力を、タンパク質結合リガンドを差し違いに隔離するために展開しています。タンパク質などの分子はニトロセルロース膜に拡散できず、NSMなどの小さなリガンドは拡散することができます。NSM(例えば、ppGpp)を検査するタンパク質と混合し、膜上でそれらを発見することによって、2つのシナリオが期待できる(図1):(p)ppGppがタンパク質に結合した場合、放射標識された(p)ppGppはタンパク質によってスポットの中央に保持され、外側に拡散せず、激しいドットを与える( 強い放射性信号)リンイメージャーの下で。しかし、(p)ppGppがタンパク質に結合しない場合、放射能シグナルの背景が均一な大きなスポットを作り出すために、自由に外側に拡散する。
さらに、DRaCALAは、タンパク質が十分な量で存在する場合、細胞全体のライセートにおける小分子と未精製タンパク質との相互作用を検出することができる。このシンプルさは、ORFeome発現ライブラリを使用して、タンパク質標的を迅速に同定する際にDRaCALAを使用することを可能にする。実際、cAMP7、環状di-AMP8、環状ジGMP9、10、および(p)ppGpp11、12、13の標的タンパク質は、DRaCALAを用いて系統的に同定されている。このビデオ記事では、(p)ppGpp を例として、DRaCALA スクリーニングを成功させるために重要な手順と考慮事項を示し、説明します。DRaCALA14の詳細については、DRaCALA を実行する前に、この記事と組み合わせて読むことを強くお勧めします。
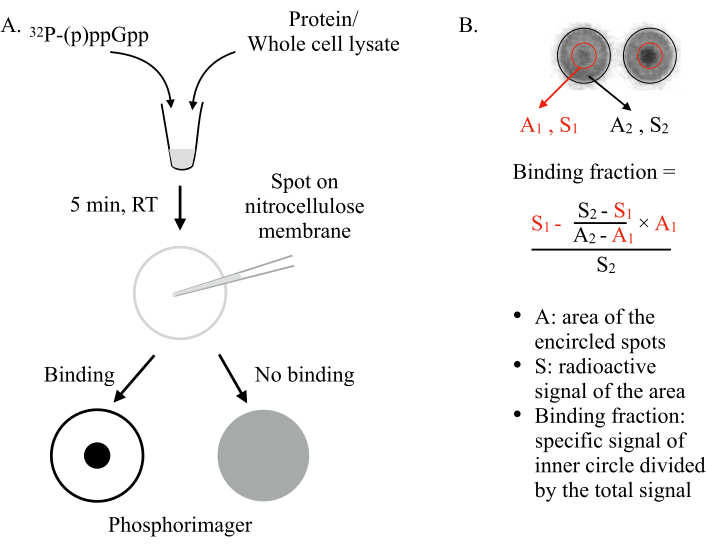
図1: ドラカラの原理(A) DRaCALAアッセイの概略図詳細については、テキストを参照してください。(B) 結合分数の定量化と計算。詳細については、テキストを参照してください。簡単に言えば、DRaCALAスポットは、スポット全体と内側の暗い点(すなわち、試験されたタンパク質の結合に起因する保持(p)ppGpp)を囲む2つの円を描くことによって分析される。特定結合信号は、非特異的なバックグラウンド信号を減算した後の内円(S1)の放射性信号(((S2-S ×1)/(A2-A1))で計算された非特異的なバックグラウンド信号(((S2-S1)/(A2-A1)です。 結合分率は、特定の結合信号を全放射性信号で割った(S2)。略語: DRaCALA = リガンドアッセイの差動ラジアル毛細管作用;(p)ppGpp =グアノシンペンタ-およびテトラリン酸塩;RT = 室温。この図の大きなバージョンを表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 細胞全体のライセートの調製
- 大腸菌K-12 ASKA ORFeomeコレクションは、96ウェル深ウェルプレートに25 μg/mLクロラムフェニコールを含む15から1.5 mLリソジニーブロス(LB)を取り込みます。一晩(O/N)を30°Cで18時間成長させ、160rpmで振る。翌日、イソプロピルβ-d-1-チオガラクトピラノシド(IPTG)(最終0.5mM)をO/N培養物に加え、30°Cで6時間タンパク質発現を誘導する。
- ペレット細胞は500xgで10分間用いった。ペレットを使用するまで-80 °Cで凍結します。細胞をリゼするには、150 μLのリシスバッファーL1(40 mM Tris pH 7.5、100 mM NaCl、10 mM MgCl2、2mM フェニルメチルスルホニルフッ化物(PMSF)、40 μg/mL DNase 1、および0.5 mg/mLリゼを添加して、レセリュードにリゼスバッファーを加えます。
- 細胞を-80°Cで30分間凍結し、37°Cで20分間解凍します。細胞をライスするためにこのサイクルを3回繰り返します。使用前に-80 °Cでライセートを保管してください。
2. Relセク とGppAの精製
注:レンサ球菌の等価性体と大腸菌K-12のGppAの組換えタンパク質Relseqは、それぞれ放射標識pppGppとppGppを合成するために使用されます。
- 成長し、各タンパク質を過剰発現する細胞を収集します。
- 大 腸菌 BL21 DE3株をLBブロスで指数位相(光学密度(OD)〜0.3-0.4)まで成長させ、5分間6000 x g で1mLの培養液をスピンダウンします。上清をデカントし、100 μLの氷冷性TSBブロス(0.1 g/mL PEG3350、0.05 mL/mLジメチルスルホキシド、20 mM MgCl2を添加したLBスープ)で細胞を再懸濁する。
- ヒスチジンタグ付き relseq と gppAを持つプラスミドを混合し、それぞれ100ng、上記の細胞懸濁液をTSBで混合し、氷上で30分間インキュベートします。40sの場合は42°Cで細胞に熱ショックを与える。混合物を氷の上に2分間置き、室温で1mLのLBスープを加え、細胞が160rpmで攪拌して37°Cで1時間回収できるようにします。
- LB寒天プレートに回収した細胞を、対応する抗生物質を補充してプレート(Relseq:100 μg/mLアンピシリン;GppA:30 μg/mL カナマイシン)。翌日、LBスープでコロニーを接種し、37°Cで両方の株のO/N前培養を開始する。
- 18時間後、O/N培養物の10mLとそれに対応する抗生物質を用いてLB培地500mLを接種する。37°Cで160rpmで振って培養を成長させます。 OD600nm が0.5~0.7に達すると、0.5 mMのIPTGを加えてタンパク質発現を誘導し、30°Cで3時間、160rpmで振る。
- 6084 x g で 4 °C で 10 分間回転させて細胞を回収します。 ペレットを氷冷1xリン酸緩衝生理食塩水(PBS)の20mLで再懸濁し、1912 x g で4°Cで20分間遠心分離を再び行う。 上清をデカントし、使用前に-20°Cでペレットを凍結します。
- ニッケルニトリロトリアセク酸(Ni-NTA)親和性精製
注: この時点以降は、サンプルが冷たくなっていることを確認してください。- 氷冷ライシスバッファーL2(50 mM Tris pH 7.5,150 mM NaCl,5%グリセロール,10 mMイミダゾール,10 mM β-メルカプトエタノールをプロテアーゼ阻害剤(EDTAフリー錠剤; 材料表を参照)を加え、ペレットを再中断します。超音波処理(60%振幅、2 s ON / 4 s OFF 8分オン)を介して細胞をlyse。4°Cで40分間23,426 x g で回転させてリセートをクリアし、精製のために上清を続けます。
- 上記遠心中、Ni-NTA樹脂を準備します。
- 500 μLの均質化 Ni-NTA 樹脂を立ち位置のポリプロピレンクロマトグラフィーカラムに移し、15分間落ち着かせ、保管液を水流にします。15mLの超純水で樹脂を2回洗浄し、15mLの溶精バッファーL2でカラムを洗浄します。
- ステップ 1 からセルライセートのクリアした上清をカラムにロードし、それを通して流れます。30 mLの洗浄バッファー(50 mM トリス pH 7.5、150 mM NaCl、5% グリセロール、20 mM イミダゾール)でカラムを洗浄します。
- 溶出バッファーの 400 μL (50 mM トリス pH 7.5, 150 mM NaCl, 5% グリセロール, 500 mM イミダゾール) を 3 回溶出させます。次に、溶出バッファーの別の 300 μL で溶出を繰り返します。溶出したタンパク質を最終的な体積700 μLに組み合わせます。
- ゲル濾過
- ゲル濾過バッファー(50 mMトリス、pH 7.5;200 mM NaCl;5%グリセロール)を準備します。サイズ除外カラムをゲル濾過バッファーの1カラム体積(25 mL)で洗浄します。
- 上記の700 μLサンプルを500 μLループでロードし、0.5 mL/minで実行し、各タンパク質を含む0.5 mLの各体積の2~3個の分画を収集します。
- スピンカラムを使用して各タンパク質を含む分画を組み合わせて濃縮し、ブラッドフォードアッセイを使用してタンパク質濃度を測定します。
3. 32PラベルのpppGppとppGppの合成
- スクリューキャップチューブに小さなRelseq 反応を組み立てます( 表1参照)。
注意:放射性試薬は、ライセンスを受けた場所と個人用保護具でのみ使用してください。
| ボリューム(μL) | ||
| 小規模 | 大幅 | |
| 純水 | ||
| 10x Relseq バッファ* | 2 | 50 |
| ATP(8mM決勝) | ||
| レル・セク(4 μ M決勝) | ||
| 32P-α-グアノシン三リン酸 (GTP) (最終 120 nM) (注意) | 0.2 | 5 |
| トータル | 20 | 500 |
表1:32P標識pppGppの小規模および大規模合成反応の組み立て情報*10x Relseqバッファには、250 mM Tris-HCl、pH 8.6が含まれています。1M NaCl;80 mM MgCl2.略語: pppGpp = グアノシン五タン酸。
- 37°Cで1時間熱ミキサーでチューブをインキュベートし、95°Cで5分間、氷の上に5分間置きます。沈殿したタンパク質を15,700 x g で5分間スピンダウンし、上清(合成 32P-pppGpp)を新しいスクリューキャップチューブに移します。
- 32P-pppGppから32P-ppGppを合成するには、32p-pppGpp製品の半分を新しいスクリューキャップチューブに移し、1 μM GppAを追加します。37°Cで10分間、95°Cで5分間インキュベートし、5分間氷の上に置きます。
- 沈殿したタンパク質を15,700 x g で5分間スピンダウンし、上清(合成 32P-ppGpp)を新しいスクリューキャップチューブに移します。
- 1.5 M KH 2 PO 4、pH3.4を移動相として使用し、薄層クロマトグラフィー(TLC)プレート(ポリエチレンイミン修飾セルロースTLCプレート)上でサンプル1μLを実行して、32P-pppGppおよび32P-ppGppを分析します。
注意:α-32Pラベルグアノシン5'-三リン酸(32P-α-GTP)をコントロールとして使用します。 - TLCプレートを完全に乾燥させ、透明なプラスチックフォルダの間に置き、5分間保管用蛍光体スクリーンに露出させます。リンイメージャーを使用して信号を視覚化し、定量化します。
注 :32P-pppGppと 32P-ppGppの比率が85%を超える場合、 表1を用いて大規模反応(500 μL、スクリーニングに十分な20 96ウェルプレート)を組み立てて合成することができます。
4. (p)ppGppの標的タンパク質のDRaCALAスクリーニング
- 全細胞ライセートの20 μLを96ウェルVボトムマイクロティタープレートに解凍して移送します。 セラチア・マルセセンスからエンドヌクレアーゼの2.5 U/ウェルを加え、37°Cで15分間インキュベートして、リゼートの粘度を下げます。氷の上に20分間ライセートを置きます。
- 32P-pppGpp と32P-ppGpp を 1:1 の比率で混合し、1x リシス バッファー L1 を加えて(p)ppGpp の最終濃度を 4 nM にします。
注:pppGppとppGppの化学的類似性を考えると、両方の化学物質を混合するとスクリーニングプロセスが簡素化されます。 - マルチチャンネルピペットとフィルター処理されたピペットチップを使用して、(p)ppGpp混合物を細胞のリセートと10 μL混合します。室温(RT)で5分間インキュベートします。
- 96 xピンツールを、非イオン洗剤の0.01%溶液に30sの溶液を入れ、ティッシュペーパーで30sで乾かします。ピンツール3xの洗浄を繰り返します。
- 上記の96ウェルサンプルプレートにピンツールを置き、30 s待ちます。ピンツールをまっすぐ上に持ち上げ、ニトロセルロース膜の上にまっすぐに30s置きます。
注:スポットが欠落している場合は、ピペットとフィルタリングされたチップで対応するサンプルの2 μLを見つけます。以下に示すように同じサンプルの重複スポットを作ることをお勧めします。 - 透明なプラスチック製のフォルダの間に膜を5分間乾燥させ、5分間保管用蛍光体スクリーンに露出させます。リンイメージャーを使用して視覚化します。
5. 潜在的標的タンパク質の定量と同定
- リンイメージャーに関連付けられた解析ソフトウェアを使用して、視覚化されたプレートの .gel ファイルを開きます。 配列解析 機能を使用して、12 列 x 8 行のグリッドを設定して 96 個のスポットを定義します。
- スポット全体の外側の端を囲む大きな円を定義します ( 図 1Bを参照)。定義された大きな円の Volumn+背景 と 領域 をエクスポートし、スプレッドシートに保存します。
注:必要に応じて、個々の円をスポットと完全に重なるように位置を変更し、個々の円のサイズを変更して実際のスポットよりもわずかに大きくします。 - 小さな内側の点を囲むには、定義された円を下にサイズします。 Volumn + 背景、 定義された小さな円の 領域 をエクスポートし、スプレッドシートに保存します。
- 図 1Bの式を使用してスプレッドシートの結合分数を計算し、データをプロットします。他のウェルの大部分と比較して高い結合画分を示すウェル内の潜在的な結合タンパク質を同定する。

図2:DRaCALAスクリーニングプロセスの全体的なワークフロー大腸菌ASKAコレクションからのタンパク質産生が誘導され、細胞が細胞を取り除きます。一方、組換えタンパク質Relseq-HisとGppA-Hisは精製され、32P-α-GTPから32P標識pppGppおよびppGppを合成するために使用されます。放射性標識(p)ppGpp分子は、その後、ライゼートと混合され、96ピンツールは、その後の蛍光体の貯蔵画面への暴露、イメージング、および放射性信号の定量のためにニトロセルロース膜上の混合物を発見するために使用されます。略語: DRaCALA = リガンドアッセイの差動ラジアル毛細管作用;(p)ppGpp =グアノシンペンタ-およびテトラリン酸塩;RT = 室温;IPTG = イソプロピル β-d-1-チオガラクトピラノシド;GTP = グアノシン 5'-三リン酸;SDS-PAGE = ドデシルスルフェート -ポリアクリルアミドゲル電気泳動;TLC = 薄層クロマトグラフィー。この図の大きなバージョンを表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
結果
上記のプロトコルに従うと、通常は2種類の結果が得られます(図3)。
図3Aは、大部分のウェルからのバックグラウンド結合信号(0.025<結合分率)が比較的低いプレートを示す。ウェルH3からの正の結合シグナルは、他のウェルに対して観察されたものよりもはるかに高い〜0.35の結合分率を与える。定量化がなくても、H3が顕著であ?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
DRaCALAスクリーニングを行う上で重要なステップの1つは、良好な全細胞のライセートを得る。まず、試験されたタンパク質を大量に、可溶性の形態で産生すべきである。第二に、細胞のリシスは完全であり、そして、リセートの粘度は最小限でなければならない。リソザイムの包含と凍結融解の3サイクルの使用は、多くの場合、細胞を完全にライゼするのに十分です。しかし、放出された染?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは開示する利益相反を持っていません。
謝辞
この研究は、YEZへのNNFプロジェクトグラント(NNF19OC0058331)と、MLSへのマリー・スクウォトフスカ・キュリー交付金協定(Nº 801199)に基づく欧州連合(EU)のホライゾン2020研究・イノベーションプログラムによって支援されています。

Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 32P-α-GTP | Perkinelmer | BLU006X250UC | |
| 96 x pin tool | V&P Scientific | VP 404 | 96 Bolt Replicator, on 9 mm centers, 4.2 mm Bolt Diameter, 24 mm long |
| 96-well V-bottom microtiter plate | Sterilin | MIC9004 | Sterilin Microplate V Well 611V96 |
| Agar | OXOID - Thermo Fisher | LP0011 | Agar no. 1 |
| ASKA collection strain | NBRP, SHIGEN, JAPAN | Ref: DNA Research, Volume 12, Issue 5, 2005, Pages 291–299. https://doi.org/10.1093/dnares/dsi012 | |
| Benzonase | SIGMA | E1014-25KU | genetically engineered endonuclease from Serratia marcescens |
| Bradford Protein Assay Dye | Bio-Rad | 5000006 | Reagent Concentrate |
| DMSO | SIGMA | D8418 | ≥99.9% |
| DNase 1 | SIGMA | DN25-1G | |
| gel filtration10x300 column | GE Healthcare | 28990944 | contains 20% ethanol as preservative |
| Glycerol | PanReac AppliChem | 122329.1214 | Glycerol 87% for analysis |
| Hypercassette | Amersham | RPN 11647 | 20 x 40 cm |
| Imidazole | SIGMA | 56750 | puriss. p.a., ≥ 99.5% (GC) |
| IP Storage Phosphor Screen | FUJIFILM | 28956474 | BAS-MS 2040 20x 40 cm |
| Isopropyl β-d-1-thiogalactopyranoside (IPTG) | SIGMA | I6758 | Isopropyl β-D-thiogalactoside |
| Lysogeny Broth (LB) | Invitrogen - Thermo Fisher | 12795027 | Miller's LB Broth Base |
| Lysozyme | SIGMA | L4949 | from chicken egg white; BioUltra, lyophilized powder, ≥98% |
| MgCl2 (Magnesium chloride) | SIGMA | 208337 | |
| MilliQ water | ultrapure water | ||
| multichannel pipette | Thermo Scientific | 4661110 | F1 - Clip Tip; 1-10 ul, 8 x channels |
| NaCl | VWR Chemicals | 27810 | AnalaR NORMAPUR, ACS, Reag. Ph. Eur. |
| Ni-NTA Agarose | Qiagen | 30230 | |
| Nitrocellulose Blotting Membrane | Amersham Protran | 10600003 | Premium 0.45 um 300 mm x 4 m |
| PBS | OXOID - Thermo Fisher | BR0014G | Phosphate buffered saline (Dulbecco A), Tablets |
| PEG3350 (Polyethylene glycol 3350) | SIGMA | 202444 | |
| phenylmethylsulfonyl fluoride (PMSF) | SIGMA | 93482 | Phenylmethanesulfonyl fluoride solution - 0.1 M in ethanol (T) |
| Phosphor-imager | GE Healthcare | 28955809 | Typhoon FLA-7000 Phosphor-imager |
| Pipette Tips, filtered | Thermo Scientific | 94410040 | ClipTip 12.5 μl nonsterile |
| Poly-Prep Chromatography column | Bio-Rad | 7311550 | polypropylene chromatography column |
| Protease inhibitor Mini | Pierce | A32955 | Tablets, EDTA-free |
| screw cap tube | Thermo Scientific | 3488 | Microcentifuge Tubes, 2.0 ml with screw cap, nonsterile |
| SLS 96-deep Well plates | Greiner | 780285 | MASTERBLOCK, 2 ML, PP, V-Bottom, Natural |
| spin column | Millipore | UFC500396 | Amicon Ultra -0.5 ml Centrifugal Filters |
| Thermomixer | Eppendorf | 5382000015 | Thermomixer C |
| TLC plate (PEI-cellulose F TLC plates) | Merck Millipore | 105579 | DC PEI-cellulose F (20 x 20 cm) |
| Tris | SIGMA | BP152 | Tris Base for Molecular Biology |
| Tween 20 | SIGMA | P1379 | viscous non-ionic detergent |
| β-mercaptoethanol | SIGMA | M3148 | 99% (GC/titration) |
参考文献
- Kalia, D., et al. Nucleotide, c-di-GMP, c-di-AMP, cGMP, cAMP, (p)ppGpp signaling in bacteria and implications in pathogenesis. Chemical Society Reviews. 42 (1), 305-341 (2013).
- Camilli, A., Bassler, B. L. Bacterial small-molecule signaling pathways. Science. 311 (5764), 1113-1116 (2006).
- Luo, Y., et al. The cAMP capture compound mass spectrometry as a novel tool for targeting cAMP-binding proteins: from protein kinase A to potassium/sodium hyperpolarization-activated cyclic nucleotide-gated channels. Molecular & Cellular Proteomics. 8 (12), 2843-2856 (2009).
- Nesper, J., Reinders, A., Glatter, T., Schmidt, A., Jenal, U. A novel capture compound for the identification and analysis of cyclic di-GMP binding proteins. Journal of Proteomics. 75 (15), 4874-4878 (2012).
- Laventie, B. J., et al. Capture compound mass spectrometry--a powerful tool to identify novel c-di-GMP effector proteins. Journal of Visual Experiments. (97), e51404(2015).
- Roelofs, K. G., Wang, J., Sintim, H. O., Lee, V. T. Differential radial capillary action of ligand assay for high-throughput detection of protein-metabolite interactions. Proceedings of the National Academy of Sciences of the United States of America. 108 (37), 15528-15533 (2011).
- Zhang, Y., et al. Evolutionary adaptation of the essential tRNA methyltransferase TrmD to the signaling molecule 3 ',5 '-cAMP in bacteria. Journal of Biological Chemistry. 292 (1), 313-327 (2017).
- Corrigan, R. M., et al. Systematic identification of conserved bacterial c-di-AMP receptor proteins. Proceedings of the National Academy of Sciences of the United States of America. 110 (22), 9084-9089 (2013).
- Roelofs, K. G., et al. Systematic identification of cyclic-di-GMP binding proteins in Vibrio cholerae reveals a novel class of cyclic-di-GMP-binding ATPases associated with type II secretion systems. PLoS Pathogen. 11 (10), 1005232(2015).
- Fang, X., et al. GIL, a new c-di-GMP-binding protein domain involved in regulation of cellulose synthesis in enterobacteria. Molecular Microbiology. 93 (3), 439-452 (2014).
- Corrigan, R. M., Bellows, L. E., Wood, A., Grundling, A. ppGpp negatively impacts ribosome assembly affecting growth and antimicrobial tolerance in Gram-positive bacteria. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1710-1719 (2016).
- Zhang, Y., Zbornikova, E., Rejman, D., Gerdes, K. Novel (p)ppGpp binding and metabolizing proteins of Escherichia coli. Mbio. 9 (2), 02188(2018).
- Yang, J., et al. The nucleotide pGpp acts as a third alarmone in Bacillus, with functions distinct from those of (p) ppGpp. Nature Communications. 11 (1), 5388(2020).
- Orr, M. W., Lee, V. T. Differential radial capillary action of ligand assay (DRaCALA) for high-throughput detection of protein-metabolite interactions in bacteria. Methods in Molecular Biology. 1535, 25-41 (2017).
- Kitagawa, M., et al. Complete set of ORF clones of Escherichia coli ASKA library (A complete Set of E. coli K-12 ORF archive): Unique resources for biological research. DNA Research. 12 (5), 291-299 (2005).
- Hochstadt-Ozer, J., Cashel, M. The regulation of purine utilization in bacteria. V. Inhibition of purine phosphoribosyltransferase activities and purine uptake in isolated membrane vesicles by guanosine tetraphosphate. Journal of Biological Chemistry. 247 (21), 7067-7072 (1972).
- Zhang, Y. E., et al. p)ppGpp regulates a bacterial nucleosidase by an allosteric two-domain switch. Molecular Cell. 74 (6), 1239-1249 (2019).
- Wang, B., et al. Affinity-based capture and identification of protein effectors of the growth regulator ppGpp. Nature Chemical Biology. 15 (2), 141-150 (2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved