Method Article
نهج قابل للتطوير للغاية لإجراء مسوحات بيئية للديدان الخيطية Caenorhabditis الذاتية
In This Article
Summary
يمكن استخدام هذا البروتوكول لإجراء مسوحات بيئية واسعة النطاق للديدان الخيطية Caenorhabditis الذاتية. الميزة الأساسية لهذه الطريقة هي التنظيم الفعال وتحليل البيانات البيئية والجزيئية المرتبطة بالديدان الخيطية التي تم جمعها من الطبيعة.
Abstract
Caenorhabditis elegans هي واحدة من الكائنات الحية النموذجية الرئيسية في علم الأحياء ، ولكن في الآونة الأخيرة فقط ركز الباحثون على بيئتها الطبيعية. التباين النسبي للمعلومات حول C. elegans في سياقها الطبيعي يأتي من التحديات التي ينطوي عليها تحديد الديدان الخيطية الصغيرة في الطبيعة. على الرغم من هذه التحديات ، تسبب التركيز المتزايد على بيئة C. elegans في ثروة من المعلومات الجديدة المتعلقة بحياتها خارج المختبر. ساهم البحث المكثف عن C. elegans في الطبيعة في اكتشاف العديد من أنواع Caenorhabditis الجديدة وكشف أن الديدان الخيطية المتجانسة غالبا ما تتعايش في البرية ، حيث تتغذى على الأزهار الميكروبية المرتبطة بالمواد النباتية المتعفنة. كما كشف تحديد الأنواع الجديدة أن نظام التزاوج الأندروديوكي للذكور والخنثى ذاتي الإخصاب قد تطور ثلاث مرات بشكل مستقل داخل التهاب Caenorhabditis. أما النوعان الآخران ذاتيان، وهما C. briggsae و C. tropicalis، فإنهما يشتركان في المزايا التجريبية ل C. elegans وقد مكنا من إجراء دراسات مقارنة على الأساس الميكانيكي للسمات الهامة، بما في ذلك الإخصاب الذاتي. على الرغم من هذه التطورات ، لا يزال هناك الكثير مما يجب تعلمه حول البيئة والتنوع الطبيعي لهذه الأنواع المهمة. على سبيل المثال ، ما زلنا نفتقر إلى المعلومات الوظيفية للعديد من جيناتهم ، والتي لا يمكن تحقيقها إلا من خلال فهم بيئتهم الطبيعية. لتسهيل البحث البيئي عن الديدان الخيطية Caenorhabditis الذاتية ، طورنا طريقة قابلة للتطوير للغاية لجمع الديدان الخيطية من البرية. تستخدم طريقتنا منصات جمع البيانات المتنقلة وقواعد البيانات المستندة إلى السحابة وبيئة برامج R لتعزيز قدرة الباحثين على جمع الديدان الخيطية من البرية وتسجيل البيانات البيئية المرتبطة بها وتحديد الديدان الخيطية البرية باستخدام الباركود الجزيئي.
Introduction
جلب العقدان الماضيان اهتماما متزايدا ببيئة الديدان الخيطية Caenorhabditis. من هذه الدراسات ، نعلم أن أنواع Caenorhabditis التي تعيش بحرية يمكن عزلها من الموائل الدقيقة سريعة الزوال في كل من المناطق المعتدلة والمدارية ، حيث تتغذى على الأزهار الميكروبية المرتبطة بالمواد النباتية المتحللة ، وأحيانا في التماثل 1،2،3،4،5،6،7،8 . لقد تعلمنا أيضا أن التطور المتقارب للإخصاب الذاتي قد حدث في الجنس ثلاث مرات ، وأن الذات هي الطريقة السائدة للتكاثر ل C. briggsae و C. elegans و C. tropicalis9,10. من بين هؤلاء المصورين الذاتيين ، C. elegans هي واحدة من أكثر الحيوانات التي تمت دراستها على نطاق واسع على الأرض وقد استخدمها الباحثون لتحقيق تقدم نقدي في علم الأحياء. الأهم من ذلك ، أن الأنواع الأخرى من Caenorhabditis تشترك في العديد من المزايا التجريبية C. elegans وتتقدم بسرعة في الدراسات المقارنة في الجنس. ومع ذلك، فإن الطبيعة المبهمة لهذه الديدان الخيطية في البرية تجعل من الصعب دراسة بيئتها وتنوعها الطبيعي، وهو أمر بالغ الأهمية لفهم الوظائف البيولوجية لجيناتها والطرق التي شكل بها التطور التنوع الجيني بين الأنواع10،11.
التحدي الأكبر لدراسة بيئة الديدان الخيطية Caenorhabditis في البرية هو صغر حجمها. غالبا ما يبلغ طول الديدان الخيطية البالغة 1 مم أو أقل. يتطلب هذا التحدي أن يقوم الباحثون بأخذ عينات من الركائز من البرية ومحاولة فصل الديدان الخيطية ذات الأهمية عن الركائز في المختبر دون القدرة على مراقبة الحيوانات في البرية. نظرا لأنه حتى الخبراء المدربين يجدون صعوبة في التمييز بين الديدان الخيطية Caenorhabditis من الديدان الخيطية الأخرى التي تعيش بحرية تحت المجهر ، تتم إزالة الديدان الخيطية عادة من الركيزة ، وعزلها ، وتركها للتكاثر قبل أن يتم تحديدها بواسطة هوية التسلسل باستخدام الباركود الجزيئي المعمول به3،12،13،14 . يمثل الوقت والجهد اللازمان لمعالجة كل ديدان خيطية بهذه الطريقة تحديا تنظيميا ، حيث يجب أن يكون الباحثون قادرين على تتبع هوية كل ديدان خيطية معزولة في المختبر إلى الركيزة الدقيقة والبيانات الإيكولوجية المرتبطة بها التي تم أخذ عينات منها في الميدان. هنا ، نصف عملية خطوة بخطوة لجمع وتحديد الديدان الخيطية Caenorhabditis الذاتية بكفاءة من الحقل وربط هذه العزلات بأمانة بالبيانات المكانية والبيئية المرتبطة بها على نطاق واسع.
تزيد طريقة الجمع هذه من حجم ودقة المسوحات البيئية باستخدام منصات جمع البيانات المتنقلة وقواعد البيانات المستندة إلى السحابة وبيئة برامج R. Fulcrum عبارة عن نظام أساسي لجمع البيانات قابل للتخصيص يعمل مع معظم الأجهزة المحمولة ويسمح للمستخدمين بإنشاء تطبيقات مخصصة لجمع وتنظيم البيانات المستندة إلى الموقع (https://www.fulcrumapp.com). يوفر هذا البروتوكول تعليمات مفصلة حول كيفية استخدام تطبيقات جمع البيانات المخصصة لتنظيم البيانات الإيكولوجية الواضحة مكانيا من الميدان وربط تلك البيانات بدقة بهوية الديدان الخيطية المعزولة في المختبر. يشرح البروتوكول أيضا كيفية تحديد الديدان الخيطية Caenorhabditis الذاتية بكفاءة باستخدام الباركود الجزيئي المعمول به. يمكن معالجة البيانات من هذه الطرق ببساطة وتكرار مع حزمة برامج R المصاحبة easyFulcrum15 لاستكشاف البيئة والتنوع الجيني لمجموعات Caenorhabditis الطبيعية.
Protocol
1. إعداد المجموعة
- حدد موقعا لمسح الديدان الخيطية Caenorhabditis .
ملاحظة: في معظم المناطق المعتدلة، يمكن عزل C. elegans و C. briggsae بسهولة عن الموائل المرتبطة بالإنسان مثل الحقول الزراعية أو الحدائق الريفية والحضرية1. في المناطق شبه الاستوائية والمدارية ، يمكن العثور على C. briggsae و C. elegans و C. tropicalis في الموائل المرتبطة بالإنسان المذكورة أعلاه ، وأحيانا على مقربة من بعضها البعض. ومع ذلك ، يبدو أن C. elegans تفضل الموائل الأكثر برودة وجفافا من الأنواع الأخرى في الموائل الاستوائية7,8. يمكن أيضا عزل كل نوع من الأنواع عن الموائل البرية التي لا ترتبط بالبشر ، ولكن يتم أخذ عينات من هذه الموائل في كثير من الأحيان. - إنشاء مشروع Fulcrum لتنظيم جمع البيانات وعزلها باستخدام تطبيقات جمع البيانات المتنقلة.
- أنشئ حسابا مع Fulcrum عبر الإنترنت باستخدام اتفاقية تعليمية بدون تكلفة16. أضف تطبيق أخذ عينات حقل الديدان الخيطية إلى حساب Fulcrum بالنقر فوق الزر ADD APP 17.
- أضف تطبيق عزل Nematode إلى حساب بالنقر فوق الزر ADD APP 18.
ملاحظة: يوصى بتنظيم كل رحلة إلى موقع ما كمشروع تجميع باستخدام اصطلاح التسمية "YearMonthLocation" ، على سبيل المثال ، 2020FebruaryAustralia.
- أضف مستخدمين إلى حساب Fulcrum لمنحهم حق الوصول إلى مشروع المجموعة. تأكد من أن كل مستخدم يقوم بتنزيل تطبيق Fulcrum للهاتف المحمول للمشاركة في المشروع.
- اطبع مجموعة من ملصقات رمز الاستجابة السريعة لتتبع المجموعات (C-labels) وعزلات الديدان الخيطية (S-labels) باستخدام تطبيق الهاتف المحمول. قم بإرفاق ملصقات C بأكياس بلاستيكية ذات قفل سحابي ، ولف الأكياس الملصقة إلى مجموعات من 25 ، ولفها بشريط مطاطي للتعبئة. احتفظ بمجموعة ملصقات S لاستخدامها في المختبر.
ملاحظة: في جميع أنحاء هذا البروتوكول ، يتم تضمين المجموعات (الركائز من الحقل) في أكياس أو على لوحات ويتم تصنيفها بملصقات C. يتم تسمية الديدان الخيطية المعزولة بملصقات S. تستخدم ملصقات C لتحديد المجموعات الفريدة ، وتستخدم ملصقات S لتحديد عزلات الديدان الخيطية الفريدة. يستخدم هذان النوعان من التسميات لإجراء اتصال بين مجموعة معينة (C-label) والديدان الخيطية المعزولة من تلك المجموعة (S-labels) في قاعدة بيانات Fulcrum. اطبع ضعف عدد الملصقات S كملصقات C لمشروع جمع لأنه ، في المتوسط ، يتم عزل اثنين من الديدان الخيطية لكل مجموعة. يمكن طباعة المزيد من ملصقات S لاحقا إذا لزم الأمر. يتم توفير 2500 تسمية C فريدة (الملف التكميلي 1) و 5000 تسمية S فريدة (الملف التكميلي 2) في الملحق. - قم بإعداد ألواح NGMA مقاس 10 سم للمجموعات وألواح NGMA مقاس 3.5 سم لعزل الديدان الخيطية. اصنع صفيحة واحدة بحجم 10 سم ولوحين على الأقل بحجم 3.5 سم لكل مجموعة21. يتم زرع هذه الصفائح مع سلالة الإشريكية القولونية OP50 وفقا للبروتوكولات المعمول بها. تخزين لوحات قبل الاستخدام في 4 درجة مئوية لمدة لا تزيد عن 1 شهر.
2. جمع الحقل
ملاحظة: غالبا ما يتم عزل الديدان الخيطية Caenorhabditis من المواد النباتية المتعفنة ، بما في ذلك الفواكه والمكسرات والبذور والقرون والزهور والسيقان والقمامة النباتية والسماد العضوي 1،5،6،8. أفضل الركائز فاسدة ولا يمكن التعرف عليها تقريبا كثمار أو زهور ؛ تجنب الركائز الجافة جدا أو الرطبة (الشكل 1). يتم جمع الركائز بكفاءة أكبر من الحقل من خلال العمل في أزواج. سيقوم الفرد الذي لديه ميزان حرارة الأشعة تحت الحمراء غير الملامسة باختيار ركيزة لجمع العينة وجمعها بينما يستخدم شريكه تطبيق أخذ العينات الميدانية الخيطية في Fulcrum لتسجيل بيانات الجمع. سيكرر زوج المجمعين هذه العملية حتى يتم جمع العدد المطلوب من العينات. وترد قائمة المواد اللازمة للعمل الميداني في (الجدول التكميلي 1).
- افتح تطبيق Fulcrum للجوال، وحدد أخذ عينات حقل النيماتودا من القائمة المنسدلة. اضغط على + لبدء سجل جديد في المشروع (الشكل 2A). التقط صورة للركيزة.
- انقر فوق المربع الموجود في الوسط العلوي لتحديد مشروع التجميع الصحيح الذي تم إجراؤه في الخطوة 1.2 (الشكل 2B). اضغط على حقل C-label في أسفل سجل المجموعة واختر المسح الضوئي عند ظهور المطالبة. امسح الباركود الموجود على حقيبة التجميع باستخدام كاميرا الجهاز المحمول، ثم اضغط على تم في الجزء العلوي الأيسر من الشاشة.
- اضغط على حقل الركيزة وحدد نوع الركيزة من القائمة المنسدلة. أضف ملاحظات حول الركيزة من خلال النقر فوق حقل ملاحظات الركيزة وإدخال الملاحظات يدويا.
- اختر منظرا أفقيا من القائمة المنسدلة. اختر المناظر الطبيعية التي تمثل موقع أخذ العينات على أفضل وجه.
- اختر طريقة عرض السماء. عند اختيار عرض السماء، صف رؤية السماء في موقع أخذ العينات (على سبيل المثال، رؤية السماء بالكامل دون إعاقة الرؤية من الأشجار أو الهياكل الأخرى = ممتلئة).
- قم بقياس درجة حرارة سطح الركيزة باستخدام مقياس الحرارة غير المتصل وسجل القيمة في حقل درجة حرارة الركيزة.
ملاحظة: أمسك بمقياس الحرارة غير المتصل على مسافة لا تزيد عن 14 بوصة من الركيزة أثناء تسجيل درجة الحرارة. - قم بقياس درجة الحرارة والرطوبة المحيطة باستخدام الجهاز المحمول باليد وسجل هذه البيانات في الحقول المناسبة.
ملاحظة: تحقق من أن جهاز درجة الحرارة والرطوبة المحيطة غير معلق. ستتغير وحدة القياس عند تحرير الزر. احتفظ بالجهاز في جيب خارجي لتجنب القراءات غير المنتظمة. - احفظ السجل في Fulcrum من خلال النقر على حفظ في الجزء العلوي الأيسر من الشاشة.
- اجمع حوالي ملعقة كبيرة من الركيزة بدون عصي أو قطع صلبة أخرى عن طريق عكس كيس التجميع لاستخدامه ك "قفاز" لالتقاط الركيزة ، ثم قم بإغلاق الحقيبة. ضع منشفة ورقية في الكيس إذا كانت العينة رطبة بشكل خاص.
ملاحظة: في المناخات الحارة، ضع الأكياس في مبردات ناعمة مع عبوات أكثر برودة للحفاظ على برودة المجموعات. - بعد جمع جميع العينات لهذا اليوم ، قم بتنظيف معدات التجميع ، وإخراج البطاريات من المجسات ، وإعادة شحن البطاريات ، وإعادة تجميد حزم الفريزر. قم بمزامنة بيانات مجموعة Fulcrum عن طريق النقر فوق الزر مزامنة في الجزء العلوي الأيسر من تطبيق أخذ عينات حقل الديدان الخيطية .
ملاحظة: قد تستغرق التحميلات عدة دقائق بدون اتصال خلوي قوي، لذا قد يكون من الأفضل انتظار الوصول إلى شبكة Wi-Fi. ستبقى البيانات على الأجهزة المحمولة وسيتم مزامنتها مع السحابة. - اشحن العينات إلى مؤسسة منزلية عن طريق وضعها في صندوق شحن بين عشية وضحاها. تقليل الوقت الذي تتعرض فيه العينات لدرجات حرارة أقل من 11 درجة مئوية أو أكبر من 25 درجة مئوية عن طريق شحن الطرود في الأيام التي يتم فيها نقل البضائع.
ملاحظة: معظم مرافق الشحن لا تشحن البضائع بين عشية وضحاها في عطلات نهاية الأسبوع في مواقع نائية.
3. طلاء المجموعات الميدانية في المختبر
ملاحظة: يوضح هذا القسم بالتفصيل كيفية تنظيم نقل العينات من أكياس التجميع الموسومة إلى اللوحات الملصقة. قد تصل العينات من شحنة بين عشية وضحاها أو مباشرة من الحقل.
- استلام شحنة المجموعات وفحصها بحثا عن أكياس مكسورة أو أدلة أخرى على الضرر. إذا تم كسر الأكياس ، تخلص من المواد ونظف أكياس التجميع غير المنكسرة بالإيثانول بنسبة 70٪ ؛ تجنب ملصق C الموجود على الحقيبة مع الإيثانول لأنه سيغير لون الملصق ويجعل من الصعب قراءته.
- لكل حقيبة بقفل سحابي، لاحظ ملصق C الموجود على الحقيبة وقم بإرفاق ملصق C مطابق بغطاء لوحة مقاس 10 سم مرقطة ببكتيريا OP50.
ملاحظة: يشار إلى الألواح الموسومة مقاس 10 سم باسم "لوحات C" لبقية البروتوكول. أسهل طريقة لتنظيم العينات هي وضع أكياس التجميع على مقعد المختبر مع لوحة C المطابقة في الأعلى (الشكل 3). - لكل مجموعة، انقل حوالي ملعقة كبيرة من العينة من كيس التجميع إلى اللوحة C باستخدام ملعقة بلاستيكية نظيفة. أضف العينة حول العشب البكتيري على شكل هلال أو حلقة ، ولا تغطي العشب البكتيري بالكامل (الشكل 4).
ملاحظة: حافظ على نظافة ملعقة كبيرة عن طريق وضعها في كوب من الإيثانول بنسبة 95٪ عندما لا تكون قيد الاستخدام. استخدم منشفة ورقية لتجفيف ملعقة كبيرة قبل نقل عينات إضافية. - سجل الوقت الذي تم فيه نقل المجموعات من أكياس التجميع إلى لوحات C واحتفظ بألواح C في درجة حرارة الغرفة (RT) لمدة 24 ساعة على الأقل قبل محاولة عزل الديدان الخيطية في القسم 4.
4. عزل الديدان الخيطية من المجموعات
- افتح تطبيق Fulcrum على الجهاز المحمول واختر Nematode Isolation من قائمة التطبيق (الشكل 5A). أنشئ سجلا جديدا للعزل من خلال النقر على الرمز + في أسفل اليسار (الشكل 5B).
- في شاشة سجل العزل الجديد، قم بتأكيد مشروع التجميع الصحيح عن طريق تحديد اسم المشروع المعروض في المربع الموجود في الوسط العلوي. إذا تم عرض المشروع الخطأ، فانقر فوق اسم المشروع للتبديل إلى المشروع الصحيح (الشكل 5C).
- اضغط على زر التحديد أسفل حقل C-label للعثور على C-label المرتبط بالعينة التي يتم عزل الديدان الخيطية منها (الشكل 5D). اضغط على أيقونة البحث ، ثم اضغط على أيقونة المسح الضوئي لمسح رمز الاستجابة السريعة C-label على لوحة C باستخدام كاميرا الجهاز. بمجرد مسح رمز الاستجابة السريعة ضوئيا، سيظهر سجل C-label في حقل C-label .
- اضغط على أيقونة الكاميرا في حقل الصور لفتح كاميرا الجهاز واستخدامها لالتقاط صورة للعينة على لوحة C مع رمز الاستجابة السريعة المرئي (الشكل 5E). اضغط على تم للعودة إلى شاشة العزل.
ملاحظة: يمكن استخدام صور سجل العزل هذه لاستكشاف سمات محددة للركيزة في وقت لاحق. - استخدم مجهر تشريح للبحث عن الديدان الخيطية على اللوحة C. اضغط على حقل الديدان في العينة لتسجيل وجود الديدان الخيطية على العينة (الشكل 5F). اضغط على نعم إذا كانت الديدان الخيطية موجودة على لوحة C واضغط على لا إذا لم تكن هناك ديدان خيطية.
- اضغط على المسارات في حالة وجود مسارات الديدان الخيطية فقط. في حالة عدم وجود ديدان خيطية ، قم بتصوير لوحة C والتخلص منها في صندوق خطر بيولوجي.
ملاحظة: اقلب اللوحة C فوق صندوق النفايات الخطرة بيولوجيا واضغط برفق على الجزء الخلفي من اللوحة لإزاحة جميع الركائز التي تم أخذ عينات منها. تسهل هذه الخطوة العثور على الديدان الخيطية التي يمكن أن تكون تحت الركيزة على اللوحة C وعزلها. - في حالة وجود الديدان الخيطية ، اعزل ما يصل إلى خمسة ديدان خيطية من اللوحة C. لعزل الديدان الخيطية، انقل نيماتودا واحدة من اللوحة C إلى لوحة S باستخدام اختيار سلك من البلاتين. عزل البالغين الأصحاء والمحبوبين إن أمكن. ومع ذلك ، اعزل المراحل الأخرى إذا لم يتم العثور على البالغين.
ملاحظة: بعد العزل، سيكون لكل منها ما يصل إلى خمس لوحات S ديدان خيطية واحدة. احتفظ بهذه الصفائح S مع الديدان الخيطية المعزولة من نفس اللوحة C المنظمة معا في كومة أنيقة بعيدا عن لوحات S الأخرى حتى يتم إدخالها في Fulcrum. - اضغط على حقل اللوحات التي تحمل علامة S لإدخال لوحة (لوحات) S المستخدمة في هذا العزل. اضغط على + في أسفل اليمين. اضغط على S-label ثم انقر فوق مسح ضوئي لفتح كاميرا الجهاز. استخدم كاميرا الجهاز لمسح رمز الاستجابة السريعة S-label ضوئيا على لوحة S.
ملاحظة: تأكد من تطابق رمز S-label مع الرمز الموجود على اللوحة. إذا كان مطابقا ، فانقر فوق تم. إذا لم يحدث ذلك ، فانقر فوق إلغاء وإعادة المسح الضوئي حتى يتطابق ، ثم انقر فوق تم. في بعض الأحيان يتم مسح رموز QR لللوحات القريبة عن طريق الخطأ. - بعد إدخال كل لوحة S، احفظ الإدخال باستخدام الزر حفظ في أعلى اليمين. سيتم فقدان الإدخال إذا لم يتم حفظه. اضغط على + في أسفل اليمين لإضافة المزيد من الألواح التي تحمل علامة S إذا لزم الأمر حتى يتم إدخال جميع الديدان الخيطية المعزولة من لوحة C. بعد إضافة جميع اللوحات التي تحمل علامة S لسجل العزل ، انقر فوق الزر < في الجزء العلوي الأيسر للعودة إلى شاشة سجل العزل.
ملاحظة: لإلغاء سجل عزل لأنه لا يمكن حل الأخطاء، انقر فوق إلغاء الأمر في الجزء العلوي الأيمن. ستفتح هذه الخطوة مربع حوار يسأل عما إذا كان يمكن تجاهل السجل دون حفظه. إذا رغبت في ذلك ، انقر فوق نعم ، تجاهل. - اضغط على زر حفظ في الجزء العلوي الأيمن بمجرد إضافة جميع المعلومات بشكل صحيح. ثم قم بتصوير الألواح S بديدان خيطية معزولة ووضعها جانبا في منطقة مخصصة لحمل ألواح S مع الديدان الخيطية.
- قم بتصوير اللوحة C والتخلص منها في صندوق المخاطر البيولوجية. اضغط على أيقونة المزامنة لتحميل جميع البيانات إلى Fulcrum.
- فرز جميع لوحات S بترتيب أبجدي رقمي ، ثم ضع لوحات S في صناديق من الورق المقوى. تأكد من أن لوحات S مغطاة لأسفل ومظلية. قم بتكديس ما يصل إلى أربع لوحات S في موضع واحد في الصندوق وقم بتسمية صندوق الورق المقوى باسم المشروع والتاريخ والوقت ورقم الصندوق الفريد.
- قم بتخزين المربعات المصنفة في RT. وسيتم فحص هذه العزلات للتأكد من عدم انتشارها في الساعة 48 ومرة أخرى في الساعة 168 إذا لزم الأمر.
5. تصدير لوحات S من نقطة الارتكاز
ملاحظة: يوضح هذا القسم كيفية تصدير تسميات S المستخدمة في عملية العزل من قاعدة بيانات مشروع Fulcrum. سيتم استخدام هذه الملصقات S لتتبع الخطوط المتساوية الإناث المنتشرة أثناء تحديدها بواسطة هوية التسلسل في الأقسام 6-9.
- سجل الدخول إلى موقع Fulcrum على الويب وحدد تطبيق عزل الديدان الخيطية . انقر على المصدر من الجانب الأيسر من الشاشة. انقر لتحديد المشروع المطلوب، وحدد المربع عزل الديدان الخيطية . انقر فوق التالي لتنزيل ملف .zip يحتوي على ملف "nematode_isolation_s_labeled_plates.csv".
- افتح ملف "nematode_isolation_s_labeled_plates.csv" وقم بفرزه حسب عمود "S-label" بترتيب تصاعدي (سيكون أصغر تصنيف S-label في الأعلى). حدد جميع تسميات S وانسخها من جدول البيانات.
- انتقل إلى قالب التنميط الجيني المعزول البري ورقة جوجل (wild_isolate_genotyping_template) باستخدام متصفح ويب19.
- قم بعمل نسخة من ورقة Google هذه بالنقر بزر الماوس الأيمن فوق علامة التبويب قالب التنميط الجيني ثم تحديد الخيار نسخ إلى جدول بيانات جديد . اختر فتح جدول البيانات لعرض ورقة Google الجديدة.
- قم بتسمية هذه الورقة الجديدة باسم مشروع نقطة الارتكاز متبوعا ب "wild_isolate_genotyping" ، على سبيل المثال ، "2020FebruaryAustralia_wild_isolate_genotyping".
ملاحظة: يشار إلى هذه الورقة باسم "ورقة التنميط الجيني" في بقية البروتوكول.
- الصق تسميات S المنسوخة من عمود "nematode_isolation_s_labeled_plates.csv" "s_label" في عمود ورقة التنميط الجيني بعنوان "s_label". تحقق من عمود "s_label_repeat_error" بحثا عن "1". تعني قيمة "1" في هذا العمود أن تسمية S مكررة في مكان ما على ورقة التنميط الجيني. إذا تم اكتشاف الازدواجية ، فقم بالتحقيق فيها وتصحيحها قبل المضي قدما.
- املأ عمود "isolation_box_number" في ورقة التنميط الجيني لجميع تسميات S.
6. تحقق من الانتشار على لوحات S
- تحقق من انتشار الحيوانات على لوحات S بعد 48 ساعة من العزل (استخدم تاريخ ووقت آخر عزل على المربع من الخطوة 4.11 لتوجيه التوقيت).
ملاحظة: تتميز الديدان الخيطية المنتشرة بنسل على لوحة S. - إذا كانت الصفيحة S تتكاثر، فأدخل "1" في العمود proliferation_48 على ورقة التنميط الجيني، ثم انقل اللوحة S إلى مربع يحمل علامة "انتشار 48 ساعة، المربع 1". ضع 88 لوحة S كحد أقصى في صندوق الانتشار ، ثم ابدأ في ملء صندوق جديد يحمل اسم "انتشار 48 ساعة ، الصندوق 2". تأكد من تنظيم ملصقات S بترتيب أبجدي رقمي في صناديق الانتشار لمدة 48 ساعة.
ملاحظة: لا تتخلص من لوحات S غير المنتشرة. سيتم فحص هذه اللوحات مرة أخرى في الساعة 168 بعد العزل. إذا رغبت في ذلك ، قم بدمج لوحات S هذه بترتيب رقمي في مربعات تحمل علامة "48 h غير قابلة للتكاثر ، المربع X" ، ولكن تذكر أن تسجل متى يجب أن يحدث الاختيار 168 h في المربع الجديد. - بعد تحديد جميع الملصقات S المنتشرة في 48 ساعة ، انتقل إلى القسم 7 للألواح S مع الانتشار عند 48 ساعة.
- تحقق من لوحات S التي لم تكن تتكاثر عند 48 ساعة بعد العزل مرة أخرى عند 168 ساعة بعد العزل.
- إذا كانت الصفيحة S تتكاثر الآن، فأدخل "1" في العمود proliferation_168 على ورقة التنميط الجيني ثم انقل الصفيحة S إلى مربع يحمل علامة "انتشار 168 ساعة، المربع 1".
- ضع 88 لوحة S كحد أقصى في صندوق الانتشار ، ثم ابدأ في ملء صندوق جديد يحمل اسم "انتشار 168 ساعة ، الصندوق 2". تأكد من تنظيم ملصقات S بترتيب أبجدي رقمي في مربعات الانتشار 168 ساعة.
- تخلص من الصفائح S التي ليس لها انتشار بعد 168 ساعة. انتقل إلى القسم 7 للألواح S ذات الانتشار عند 168 ساعة.
7. تحلل خطوط متساوي الإناث
ملاحظة: ستستخدم هذه الخطوة أداة تصفية البيانات في جداول بيانات Google للمساعدة في طباعة أوراق عمل التحلل للوحات S في مربعات الانتشار. الغرض من أوراق عمل التحلل هو تزويد الموظفين بالمواضع الصحيحة لملصقات S في أنابيب شريط التحلل على مقاعد البدلاء.
- افتح ورقة التنميط الجيني للمشروع المطلوب وحدد جميع الخلايا عن طريق كتابة Cmd + A. انقر فوق البيانات > إنشاء عامل تصفية لإضافة زر تصفية إلى كل رأس عمود. استخدم أزرار التصفية لعرض لوحات S التي سيتم تصنيفها وراثيا فقط. على سبيل المثال ، إذا كان سيتم تحليل جميع لوحات S ذات الانتشار عند 48 ساعة: انقر فوق الزر تصفية في عمود "proliferation_48" وحدد "1".
- بمجرد تصفية ورقة google للتنميط الجيني، راجع قائمة ملصقات S المعروضة للتأكد من أنها ملصقات S المراد طباعتها على ورقة العمل.
- في عمود "strip_tube_number" في ورقة Google للتنميط الجيني، أدخل رقما فريدا كل 11 صفا.
- أدخل أرقام أنبوب الشريط لمشروع بترتيب متتالي يبدأ من 1 ولا يتم تكرارها أبدا. في "strip_tube_position"، أدخل من 2 إلى 12 لكل رقم أنبوب شريط.
ملاحظة: استخدم أنابيب الشريط المكونة من 12 أنبوبا للتحلل. سيتم التحكم في الموضع الأول (strip_tube_position 1) ، ولكن لا تتم إضافة عناصر التحكم إلى أوراق عمل التحلل (تتم إضافة strip_tube_positions فقط ، 2-12). في وقت التحلل ، ستتم إضافة سلالة التحكم الإيجابية "N2" إلى الموضع 1 من كل أنبوب شريط زوجي مرقم كعنصر تحكم إيجابي. لا تتم إضافة أي ديدان إلى الموضع 1 من كل أنبوب شريط فردي كعنصر تحكم سلبي.
- قم بتصفية ورقة Google للتنميط الجيني بشكل أكبر لتضمين ملصقات S فقط في مربع انتشار واحد سيتم تحليله ، ثم حدد الأعمدة "s_label" من خلال "lysis_notes". طباعة ورقة عمل تحلل لكل مربع انتشار ليتم تحليلها.
- انقر على القائمة المنسدلة في حقل الطباعة وحدد الخلايا المحددة. انقر فوق التالي في الجزء العلوي الأيسر، ثم استخدم مربع الحوار لطباعة ورقة عمل التحلل لمربع الانتشار.
- كرر الخطوات من 7.3 إلى 7.5 لطباعة ورقة عمل تحلل لكل مربع انتشار.
ملاحظة: يحتوي كل صندوق انتشار على ما يصل إلى 88 صفيحة S، وهو ما يتوافق مع ثمانية أنابيب شريطية من 12 بئرا. - قم بإعداد أنابيب شريط 12 بئرا لجميع العينات التي سيتم تحليلها. قم بتسمية أنبوب شريطي ب "strip_tube_number" فريد تم تعيينه في ورقة عمل التحلل. يجب كتابة هذا الملصق على شريط الغطاء وأنبوب الشريط لتجنب الارتباك إذا تم فصلهما. تحتوي أنابيب الشريط EVEN على تحكم إيجابي (ديدان N2) في الموضع 1. تحتوي أنابيب شريط ODD على تحكم سلبي (بدون ديدان) في الموضع 1.
- قم بتكوين ما يكفي من المخزن المؤقت للتحلل (100 mM KCl ، 20 mM Tris pH 8.2 ، 5 mM MgCl2 ، 0.9٪ IGEBAL ، 0.9٪ Tween 20 ، 0.02٪ الجيلاتين مع إضافة بروتين K إلى تركيز نهائي قدره 0.4 mg / mL) لجميع العينات وإضافة 5٪ إضافية لخطأ الماصة. مقياس حسب الضرورة.
ملاحظة: من الأفضل تحضير المخزن المؤقت للتحلل من خلال الجمع بين جميع المكونات باستثناء البروتين K والتجميد في 10-50 مل من الأليكوتات عند -20 درجة مئوية. إذابة aliquots والحفاظ عليها في 4 درجة مئوية قبل الاستخدام ؛ مباشرة قبل الاستخدام ، أضف بروتين K واخلطه جيدا. احتفظ بمخزن التحلل المؤقت على الجليد أثناء العمل. - رتب لوحات S لأنبوب شريط معين بالترتيب باستخدام ورقة عمل التحلل المطبوعة كدليل.
- قم بفك غطاء أنبوب شريط واحد وأضف 8 ميكرولتر من المخزن المؤقت للتحلل إلى كل غطاء باستخدام ماصة متكررة. أضف المخزن المؤقت للتحلل إلى شريط واحد من الأحرف الكبيرة في كل مرة لأن المخزن المؤقت للتحلل سيتبخر إذا ترك في RT وتم الكشف عنه. اختر 3-5 من لوحات المصدر (لوحة S أو لوحة مخزون N2 للتحكم الإيجابي) في مواضع الغطاء المناسبة الموضحة في ورقة عمل التحلل. سجل الملاحظات لأي لوحة S تحتوي على أقل من 5 ديدان تم اختيارها للتحلل في القسم lysis_notes من ورقة عمل التحلل.
- بعد تحميل الديدان الخيطية في كل موضع من موضع أنبوب الشريط، ضع شريط الغطاء مرة أخرى على أنبوب الشريط. قم بمطابقة الغطاء المميز (الموضع 1) مع الأنبوب المميز (الموضع 1). بمجرد تغطيتها ، قم بالطرد المركزي لأنبوب الشريط لفترة وجيزة حتى تصبح الديدان الخيطية في أسفل الأنبوب.
- ضع الشريط في الفريزر -80 درجة مئوية حتى يتم تجميده تماما (10 دقائق على الأقل). كرر الخطوات من 7.9 إلى 7.11 حتى تحتوي جميع الشرائط على ديدان خيطية مضافة للتحلل. تنظيم شرائط الأنابيب بترتيب رقمي.
- قم بإزالة مجموعات أنابيب الشريط وتشغيل برنامج التحلل في جهاز تدوير حراري: 1 ساعة عند 60 درجة مئوية ، 15 دقيقة عند 95 درجة مئوية ، امسك عند 12 درجة مئوية. عند الانتهاء من برنامج التحلل ، قم بتدوير العينات عند 300 × g لمدة 15 ثانية في RT وقم بتخزين الليزات عند -80 درجة مئوية لمدة تصل إلى أسبوع واحد.
- قم بتنظيم شرائط الأنبوب بترتيب رقمي باستخدام حاملات لوحات 96 بئرا وقم بتضمين ملصق يحتوي على رقم صندوق الانتشار ونطاق رقم أنبوب الشريط والتاريخ والأحرف الأولى من اسم الباحث. قم بتحديث عمودي ورقة التنميط الجيني "lysis_date" و "lysis_notes" بمعلومات من ورقة عمل التحلل.
8. PCR من تسلسلات SSU و ITS2
ملاحظة: سيوفر هذا القسم إرشادات حول كيفية إجراء اثنين من PCR منفصلة لكل لوحة S محللة. تقوم المجموعة التمهيدية الأولى بتضخيم جزء 500-bp من جين الوحدة الفرعية الصغيرة 18S rDNA (SSU) ؛ oECA1271 = التمهيدي الأمامي TACAATGGAAGGCAGCAGGC, oECA1272 = التمهيدي العكسي CCTCTGACTTTCGTTCTTGATTAA 12. يستخدم PCR هذا للتحقق من جودة الحمض النووي للقالب. يعمل PCR على تضخيم منطقة SSU لجميع أنواع الديدان الخيطية تقريبا. إذا فشل SSU PCR في التضخيم ، فإن هذه النتيجة تشير إلى أن جودة التحلل رديئة ويجب تكرار التحلل لهذه اللوحة S. تعمل المجموعة التمهيدية الثانية على تضخيم جزء 2000 نقطة أساس من منطقة الفاصل المنسوخة الداخلية بين جينات rDNA 5.8S و 28S (ITS2) ؛ oECA1687 = التمهيدي الأمامي CTGCGTTACTTACCACGAATTGCARAC, oECA202 = التمهيدي العكسي GCGGTATTTGCTACTACCAYYAMGATCTGC3. منتج ITS2 PCR هو تسلسل Sanger ويستخدم التسلسل لتحديد الديدان الخيطية في جنس Caenorhabditis إلى مستوى الأنواع حسب تشابه التسلسل.
- استخدم أداة التصفية في ورقة التنميط الجيني لعرض ملصقات S فقط لاستخدامها في تفاعل البوليميراز المتسلسل.
- قم بتحديث أعمدة pcr_plate_number pcr_well في ورقة التنميط الجيني. لمنع تدهور مواد التحلل ، يتم تشغيل SSU و ITS2 PCRs في نفس الوقت.
- استخدم نفس pcr_plate_number ل ITS2 و SSU PCRs على الرغم من أن هذه تفاعلات منفصلة في لوحات منفصلة. سيتم تمييزها بعلامات "SSU" أو "ITS2".
- قم بتعيين pcr_plate_number إلى ثمانية أنابيب شريطية أو أقل (أنبوب شريط واحد لكل صف من لوحة PCR ذات 96 بئرا ، مرتبة بترتيب تصاعدي ، على سبيل المثال ، أدنى رقم أنبوب شريط في الأعلى). ثم قم بتعيين pcr_plate_well لكل ملصق S في أنابيب الشريط.
ملاحظة: يتم ترتيب الأنابيب الشريطية بترتيب تصاعدي، مع تعيين أدنى رقم أنبوب شريطي للصف A وأعلى رقم في الصف H. يتم تعيين الموضع 1 من جميع أنابيب الشريط إلى العمود 1. لذلك ، سيتم تعيين أنبوب الشريط رقم 1 ، الموضع 1 إلى لوحة PCR رقم 1 ، البئر A01. - قم بتسمية لوحة (لوحات) PCR 96-well لاستيعاب العينات التي سيتم استخدامها ل PCR. قم بتسمية كل لوحة PCR بالمعلومات التالية: اسم المشروع ونوع PCR ورقم لوحة PCR وتاريخ PCR (على سبيل المثال ، 2020FebruaryAustralia_SSU_1_20200304). أيضا ، قم بتسمية اللوحة بأرقام أنبوب الشريط التي سيتم تحميلها في كل صف.
- قم بإزالة مادة التحلل من الفريزر -80 درجة مئوية وقم بإذابة أنابيب الشريط التي تحتوي على مادة التحلل على الجليد. بينما تذوب مادة التحلل ، قم بإعداد مزيج ITS2 و SSU الرئيسي في أنابيب منفصلة على الجليد. تم العثور على وصفات SSU و ITS2 PCR في الجدول التكميلي 2.
ملاحظة: قم بإعداد 100 تفاعل من المزيج الرئيسي PCR لكل لوحة 96 بئرا للسماح بخطأ السحب. استخدم مخروطي 15 مل أو 50 مل للاحتفاظ بالمزيج الرئيسي إذا كان سيتم استخدام كميات كبيرة. - دوامة المزيج الرئيسي بلطف حتى يتم توزيع طق في جميع أنحاء المزيج. بمجرد خلطها ، قم بخلط 38 ميكرولتر من المزيج الرئيسي إلى الآبار المناسبة لألواح PCR على الجليد. استخدم أحواض V-bottom المعقمة ذات الاستخدام الواحد وماصة متعددة القنوات بسعة 12 بئرا لنقل المزيج الرئيسي إلى ألواح PCR.
- قم بتدوير أنابيب شريط التحلل المذاب لإزالة مادة التحلل من القبعات. قم بإزالة أغطية جميع أنابيب الشريط التي سيتم تحميلها في لوحة PCR الأولى بعناية. استخدم ماصة متعددة القنوات منخفضة الحجم (إما 12 بئرا أو 8 آبار) لإضافة 2 ميكرولتر من الليزات إلى البئر المناسب في لوحة PCR. ماصة بلطف الليزات لأعلى ولأسفل مرة واحدة قبل إزالة 2 ميكرولتر.
ملاحظة: تحقق من النصائح للتأكد من أنها تحتوي على التحلل قبل النقل. تذكر تغيير التلميحات بين الصفوف أو الأعمدة. - قم بتغطية لوحة PCR بورق لاصق PCR واستخدم الأسطوانة لإنشاء ختم محكم. بعد تطبيق الرقاقة ، قم بتدوير لوحات PCR لفترة وجيزة في جهاز طرد مركزي. احتفظ باللوحة على الجليد حتى تصبح جاهزة للتشغيل في جهاز التدوير الحراري.
- قم بتشغيل PCRs باستخدام برنامج الدورة الحرارية المناسب. ارجع إلى الجدول التكميلي 2 للحصول على تفاصيل برامج SSU و ITS2 PCR.
- كرر الخطوات 8.4- 8.8 حتى يتم تشغيل كافة PCRs.
- أثناء تشغيل تفاعلات PCR ، صب هلام الأغاروز 100 مل 1.5٪. كل هلام سوف يحمل عينات أو لوحة PCR واحدة.
- أضف 1.5 غرام من الأغاروز إلى قارورة سعة 500 مل ، ثم أضف 100 مل من 1x TAE buffer (الجدول التكميلي 3) وقم بالدوران للخلط. الميكروويف لإذابة وتبريد الجل.
- بمجرد تبريد المحلول ، أضف 5 ميكرولتر من محلول بروميد الإيثيديوم 10 ملغ / مل واخلطه حتى يمتزج. صب المحلول في صينية صب مع أربعة أمشاط 25 بئرا بحيث يمكن للهلام استيعاب 96 عينة بالإضافة إلى سلم لكل صف في الجل.
ملاحظة: بروميد الإيثيديوم هو طفرة قوية. عند التعامل مع بروميد الإيثيديوم ، استخدم معطفا مختبريا وقفازات مقاومة للمواد الكيميائية ونظارات السلامة الكيميائية.
- قبل الانتهاء من PCR مباشرة ، أضف صبغة تحميل 6x إلى حوض يمكن التخلص منه واستخدم ماصة متعددة القنوات لإضافة 2 ميكرولتر من صبغة تحميل 6x إلى كل بئر من لوحة PCR جديدة 96 بئرا. سيتم استخدام هذه اللوحة لتحميل العينات في الجل. اصنع ما يكفي من هذه اللوحات لاستيعاب جميع العينات.
- عند الانتهاء من PCR ، قم بإزالة لوحات PCR وقم بطردها مركزيا لفترة وجيزة عند 300 × g لمدة 15 ثانية في RT. قم بتخزين لوحات PCR على الجليد حتى يمكن نفاد منتجات PCR على هلام.
- لتشغيل المنتجات على هلام ، استخدم ماصة متعددة القنوات من 12 بئرا لإضافة 5 ميكرولتر من كل عينة إلى البئر المناسب للوحة 96 بئرا تحتوي على 2 ميكرولتر من صبغة تحميل 6x.
- ثم قم بتحميل 6 ميكرولتر من هذا الخليط في كل بئر من هلام تم صبه مؤخرا. قم بتحميل 6 ميكرولتر من 1 كيلو بايت بالإضافة إلى سلم في البئر الأول من كل صف من الجل.
ملاحظة: لملء آبار الجل ، قد يكون من الضروري تخلل الصف A والصف B من لوحة PCR في الصف الأول من الجل. لتجنب الارتباك ، سجل gel_number gel_position في ورقة التنميط الجيني لكل عينة PCR.
- ضع غطاء رقائق جديد على PCR المتبقي في اللوحة (اللوحات) وخزنها عند 4 درجات مئوية. سيتم استخدام منتجات التفاعل هذه للتسلسل في الخطوة 9.
- قم بتشغيل منتجات PCR على الجل على 120 فولت لمدة 20 دقيقة. قم بتصوير الجل والسجل الذي تنتج عنه ملصقات S منتجات ITS2 و / أو SSU PCR في عمودي "pcr_product_its2" و "prc_product_ssu" من ورقة التنميط الجيني. ضع علامة على وجود فرقة مع "1" ؛ ضع علامة "0" بدون فرقة.
9. تحديد الديدان الخيطية مع تسلسل سانجر وتسلسل انفجار
ملاحظة: يوفر هذا القسم تعليمات لتسلسل أمبليونات ITS2 من ملصقات S، ومحاذاة تلك التسلسلات مع قاعدة بيانات المركز الوطني لمعلومات التكنولوجيا الحيوية (NCBI) باستخدام خوارزمية BLAST، وتحليل نتائج BLAST لتحديد الديدان الخيطية على لوحات S.
- لكل عينة إيجابية ITS2 ، استخدم منتج ITS2 PCR المتبقي لتسلسل Sanger باستخدام التمهيدي الأمامي oECA306 (CACTTTCAAGCAACCCGAC). رتب لربط ملفات مخرجات التسلسل بسهولة ب S-label عن طريق تسجيل عمودي "sequencing_plate" و "sequencing_well" لكل علامة S في ورقة التنميط الجيني.
- احصل على ملفات الإخراج .seq لكل تسمية S من النظام الأساسي للتسلسل. ترتيب ملفات .seq لمشروع في دليل واحد مع ملفات .seq لكل دفعة من التسلسل الموجود في الدلائل الفرعية.
- افتح أداة واجهة سطر الأوامر وانتقل إلى الدليل العلوي الذي يحتوي على ملفات .seq عن طريق إدخال الأمر: cd . إذا لم يكن موجودا بالفعل ، فقم بإنشاء FASTA مدمج لجميع ملفات .seq عن طريق إدخال الأمر التالي: ل dir in */; do cd $dir; for file in *.seq; do echo ">"$file; cat $file; done >>.. /all_seqs.fa; مؤتمر نزع السلاح ..; انتهى.
ملاحظة: ستقوم هذه التعليمة البرمجية بإنشاء ملف FASTA مدمج باسم 'all_seqs.fa' من كافة ملفات.seq في دليل المشروع. يمكن استخدام هذا الملف في أداة انفجار النيوكليوتيدات عبر الإنترنت NCBI لمحاذاة تسلسل ITS2 لكل تسمية S بسرعة مع قاعدة بيانات تسلسل NCBI. - في متصفح الويب، انتقل إلى موقع NBCI BLAST على الويب20 وانقر على زر اختيار ملف . حدد ملف all_seqs.fa الذي تم إنشاؤه للتو ، ثم انقر فوق الزر تسلسلات مشابهة إلى حد ما (BLASTn). انقر فوق الزر BLAST في أسفل الصفحة لبدء البحث عن BLAST.
- قم بتحديث ورقة التنميط الجيني بنتائج BLAST لكل تسمية S. استخدم أداة التصفية لتسهيل تحديث ورقة Google للتنميط الجيني. انقر فوق البيانات > إنشاء عامل تصفية لإضافة زر تصفية إلى كل رأس عمود. قم بتصفية عمود sequencing_plate لتحديد لوحات التسلسل التي سيتم تحديثها بنتائج BLAST.
- استخدم القائمة المنسدلة في صفحة نتائج NCBI BLAST للتحقق من النتائج لكل تسلسل من تسلسل S-plate ITS2 (الشكل 6).
- تحقق من عدم وقوع أي انفجارات. معرف التسلسل في القائمة المنسدلة المسبوقة ب * لا يحتوي على أي ضربات انفجار. بالنسبة إلى تسميات S هذه، أدخل "لم يتم التوصل إلى نتيجة <التاريخ الحالي>" في العمود manual_blast_notes من ورقة التنميط الجيني.
- تحقق من وجود أنواع جديدة محتملة من التهاب Caenorhabditis. انقر على الرابط الموجود في أعلى الضربة لتصور المحاذاة (الشكل 6). إذا كانت أعلى ضربة هي (1) نوع Caenorhabditis ، (2) تحتوي المحاذاة على أكثر من خمسة عدم تطابق في وسط التسلسل ، و (3) تغطية الاستعلام أكبر من 50٪ ، تشير هذه النتيجة إلى أن المعزول قد يكون نوعا جديدا من Caenorhabditis (الشكل 7). بالنسبة للوحات S هذه ، فإن أنواع أعلى BLAST تصل إلى عمود "species_id" ، وتدخل 1 في عمود "possible_new_caeno_sp" ، و "Caeno sp جديد محتمل" في عمود "manual_blast_notes" إلى جانب النسبة المئوية للهوية ، (على سبيل المثال ، "هوية Caeno sp. 89٪ الجديدة المحتملة").
- بالنسبة لتسلسلات S-plate التي تنفجر إلى نوع Caenorhabditis ، أدخل الجنس الكامل واسم الأنواع لأعلى ضربة BLAST في عمود "species_id". على سبيل المثال ، "Caenorhabditis elegans".
- بالنسبة لتسلسلات لوحة S التي تنفجر إلى نوع غير Caenorhabditis ، أدخل فقط جنس أعلى ضربة BLAST متبوعا ب "sp" في عمود "species_id". هذا الترميز يعني أن المعزول هو نوع غير معروف داخل الجنس المسمى. على سبيل المثال ، "Oscheius sp.".
ملاحظة: لا يمكن استخدام تسلسل ITS2 لتحديد العزلات بشكل موثوق إلى مستوى الأنواع خارج جنس Caenorhabditis 3,13. - أدخل 1 في عمود "make_strain_name" من ورقة التنميط الجيني إذا كان "species_id" = "Caenorhabditis elegans" أو "Caenorhabditis briggsae" أو "Caenorhabditis tropicalis" أو "possible_new_caeno_sp" = 1.
- قم بتسمية السلالات بأسماء فريدة وفقا لاصطلاحات تسمية Caenorhabditis ، أي تسمية مختبرية فريدة تتكون من 2-3 أحرف كبيرة متبوعة برقم لكل سلالة فريدة 23. أدخل أسماء السلالات في عمود "strain_name".
- بعد تسمية السلالات ، يمكن حفظها بالتجميد باستخدام البروتوكولات المعمول بها 24.
10. معالجة بيانات التجميع باستخدام حزمة easyFulcrum في R
ملاحظة: توضح هذه الخطوة كيفية ربط بيانات التجميع (C-labels) وبيانات عزل الديدان الخيطية (S-labels) معا باستخدام حزمة easyFulcrum R. يحتوي البرنامج على وظائف من شأنها أن تنضم إلى بيانات Fulcrum مع بيانات التنميط الجيني من ورقة التنميط الجيني بحيث يتم تنظيم هويات الأنواع S-label وأسماء السلالات في إطار بيانات واحد.
- إنشاء دليل جديد باسم مشروع المجموعة. ترتيب بنية المجلد داخل الدليل لمطابقة المتطلبات الموضحة في حزمة R easyFulcrum15.
- انتقل إلى موقع Fulcrum على الويب وقم بتسجيل الدخول. قم بتصدير بيانات المشروع الخام من قاعدة بيانات Fulcrum باستخدام أداة تصدير البيانات الخاصة بموقع Fulcrum على الويب على اليسار وحدد خانات الاختيار التالية: المشروع ، تضمين الصور ، تضمين بيانات GPS ، أخذ عينات الحقل ، والعزل.
ملاحظة: سيتم تصدير بيانات Fulcrum للمشروع كخمسة ملفات قيمة (.csv) مفصولة بفواصل. سيتم ضم بيانات المشروع الكاملة معا في إطار بيانات واحد باستخدام حزمة easyFulcrum في R. - نقل الملفات الخمسة .csv المصدرة من Fulcrum إلى دليل المشروع الذي تم إنشاؤه في الخطوة 10.1 كما هو موضح في المقالة القصيرة easyFulcrum vignette21.
- افتح جلسة عمل Rstudio وقم بتثبيت حزمة easyfulcrum في R عن طريق إدخال الأوامر التالية في وحدة تحكم R 'install.packages("devtools")' و 'devtools::install_github("AndersenLab/easyfulcrum")'.
- افتح برنامج نصي R جديدا واتبع الإرشادات الواردة في المقالة القصيرة easyfulcrum لمعالجة بيانات التجميع21.
النتائج
تم استخدام هذا البروتوكول لجمع الديدان الخيطية Caenorhabditis من مواقع متعددة ، بما في ذلك هاواي وكاليفورنيا. يختلف معدل نجاح العزل للديدان الخيطية Caenorhabditis باختلاف موقع التجميع والمناخ وتجربة أخذ العينات وأنواع الركيزة التي تم أخذ عينات منها. وقد استخدم البروتوكول لأخذ عينات واسعة النطاق من جزر هاواي، حيث أجريت تسعة مشاريع لجمع العينات على مدى سنوات ومواسم متعددة. معدلات نجاح العزل لأنواع Caenorhabditis الذاتية متطابقة تقريبا بالنسبة ل C. briggsae (162 من 4,506 عينات ، 3.6٪) و C. elegans (163 من 4,506 عينات ، 3.6٪) ، وأقل بكثير ل C. tropicalis (26 من 4,506 عينات ، 0.58٪)8. يتم إثراء كل نوع من الأنواع الذاتية على ركائز الفاكهة والزهور المتعفنة بالنسبة لفئات الركيزة الأخرى. قم بأخذ عينة من ركائز الفاكهة والزهور المتعفنة إذا كان الباحث يحاول زيادة معدل النجاح إلى أقصى حد بدلا من توصيف تفضيلات الركيزة. ومع ذلك ، يختلف معدل النجاح مع جودة الركيزة المحددة. على سبيل المثال ، بين ركائز الفاكهة والزهور ، من المحتمل ألا تنتج تلك الركائز الجافة جدا أو الرطبة أو الطازجة الديدان الخيطية Caenorhabditis .
تتجلى قابلية بروتوكول الجمع هذا للتوسع من خلال عدد المجموعات التي يمكن لزوج واحد من الباحثين جمعها من البرية. على سبيل المثال ، في أكتوبر من عام 2018 ، تمكن زوج من الباحثين الذين يستخدمون بروتوكول الجمع هذا من جمع ما مجموعه أكثر من 1000 عينة في 7 أيام من مواقع متعددة في جزيرتين من جزر هاواي. قام هذا الفريق الميداني بشحن العينات بين عشية وضحاها إلى المختبر، حيث قام فريق من ثمانية باحثين بعزل أكثر من 2000 ديدان خيطية من العينات عند وصولها. ومن المزايا الرئيسية لهذا البروتوكول أنه يقلل إلى أدنى حد من التكلفة المرتبطة بأخذ العينات في المواقع النائية عن طريق تقليل المعدات والموظفين اللازمين في الميدان. باستخدام هذا البروتوكول ، يمكن لفريق ميداني صغير التركيز على أخذ العينات بينما يمكن لفريق العزل معالجة العينات في مؤسستهم المنزلية باستخدام معدات هشة وثقيلة مثل تشريح المجاهر وألواح الأجار لعزل الديدان الخيطية. وعلاوة على ذلك، فإن تنفيذ تطبيق جمع البيانات المتنقلة يسمح بربط جميع البيانات الميدانية المرتبطة بالعينات مباشرة بالعلامة C، مما يمكن فريق العزل من العمل بشكل مستقل عن الفريق الميداني أثناء معالجة العينات.
يجب على الباحثين الذين يستخدمون بروتوكول الجمع هذا النظر في الجهد المطلوب لعزل الديدان الخيطية قبل مشروع الجمع. خطوات العزل وتحديد الهوية تحد من المعدل ، ويمكن لفريق جمع صغير أن يطغى بسرعة على العوازل بالعينات. وعلاوة على ذلك، فإن حيز المختبر اللازم لمعالجة العديد من المجموعات يمكن أن يتداخل مع البحوث الجارية (الشكل 3). بالإضافة إلى ذلك ، تتطلب بعض الديدان الخيطية المعزولة جهدا إضافيا للنمط الوراثي. على سبيل المثال ، يفشل ما يقرب من 2٪ من العزلات في التضخيم مع مجموعة التمهيدي SSU PCR بعد محاولة التحلل الأولى ويجب إعادة تحليلها للتأكد من أن مادة التحلل مناسبة للتضخيم باستخدام مجموعة التمهيدي ITS2 (الشكل 8). علاوة على ذلك ، فإن ما يقرب من 3٪ من العزلات تفشل في إنتاج تسلسلات عالية الجودة بعد جولة أولية من تسلسل سانجر. بالنسبة لهذه العزلات ، غالبا ما تكون هناك حاجة إلى جولة أخرى من التحلل و ITS2 PCR وتسلسل Sanger ، مما قد يزيد من وقت التسليم لفريق العزل. الأهم من ذلك ، أن هوية التسلسل وحدها ليست دليلا كافيا لتبرير نوع جديد من التهاب Caenorhabditis (الشكل 7). لتبرير رفع العزل بشكل صحيح كنوع جديد من Caenorhabditis ، يجب بذل جهد إضافي لإجراء تجارب التزاوج وإنشاء عينة مطبوعة13. ويفضل أيضا وصف مورفولوجي رسمي للعينة المكتوبة ولكنه غير مطلوب3. وتشير هذه الاعتبارات مجتمعة إلى أن الباحثين الذين يعتمدون بروتوكول الجمع هذا سيستفيدون من الاختبارات التجريبية لخطوات العزل وتحديد الهوية لضمان تخصيص الموارد بشكل صحيح قبل بدء مشروع الجمع. الأهم من ذلك ، حتى مشاريع الجمع الصغيرة يمكن أن تستفيد من هذا البروتوكول لأن العملية قابلة للتكرار بشكل كبير ، ويمكن بسهولة تدقيق البيانات لأغراض مراقبة الجودة عبر مجموعات المختبرات.

الشكل 1: أمثلة الركيزة. (أ) تظهر فاكهة متعفنة مثالية في وسط الصورة (1) ، ولا يمكن التعرف على الثمرة تقريبا. تظهر الفاكهة الأقل تعفنا في مكان قريب ؛ تجنب أخذ عينات من الفواكه الطازجة الساقطة (2). (ب) تظهر زهرة متحللة بشكل مثالي في الأعلى (3). تجنب أخذ عينات من الزهور المتساقطة حديثا (4). (ج) تعتبر فضلات الأوراق الداكنة تحت الطبقة العليا من الأوراق الجافة مثالية عند أخذ العينات لأخذ الديدان الخيطية Caenorhabditis (5). تجنب أخذ عينات من فضلات الأوراق الجافة (6). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: تطبيق الهاتف المحمول لأخذ العينات الميدانية من الديدان الخيطية . (أ) الشاشة الأولية بعد فتح تطبيق أخذ العينات الميدانية الخيطية على جهاز Apple في Fulcrum. يشير السهم الأحمر في أسفل اليسار إلى الزر + المستخدم لإنشاء سجل مجموعة جديد. (ب) مثال على سجل جمع جديد يظهر على جهاز Apple. يشير السهم الأحمر إلى حقل "المشروع" أعلى شاشة سجل المجموعة. تأكد من تحديد المشروع الصحيح عند أخذ العينات في الحقل. سيتم تعيين حقل المشروع افتراضيا إلى آخر مشروع تم استخدامه عند إنشاء سجلات تجميع لاحقة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 3: أكياس التجميع ولوحات التجميع المنظمة قبل طلاء العينات. يوضح هذا الشكل العينات الموجودة في أكياس التجميع التي تحمل علامة C على اليسار. تحتوي كل حقيبة تجميع على لوحة مطابقة بطول 10 سم تحمل علامة C فوقها. على اليمين توجد لوحات تجميع 10 سم تحتوي على عينات من المواد بعد نقلها من أكياس التجميع. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 4: لوحة تجميع (لوحة C) مع عينة منقولة بشكل صحيح. صفيحة C مقاس 10 سم مع فاكهة متحللة موضوعة على حافة العشب البكتيري. يتم إرفاق ملصق C بغطاء اللوحة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
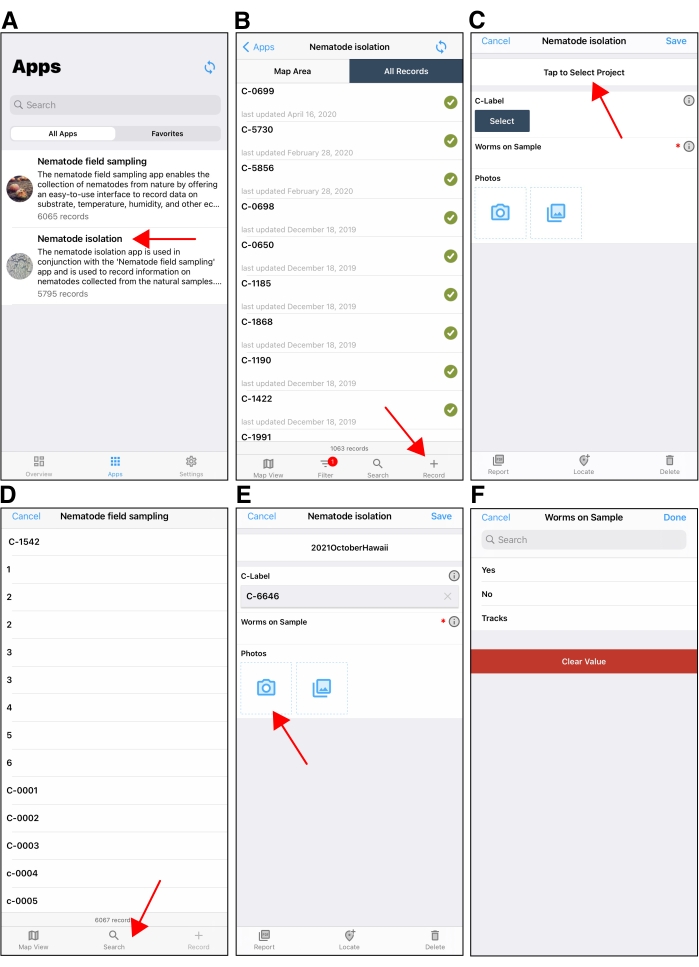
الشكل 5: تطبيق الهاتف المحمول المعزول Nematode . (أ) شاشة اختيار التطبيق في تطبيق Fulcrum للجوال. يشير السهم الأحمر إلى تطبيق عزل الديدان الخيطية . (ب) الشاشة الأولية بعد فتح تطبيق عزل الديدان الخيطية على جهاز Apple في Fulcrum. يشير السهم الأحمر في أسفل اليسار إلى الزر + المستخدم لإنشاء سجل عزل جديد. (C) مثال على سجل عزل جديد يظهر على جهاز Apple. يشير السهم الأحمر إلى حقل "المشروع" أعلى شاشة سجل العزل. تأكد من تحديد المشروع الصحيح عند العزل. سيتم تعيين حقل المشروع افتراضيا إلى آخر مشروع تم استخدامه عند إنشاء سجلات عزل لاحقة. (D) بعد النقر على حقل Select ضمن C-label ، سيضغط المستخدمون على زر البحث (سهم أحمر) للعثور على C-label الذي يعزلون منه الديدان الخيطية. (ه) بعد تحديد الملصق C، سيقوم المستخدمون بتصوير اللوحة C باستخدام كاميرا الجهاز. (و) يقوم المستخدمون بعد ذلك بإدخال ما إذا كانت هناك ديدان خيطية على اللوحة C أم لا. تتم إضافة ملصقات S إلى سجل العزل إذا كانت هناك ديدان خيطية يجب عزلها. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
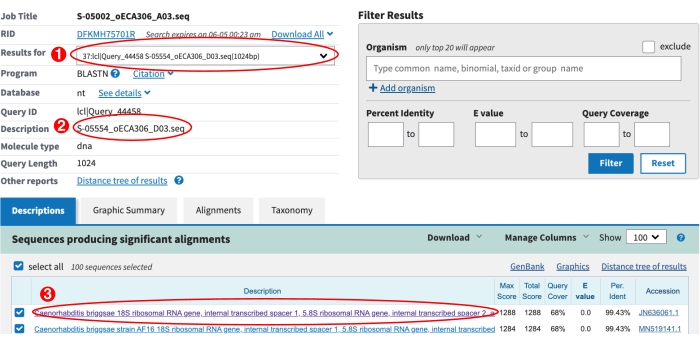
الشكل 6: صفحة نتائج NCBI BLAST (1) القائمة المنسدلة المستخدمة لعرض نتائج BLAST لجميع التسلسلات. (2) وصف التسلسل الحالي المحدد من القائمة المنسدلة. في هذه الحالة يتم عرض نتائج S-label S-05554. (3) يتم عرض أعلى ضربة انفجار ل S-05554. يشير النص الأرجواني إلى أنه تم النقر فوق الرابط الخاص بتصور هذه المحاذاة. يرجى التأكد من فحص المحاذاة بالعين لتحديد أنواع Caenorhabditis الجديدة المحتملة ، انظر الخطوة 9.8 أعلاه. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 7: أمثلة تصور محاذاة انفجار NCBI . (أ) مثال على تسلسل استعلام ITS2 الخاص بالعزل محاذاة لتسلسل موضوع C. kamaaina. (1) النسبة المئوية لهوية المحاذاة (89٪) ، وهي منخفضة بالنسبة لأعلى ضربة BLAST. (2) عدم تطابق بين الاستعلام وتسلسل الموضوع (G إلى A). (3) فجوة زوج من أربعة قواعد في تسلسل الموضوع الذي تم إجراؤه بواسطة خوارزمية المحاذاة ؛ تشير الفجوات في الاستعلام أو الموضوع إلى ضعف المحاذاة. (4) منطقة معممة في وسط المحاذاة مع العديد من حالات عدم التطابق والفجوات. تشير منطقة مثل هذه إلى أن تسلسل الاستعلام قد يأتي من نوع جديد من Caenorhabditis . يظهر مثال محاذاة فعلي لنوع جديد ، C. oiwi ، تم اكتشافه في عام 2017. (ب) مثال على المحاذاة الجيدة بين تسلسل استعلام ITS2 الخاص بالعزل وتسلسل الموضوع. (5) النسبة المئوية لهوية المحاذاة (99٪) ، مما يعني عادة أن تسلسل الاستعلام يأتي من عزل من نفس النوع مثل الموضوع. (6) منطقة مركزية من المواءمة مع الهوية الكاملة. تشير منطقة مثل هذه إلى أن عزل الاستعلام من المحتمل أن يكون نفس النوع مثل الموضوع. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
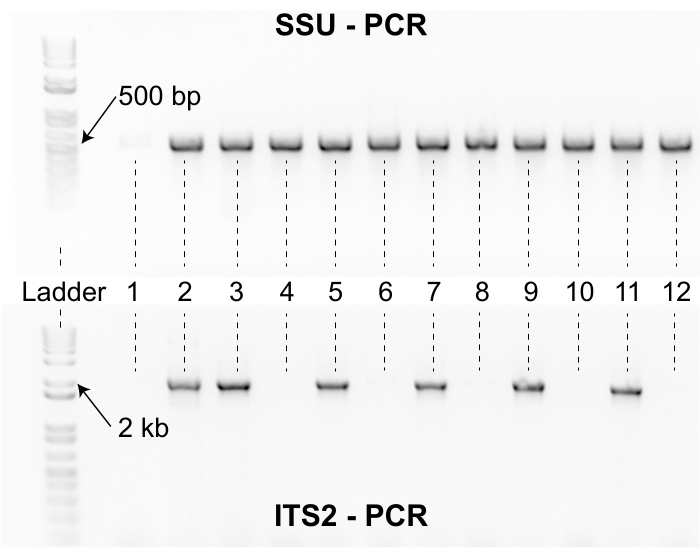
الشكل 8: منتجات SSU و ITS2 PCR. يعرض الجل العلوي منتجات PCR التي تم إنشاؤها باستخدام مجموعة SSU التمهيدية ل 12 عينة تمثيلية. يتم تضمين سلم الحمض النووي على اليسار كمرجع. يبلغ طول منتجات SSU PCR للديدان الخيطية Caenorhabditis حوالي 500 نقطة أساس. تم تضخيم العينات 2-12 باستخدام مجموعة SSU التمهيدية ولكن العينة الأولى لم تفعل ذلك. يشير عدم وجود 500 نقطة أساس SSU amplicon للعينة الأولى إلى أن مادة التحلل كانت ذات نوعية رديئة ويجب إعادة تحليل العينة. يعرض الجل السفلي منتجات تفاعل البوليميراز المتسلسل التي تم إنشاؤها باستخدام مجموعة التمهيدي ITS2 لنفس العينات ال 12 الموضحة في الجل العلوي. السلم والعينات في نفس الاتجاه لكلا الهلام. ستة من العينات ال 12 لم تتضخم مع مجموعة التمهيدي ITS2. العينات ذات نطاقات SSU و ITS2 هي Sanger متسلسلة ومحددة بواسطة تشابه التسلسل باستخدام خوارزمية NCBI BLAST. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الملف التكميلي 1: تسميات C. ملف PDF يحتوي على 2500 تسمية C فريدة. يرجى النقر هنا لتنزيل هذا الملف.
الملف التكميلي 2: تسميات S. ملف PDF يحتوي على 5000 تسمية S فريدة. يرجى النقر هنا لتنزيل هذا الملف.
الجدول التكميلي 1: المواد الميدانية. قائمة التعبئة والمواد المستخدمة في هذا المجال لأخذ عينات من الديدان الخيطية. يرجى النقر هنا لتنزيل هذا الجدول.
الجدول التكميلي 2: وصفات تفاعل البوليميراز المتسلسل وظروف الدورة الحرارية. جدول بوصفات PCR وشروط الدورة الحرارية ل ITS2 و SSU PCRs. يرجى النقر هنا لتنزيل هذا الجدول.
الجدول التكميلي 3: وصفات الرحلان الكهربائي المؤقتة. وصفة لمحلول حمض الإيثيلين ديامينيتراسيتيك 0.5 م درجة الحموضة 8.0 (EDTA) ومحلول المخزن المؤقت TRIS-acetate-EDTA (TAE). يرجى النقر هنا لتنزيل هذا الجدول.
Discussion
يحتوي هذا البروتوكول على خطوات حاسمة يجب تنفيذها بحذر. فعلى سبيل المثال، من المهم أن تحرص الأفرقة الميدانية وفرق العزل على اختيار مشروع التجميع الصحيح في التطبيق قبل جمع العينات من الميدان أو عزل الديدان الخيطية من العينات الموجودة في المختبر. في حالة تحديد مشروع التجميع الخاطئ ، من الأفضل تصحيح سجلات البيانات الخاطئة في قاعدة بيانات Fulcrum باستخدام أدوات تحرير السجلات عبر الإنترنت. يمكن أن تكون هذه العملية مملة للعديد من السجلات في غير محلها. ومع ذلك، تحتفظ قاعدة البيانات بأي تغييرات تطرأ على السجلات بحيث يمكن إجراء تدقيق كامل لسجلات التجميع والعزل. وتشمل الخطوات الحاسمة الأخرى في هذا البروتوكول معالجة العينات من الميدان والديدان الخيطية المعزولة من تلك العينات. لضمان بقاء الديدان الخيطية Caenorhabditis على قيد الحياة في خطوات أخذ العينات والشحن ، يجب الحفاظ على درجة حرارة العينات بين 4 درجات مئوية و 25 درجة مئوية. درجات الحرارة فوق 25 درجة مئوية يمكن أن تسبب العقم في C. elegans14. تأكد من نقل العينات من أكياس التجميع إلى لوحات التجميع في غضون خمسة أيام كلما أمكن ذلك لتقليل الخسارة إلى الديدان الخيطية. بعد عزل الديدان الخيطية ، من الأهمية بمكان أن يتم تنميطها وراثيا وحفظها بالتجميد قبل أن تهلك. من الصعب العثور على الديدان الخيطية الحية على ألواح S التي يزيد عمرها عن أسبوعين إلى ثلاثة أسابيع لأن التلوث الفطري والبكتيري يمكن أن يجعل لوحات S غير مضيافة.
يمكن تعديل هذا البروتوكول بسهولة لاستيعاب أنواع مختلفة من البيانات التي قد يرغب الباحثون في جمعها أثناء وجودهم في الميدان. على سبيل المثال ، من السهل تخصيص تطبيق "أخذ عينات حقل Nematode" باستخدام حقول إدخال بيانات جديدة باستخدام واجهة المستخدم الرسومية عبر الإنترنت من Fulcrum لتحرير التطبيقات. علاوة على ذلك، يمكن لحزمة تحليل البيانات، easyFulcrum، استيعاب هذه التعديلات عند معالجة البيانات الجديدة15. تعديل آخر قد يجده المستخدمون جذابا هو استخدام طريقة مختلفة لأخذ العينات في هذا المجال. بدلا من أخذ عينات من الركائز المنفصلة ، قد يرغب الباحثون في أخذ عينات من مناطق أكبر تحتوي على أنواع متعددة من الركائز. ومن الأفضل معالجة هذه العينات الأكبر حجما في المختبر باستخدام طرق استخراج قمع أو صينية بيرمان13. الأهم من ذلك ، أن استخدام C-labels و S-labels لا يزال ينطبق على هذه التقنيات وبالتالي فهي متوافقة مع تطبيقات الهاتف المحمول.
تتعلق القيود الأساسية لهذا البروتوكول بوقت مناولة الديدان الخيطية قبل عزلها في المختبر. أولا، إن الفارق الزمني بين جمع العينات وعزل الديدان الخيطية يجعل من المستحيل تسجيل مراحل تطور الديدان الخيطية على عينة معينة في وقت جمعها. ثانيا، تواتر الذكور والعبور الخارجي في الطبيعة هي أسئلة تطورية رئيسية لأنانية Caenorhabditis nematodes10. هذه الطريقة ليست مناسبة تماما لمعالجة هذه الأسئلة لأن الديدان الخيطية من المحتمل أن تكون قد مرت عبر أجيال متعددة قبل العزلة. العزلة المتأخرة تعني أن الدليل المباشر على تواتر الذكور في الطبيعة أمر مستحيل. وعلاوة على ذلك، فإن التأخير المتعدد الأجيال أثناء خطوات التنميط الجيني يعني أن الأدلة الجينومية على العبور الخارجي (التغايرية الزيجوتية) سوف تتآكل قبل أن يتسنى تسلسل سلالة الديدان الخيطية. لتحديد تباين الزيجوت في الطبيعة ، يتم استخدام النسل الذي تنتجه الديدان الخيطية المعزولة مباشرة عن الطبيعة للتسلسل2. هناك قيد آخر محتمل لهذا البروتوكول وهو أنه منحاز نحو تحديد التهاب Caenorhabditis. وذلك لأن الديدان الخيطية المعزولة من الأنواع الذاتية لديها فرصة أكبر للتكاثر من العابرين الخارجيين الملزمين ، والتي لن تتكاثر إلا إذا تم عزل الأنثى المخصبة.
تعتمد طريقة التجميع هذه على بروتوكولات التجميع الموجودة13,14. التقدم الرئيسي لهذه التقنية هو استخدام التكنولوجيا المتنقلة والبرامج المخصصة لتسهيل تنظيم كميات هائلة من البيانات البيئية والجزيئية المرتبطة بمشاريع الجمع واسعة النطاق. يمكن استخدام البيانات البيئية الناتجة باستخدام بروتوكول الجمع هذا لمعالجة الأسئلة المعلقة للمجموعات الطبيعية لأنواع Caenorhabditis. على سبيل المثال ، تم استخدام البيانات التي تم إنشاؤها باستخدام هذه الطريقة لاكتشاف التفضيلات المتخصصة للأنواع عبر جزر هاواي. علاوة على ذلك ، من خلال تسلسل جينومات الديدان الخيطية المحفوظة بالتجميد ، يمكن للباحثين التحقيق في كيفية ارتباط أنماط التباين الجيني بالبيانات البيئية. ويمكن لبحوث من هذا النوع أن تكشف عن بصمات التكيف المحلي لدى مجموعات التهاب كاينورهابديتس وأن تقدم رؤى مهمة حول أهمية التباين الجيني في السياقات الطبيعية8. للحصول على فهم وظيفي للعديد من الجينات في الديدان الخيطية Caenorhabditis ، من المحتمل أن تكون الدراسات البيئية مطلوبة11. حتى بالنسبة ل C. elegans ، يفتقر جزء كبير من الجينات إلى التعليقات التوضيحية الوظيفية ، على الرغم من كونه أول متعدد الخلايا يتم تسلسله وأحد أكثر الحيوانات التي تمت دراستها بدقة على الأرض. تم تطوير بروتوكول الجمع هذا للمساعدة في معالجة هذه الفجوة المعرفية من خلال تسهيل جمع الديدان الخيطية Caenorhabditis البرية ودراسة بيئتها وتنوعها الجيني الطبيعي.
Disclosures
ولم يبلغ المؤلفون عن أي تضارب في المصالح.
Acknowledgements
تم دعم هذا البحث من خلال أموال بدء التشغيل من جامعة نورث وسترن وجائزة CAREER من مؤسسة العلوم الوطنية (IOS-1751035) ، وكلاهما يمنح ل E.C.A.
Materials
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
References
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved