Method Article
Selfing caenorhabditis 선충류의 생태 조사를 수행하기위한 고도로 확장 가능한 접근법
요약
이 프로토콜은 자아 Caenorhabditis 선충류의 대규모 생태 조사를 수행하는 데 사용할 수 있습니다. 이 방법의 주요 장점은 자연에서 수집 된 선충류와 관련된 생태 및 분자 데이터의 효율적인 조직 및 분석입니다.
초록
Caenorhabditis elegans 는 생물학의 주요 모델 유기체 중 하나이지만 최근에야 연구자들은 자연 생태학에 중점을 두었습니다. 자연적 맥락에서 C. elegans 에 대한 정보의 상대적 희소성은 자연에서 작은 선충류의 확인과 관련된 도전에서 비롯됩니다. 이러한 도전에도 불구하고 C. elegans 의 생태학에 대한 관심이 높아짐에 따라 실험실 외부의 삶에 관한 풍부한 새로운 정보가 생겨났습니다. 자연에서 C. elegans 에 대한 강화 된 검색은 많은 새로운 Caenorhabditis 종의 발견에 기여했으며 congeneric 선충류가 썩어가는 식물 물질과 관련된 미생물 꽃을 먹는 야생에서 자주 서식한다는 것을 밝혀 냈습니다. 새로운 종의 확인은 또한 수컷과 자기 수정 헤르마프로디테스의 안드로디오시스 교미 시스템이 Caenorhabditis에서 독립적으로 세 번 진화했다는 것을 밝혀 냈습니다. 다른 두 종의 자가종인 C. briggsae 와 C. tropicalis는 C. elegans 의 실험적 이점을 공유하고 자기 수정을 포함한 중요한 특성의 기계론적 기초에 대한 비교 연구를 가능하게 했다. 이러한 발전에도 불구하고,이 중요한 종의 생태와 자연 다양성에 대해 많은 것을 배워야합니다. 예를 들어, 우리는 여전히 많은 유전자에 대한 기능적 정보가 부족하며, 이는 자연 생태학에 대한 이해를 통해서만 얻을 수 있습니다. Caenorhabditis 선충류를 자칭하는 생태 연구를 용이하게하기 위해, 우리는 야생에서 선충류를 수집 할 수있는 고도로 확장 가능한 방법을 개발했습니다. 우리의 방법은 모바일 데이터 수집 플랫폼, 클라우드 기반 데이터베이스 및 R 소프트웨어 환경을 사용하여 야생에서 선충류를 수집하고, 관련 생태 데이터를 기록하고, 분자 바코드를 사용하여 야생 선충류를 식별하는 연구자의 능력을 향상시킵니다.
서문
지난 이십 년 동안 Caenorhabditis 선충류의 생태학에 대한 관심이 높아졌습니다. 이 연구들로부터 우리는 자유 살아있는 Caenorhabditis 종들이 온대 및 열대 지방의 일시적인 미세 서식지에서 분리 될 수 있다는 것을 알고 있으며, 때로는 대칭에서 식물 물질을 분해하는 것과 관련된 미생물 꽃을 먹습니다.1,2,3,4,5,6,7,8 . 우리는 또한 자기 수정의 수렴 진화가 속 (genus )에서 세 번 발생했으며, 자가화는 C. briggsae, C. elegans 및 C. tropicalis9,10의 지배적 인 번식 방식이라는 것을 배웠습니다. 이 셀커 중 C. elegans는 지구상에서 가장 널리 연구 된 동물 중 하나이며 생물학에서 중요한 발전을 이루기 위해 연구원들에 의해 사용되었습니다. 중요하게도, 다른 자가성 Caenorhabditis 종은 많은 C. elegans 실험 이점을 공유하고 있으며 속의 비교 연구를 빠르게 진행하고 있습니다. 그러나 야생에서 이러한 선충류의 비밀스러운 특성으로 인해 생태학과 자연 다양성을 연구하기가 어려워 유전자의 생물학적 기능과 진화가 종 간의 유전 적 다양성을 형성하는 방법을 이해하는 데 중요합니다.10,11.
야생에서 Caenorhabditis 선충류를 자행하는 생태학을 연구하는 데 가장 큰 도전은 작은 크기입니다. 성인 선충류는 종종 길이가 1mm 이하입니다. 이 도전은 연구자가 야생에서 기질을 샘플링하고 야생에서 동물을 관찰 할 수있는 능력없이 실험실의 기질에서 관심있는 선충류를 분리하려고 시도해야합니다. 숙련 된 전문가조차도 현미경으로 다른 자유 살아있는 선충류와 자가하는 Caenorhabditis 선충류를 구별하기가 어렵다는 것을 알기 때문에 선충류는 일반적으로 기질에서 제거되고 격리되어 확립 된 분자 바코드를 사용하여 서열 동일성에 의해 확인되기 전에 증식하도록 방치됩니다.3,12,13,14 . 이러한 방식으로 각 선충류를 처리하는 데 필요한 시간과 노력은 연구자들이 실험실에서 분리 된 각 선충류의 신원을 현장에서 샘플링 된 정확한 기질 및 관련 생태 데이터로 추적 할 수 있어야하기 때문에 조직적 도전을 제시합니다. 여기에서는 현장에서 자가하는 Caenorhabditis 선충류를 효율적으로 수집하고 식별하고이 격리 물을 관련 공간 및 생태 데이터와 대규모로 충실하게 연결하기위한 단계별 프로세스를 설명합니다.
이 수집 방법은 모바일 데이터 수집 플랫폼, 클라우드 기반 데이터베이스 및 R 소프트웨어 환경을 사용하여 생태 조사의 규모와 정확성을 높입니다. Fulcrum은 대부분의 모바일 장치에서 작동하며 사용자가 https://www.fulcrumapp.com(위치 기반 데이터)를 수집하고 구성하는 사용자 지정 응용 프로그램을 빌드할 수 있는 사용자 지정 데이터 수집 플랫폼입니다. 이 프로토콜은 맞춤형 데이터 수집 응용 프로그램을 사용하여 현장에서 공간적으로 명시 적 생태 데이터를 구성하고 해당 데이터를 실험실에서 격리 된 선충류의 신원과 정확하게 연결하는 방법에 대한 자세한 지침을 제공합니다. 이 프로토콜은 또한 확립 된 분자 바코드를 사용하여 자아 Caenorhabditis 선충류를 효율적으로 식별하는 방법을 설명합니다. 이러한 방법의 데이터는 동반 된 R 소프트웨어 패키지 easyFulcrum15를 사용하여 간단하고 재현 가능하게 처리하여 자연 Caenorhabditis 집단의 생태학 및 유전 적 다양성을 탐구 할 수 있습니다.
프로토콜
1. 수집 준비
- Caenorhabditis 선충류를 조사 할 위치를 확인하십시오.
참고 : 대부분의 온대 지역에서 C. elegans 및 C. briggsae는 농경지나 농촌 및 도시 정원과 같은 인간 관련 서식지에서 쉽게 격리 될 수 있습니다1. 아열대 및 열대 지방에서 C. briggsae, C. elegans 및 C. tropicalis는 모두 위에 나열된 인간 관련 서식지에서 발견 될 수 있으며 때로는 서로 매우 가깝습니다. 그러나 C. elegans는 열대 서식지의 다른 종보다 더 시원하고 건조한 서식지를 선호하는 것으로 보입니다7,8. 각 종은 인간과 관련이없는 야생 서식지에서 분리 될 수도 있지만 이러한 서식지는 덜 자주 샘플링됩니다. - Fulcrum 프로젝트를 만들어 모바일 데이터 수집 응용 프로그램을 사용하여 수집 및 격리 데이터를 구성합니다.
- 무료 교육 계약을 사용하여 Fulcrum 온라인 계정 만들기16. 선충류 필드 샘플링 응용 프로그램을 ADD APP 버튼 17을 클릭하여 Fulcrum 계정에 추가하십시오.
- 선충류 격리 응용 프로그램을 ADD APP 버튼 18을 클릭하여 계정에 추가하십시오.
참고: 한 장소로의 각 여행은 명명 규칙 'YearMonthLocation'(예: 2020FebruaryAustralia)을 사용하여 수집 프로젝트로 구성하는 것이 좋습니다.
- 사용자를 Fulcrum 계정에 추가하여 컬렉션 프로젝트에 대한 액세스 권한을 부여합니다. 각 사용자가 Fulcrum 모바일 응용 프로그램을 다운로드하여 프로젝트에 참여하는지 확인합니다.
- QR 코드 레이블 세트를 인쇄하여 모바일 애플리케이션으로 컬렉션(C-레이블) 및 선충류 격리(S-레이블)를 추적합니다. C 라벨을 지퍼 잠금 비닐 봉지에 부착하고 라벨이 붙은 봉지를 25 개의 그룹으로 굴린 다음 포장을위한 고무 밴드로 포장하십시오. 실험실에서 사용할 수 있도록 S-라벨 세트를 보관하십시오.
참고: 이 프로토콜 전체에서 컬렉션(현장의 기판)은 가방이나 접시에 포함되어 있으며 C-라벨로 표시되어 있습니다. 분리 된 선충류는 S 라벨로 표시되어 있습니다. C 라벨은 고유 한 컬렉션을 식별하는 데 사용되며 S 라벨은 고유 한 선충류 분리물을 식별하는 데 사용됩니다. 이 두 가지 유형의 레이블은 Fulcrum 데이터베이스의 특정 컬렉션(C-레이블)과 해당 컬렉션에서 격리된 선충류(S-레이블) 간의 연결을 만드는 데 사용됩니다. 컬렉션 프로젝트의 C 레이블 수의 두 배를 C 레이블로 인쇄하면 평균적으로 컬렉션 당 두 개의 선충류가 격리되어 있기 때문입니다. 필요한 경우 나중에 더 많은 S-레이블을 인쇄할 수 있습니다. 보충 자료에는 2,500개의 고유한 C-레이블(보충 파일 1)과 5,000개의 고유한 S-레이블(보충 파일 2)이 제공됩니다. - 수집을 위해 10cm NGMA 플레이트를 준비하고 선충류를 분리하기 위해 3.5cm NGMA 플레이트를 준비하십시오. 컬렉션 당 하나의 10cm 플레이트와 적어도 두 개의 3.5cm 플레이트를 만드십시오21. 이들 플레이트는 확립된 프로토콜에 따라 에스케리치아 콜라이 균주 OP50으로 시딩된다. 플레이트를 사용하기 전에 4°C에서 1개월 이상 보관하지 마십시오.
2. 필드 수집
참고 : Caenorhabditis 선충류는 과일, 견과류, 씨앗, 꼬투리, 꽃, 줄기, 식물 깔짚 및 퇴비를 포함한 썩어가는 식물성 물질로부터 가장 자주 분리됩니다1,5,6,8. 최고의 기질은 썩어서 과일이나 꽃으로 거의 인식 할 수 없습니다. 너무 건조하거나 젖은 기판은 피하십시오(그림 1). 기판은 쌍으로 작업하여 현장에서 가장 효율적으로 수집됩니다. 비접촉 적외선 온도계를 가진 개인은 수집 할 기판을 선택하고 샘플을 수집하는 반면 파트너는 Fulcrum의 선충류 필드 샘플링 응용 프로그램을 사용하여 수집 데이터를 기록합니다. 한 쌍의 컬렉터는 원하는 수의 샘플이 수집될 때까지 이 과정을 반복합니다. 현장 조사에 필요한 재료 목록은 (보충 표 1)에서 찾을 수 있습니다.
- Fulcrum 모바일 앱을 열고 드롭다운 메뉴에서 선충류 필드 샘플링을 선택합니다. + 키를 눌러 프로젝트에서 새 레코드를 시작합니다(그림 2A). 기판의 사진을 찍으십시오.
- 위쪽 중앙에 있는 상자를 클릭하여 1.2단계에서 만든 올바른 컬렉션 프로젝트를 선택합니다(그림 2B). 컬렉션 레코드 하단의 C-label 필드를 탭하고 프롬프트가 나타나면 스캔을 선택합니다. 모바일 장치 카메라를 사용하여 컬렉션 백의 바코드를 스캔 한 다음 화면 오른쪽 상단의 완료 를 누릅니다.
- 기판 필드를 탭하고 드롭다운 메뉴에서 기판 유형을 선택합니다. 기판 메모 필드를 탭하고 수동으로 메모를 입력하여 기판에 대한 메모를 추가합니다.
- 드롭다운 메뉴에서 풍경을 선택합니다. 샘플링 사이트를 가장 잘 나타내는 풍경을 선택합니다.
- 하늘 보기를 선택합니다. 하늘 보기를 선택할 때, 샘플링 사이트에서의 하늘 가시성을 기술한다(예를 들어, 나무 또는 다른 구조물로부터 방해받지 않는 전체 하늘 보기 = 전체).
- 비접촉 온도계를 사용하여 기판의 표면 온도를 측정하고 기판 온도 필드에 값을 기록하십시오.
주: 온도를 기록하는 동안 비접촉 온도계를 기판에서 14인치 이상 유지하지 마십시오. - 핸드헬드 장치로 주변 온도와 습도를 측정하고 이러한 데이터를 적절한 필드에 기록합니다.
주: 주변 온도 및 습도 장치가 대기 상태에 있지 않은지 확인하십시오. 측정 단위는 버튼을 놓으면 변경됩니다. 불규칙한 판독 값을 피하기 위해 장치를 외부 주머니에 보관하십시오. - 화면 왼쪽 상단의 저장을 탭하여 Fulcrum에 레코드를 저장합니다 .
- 스틱이나 다른 단단한 조각이없는 기질의 큰 스푼을 수집 가방을 뒤집어서 기판을 집어 들기위한 "장갑"으로 사용한 다음 가방을 밀봉하십시오. 샘플이 특히 촉촉한 경우 종이 타월을 가방에 넣으십시오.
참고 : 더운 기후에서는 컬렉션을 시원하게 유지하기 위해 쿨러 팩이있는 부드러운 쿨러에 가방을 넣으십시오. - 하루 동안 모든 샘플을 수집 한 후 수집 장비를 청소하고 프로브에서 배터리를 꺼내고 배터리를 충전하고 냉동고 팩을 다시 고정하십시오. 선충류 필드 샘플링 응용 프로그램의 왼쪽 상단에 있는 동기화 단추를 탭하여 Fulcrum 수집 데이터를 동기화합니다.
참고: 강력한 셀룰러 연결 없이 업로드하는 데 몇 분 정도 걸릴 수 있으므로 WiFi 액세스를 기다리는 것이 가장 좋습니다. 데이터는 모바일 장치에 남아 있으며 클라우드로 동기화됩니다. - 샘플을 하룻밤 사이에 배송 상자에 넣어 가정 기관에 배송하십시오. 화물이 운송되는 날에 포장물을 운송하여 샘플이 11°C 미만 또는 25°C 이상의 온도에 노출되는 시간을 최소화합니다.
참고: 대부분의 운송 시설은 주말에 외딴 곳에서 하룻밤 사이에 화물을 운송하지 않습니다.
3. 실험실에서 현장 수집 도금
참고: 이 섹션에서는 라벨이 붙은 수집 백에서 라벨이 붙은 플레이트로 샘플을 옮기는 방법을 자세히 설명합니다. 샘플은 하룻밤 사이에 선적되거나 현장에서 직접 도착할 수 있습니다.
- 수거품 배송을 받고 파손 된 가방 또는 기타 손상 증거를 검사하십시오. 가방이 파손되면 재료를 버리고 깨지지 않은 수집 가방을 70 % 에탄올로 청소하십시오. 에탄올이 든 가방의 C 라벨은 라벨을 변색시키고 읽기가 어려워지기 때문에 피하십시오.
- 각 지퍼 잠금 백에 대해 가방의 C 라벨을 기록하고 OP50 박테리아가 발견 된 10cm 플레이트의 뚜껑에 일치하는 C 라벨을 부착하십시오.
참고: 라벨이 붙은 10cm 플레이트는 나머지 프로토콜에 대해 'C-플레이트'라고 합니다. 샘플을 구성하는 가장 쉬운 방법은 일치하는 C-플레이트가 있는 실험실 벤치에 수집 백을 놓는 것입니다(그림 3). - 각 컬렉션에 대해 깨끗한 플라스틱 스푼을 사용하여 수집 백에서 C 플레이트로 약 한 스푼의 샘플을 옮깁니다. 초승달 또는 고리 모양으로 박테리아 잔디밭 주위에 샘플을 추가하고 박테리아 잔디를 완전히 덮지 마십시오 (그림 4).
참고 : 큰 스푼을 사용하지 않을 때 95 % 에탄올의 비커에 넣어 깨끗하게 유지하십시오. 추가 샘플을 옮기기 전에 종이 타월을 사용하여 큰 스푼을 말립니다. - 컬렉션이 수집 백에서 C 플레이트로 옮겨진 시간을 기록하고 섹션 4에서 선충류를 분리하기 전에 C 플레이트를 실온 (RT)에 적어도 24 시간 동안 보관하십시오.
4. 컬렉션에서 선충류를 격리
- 모바일 장치에서 Fulcrum 응용 프로그램을 열고 응용 프로그램 메뉴에서 선충류 격리 를 선택합니다(그림 5A). 오른쪽 아래에 있는 + 아이콘을 탭하여 새 격리 레코드를 만듭니다(그림 5B).
- 새 격리 레코드 화면에서 위쪽 중앙의 상자에 표시된 프로젝트 이름을 확인하여 올바른 컬렉션 프로젝트를 확인합니다. 잘못된 프로젝트가 표시되면 프로젝트 이름을 탭하여 올바른 프로젝트로 전환합니다(그림 5C).
- C 레이블 필드 아래의 선택 단추를 탭하여 선충류가 격리된 샘플과 연결된 C 레이블 을 찾습니다(그림 5D). 검색 아이콘을 탭한 다음 스캔 아이콘을 탭하여 장치 카메라로 C 플레이트의 C 라벨 QR 코드를 스캔 합니다. QR 코드를 스캔하면 C 레이블 레코드가 C 레이블 필드에 나타납니다.
- 사진 필드에서 카메라 아이콘을 탭하여 장치 카메라를 열고 QR 코드가 표시된 C-플레이트에서 샘플 사진을 찍는 데 사용합니다(그림 5E). 완료를 탭하여 격리 화면으로 돌아갑니다.
참고: 이러한 격리 레코드 사진은 나중에 기판의 특정 속성을 탐색하는 데 사용할 수 있습니다. - 해부 현미경을 사용하여 C 플레이트에서 선충류를 찾으십시오. 샘플의 웜 필드를 탭하여 샘플에 선충류가 있는지 기록합니다(그림 5F). 선충류가 C 플레이트에 있으면 예를 누르고 선충류가 없으면 아니오 를 누릅니다.
- 선충류 트랙 만 있으면 트랙을 누릅니다. 선충류가 없으면 C 플레이트를 파라 필름으로 만들고 생물학적 위험 쓰레기통에 버리십시오.
참고: C-플레이트를 생물학적 위험 폐기물 통 위로 뒤집고 플레이트 뒷면을 부드럽게 탭하여 샘플링된 모든 기판을 제거합니다. 이 단계를 통해 C 플레이트의 기질 아래에있을 수있는 선충류를 쉽게 찾고 분리 할 수 있습니다. - 선충류가 있으면 C 플레이트에서 최대 다섯 개의 선충류를 분리하십시오. 선충류를 분리하려면 백금 와이어 픽을 사용하여 선충류 하나를 C 플레이트에서 S 플레이트로 옮깁니다. 가능한 경우 건강하고 육즙이 많은 성인을 격리하십시오. 그러나 성인이 발견되지 않으면 다른 단계를 격리하십시오.
참고 : 격리 후 최대 다섯 개의 S 플레이트에는 각각 하나의 선충류가 있습니다. 이 S 플레이트는 Fulcrum에 들어갈 때까지 다른 S 플레이트에서 멀리 떨어진 깔끔한 스택에 함께 조직 된 동일한 C 플레이트의 격리 된 선충류와 함께 보관하십시오. - S -레이블이 지정된 플레이트 필드를 탭하여 이 분리에 사용되는 S-플레이트로 들어갑니다. 오른쪽 하단의 + 를 누릅니다. S 레이블 을 탭한 다음 스캔 을 클릭하여 장치 카메라를 엽니 다. 장치 카메라를 사용하여 S 플레이트의 S 라벨 QR 코드를 스캔합니다.
주: S-라벨 코드가 플레이트의 코드와 일치하는지 확인하십시오. 일치하면 완료를 누릅니다. 그렇지 않은 경우 취소 를 탭하고 일치 할 때까지 다시 검색 한 다음 완료를 클릭하십시오. 때로는 근처 플레이트의 QR 코드가 실수로 스캔됩니다. - 각 S-플레이트를 입력한 후 오른쪽 상단의 저장 버튼으로 항목을 저장합니다 . 항목이 저장되지 않으면 손실됩니다. 오른쪽 하단의 + 를 탭하여 필요한 경우 C 플레이트에서 분리 된 모든 선충류가 입력 될 때까지 S 라벨이 붙은 플레이트를 더 추가하십시오. 격리 레코드에 S-레이블이 지정된 플레이트를 모두 추가한 후 왼쪽 상단의 < 버튼을 탭하여 격리 기록 화면으로 돌아갑니다.
참고: 실수를 해결할 수 없기 때문에 격리 레코드를 취소하려면 왼쪽 상단의 취소 를 클릭하십시오. 이 단계에서는 저장하지 않고 레코드를 삭제할 수 있는지 여부를 묻는 대화 상자가 열립니다. 원하는 경우 예, 폐기를 클릭합니다. - 격리 레코드에 모든 정보가 올바르게 추가되면 오른쪽 상단의 저장 버튼을 누릅니다. 그런 다음 격리 된 선충류로 S 플레이트를 파라 필름으로 만들고 선충류가있는 S 플레이트를 보관하도록 지정된 지역에 따로 보관하십시오.
- C 플레이트를 파라 필름으로 만들고 생물학적 위험 쓰레기통에 버립니다. 동기화 아이콘을 탭하여 모든 데이터를 지렛대에 업로드합니다.
- 모든 S-플레이트를 영숫자 순서로 정렬한 다음 S-플레이트를 골판지 상자에 넣습니다. S-플레이트가 뚜껑 쪽을 아래로 내리고 파라 필름으로 되어 있는지 확인하십시오. 최대 네 개의 S-플레이트를 상자의 한 위치에 쌓고 골판지 상자에 프로젝트 이름, 날짜, 시간 및 고유한 상자 번호로 레이블을 지정합니다.
- 레이블이 지정된 상자를 RT에 저장합니다. 이들 단리물은 필요한 경우 48 h에서 그리고 168 h에서 다시 증식을 검사할 것이다.
5. 지렛대에서 S 플레이트 수출
참고: 이 섹션에서는 격리 프로세스에 사용되는 S-레이블을 Fulcrum 프로젝트 데이터베이스에서 내보내는 방법에 대해 자세히 설명합니다. 이들 S-라벨은 섹션 6-9에서 서열 동일성에 의해 식별되는 동안 증식하는 동위원소 선을 추적하는 데 사용될 것이다.
- Fulcrum 웹 사이트에 로그인하고 선충류 격리 응용 프로그램을 선택하십시오. 화면 왼쪽에서 내보내기 를 클릭합니다. 원하는 프로젝트를 클릭하여 선택하고 선충류 격리 상자를 선택하십시오. 다음을 클릭하여 '.zip' 파일이 포함된 nematode_isolation_s_labeled_plates.csv 파일을 다운로드합니다.
- 'nematode_isolation_s_labeled_plates.csv'파일을 열고 'S-label'열별로 오름차순으로 정렬합니다 (가장 작은 S 레이블이 맨 위에 있음). 모든 S-레이블을 선택하고 스프레드시트에서 복사합니다.
- 웹 브라우저를 사용하여 야생 격리 지노타이핑 템플릿 Google 시트 (wild_isolate_genotyping_template)로 이동하십시오19.
- Genotyping 템플릿 탭을 마우스 오른쪽 버튼으로 클릭 한 다음 새 스프레드 시트로 복사 옵션을 선택하여이 Google 시트의 사본을 만드십시오. 스프레드시트 열기를 선택하여 새 Google 스프레드시트를 봅니다.
- 이 새 시트의 이름을 지지점 프로젝트 이름 뒤에 'wild_isolate_genotyping'(예: '2020FebruaryAustralia_wild_isolate_genotyping')로 지정합니다.
참고: 이 시트는 프로토콜의 나머지 부분에서 '지노타이핑 시트'라고 합니다.
- 'nematode_isolation_s_labeled_plates.csv' 's_label' 열에서 복사한 S-레이블을 's_label'이라는 제목의 지노타이핑 시트 열에 붙여넣습니다. 's_label_repeat_error'열에서 '1'을 확인합니다. 이 열에서 '1' 값은 S-레이블이 지노타이핑 시트의 어딘가에 중복된다는 것을 의미합니다. 중복이 발견되면 앞으로 진행하기 전에 조사하고 수정하십시오.
- 모든 S-레이블에 대한 지노타이핑 시트 'isolation_box_number' 열을 입력합니다.
6. S판의 증식 확인
- 격리 후 48시간 후에 S-플레이트에서 증식하는 동물이 있는지 확인합니다(타이밍을 안내하기 위해 4.11단계의 박스에서 마지막 격리 날짜와 시간을 사용).
참고 : 증식하는 선충류는 S 판의 자손이 특징입니다. - S판이 증식하는 경우 지노타이핑 시트의 proliferation_48 열에 '1'을 입력한 다음 S-플레이트를 '48시간 증식, 상자 1'이라고 표시된 상자로 이동합니다. 증식 상자에 최대 88 개의 S 플레이트를 넣은 다음 '48 시간 증식, 상자 2'라고 표시된 새 상자를 채우기 시작하십시오. S-레이블이 48시간 확산 상자에 영숫자 순서로 구성되어 있는지 확인합니다.
참고: 비확산 S-플레이트는 폐기하지 마십시오. 이 플레이트는 격리 후 168 시간에 다시 점검됩니다. 원하는 경우 이러한 S-플레이트를 숫자 순서로 '48시간 비확산, 상자 X'라고 표시된 상자에 통합하되 새 상자에서 168h 확인이 언제 발생해야 하는지 기록해야 합니다. - 48 h에서 모든 증식하는 S-라벨을 확인한 후, 48 h에서 증식하는 S-플레이트에 대해 섹션 7로 이동하십시오.
- 격리 후 168 h에서 48 시간 후에 다시 증식하지 않은 S- 플레이트를 확인하십시오.
- S판이 현재 증식 중인 경우 지노타이핑 시트의 proliferation_168 열에 '1'을 입력한 다음 S-플레이트를 '168시간 증식, 상자 1'이라고 표시된 상자로 이동합니다.
- 증식 상자에 최대 88 개의 S 플레이트를 넣은 다음 '168 시간 증식, 상자 2'라고 표시된 새 상자를 채우기 시작하십시오. 168시간 확산 상자에 S-레이블을 영숫자 순서로 구성해야 합니다.
- 168 시간 후에 증식이없는 S 플레이트를 버리십시오. 168 h에서 증식하는 S-플레이트의 경우 섹션 7로 이동하십시오.
7. 등암선의 용해
참고: 이 단계에서는 Google 스프레드시트의 데이터 필터 도구를 사용하여 확산 상자에 있는 S-플레이트에 대한 용해 워크시트를 인쇄하는 데 도움을 줍니다. 용해 워크 시트의 목적은 벤치의 용해 스트립 튜브에서 S 라벨에 대한 올바른 위치를 직원에게 제공하는 것입니다.
- 원하는 프로젝트의 지노타이핑 시트를 열고 Cmd+A를 입력하여 모든 셀을 선택합니다. 데이터 > 필터 만들기를 클릭하여 각 열 머리글에 필터 단추를 추가합니다. 필터 버튼을 사용하여 유전자형화될 S-플레이트만 표시합니다. 예를 들어, 48h에서 증식하는 모든 S-플레이트를 용해시킬 경우: 'proliferation_48' 열의 필터 버튼을 클릭하고 '1'을 선택합니다.
- 지노타이핑 Google 시트가 필터링되면 표시된 S-레이블 목록을 검토하여 워크시트에 인쇄할 S-레이블인지 확인합니다.
- 지노타이핑 Google 시트의 'strip_tube_number'열에 11 행마다 고유 한 번호를 입력하십시오.
- 프로젝트의 스트립 튜브 번호를 1부터 시작하여 연속적인 순서로 입력하고 중복되지 않습니다. 'strip_tube_position'에 각 스트립 튜브 번호에 대해 2~12를 입력합니다.
참고: 용해를 위해 12튜브 스트립 튜브를 사용하십시오. 첫 번째 위치 (strip_tube_position 1)는 제어되지만 컨트롤은 용해 워크 시트에 추가되지 않습니다 (strip_tube_positions 만 추가, 2-12). 용해시에, 양성 대조군 균주 'N2'는 양성 대조군으로서 모든 짝수 스트립 튜브의 위치 1에 첨가될 것이다. 모든 홀수 번호 스트립 튜브의 위치 1에 웜이 음성 대조군으로 추가되지 않습니다.
- 지노타이핑 Google 시트를 추가로 필터링하여 용해 될 하나의 증식 상자에 S 레이블 만 포함 한 다음 's_label'열에서 'lysis_notes'열을 선택하십시오. 용해될 각 증식 상자에 대한 용해 워크시트를 인쇄하십시오.
- 인쇄 필드의 드롭다운 메뉴를 클릭하고 선택한 셀을 선택합니다. 오른쪽 상단의 다음을 클릭 한 다음 대화를 사용하여 확산 상자의 용해 워크 시트를 인쇄하십시오.
- 7.3-7.5단계를 반복하여 각 증식 상자에 대한 용해 워크시트를 인쇄합니다.
참고: 각 증식 상자에는 최대 88개의 S-플레이트가 들어 있으며, 이는 8개의 12웰 스트립 튜브에 해당합니다. - 용해 될 모든 샘플에 대해 12 웰 스트립 튜브를 준비하십시오. 스트립 튜브에 용해 워크시트에 지정된 고유한 'strip_tube_number'로 레이블을 지정합니다. 이 라벨은 캡 스트립과 스트립 튜브에 적셔야 분리될 경우 혼동을 피할 수 있습니다. EVEN 스트립 튜브는 위치 1에 포지티브 컨트롤 (N2 웜)을 가지고 있습니다. ODD 스트립 튜브는 위치 1에 음성 제어 (웜 없음)를 가지고 있습니다.
- 모든 샘플에 대해 충분한 용해 완충액 (100 mM KCl, 20 mM 트리스 pH 8.2, 5 mM MgCl2, 0.9 % IGEPAL, 0.9 % 트윈 20, 0.02 % 젤라틴 0.4 mg / mL의 최종 농도로 프로테이나제 K를 첨가)을 만들고 피펫 오류를 위해 5 % 추가로 첨가하십시오. 필요에 따라 크기를 조정합니다.
참고: 용해 완충액은 프로테이나제 K를 제외한 모든 성분을 조합하고 -20°C에서 10-50 mL 분취량으로 동결시킴으로써 제조되는 것이 가장 좋다. 분취량을 해동하고 사용 전에 4°C에서 유지; 사용 직전에 프로테이나제 K를 넣고 완전히 섞는다. 작업하는 동안 용해 완충액을 얼음에 보관하십시오. - 인쇄된 용해 워크시트를 가이드로 사용하기 위해 특정 스트립 튜브에 대한 S-플레이트를 배열합니다.
- 하나의 스트립 튜브의 뚜껑을 풀고 반복 피펫터로 각 캡에 8μL의 용해 버퍼를 첨가하십시오. 용해 버퍼를 한 번에 하나의 캡 스트립에 추가하면 용해 버퍼가 RT에 방치되어 풀리면 증발하기 때문입니다. 소스 플레이트 (양성 대조군의 경우 S-플레이트 또는 N2 스톡 플레이트)에서 3-5 마리의 동물을 용해 워크 시트에 표시된 적절한 캡 위치로 선택하십시오. 용해 워크 시트의 lysis_notes 섹션에서 용해에 선택된 5 개 미만의 웜이있는 S 플레이트에 대한 메모를 기록하십시오.
- 선충류를 스트립 튜브의 각 위치에 적재 한 후 캡 스트립을 스트립 튜브에 다시 놓습니다. 표시된 캡(위치 1)을 표시된 튜브(위치 1)와 일치시킵니다. 일단 뚜껑을 덮으면 선충류가 튜브 바닥에 올 때까지 스트립 튜브를 간단히 원심 분리하십시오.
- 스트립을 완전히 동결될 때까지(적어도 10분) -80°C 냉동고에 놓는다. 모든 스트립에 용해를 위해 선충류가 첨가 될 때까지 7.9 ~ 7.11 단계를 반복하십시오. 튜브 스트립을 숫자 순서로 구성합니다.
- 스트립 튜브 세트를 제거하고 열순환기에서 용해 프로그램을 실행하십시오 : 60 °C에서 1 시간, 95 °C에서 15 분, 12 °C에서 유지. 용해 프로그램이 완료되면, 샘플을 RT에서 15초 동안 300 x g 에서 스핀다운하고, 용해물을 -80°C에서 최대 1주일 동안 저장한다.
- 96웰 플레이트 홀더를 사용하여 튜브 스트립을 숫자 순서로 구성하고 확산 상자 번호, 스트립 튜브 번호 범위, 날짜 및 연구원의 이니셜이 있는 라벨을 포함합니다. 지노타이핑 시트 열 'lysis_date' 및 'lysis_notes'을 용해 워크시트의 정보로 업데이트합니다.
8. SSU 및 ITS2 서열의 PCR
참고: 이 섹션에서는 용해된 각 S-플레이트에 대해 두 개의 개별 PCR을 수행하는 방법에 대한 지침을 제공합니다. 첫 번째 프라이머 세트는 18S rDNA 소형 서브유닛 유전자 (SSU)의 500-bp 단편을 증폭시키고; oECA1271 = 정방향 프라이머 TACAATGGAAGGCAGAGGC, oECA1272 = 역방향 프라이머 CCTCTGACTTTCGTTCTTGATTAA 12. 이 PCR은 주형 DNA의 품질을 확인하는 데 사용됩니다. PCR은 거의 모든 선충류 종에 대한 SSU 영역을 증폭시킨다. SSU PCR이 증폭되지 않으면, 이 결과는 용해 품질이 좋지 않으며 이 S-플레이트에 대해 용해를 반복해야 함을 시사한다. 제2 프라이머 세트는 5.8S 및 28S rDNA 유전자 (ITS2) 사이의 내부 전사된 스페이서 영역의 2,000-bp 단편을 증폭시키고; oECA1687 = 정방향 프라이머 CTGCGTTACTTACCACGAATTGCARAC, oECA202 = 역방향 프라이머 GCGGTATTTGCTACTACCAYYAMGATCTGC3. ITS2 PCR 산물은 생어 서열화되고 서열은 서열 유사성에 의해 종 수준으로 예쁜 선충류를 확인하기 위해 사용된다.
- 지노타이핑 시트의 필터링 도구를 사용하여 PCR에 사용할 S-레이블만 볼 수 있습니다.
- pcr_plate_number을 업데이트하고 지노타이핑 시트에 열을 pcr_well니다. 용해 물질의 분해를 방지하기 위해 SSU 및 ITS2 PCR이 동시에 실행됩니다.
- ITS2 및 SSU PCR에 대해 동일한 pcr_plate_number를 사용하더라도 별도의 플레이트에서 별도의 반응이 발생하더라도 사용하십시오. 그들은 'SSU'또는 'ITS2'레이블로 구별됩니다.
- pcr_plate_number를 여덟 개 이하의 스트립 튜브(96-웰 PCR 플레이트의 행당 하나의 스트립 튜브, 오름차순으로 배열, 예를 들어, 상단의 가장 낮은 스트립 튜브 번호)에 할당한다. 그런 다음 스트립 튜브의 각 S-레이블에 pcr_plate_well을 할당합니다.
참고: 스트립 튜브는 오름차순으로 배열되며, 가장 낮은 스트립 튜브 번호는 행 A에 할당되고 모든 스트립 튜브의 행 H. 위치 1에서 가장 높은 숫자는 열 1에 할당됩니다. 따라서, 스트립 튜브 번호 1, 위치 1은 PCR 플레이트 번호 1, 웰 A01에 할당될 것이다. - 96-웰 PCR 플레이트(들)를 표지하여 PCR에 사용될 샘플을 수용한다. 각 PCR 플레이트에 프로젝트 이름, PCR 유형, PCR 플레이트 번호 및 PCR 날짜(예: 2020FebruaryAustralia_SSU_1_20200304)로 레이블을 지정합니다. 또한 각 행에 로드될 스트립 튜브 번호로 플레이트에 레이블을 지정합니다.
- 용해 물질을 -80°C 냉동고로부터 제거하고, 용해 물질을 함유하는 스트립 튜브를 얼음 상에서 해동시킨다. 용해 물질이 해동되는 동안 ITS2 및 SSU 마스터 믹스를 얼음 위의 별도의 튜브에 준비하십시오. SSU 및 ITS2 PCR 레시피는 보충 표 2에서 찾을 수 있다.
참고: 피펫팅 오류를 허용하기 위해 각 96웰 플레이트에 대해 PCR 마스터 믹스의 100회 반응을 준비하십시오. 대량으로 사용할 경우 15mL 또는 50mL 원뿔형을 사용하여 마스터 믹스를 보관하십시오. - Taq가 믹스 전체에 분포될 때까지 마스터 믹스를 부드럽게 소용돌이치십시오. 일단 혼합되면, 마스터 믹스의 분취량 38 μL를 얼음 상의 PCR 플레이트의 적절한 웰에 혼합한다. 멸균 일회용 v-bottom 트로프와 12웰 다채널 피펫을 사용하여 마스터 믹스를 PCR 플레이트로 전송하십시오.
- 해동된 용해 스트립 튜브를 아래로 회전시켜 캡에서 용해 물질을 제거합니다. 첫 번째 PCR 플레이트에 적재될 모든 스트립 튜브의 뚜껑을 조심스럽게 제거하십시오. 저용량 다중 채널 피펫(12웰 또는 8웰)을 사용하여 2μL의 용해물을 PCR 플레이트의 적절한 웰에 첨가한다. 용해물을 2 μL를 제거하기 전에 한 번 위아래로 부드럽게 피펫한다.
참고 : 이송 전에 용해가 포함되어 있는지 확인하기 위해 팁을 확인하십시오. 행이나 열 사이의 팁을 변경해야 합니다. - PCR 플레이트를 PCR 접착 호일로 덮고 롤러를 사용하여 단단한 밀봉을 만듭니다. 호일을 도포한 후, PCR 플레이트를 원심분리기에서 간단히 스핀다운한다. 열순환기에서 달릴 준비가 될 때까지 접시를 얼음 위에 보관하십시오.
- 적절한 열순환 프로그램으로 PCR을 실행하십시오. SSU 및 ITS2 PCR 프로그램의 세부사항은 보충표 2를 참조한다.
- 모든 PCR이 실행될 때까지 8.4-8.8단계를 반복합니다.
- PCR 반응이 실행되는 동안, 1.5% 아가로스 겔 100 mL를 부었다. 각 젤은 샘플 또는 단일 PCR 플레이트를 보유합니다.
- 1.5 g의 아가로스를 500 mL 플라스크에 첨가한 다음, 100 mL의 1x TAE 완충액 (보충 표 3)을 첨가하고 소용돌이 치며 혼합한다. 마이크로웨이브를 사용하여 젤을 용해시키고 냉각시킨다.
- 용액이 냉각되면 10mg/mL 에티듐 브로마이드 용액 5μL를 넣고 혼합하여 결합합니다. 용액을 네 개의 25웰 빗이 있는 캐스팅 트레이에 붓고 젤이 96개의 샘플과 젤의 각 행에 대한 사다리를 수용할 수 있도록 합니다.
참고 : 에티듐 브로마이드는 강력한 돌연변이 유발 물질입니다. 에티듐 브로마이드를 취급 할 때는 실험실 코트, 내화학성 장갑 및 화학 안전 고글을 사용하십시오.
- PCR이 완료되기 직전에 일회용 트로프에 6x 로딩 염료를 첨가하고 다중 채널 피펫을 사용하여 2μL의 6x 로딩 염료를 새로운 96-웰 PCR 플레이트의 각 웰에 첨가한다. 이 플레이트는 샘플을 겔에 로딩하는데 사용될 것이다. 모든 샘플을 수용 할 수 있도록이 플레이트를 충분히 만드십시오.
- PCR이 완료되면 PCR 플레이트를 제거하고 RT에서 15초 동안 300 x g 에서 간단히 원심분리합니다. PCR 산물이 젤 상에서 소진될 때까지 PCR 플레이트를 얼음 위에 보관하십시오.
- 제품을 겔 상에서 실행하려면, 12-웰 다중 채널 피펫을 사용하여 2 μL의 6x 로딩 염료를 함유하는 96-웰 플레이트의 적절한 웰에 각 샘플의 5 μL를 첨가한다.
- 그런 다음 이 혼합물 6 μL를 최근에 주조된 겔의 각 웰에 로딩한다. 6μL의 1KB 플러스 사다리를 젤의 각 행의 첫 번째 웰에 로딩하십시오.
참고: 젤의 웰을 채우려면 젤의 첫 번째 행에 있는 PCR 플레이트에서 행 A와 행 B를 삽입해야 할 수 있습니다. 혼동을 피하려면 gel_number 및 gel_position을 각 PCR 샘플의 지노타이핑 시트에 기록하십시오.
- 플레이트(들) 내의 나머지 PCR들 상에 새로운 호일 뚜껑을 놓고 4°C에서 저장한다. 이들 반응 생성물은 단계 9에서 시퀀싱에 사용될 것이다.
- PCR 산물을 20분 동안 120V에서 겔 상에서 실행한다. 젤을 이미지화하고 지노타이핑 시트의 'pcr_product_its2' 및 'prc_product_ssu' 열에 어떤 S-라벨이 ITS2 및/또는 SSU PCR 산물을 산출하는지 기록한다. '1'로 밴드의 존재를 표시하십시오. 밴드가 없는 경우 '0'을 표시합니다.
9. Sanger 시퀀싱 및 서열 블라스트를 사용한 선충류 확인
참고: 이 섹션에서는 S-레이블에서 ITS2 앰플리콘을 시퀀싱하고, BLAST 알고리즘을 사용하여 이러한 시퀀스를 NCBI(National Center for Biotechnology Information) 데이터베이스에 정렬하고, BLAST 결과를 구문 분석하여 S-플레이트의 선충류를 식별하는 방법에 대한 지침을 제공합니다.
- ITS2 양성인 각 샘플에 대해, 정방향 프라이머 oECA306 (CACTTTCAAGCAACCCGAC)을 사용하여 생어 시퀀싱을 위해 나머지 ITS2 PCR 산물을 사용하십시오. 지노타이핑 시트에 각 S-레이블의 'sequencing_plate' 및 'sequencing_well' 열을 기록하여 시퀀싱 출력 파일을 S-레이블에 쉽게 연결할 수 있도록 정렬합니다.
- 시퀀싱 플랫폼에서 각 S-레이블에 대한 .seq 출력 파일을 가져옵니다. 프로젝트의 .seq 파일을 하위 디렉토리에 있는 각 시퀀싱 배치에 대해 .seq 파일이 있는 단일 디렉토리에 정렬합니다.
- 명령줄 인터페이스 도구를 열고 cd 명령을 입력하여 .seq 파일이 들어 있는 맨 위 디렉터리로 이동합니다. 아직 존재하지 않는 경우 다음 명령을 입력하여 모든 .seq 파일에 대해 병합 된 FASTA를 만듭니다 : */의 dir에 대해; cd $dir을 수행; * .seq의 파일에 대해; 에코 ">"를 수행$file; 고양이 $file; >> 완료. /all_seqs.fa; cd ..; 완료.
참고: 이 코드는 프로젝트 디렉터리의 모든 .seq 파일에서 'all_seqs.fa'라는 병합된 FASTA 파일을 만듭니다. 이 파일은 NCBI 온라인 뉴클레오티드 BLAST 도구에서 각 S-라벨의 ITS2 서열을 NCBI의 서열 데이터베이스에 신속하게 정렬하는 데 사용할 수 있습니다. - 웹 브라우저에서 NBCI BLAST 웹 사이트20으로 이동하여 파일 선택 버튼을 클릭하십시오. 방금 만든 all_seqs.fa 파일을 선택한 다음 다소 유사한 시퀀스 (BLASTn) 버튼을 클릭하십시오. 페이지 하단의 BLAST 버튼을 클릭하여 BLAST 검색을 시작합니다.
- 지노타이핑 시트를 각 S-레이블에 대한 BLAST 결과로 업데이트합니다. 필터 도구를 사용하여 지노타이핑 Google 시트를 쉽게 업데이트 할 수 있습니다. 데이터 > 필터 만들기를 클릭하여 각 열 머리글에 필터 단추를 추가합니다. sequencing_plate 열을 필터링하여 BLAST 결과로 업데이트할 시퀀싱 플레이트를 선택합니다.
- NCBI BLAST 결과 페이지의 드롭다운 메뉴를 사용하여 각 S-플레이트 ITS2 시퀀스에 대한 결과를 확인합니다(그림 6).
- BLAST 히트가 없는지 확인합니다. *로 접두사가 붙은 드롭다운의 시퀀스 ID에는 폭발 히트가 없습니다. 이러한 S-레이블의 경우 지노타이핑 시트의 < 열에 '적중 > 없음 manual_blast_notes현재 날짜'를 입력합니다.
- 가능한 새로운 Caenorhabditis 종을 확인하십시오. 위쪽 히트에 있는 링크를 클릭하여 정렬을 시각화합니다(그림 6). 상단 히트가 (1) Caenorhabditis 종이고, (2) 정렬이 서열의 중심에 5개 이상의 불일치를 포함하고, (3) 질의 커버리지가 50%를 초과하는 경우, 이 결과는 단리물이 새로운 Caenorhabdis 종 일 수 있음을 시사한다(도 7). 이러한 S-플레이트 진입을 위해, 'species_id' 열에 명중한 상위 블라스트의 종은 'possible_new_caeno_sp 열'에 1을 입력하고, '가능한 새로운 Caeno sp.' 열에 퍼센트 동일성과 함께 'manual_blast_notes' 열에 '가능한 새로운 Caeno sp. 89% 동일성'을 입력합니다.
- Caenorhabditis 종에 BLAST하는 S-플레이트 서열의 경우, 'species_id'열에 히트 된 상단 BLAST의 전체 속 및 종 이름을 입력하십시오. 예를 들어, 'Caenorhabditis elegans'.
- 비 Caenorhabditis 종에 BLAST 하는 S-플레이트 서열의 경우, 'species_id'열에 'sp'가 뒤따르는 상위 BLAST 히트의 속만 입력하십시오. 이 표기법은 격리물이 명명 된 속 내에서 알려지지 않은 종임을 의미합니다. 예를 들어, ' Oscheius sp.'.
참고: ITS2 서열은 Caenorhabditis 속3,13 외부의 종 수준으로의 분리물을 확실하게 식별하는데 사용될 수 없다. - 지노타이핑 시트의 'make_strain_name 열'에 'species_id' = 'Caenorhabditis elegans', 'Caenorhabditis briggsae' 또는 'Caenorhabditis tropicalis', OR 'possible_new_caeno_sp' = 1인 경우 1을 입력합니다.
- Caenorhabditis 명명법 규칙에 따라 고유 한 이름으로 균주의 이름을 지정하십시오 (즉, 2-3 개의 대문자로 구성된 고유 한 실험실 지정과 각 고유 한 균주에 대한 숫자로 구성된 23). 'strain_name' 열에 변형 이름을 입력합니다.
- 균주가 명명된 후에, 이들은 확립된 프로토콜 (24)을 사용하여 동결보존될 수 있다.
10. R의 easyFulcrum 패키지로 수집 데이터 처리
참고: 이 단계에서는 easyFulcrum R 패키지를 사용하여 수집 데이터(C-레이블)와 선충류 격리 데이터(S-레이블)를 함께 연결하는 방법을 설명합니다. 이 소프트웨어에는 Fulcrum 데이터를 지노타이핑 시트의 지노타이핑 데이터와 추가로 조인하여 S-라벨 종 ID 및 균주 이름이 단일 데이터 프레임으로 구성되도록 하는 기능이 포함되어 있습니다.
- 컬렉션 프로젝트에 대해 이름이 지정된 새 디렉터리를 만듭니다. R 패키지 easyFulcrum15에 설명된 요구 사항과 일치하도록 디렉터리 내의 폴더 구조를 정렬합니다.
- Fulcrum 웹 사이트로 이동하여 로그인합니다. 왼쪽에 있는 Fulcrum 웹 사이트의 데이터 내보내기 도구를 사용하여 Fulcrum 데이터베이스에서 원시 프로젝트 데이터를 내보내고 프로젝트, 사진 포함, GPS 데이터 포함, 필드 샘플링 및 격리 확인란을 선택합니다.
참고: 프로젝트의 Fulcrum 데이터는 다섯 개의 쉼표로 구분된 값(.csv) 파일로 내보내집니다. 전체 프로젝트 데이터는 R의 easyFulcrum 패키지를 사용하여 단일 데이터 프레임으로 결합됩니다. - Fulcrum에서 내보낸 다섯 개의 .csv 파일을 easyFulcrum vignette21의 지시에 따라 10.1단계에서 만든 프로젝트 디렉토리로 이동합니다.
- Rstudio 세션을 열고 R 콘솔 'install.packages("devtools")' 및 'devtools::install_github("AndersenLab/easyfulcrum")'에 다음 명령을 입력하여 R에 easyfulcrum 패키지를 설치합니다.
- 새 R 스크립트를 열고 easyfulcrum 비네트의 지시에 따라 수집 데이터를 처리합니다21.
결과
이 프로토콜은 하와이와 캘리포니아를 포함한 여러 지역에서 Caenorhabditis 선충류를 수집하는 데 사용되었습니다. Caenorhabditis 선충류의 분리 성공률은 수집 위치, 기후, 샘플링 경험 및 샘플링 된 기질 유형에 따라 다릅니다. 이 프로토콜은 하와이 제도를 광범위하게 샘플링하는 데 사용되어 왔으며, 아홉 개의 수집 프로젝트가 수년 및 계절에 걸쳐 수행되었습니다. Caenorhabditis 종을 자가하는 격리 성공률은 C. briggsae (4,506 샘플 중 162 개, 3.6 %)와 C. elegans (4,506 개 샘플 중 163 개, 3.6 %)와 거의 동일하며 C. tropicalis (4,506 개 샘플 중 26 개, 0.58 %)8의 경우 훨씬 낮습니다. 각각의 자아 종은 다른 기질 범주에 비해 썩어가는 과일과 꽃 기질에 풍부합니다. 시료 썩은 과일과 꽃 기질 연구자가 기질 선호도를 특성화하기보다는 성공률을 극대화하려고 시도하는 경우. 그러나, 성공률은 선택된 기판의 품질에 따라 달라진다. 예를 들어, 과일과 꽃 기질 중에서 너무 건조하거나 젖거나 신선한 기질 은 Caenorhabditis 선충류를 생성하지 못할 가능성이 큽니다.
이 수집 프로토콜의 확장성은 한 쌍의 연구원이 야생에서 수집 할 수있는 컬렉션 수에서 분명합니다. 예를 들어, 2018년 10월, 이 수집 프로토콜을 사용하는 한 쌍의 연구원은 하와이 제도 두 곳의 여러 위치에서 7일 동안 총 1,000개 이상의 샘플을 수집할 수 있었습니다. 이 현장 팀은 하룻밤 사이에 샘플을 실험실로 운송했으며, 여덟 명의 연구원 팀이 샘플에서 2,000 개 이상의 선충류를 분리했습니다. 이 프로토콜의 주요 장점은 현장에서 필요한 장비 및 인력을 줄임으로써 원격 위치에서의 샘플링과 관련된 비용을 최소화한다는 것입니다. 이 프로토콜을 사용하면 소규모 현장 팀이 샘플링에 집중할 수 있으며 격리 팀은 선충류를 분리하기 위해 현미경 및 한천 플레이트를 해부하는 것과 같은 깨지기 쉽고 무거운 장비를 사용하여 가정 기관에서 샘플을 처리 할 수 있습니다. 또한 모바일 데이터 수집 응용 프로그램의 구현을 통해 샘플과 관련된 모든 필드 데이터를 C 레이블에 직접 연결할 수 있으므로 격리 팀이 샘플을 처리하는 동안 현장 팀과 독립적으로 작업 할 수 있습니다.
이 수집 프로토콜을 사용하는 연구원은 수집 프로젝트 전에 선충류를 분리하는 데 필요한 노력을 고려해야합니다. 격리 및 식별 단계는 속도 제한이 있으며 소규모 수집 팀은 샘플로 절연기를 빠르게 압도 할 수 있습니다. 또한 많은 컬렉션을 처리하는 데 필요한 실험실 공간은 진행중인 연구를 방해 할 수 있습니다 (그림 3). 또한, 일부 고립 된 선충류는 유전자형에 대한 추가 노력이 필요합니다. 예를 들어, 분리물의 약 2%는 첫 번째 용해 시도 후에 SSU PCR 프라이머 세트로 증폭되지 않으며, 용해 물질이 ITS2 프라이머 세트를 사용한 증폭에 적합한지 확인하기 위해 재용해되어야 한다(그림 8). 또한, 분리물의 약 3%는 Sanger 시퀀싱의 초기 라운드 후에 품질 서열을 생성하지 못합니다. 이러한 격리물의 경우, 또 다른 용해 라운드, ITS2 PCR 및 생어 시퀀싱이 종종 필요하며, 이는 격리 팀의 핸드 타임을 증가시킬 수 있습니다. 중요하게도, 서열 동일성만으로는 새로운 Caenorhabditis 종을 정당화하기에 충분한 증거가 아니다(그림 7). 분리물을 새로운 Caenorhabditis 종으로 올리는 것을 적절하게 정당화하기 위해서는 짝짓기 실험을 수행하고 형식화 된 표본을 확립하기 위해 추가적인 노력이 이루어져야합니다13. 타이핑된 표본에 대한 공식적인 형태학적 설명도 바람직하지만 필수는 아니다3. 함께, 이러한 고려 사항은이 수집 프로토콜을 채택 한 연구원이 수집 프로젝트가 시작되기 전에 자원이 적절하게 할당되었는지 확인하기 위해 격리 및 식별 단계에 대한 시험 테스트의 이점을 누릴 수 있음을 시사합니다. 중요한 것은 소규모 수집 프로젝트조차도 프로세스가 재현성이 뛰어나고 실험실 그룹 전체에서 품질 관리 목적으로 데이터를 쉽게 감사 할 수 있기 때문에이 프로토콜의 이점을 누릴 수 있다는 것입니다.

그림 1 : 기질 예. (A) 이상적인 썩는 과일은 이미지 (1)의 중앙에 나타나 있으며, 열매는 거의 인식 할 수 없습니다. 덜 썩은 과일이 근처에 표시됩니다. 갓 떨어진 과일을 샘플링하지 마십시오 (2). (B) 이상적으로 분해 된 꽃이 상단 (3)에 표시됩니다. 갓 떨어진 꽃을 샘플링하지 마십시오 (4). (C) 마른 잎의 최상층 아래의 어두운 잎 깔짚은 Caenorhabditis 선충류를 자칭하기 위해 샘플링 할 때 이상적입니다 (5). 마른 잎 깔짚을 샘플링하지 마십시오 (6). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 선충류 필드 샘플링 모바일 응용 프로그램. (A) Fulcrum의 Apple 장치에서 선충류 필드 샘플링 응용 프로그램을 연 후의 초기 화면입니다. 오른쪽 하단의 빨간색 화살표는 새 컬렉션 레코드를 만드는 데 사용되는 + 단추를 가리킵니다. (B) Apple 장치에 표시된 새로운 컬렉션 레코드의 예. 빨간색 화살표는 컬렉션 레코드 화면 상단의 '프로젝트' 필드를 가리킵니다. 현장에서 샘플링할 때 올바른 프로젝트를 선택해야 합니다. 프로젝트 필드는 기본적으로 후속 컬렉션 레코드를 만들 때 사용된 마지막 프로젝트로 설정됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: 샘플을 도금하기 전에 정리된 수집 가방 및 수집 플레이트. 이 그림은 왼쪽의 C 라벨이 붙은 수집 백의 샘플을 보여줍니다. 각 수집 가방에는 그 위에 일치하는 C 라벨이 붙은 10cm 플레이트가 있습니다. 오른쪽에는 수집 가방에서 옮겨진 후 샘플 재료가 들어있는 10cm 수집 플레이트가 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: 적절하게 이송된 샘플이 있는 수집 플레이트(C-플레이트). 박테리아 잔디밭의 가장자리에 놓인 분해 과일이있는 10cm C 플레이트. C 라벨은 플레이트 뚜껑에 부착되어 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
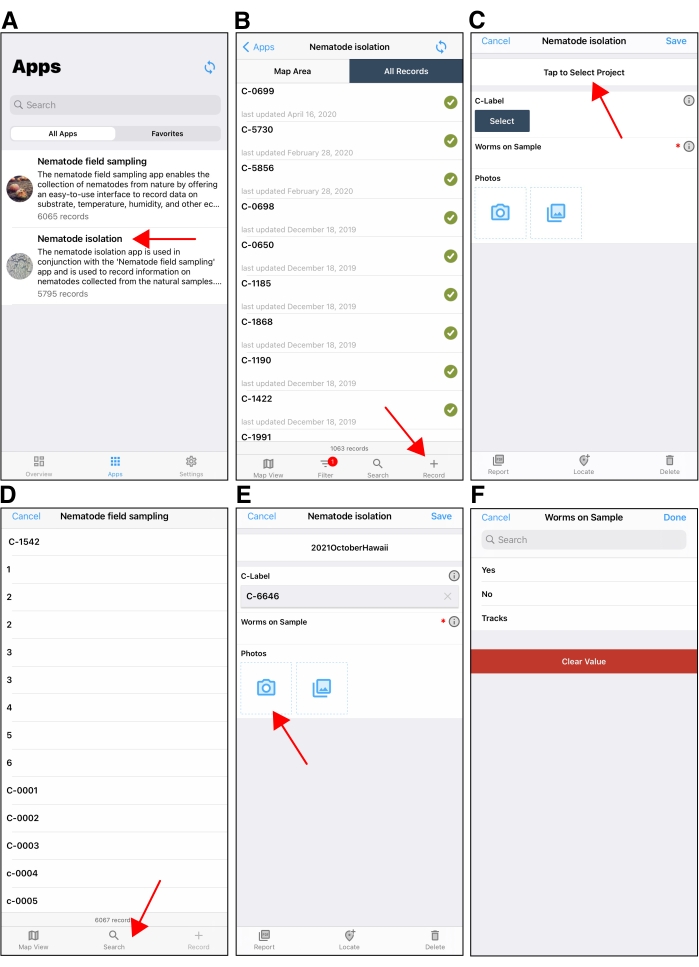
그림 5: 선충류 격리 모바일 응용 프로그램. (A) Fulcrum 모바일 응용 프로그램의 응용 프로그램 선택 화면입니다. 빨간색 화살표는 선충류 격리 응용 프로그램을 가리 킵니다. (B) Fulcrum의 Apple 장치에서 선충류 격리 응용 프로그램을 연 후 초기 화면. 오른쪽 아래에 있는 빨간색 화살표는 새 격리 레코드를 만드는 데 사용되는 + 단추를 가리킵니다. (C) Apple 장치에 표시된 새로운 격리 레코드의 예. 빨간색 화살표는 격리 기록 화면 상단의 '프로젝트' 필드를 가리킵니다. 격리할 때 올바른 프로젝트를 선택해야 합니다. 프로젝트 필드는 기본적으로 후속 격리 레코드를 만들 때 사용된 마지막 프로젝트로 설정됩니다. (D) C-라벨 아래의 선택 필드를 탭한 후 사용자는 검색 버튼(빨간색 화살표)을 탭하여 선충류를 격리하는 C 라벨을 찾습니다. (E) C 라벨을 선택한 후 사용자는 장치 카메라를 사용하여 C 플레이트를 촬영합니다. (F) 사용자는 C 플레이트에 선충류가 있는지 여부를 입력합니다. S-레이블은 격리할 선충류가 있는 경우 격리 레코드에 추가됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
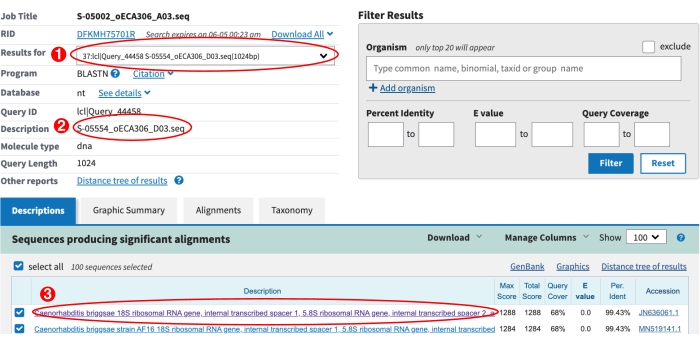
그림 6: NCBI BLAST 결과 페이지 . (1) 모든 시퀀스의 BLAST 결과를 보는 데 사용되는 드롭다운 메뉴입니다. (2) 드롭 다운에서 선택한 현재 시퀀스에 대한 설명. 이 경우 S-label S-05554에 대한 결과가 표시됩니다. (3) S-05554의 최고 블래스트 히트가 표시됩니다. 자주색 텍스트는 이 정렬을 시각화하는 링크가 클릭되었음을 나타냅니다. 가능한 새로운 Caenorhabditis 종을 확인하기 위해 눈으로 정렬을 검사하십시오, 위의 9.8 단계를 참조하십시오. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 7: NCBI BLAST 정렬 시각화 예. (A) C. kamaaina 대상 시퀀스에 정렬된 격리물의 ITS2 쿼리 시퀀스의 예. (1) 정렬의 동일성 비율 (89 %)으로, 상위 BLAST 히트에 대해 낮습니다. (2) 질의와 주제 서열 (G 내지 A) 사이의 불일치. (3) 정렬 알고리즘에 의해 만들어진 피사체 서열에서 네 개의 염기쌍 갭; 쿼리 또는 주제의 간격은 정렬이 불량함을 나타냅니다. (4) 많은 불일치와 틈새가있는 정렬의 중심에있는 일반화 된 영역. 이와 같은 영역은 쿼리 서열이 새로운 Caenorhabditis 종에서 나올 수 있음을 시사합니다. 표시된 것은 2017 년에 발견 된 새로운 종 인 C. oiwi의 실제 정렬 예입니다. (B) 격리물의 ITS2 질의 서열과 피사체 서열 사이의 양호한 정렬의 예. (5) 정렬의 퍼센트 동일성 (99 %)은 일반적으로 쿼리 시퀀스가 피험자와 동일한 종의 분리에서 비롯된 것임을 의미합니다. (6) 완벽한 정체성을 가진 정렬의 중심 영역. 이와 같은 영역은 쿼리 격리가 주제와 동일한 종일 가능성이 있음을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
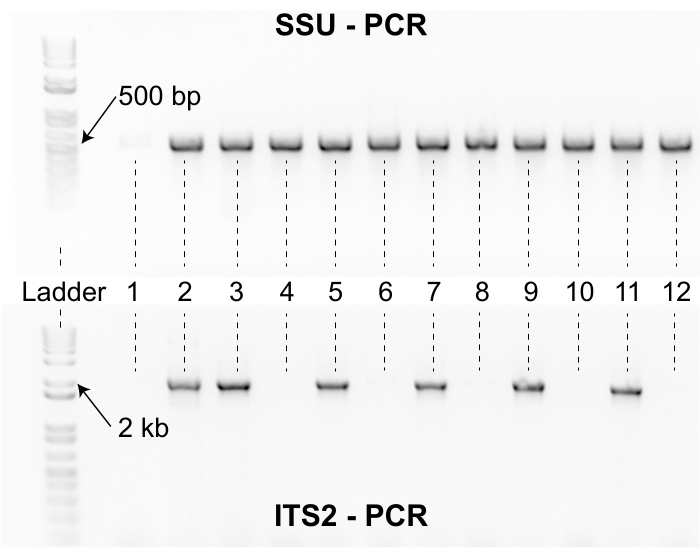
그림 8: SSU 및 ITS2 PCR 산물. 상부 겔은 12개의 대표적인 샘플에 대해 SSU 프라이머 세트로 생성된 PCR 산물을 보여준다. DNA 사다리는 참조로 왼쪽에 포함되어 있습니다. Caenorhabditis 선충류에 대한 SSU PCR 산물은 길이가 약 500 bp이다. 샘플 2-12는 SSU 프라이머 세트로 증폭되었으나 샘플 하나는 증폭되지 않았다. 샘플에 대한 500 bp SSU 앰플리콘의 부재는 용해 물질이 품질이 좋지 않았고 샘플을 재용해해야한다는 것을 암시한다. 하단 겔은 상부 겔에 표시된 동일한 12개의 샘플에 대해 ITS2 프라이머 세트로 생성된 PCR 산물을 나타낸다. 사다리와 샘플은 두 겔 모두에 대해 동일한 방향입니다. 12개의 샘플 중 여섯 개는 ITS2 프라이머 세트로 증폭되지 않았다. SSU 및 ITS2 밴드를 갖는 샘플은 생어 시퀀싱되고 NCBI BLAST 알고리즘을 사용하여 서열 유사성에 의해 식별된다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1 : C 레이블. 2500개의 고유한 C-레이블이 포함된 PDF 파일입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2: S-레이블. 5000개의 고유한 S-레이블이 포함된 PDF 파일입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 1 : 현장 자료. 선충류를 샘플링하기 위해 현장에서 사용되는 재료의 포장 목록. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
보충 표 2: PCR 조리법 및 열순환기 조건. ITS2 및 SSU PCR에 대한 PCR 레시피 및 열주기 조건 표입니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 표 3 : 전기 영동 버퍼 조리법. 0.5 M pH 8.0 에틸렌디아민테트라아세트산 용액(EDTA) 및 TRIS-아세테이트-EDTA(TAE) 완충용액에 대한 레시피이다. 이 테이블을 다운로드하려면 여기를 클릭하십시오.
토론
이 프로토콜에는 주의해서 실행해야 하는 중요한 단계가 포함되어 있습니다. 예를 들어, 현장 및 격리 팀은 현장에서 샘플을 수집하거나 실험실의 샘플에서 선충류를 분리하기 전에 응용 프로그램에서 올바른 수집 프로젝트를 신중하게 선택하는 것이 중요합니다. 잘못된 컬렉션 프로젝트를 선택한 경우 잘못된 데이터 레코드는 온라인 레코드 편집 도구를 사용하여 Fulcrum 데이터베이스에서 가장 잘 수정됩니다. 이 프로세스는 잘못 배치 된 많은 레코드에 지루할 수 있습니다. 그러나 데이터베이스는 레코드에 대한 변경 사항을 유지하므로 수집 및 격리 레코드에 대한 완전한 감사가 가능합니다. 이 프로토콜의 다른 중요한 단계는 현장에서 샘플을 처리하고 해당 샘플에서 분리 된 선충류를 처리하는 것입니다. Caenorhabditis 선충류가 샘플링 및 선적 단계에서 생존하도록하려면 샘플의 온도를 4 °C와 25 ° C 사이에서 유지해야합니다. 25 °C 이상의 온도는 C. elegans14에서 불임을 일으킬 수 있습니다. 선충류에 대한 손실을 최소화하기 위해 가능한 한 오일 이내에 샘플을 수집 가방에서 수집 접시로 옮기십시오. 선충류가 격리 된 후에는 멸망하기 전에 유전자형화되고 냉동 보존되는 것이 중요합니다. 곰팡이와 박테리아 오염으로 인해 S-플레이트가 불쾌해질 수 있기 때문에 두 주에서 세 주 이상 된 S 플레이트에서 살아있는 선충류를 찾기가 어렵습니다.
이 프로토콜은 연구자가 현장에서 수집하기를 원할 수있는 다양한 유형의 데이터를 수용하기 위해 쉽게 수정할 수 있습니다. 예를 들어, 응용 프로그램 편집을 위해 Fulcrum의 온라인 GUI를 사용하여 새로운 데이터 입력 필드로 'Nematode 필드 샘플링'응용 프로그램을 쉽게 사용자 정의 할 수 있습니다. 또한, 데이터 분석 패키지인 easyFulcrum은 새로운 데이터를 처리할 때 이러한 편집을 수용할 수 있습니다15. 사용자가 매력적으로 생각할 수 있는 또 다른 수정은 현장에서 다른 샘플링 방법을 사용하는 것이다. 연구자들은 개별 기판을 샘플링하는 대신 여러 기판 유형을 포함하는 더 큰 영역을 샘플링하기를 원할 수 있습니다. 이 더 큰 샘플은 Baermann 깔때기 또는 트레이 추출 방법을 사용하여 실험실에서 가장 잘 처리됩니다13. 중요한 것은 C 레이블 및 S- 레이블의 사용이 여전히 이러한 기술에 적용되므로 모바일 응용 프로그램과 호환된다는 것입니다.
이 프로토콜의 주요 한계는 실험실에서 격리되기 전에 선충류의 처리 시간과 관련이 있습니다. 첫째, 샘플 수집과 선충류 분리 사이의 지연 시간은 수집 시점에 주어진 샘플에 선충류의 발달 단계를 기록하는 것을 불가능하게 만듭니다. 둘째, 남성의 빈도와 자연에서의 교차는 Caenorhabditis 선충류를 자아 짓기위한 주요 진화론 적 질문입니다10. 이 방법은 선충류가 격리되기 전에 여러 세대를 거쳤을 가능성이 높기 때문에 이러한 질문을 해결하는 데 적합하지 않습니다. 지연된 격리는 본질적으로 남성 빈도에 대한 직접적인 증거가 불가능하다는 것을 의미합니다. 더욱이, 유전자형 형성 단계 동안의 다세대 지연은 선충류 균주가 서열화되기 전에 아웃크로스(heterozygosity)의 게놈 증거가 침식될 것임을 의미한다. 자연에서 이형접합체를 확인하기 위해, 자연으로부터 직접 분리된 선충류에 의해 생성된 자손이 시퀀싱2에 사용된다. 이 프로토콜의 또 다른 잠재적 한계는 그것이 자아 Caenorhabditis의 확인에 편향되어 있다는 것입니다. 이것은 자아 종의 고립 된 선충류가 수정 된 여성이 격리 된 경우에만 증식 할 수있는 의무적 인 아웃 크로스 (outcrossers)보다 증식 할 가능성이 더 높기 때문입니다.
이 수집 방법은 기존의 수집 프로토콜13,14를 기반으로 한다. 이 기술의 주요 발전은 대규모 수집 프로젝트와 관련된 방대한 양의 생태 및 분자 데이터를 쉽게 구성 할 수 있도록 모바일 기술 및 맞춤형 소프트웨어를 사용하는 것입니다. 이 수집 프로토콜을 사용하여 생성 된 생태 데이터는 Caenorhabditis 종의 자연 개체군에 대한 뛰어난 질문을 해결하는 데 사용할 수 있습니다. 예를 들어,이 방법으로 생성 된 데이터는 하와이 제도 전역의 종에 대한 틈새 선호도를 발견하는 데 사용되었습니다. 또한, 냉동 보존 선충류의 게놈을 시퀀싱함으로써 연구자들은 유전 적 변이의 패턴이 생태 데이터와 어떻게 상호 관련되어 있는지 조사 할 수 있습니다. 이러한 종류의 연구는 Caenorhabditis 집단에서 지역 적응의 시그니처를 밝혀 낼 수 있으며 자연 맥락에서 유전 적 변이의 관련성에 대한 중요한 통찰력을 제공 할 수 있습니다8. Caenorhabditis 선충류의 많은 유전자에 대한 기능적 이해를 얻으려면 생태 연구가 필요할 수 있습니다11. C. elegans의 경우에도 유전자의 상당 부분이 기능적 주석이 부족하지만, 시퀀싱되는 최초의 다세포 동물이자 지구상에서 가장 철저히 연구 된 동물 중 하나임에도 불구하고. 이 수집 프로토콜은 야생 Caenorhabditis 선충류의 수집과 생태학 및 자연 유전 적 다양성에 대한 연구를 촉진함으로써 이러한 지식 격차를 해결하는 데 도움이되도록 개발되었습니다.
공개
저자는 이해 상충을보고하지 않습니다.
감사의 말
이 연구는 노스웨스턴 대학교의 창업 기금과 국립 과학 재단 경력 상 (IOS-1751035)에 의해 지원되었으며, 둘 다 E.C.A.에 수여되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
참고문헌
- Frézal, L., Félix, M. -. A. C. elegans outside the Petri dish. eLife. 4, 05849 (2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339 (2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -. A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43 (2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465 (2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587 (2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and Related Nematodes. WormBook. , (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293 (2021).
- . Mobile data collection & workflow automation in NCAP Available from: https://www.fulcrumapp.com/ (2021)
- . Nematode Isolation Application Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021)
- . Nematode Field Sampling Application Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021)
- . JOVE wild_isolate-genotyping-template Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021)
- . Nucleotide BLAST: Search nucleotide databases using a nucleotide query Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021)
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156 (2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유