Method Article
גישה מדרגית מאוד לביצוע סקרים אקולוגיים של נמטודות קאנורהבדיטיס Selfing
In This Article
Summary
פרוטוקול זה יכול לשמש לביצוע סקרים אקולוגיים בקנה מידה גדול של נמטודות Caenorhabditis עצמית. היתרון העיקרי של שיטה זו הוא ארגון יעיל וניתוח של נתונים אקולוגיים ומולקולריים הקשורים לנמטודות שנאספו מהטבע.
Abstract
אלגנים Caenorhabditis הוא אחד האורגניזמים המודל העיקרי בביולוגיה, אבל רק לאחרונה יש חוקרים התמקדו באקולוגיה הטבעית שלה. העיוות היחסית של מידע על C. elegans בהקשר הטבעי שלה נובעת מהאתגרים הכרוכים בזיהוי הנמטודה הקטנה בטבע. למרות אתגרים אלה, התמקדות גוברת באקולוגיה של C. elegans גרמה שפע של מידע חדש לגבי חייו מחוץ למעבדה. החיפוש המוגבר אחר C. elegans בטבע תרם לגילוי של מינים חדשים רבים של Caenorhabditis וחשף כי נמטודות מולדות לעתים קרובות לחיות בטבע, שם הם ניזונים מפריחות מיקרוביות הקשורות לחומר צמחי נרקב. זיהוי של מינים חדשים גילה גם כי מערכת ההזדווגות androdioecious של זכרים והרמפרודיטים להפריה עצמית התפתחה שלוש פעמים באופן עצמאי בתוך Caenorhabditis. שני המינים האחרים של selfing, C. briggsae ו - C. tropicalis, חולקים את היתרונות הניסיוניים של C. elegans ומאפשרים מחקרים השוואתיים לבסיס המכני של תכונות חשובות, כולל הפריה עצמית. למרות התקדמות זו, נותר ללמוד רבות על האקולוגיה והמגוון הטבעי של מינים חשובים אלה. לדוגמה, עדיין חסר לנו מידע פונקציונלי עבור רבים מהגנים שלהם, אשר ניתן להשיג רק באמצעות הבנה של האקולוגיה הטבעית שלהם. כדי להקל על מחקר אקולוגי של נמטודות Caenorhabditis עצמית, פיתחנו שיטה מדרגית מאוד לאיסוף נמטודות מהטבע. השיטה שלנו עושה שימוש בפלטפורמות איסוף נתונים ניידים, במסדי נתונים מבוססי ענן ובסביבת התוכנה R כדי לשפר את יכולתם של החוקרים לאסוף נמטודות מהטבע, לתעד נתונים אקולוגיים הקשורים ולזהות נמטודות פראיות באמצעות ברקודים מולקולריים.
Introduction
שני העשורים האחרונים הביאו עניין מוגבר באקולוגיה של נמטודות Caenorhabditis. ממחקרים אלה, אנו יודעים כי מינים חיים חופשיים Caenorhabditis ניתן לבודד מן מיקרו בתי גידול ארעיים באזורים ממוזגים וטרופיים כאחד, שבו הם ניזונים מפריחות מיקרוביאליות הקשורות חומר צמחי נרקב, לפעמים בסימפטריה1,2,2,3,4,5,6,7,8 . למדנו גם כי אבולוציה מתכנסת של הפריה עצמית התרחשה בסוג שלוש פעמים, ו selfing הוא אופן הדומיננטי של רבייה עבור C. briggsae, C. elegans, ו C. tropicalis9,10. בין selfers אלה, C. elegans הוא אחד החיות הנחקרות ביותר על פני כדור הארץ, שימש על ידי חוקרים כדי לבצע התקדמות קריטית בביולוגיה. חשוב לציין, מינים אחרים של Caenorhabditis עצמי חולקים רבים של C. elegans יתרונות ניסיוניים והם מקדמים במהירות מחקרים השוואתיים בסוג. עם זאת, האופי המסתורי של נמטודות אלה בטבע מקשה על חקר האקולוגיה והמגוון הטבעי שלהן, שהוא קריטי להבנת התפקודים הביולוגיים של הגנים שלהם והדרכים שבהן האבולוציה עיצבה את המגוון הגנטי בקרב המינים10,11.
האתגר הגדול ביותר ללמוד את האקולוגיה של נמטודות Caenorhabditis עצמית בטבע הוא גודלם הקטן; נמטודות למבוגרים הן לעתים קרובות 1 מ"מ אורך או פחות. אתגר זה מחייב את החוקרים לדגום מצעים מהטבע ולנסות להפריד נמטודות של עניין מהמצעים במעבדה ללא היכולת להתבונן בבעלי חיים בטבע. מכיוון שאפילו מומחים מיומנים מתקשים להפלות נמטודות קאנורהבדיטיס עצמיות מנמטוטדות אחרות שחיות חופשיות תחת המיקרוסקופ, נמטודות מוסרות בדרך כלל מהמצע, מבודדות, ונשארות להתרבות לפני שהן מזוהות על ידי זהות רצף באמצעות ברקודים מולקולריים מבוססים3,12,13,14 . הזמן והמאמץ הנדרשים לעיבוד כל נמטודה בדרך זו מציבים אתגר ארגוני, שכן החוקרים חייבים להיות מסוגלים להתחקות אחר זהותה של כל נמטודה המבודדת במעבדה בחזרה למצע המדויק ולנתונים האקולוגיים הנלווים שנדגמו בשטח. כאן, אנו מתארים תהליך שלב אחר שלב כדי לאסוף ולזהות ביעילות נמטודות Caenorhabditis עצמית מהשטח ולקשר בנאמנות מבודדים אלה עם הנתונים המרחביים והאקולוגיים הקשורים שלהם בקנה מידה גבוה.
שיטת איסוף זו מגדילה את קנה המידה והדיוק של סקרים אקולוגיים באמצעות פלטפורמות איסוף נתונים ניידים, מסדי נתונים מבוססי ענן וסביבת התוכנה R. Fulcrum היא פלטפורמת איסוף נתונים הניתנת להתאמה אישית שעובדת עם רוב המכשירים הניידים ומאפשרת למשתמשים לבנות יישומים מותאמים אישית כדי לאסוף ולארגן נתונים מבוססי מיקום (https://www.fulcrumapp.com). פרוטוקול זה מספק הוראות מפורטות כיצד להשתמש ביישומי איסוף נתונים מותאמים אישית כדי לארגן נתונים אקולוגיים מפורשים מרחבית מהשטח ולקשר במדויק נתונים אלה עם זהות הנמטודות המבודדות במעבדה. הפרוטוקול גם מסביר כיצד לזהות ביעילות נמטודות Caenorhabditis עצמי באמצעות ברקודים מולקולריים הוקמה. הנתונים משיטות אלה ניתן לעבד בפשטות ובשחזור עם חבילת התוכנה R הנלווית easyFulcrum15 כדי לחקור את האקולוגיה ואת המגוון הגנטי של אוכלוסיות Caenorhabditis טבעיות.
Protocol
1. הכנת איסוף
- זהה מיקום לסקר נמטודות Caenorhabditis .
הערה: ברוב האזורים הממוזגים, C. elegans ו- C. briggsae ניתן לבודד בקלות מבתי גידול הקשורים לאדם כמו שדות חקלאיים או גנים כפריים ועירוניים1. באזורים סובטרופיים וטרופיים, C. briggsae, C. elegans ו- C. tropicalis ניתן למצוא את כל בתי הגידול הקשורים לאדם המפורטים לעיל, לפעמים בסמיכות זה לזה. עם זאת, נראה כי ג. אלגנס מעדיפים בתי גידול קרירים ויבשים יותר מאשר מינים אחרים בבתי גידול טרופיים7,8. כל אחד מהמינים יכול גם להיות מבודד מבתי גידול פראיים שאינם קשורים לבני אדם, אבל בתי גידול אלה נדגמים לעתים רחוקות יותר. - צור פרוייקט נקודת משען כדי לארגן את נתוני האיסוף והבידוד באמצעות יישומי איסוף הנתונים הניידים.
- צור חשבון עם נקודת המשען באופן מקוון באמצעות הסכם חינוכי ללא עלות16. הוסף את היישום דגימת שדה Nematode לחשבון נקודת המשען על ידי לחיצה על לחצן ADD APP17.
- הוסף את היישום בידוד Nematode לחשבון על-ידי לחיצה על לחצן ADD APP18.
הערה: מומלץ שכל נסיעה למיקום מאורגנת כפרויקט האיסוף שלה באמצעות מוסכמה למתן שמות 'YearMonthLocation', למשל, 2020FebruaryAustralia.
- הוסף משתמשים לחשבון נקודת המשען כדי להעניק להם גישה לפרוייקט האיסוף. ודא שכל משתמש מוריד את היישום הנייד של נקודת המשען כדי להשתתף בפרוייקט.
- הדפס קבוצה של תוויות קוד QR כדי לעקוב אחר האוספים (תוויות C) ובידודי הנמטודות (תוויות S) עם היישום הנייד. חברו את תוויות ה-C לשקיות ניילון עם רוכסן, גלגלו את השקיות המסומנות לקבוצות של 25 ועטפו אותן בגומייה לאריזה. שמור את הסט של תוויות S לשימוש במעבדה.
הערה: לאורך פרוטוקול זה, האוספים (מצעים מהשדה) כלולים בשקיות או בלוחות ומסומנים בתוויות C. הנמטודות המבודדות מסומנות בתוויות S. תוויות C משמשות לזיהוי אוספים ייחודיים, ותוויות ה- S משמשות לזיהוי מבודדי נמטודות ייחודיים. שני סוגים אלה של תוויות משמשים ליצירת הקשר בין אוסף מסוים (C-label) לבין הנמטודות המבודדות מאוסף זה (תוויות S) במסד הנתונים של נקודת המשען. הדפס מספר כפול של תוויות S כתוויות C עבור פרוייקט אוסף מכיוון שבממוצע, שתי נמטודות מבודדות לכל אוסף. ניתן להדפיס תוויות S נוספות במועד מאוחר יותר במידת הצורך. 2,500 תוויות C ייחודיות (קובץ משלים 1) ו-5,000 תוויות S ייחודיות (קובץ משלים 2) מסופקות בתוספת. - הכינו לוחות NGMA בגודל 10 ס"מ לאוספים ולוחות NGMA בגודל 3.5 ס"מ לבידוד נמטודות. הכינו צלחת אחת בקוטר 10 ס"מ ולפחות שתי צלחות בגודל 3.5 ס"מ לאוסף21. לוחות אלה הם זרעו עם Escherichia קולי זן OP50 בעקבות פרוטוקולים שנקבעו. יש לאחסן את הצלחות לפני השימוש בטמפרטורה של 4 °C (65 °F) למשך לא יותר מחודש אחד.
2. אוסף שדות
הערה: נמטודות Caenorhabditis מבודדות לרוב מחומר צמחי נרקב, כולל פירות, אגוזים, זרעים, תרמילים, פרחים, גבעולים, המלטה צמחית וקומפוסט1,5,6,8. המצעים הטובים ביותר רקובים וכמעט בלתי ניתנים לזיהוי כמו פירות או פרחים; הימנעו ממצעים יבשים או רטובים מדי (איור 1). מצעים נאספים בצורה היעילה ביותר מהשטח על ידי עבודה בזוגות. האדם עם מדחום האינפרא-אדום ללא מגע יבחר מצע לאיסוף ולאסוף את המדגם בזמן שבן זוגו משתמש ביישום דגימת שדה נמטודות ב-Fulcrum כדי לתעד את נתוני האיסוף. זוג האספנים יחזרו על תהליך זה עד שייאספו מספר הדגימות הרצוי. רשימת החומרים הדרושים לעבודת שטח נמצאת ב- (טבלה משלימה 1).
- פתחו את האפליקציה למכשירים ניידים של Fulcrum, בחרו דגימת שדה נמטודה מהתפריט הנפתח. הקש + כדי להתחיל רשומה חדשה בפרוייקט (איור 2A). צלם את המצע.
- לחץ על התיבה במרכז העליון כדי לבחור את פרוייקט האיסוף הנכון שנעשה בשלב 1.2 (איור 2B). הקש על השדה תווית C בתחתית רשומת האוסף ובחר סרוק כאשר מופיעה הבקשה. סרוק את הברקוד בתיק האיסוף באמצעות מצלמת המכשיר הנייד ולאחר מכן הקש על בוצע בפינה השמאלית העליונה של המסך.
- הקש על השדה מצע ובחר סוג מצע מהתפריט הנפתח. הוסף הערות אודות המצע על-ידי הקשה על השדה הערות מצע והזנה ידנית של הערות.
- בחר נוף מהתפריט הנפתח. בחר את הנוף המייצג בצורה הטובה ביותר את אתר הדגימה.
- בחר נוף לשמיים. בעת בחירת נוף לשמיים, תאר את הנראות של השמיים באתר הדגימה (למשל, נוף שמיים מלאים ללא נופים חסומים מעצים או מבנים אחרים = מלא).
- מדוד את טמפרטורת פני השטח של המצע באמצעות מדחום ללא מגע ורשום את הערך בשדה טמפרטורת המצע.
הערה: החזק/י את המדחום ללא מגע במרחק של לא יותר מ-14 אינץ' מהמצע בעת רישום הטמפרטורה. - מדוד את טמפרטורת הסביבה ואת הלחות באמצעות מכשיר כף היד ורשום נתונים אלה בשדות הנכונים.
הערה: ודא שטמפרטורת הסביבה והתקן הלחות אינם בהמתנה. יחידת המידה תשתנה בעת שחרור הלחצן. שמור את ההתקן בכיס חיצוני כדי למנוע קריאות לא סדירות. - שמור את הרשומה בנקודת המשען על-ידי הקשה על שמור בפינה הימנית העליונה של המסך.
- לאסוף על כף של המצע ללא מקלות או חתיכות קשות אחרות על ידי היפוך שקית האיסוף להשתמש בו כמו "כפפה" כדי להרים את המצע, ולאחר מכן לאטום את התיק. שים מגבת נייר בשקית אם המדגם לח במיוחד.
הערה: באקלים חם, הניחו את התיקים בצידניות רכות עם אריזות קרירות יותר כדי לשמור על הקולקציות קרירות. - לאחר שכל הדגימות נאספו להיום, לנקות את ציוד האיסוף, לקחת סוללות מתוך בדיקות, לטעון סוללות, להקפיא מחדש חבילות מקפיא. סנכרן את נתוני האיסוף של נקודת המשען על-ידי הקשה על לחצן סנכרן בפינה הימנית העליונה של יישום דגימת שדה Nematode .
הערה: ההעלאות יכולות להימשך מספר דקות ללא חיבור סלולרי חזק, כך שעדיף לחכות לגישה ל- WiFi. הנתונים יישארו במכשירים ניידים ויסונכרנו לענן. - שלח את הדגימות למוסד ביתי על ידי הצבתן בתיבת משלוח לילה. מזער את הזמן שבו הדגימות חשופות לטמפרטורות הנמוכות מ-11 °C (60 °F) או יותר מ-25 °C (600 °F) על ידי משלוח חבילות בימים שבהם המטען מועבר.
הערה: רוב מתקני הספנות אינם שולחים מטען במהלך הלילה בסופי שבוע במקומות מרוחקים.
3. ציפוי אוספי שדה במעבדה
הערה: סעיף זה מפרט כיצד לארגן את העברת הדגימות מתיקי אוסף מסומנים בתווית לצלחות עם תוויות. הדגימות עשויות להגיע ממשלוח בן לילה או ישירות מהשדה.
- קבל את משלוח האוספים ובדוק אם קיימים תיקים שבורים או ראיות אחרות לנזק. אם התיקים שבורים, להשליך את החומר ולנקות את שקיות האיסוף רצוף עם 70% אתנול; הימנע C-תווית על התיק עם האתנול כפי שהוא יהיה לשנות את צבע התווית ולהקשות על הקריאה.
- עבור כל שקית נעילת רוכסן, שימו לב לתווית C על השקית וצירפו תווית C תואמת למכסה של צלחת בגודל 10 ס"מ שאותרה עם חיידקי OP50.
הערה: הלוחות המסומנים בקוטר 10 ס"מ מכונים 'לוחיות C' עבור שאר הפרוטוקול. הדרך הקלה ביותר לארגן את הדגימות היא להניח את שקיות האיסוף על ספסל מעבדה עם לוחית C תואמת למעלה (איור 3). - לכל קולקציה מעבירים כף אחת של דגימה מתיק האיסוף לצלחת C באמצעות כף פלסטיק נקייה. הוסיפו את הדגימה סביב מדשאת החיידקים בצורת חצי סהר או טבעת, אל תכסו לחלוטין את מדשאת החיידקים (איור 4).
הערה: שמור על כף נקייה על ידי הצבתה בכוס של 95% אתנול כאשר הוא אינו בשימוש. השתמש במגבת נייר כדי לייבש את הכף לפני העברת דגימות נוספות. - תעד את הזמן שבו האוספים הועברו מתיקי איסוף לצלחות C ולשמור את צלחות C בטמפרטורת החדר (RT) לפחות 24 שעות לפני שתנסה לבודד נמטודות בסעיף 4.
4. בידוד נמטודות מאוספים
- פתחו את היישום 'נקודת המשען ' במכשיר הנייד ובחרו 'בידוד נמטודות ' מתפריט היישום (איור 5A). צור רשומת בידוד חדשה על-ידי הקשה על סמל + בפינה השמאלית התחתונה (איור 5B).
- במסך רשומת הבידוד החדש, אשר את פרוייקט האוסף הנכון על-ידי בדיקת שם הפרוייקט המוצג בתיבה במרכז העליון. אם מוצג פרוייקט שגוי, הקש על שם הפרוייקט כדי לעבור לפרוייקט הנכון (איור 5C).
- הקישו על הלחצן 'בחר ' תחת השדה 'תווית C' כדי למצוא את תווית ה-C המשויכת לדוגמה שממנה מבודדים נמטודות (איור 5D). הקש על סמל החיפוש ולאחר מכן הקש על סמל הסריקה כדי לסרוק את קוד ה- QR של תווית C בלוחית C באמצעות מצלמת ההתקן. לאחר סריקת קוד ה- QR, תופיע רשומת תווית C בשדה תווית C .
- הקישו על סמל 'מצלמה ' בשדה 'תמונות' כדי לפתוח את מצלמת המכשיר והשתמשו בה כדי לצלם את הדגימה בלוחית C כשקוד ה-QR גלוי (איור 5E). הקישו על 'בוצע' כדי לחזור למסך 'בידוד'.
הערה: ניתן להשתמש בתמונות רשומות בידוד אלה כדי לחקור תכונות ספציפיות של המצע במועד מאוחר יותר. - השתמש במיקרוסקופ מנתח כדי לחפש נמטודות על צלחת C. הקש על התולעים בשדה לדוגמה כדי לתעד את נוכחותן של נמטודות במדגם (איור 5F). הקש על כן אם נמטודות נמצאות על צלחת C והקש על לא אם אין נמטודות קיימות.
- הקש על רצועות אם רק מסלולי נמטודות קיימים. אם אין נמטודות קיימות, parafilm C-צלחת ולהיפטר ממנו בפח biohazard.
הערה: הפוך את לוחית C מעל פח האשפה biohazard בעדינות הקש על החלק האחורי של הצלחת כדי לחלץ את כל המצעים שנדגמו. שלב זה מקל על מציאת נמטודות שיכולות להיות מתחת למצע בלוחית C. - אם קיימות נמטודות, בודדו עד חמש נמטודות מלוחית הסי. כדי לבודד נמטודה, העבירו נמטודה אחת מצלחת C ללוחית S באמצעות פיק חוט פלטינה. לבודד מבוגרים בריאים, gravid במידת האפשר. עם זאת, לבודד שלבים אחרים אם מבוגרים לא נמצאו.
הערה: לאחר הבידוד, לכל אחת מחמש צלחות S תהיה נמטודה אחת. שמור אלה S-plate(s) עם נמטודות מבודדות מאותה צלחת C מאורגן יחד בערימה מסודרת הרחק צלחות S אחרות עד שהם נכנסים לפולקרום. - הקש על השדה לוחיות עם תווית S כדי להזין את לוחות ה- S המשמשים לבידוד זה. הקש על + בפינה השמאלית התחתונה. הקש על S-label ולאחר מכן לחץ על סרוק כדי לפתוח את מצלמת ההתקן. השתמש במצלמת ההתקן כדי לסרוק את קוד QR תווית S בלוחית S.
הערה: ודא שקוד תווית S תואם לקוד שעל הצלחת. אם הוא תואם, הקש על בוצע. אם לא, הקש על ביטול וסרוק מחדש עד שיתאים, ולאחר מכן לחץ על סיום. לפעמים קודי QR של לוחות סמוכים נסרקים בטעות. - לאחר הזנת כל לוח S, שמור את הערך עם לחצן שמור בפינה השמאלית העליונה. הערך יאבד אם הוא לא יישמר. הקש על + בפינה השמאלית התחתונה כדי להוסיף לוחות נוספים עם תווית S במידת הצורך עד הזנת כל הנמטודות המבודדות מלוחית C. לאחר הוספת כל הלוחות המסומנים בתווית S עבור רשומת הבידוד, הקש על לחצן < בפינה הימנית העליונה כדי לחזור למסך הרשומה של הבידוד.
הערה: כדי לבטל רשומת בידוד מכיוון שלא ניתן לפתור שגיאות, לחץ על ביטול בפינה הימנית העליונה. שלב זה יפתח תיבת דו-שיח השואלת אם ניתן למחוק את הרשומה מבלי לשמור אותה. אם תרצה, לחץ על כן, בטל. - הקש על לחצן שמור בפינה השמאלית העליונה לאחר שרשומת הבידוד תוסיף את כל המידע כראוי. לאחר מכן parafilm S-לוחות עם נמטודות מבודדות ולשים אותם בצד באזור המיועד להחזיק S-צלחות עם נמטודות.
- פרפילם C-צלחת ולהשליך אותו בפח biohazard. הקש על סמל הסינכרון כדי להעלות את כל הנתונים לנקודת המשען.
- מיין את כל צלחות S לפי סדר אלפאנומרי, ולאחר מכן למקם את S-צלחות לתוך קופסאות קרטון. ודא את S-צלחות הם בצד המכסה למטה ו parafilmed. יש לערום עד ארבע צלחות S במיקום אחד בתיבה ולתייג את קופסת הקרטון בשם הפרוייקט, התאריך, השעה ומספר התיבה הייחודי.
- אחסן את התיבות המסומנות בתווית ב- RT. מבודדים אלה ייבדקו להתפשטות ב 48 שעות ושוב ב 168 שעות במידת הצורך.
5. ייצוא צלחות S מנקודת המשען
הערה: סעיף זה מפרט כיצד לייצא תוויות S המשמשות בתהליך הבידוד ממסד הנתונים של פרוייקט נקודת המשען. תוויות S אלה ישמשו למעקב אחר קווי איזופל מתרבים בזמן שהם מזוהים על ידי זהות רצף בסעיפים 6-9.
- היכנס לאתר האינטרנט של נקודת המשען ובחר ביישום בידוד נמטודות . לחץ על יצואן מהצד השמאלי של המסך. לחץ כדי לבחור את הפרוייקט הרצוי וסמן את התיבה בידוד נמטודות . לחץ על הבא כדי להוריד קובץ .zip המכיל את הקובץ 'nematode_isolation_s_labeled_plates.csv'.
- פתח את הקובץ 'nematode_isolation_s_labeled_plates.csv' ומיין אותו לפי העמודה 'S-label' בסדר עולה (תווית ה- S הקטנה ביותר תהיה למעלה). בחר את כל תוויות ה-S והעתק אותן מהגיליון האלקטרוני.
- נווט אל גיליון Google של תבנית הגנוטיפינג המבודדת הפראית (wild_isolate_genotyping_template) באמצעות דפדפן אינטרנט19.
- צור עותק של גיליון Google זה על-ידי לחיצה באמצעות לחצן העכבר הימני בכרטיסיה תבנית Genotyping ולאחר מכן בחר באפשרות העתק לגיליון אלקטרוני חדש . בחר פתח גיליון אלקטרוני כדי להציג את הגיליון החדש של Google.
- תן שם לגיליון חדש זה עם שם פרוייקט נקודת המשען ואחריו 'wild_isolate_genotyping', לדוגמה, '2020FebruaryAustralia_wild_isolate_genotyping'.
הערה: גיליון זה מכונה 'גיליון genotyping' לאורך שאר הפרוטוקול.
- הדבק את תוויות ה- S שהועתקו מהעמודה 's_label' nematode_isolation_s_labeled_plates.csv' בעמודת גיליון הגנוטיפינג שכותרתה 's_label'. בדוק את העמודה 's_label_repeat_error' עבור '1'. ערך של '1' בעמודה זו פירושו שתווית ה- S משוכפלת איפשהו בגיליון genotyping. אם מתגלים כפילויות, לחקור ולתקן אותם לפני שתמשיך הלאה.
- מלא את העמודה 'isolation_box_number' של גיליון הגנוטיפינג עבור כל תוויות ה- S.
6. בדוק אם יש התפשטות על S-צלחות
- בדוק אם יש בעלי חיים מתרבים בלוחות S 48 שעות לאחר הבידוד (השתמש בתאריך ובשעה של הבידוד האחרון על האריזה משלב 4.11 כדי להנחות את התזמון).
הערה: נמטודות מתרבות מאופיינות בצאצאים על צלחת ה- S. - אם לוחית S מתרבה, הזן את '1' בעמודה proliferation_48 בגיליון הגנוטיפינג ולאחר מכן העבר את לוחית ה- S לתיבה שכותרתה 'התפשטות של 48 שעות, תיבה 1'. מניחים מקסימום של 88 S-צלחות בתיבת התפשטות, ולאחר מכן להתחיל למלא תיבה חדשה שכותרתה 'התפשטות 48 שעות, תיבה 2'. ודא שתוויות S מאורגנות בסדר אלפאנומרי בתיבות התפשטות של 48 שעות.
הערה: אין להיפטר מלוחות ה-S שאינם מתרבים; צלחות אלה ייבדקו שוב בשעה 168 שעות לאחר הבידוד. אם תרצה, איחד לוחות S אלה בסדר מספרי בתיבות שכותרתן '48 שעות ללא התפשטות, תיבה X', אך זכור לרשום מתי הבדיקה של 168 שעות חייבת להתרחש בתיבה החדשה. - לאחר זיהוי כל תוויות ה- S המתרבות במהירות של 48 שעות, עבור לסעיף 7 ללוחות S עם התפשטות של 48 שעות.
- בדוק את S-צלחות שלא התרבו ב 48 שעות לאחר הבידוד שוב ב 168 שעות לאחר הבידוד.
- אם לוחית S מתרבה כעת, הזן '1' בעמודה proliferation_168 בגיליון genotyping ולאחר מכן העבר את לוחית ה- S לתיבה שכותרתה 'התפשטות של 168 שעות, תיבה 1'.
- מניחים מקסימום של 88 S-צלחות בתיבת התפשטות, ולאחר מכן להתחיל למלא תיבה חדשה שכותרתה 'התפשטות 168 שעות, תיבה 2'. הקפד לארגן תוויות S בסדר אלפאנומרי בתיבות התפשטות של 168 שעות.
- השלך את לוחות S שאין להם התפשטות לאחר 168 שעות. עבור לסעיף 7 עבור S-צלחות עם התפשטות ב 168 שעות.
7. תמוגה של קווי איזופל
הערה: שלב זה ישתמש בכלי מסנן הנתונים בגליונות Google כדי לסייע בהדפסת גליונות עבודה תזה עבור לוחות S בתיבות ההתפשטות. מטרת גליונות העבודה של תמוגה היא לספק לאנשי הצוות את העמדות הנכונות עבור תוויות S בצינורות רצועת תמוגה על הספסל.
- פתח את גליון הגנוטיפינג עבור הפרוייקט הרצוי ובחר את כל התאים על-ידי הקלדת Cmd+A. לחץ על נתונים > צור מסנן כדי להוסיף לחצן סינון לכל כותרת עמודה. השתמש בלחצני המסנן כדי להציג רק את לוחות ה- S שיועברו genotyped. לדוגמה, אם יש להוסיף את כל לוחות ה- S עם התפשטות של 48 שעות: לחץ על לחצן סינון בעמודה 'proliferation_48' ובחר '1'.
- לאחר שגיליון google genotyping עבר סינון, סקור את רשימת תוויות ה- S המוצגות כדי לוודא שהן תוויות ה- S שיודפסו בגליון העבודה.
- בעמודה 'strip_tube_number' של גיליון google genotyping, הזן מספר ייחודי כל 11 שורות.
- הזן את מספרי שפופרת הרצועה עבור פרוייקט בסדר רציף החל מ- 1 ומעולם לא שוכפל. ב'strip_tube_position', הזן 2 עד 12 עבור כל מספר שפופרת רצועה.
הערה: השתמש בצינורות רצועת 12 צינורות לתזה. המיקום הראשון (strip_tube_position 1) יהיה בשליטה, אך הפקדים אינם מתווספים לגליונות העבודה של תמוגה (רק strip_tube_positions מתווספים, 2-12). בזמן תמוגה, זן השליטה החיובית 'N2' יתווסף למיקום 1 של כל צינור רצועה זוגי כבקרה חיובית. תולעים אינן מתווספות למיקום 1 של כל צינור רצועה אי-זוגי כפקד שלילי.
- סנן עוד יותר את גיליון google genotyping כדי לכלול רק את תוויות ה- S בתיבת התפשטות אחת שיש להוסיף, ולאחר מכן בחר את העמודות 's_label' באמצעות 'lysis_notes'. הדפס גליון עבודה תמוז עבור כל תיבת התפשטות שיש להוסיף.
- לחץ על התפריט הנפתח בשדה הדפסה ובחר תאים נבחרים. לחץ על הבא בפינה השמאלית העליונה ולאחר מכן השתמש בתיבת הדו-שיח כדי להדפיס את גליון העבודה תמוגה עבור תיבת ההתפשטות.
- חזור על שלבים 7.3-7.5 כדי להדפיס גליון עבודה תמוגה עבור כל תיבת התפשטות.
הערה: כל תיבת התפשטות מכילה עד 88 לוחות S, אשר תואמים עם שמונה צינורות רצועה 12-well. - הכן צינורות רצועה 12-well עבור כל הדגימות כי יהיה lysed. הוסף תווית לצינור רצועה עם 'strip_tube_number' ייחודי המוקצה בגליון העבודה תמוגה. תווית זו חייבת להיות כתובה על רצועת הכובע ועל צינור החשפנות כדי למנוע בלבול אם הם מופרדים. צינורות החשפה אפילו יש שליטה חיובית (תולעי N2) בעמדה 1. צינורות הרצועה ODD יש שליטה שלילית (ללא תולעים) בעמדה 1.
- להמציא מספיק מאגר תמוגה (100 mM KCl, 20 mM Tris pH 8.2, 5 מ"מ MgCl2, 0.9% IGEPAL, 0.9% Tween 20, 0.02% ג'לטין עם חלבון K הוסיף לריכוז סופי של 0.4 מ"ג / מ"ל) עבור כל הדגימות ולהוסיף 5% נוספים עבור שגיאת פיפטה. קנה מידה לפי הצורך.
הערה: מאגר תמוגה מוכן בצורה הטובה ביותר על ידי שילוב של כל המרכיבים למעט חלבון K והקפאה ב 10-50 מ"ל aliquots ב -20 °C (60 °F). להפשיר aliquots ולשמור על 4 °C (50 °F) לפני השימוש; מיד לפני השימוש, מוסיפים חלבון K ומערבבים היטב. שמור את מאגר תמוגה על קרח בזמן העבודה. - סדר את לוחות ה- S עבור צינור רצועה מסוים לפי הסדר באמצעות גליון העבודה המודפס תמוגה כמדריך.
- נקה צינור רצועה אחד והוסף 8 μL של מאגר תמוגה לכל מכסה עם pipettor חוזר. הוסף את מאגר תמוגה לרצועה אחת של כובעים בכל פעם מכיוון שמאגר התמזה יתאדה אם יישאר ב- RT וייחשף. בחר 3-5 בעלי חיים מלוחות המקור (צלחת S או צלחת מלאי N2 עבור פקדים חיוביים) לתוך עמדות המכסה המתאימות המצוינות בגליון העבודה תמוגה. רשום הערות עבור כל לוחית S עם פחות מ- 5 תולעים שנבחרו לתזה במקטע lysis_notes של גליון העבודה תמוגה.
- לאחר טעינת נמטודות לתוך כל עמדה של צינור החשפנות, מניחים את רצועת הכובע בחזרה על צינור הרצועה. התאם את המכסה המסומן (מיקום 1) עם הצינור המסומן (מיקום 1). לאחר כתרים, צנטריפוגה צינור החשפנות לזמן קצר עד nematodes הם בתחתית הצינור.
- מניחים את הרצועה במקפיא -80 °C (80 °F) עד להקפאה מלאה (לפחות 10 דקות). חזור על שלבים 7.9 עד 7.11 עד שכל הרצועות יש נמטודות הוסיף עבור תמוגה. ארגן את רצועות הצינור בסדר מספרי.
- הסר את הסטים של צינורות רצועה ולהפעיל את תוכנית תמוגה בתרמוציקלר: 1 h ב 60 °C (60 °F), 15 דקות ב 95 °C (95 °F), להחזיק ב 12 °C (60 °F). כאשר תוכנית תמוגה נעשית, לסובב את הדגימות ב 300 x g עבור 15 s ב RT ולאחסן את lysates ב -80 °C (80 °F) עד 1 שבוע.
- ארגן את רצועות הצינור בסדר מספרי באמצעות מחזיקי לוחות 96 בארות ולכלול תווית עם מספר תיבת התפשטות, טווח מספרי שפופרת רצועה, תאריך וראשי התיבות של החוקר. עדכן את עמודות גיליון הגנוטיפינג 'lysis_date' ו'lysis_notes' במידע מגליון העבודה תמוגה.
8. PCR של רצפי SSU ו- ITS2
הערה: סעיף זה יספק הוראות כיצד לבצע שני מחשבי PCR נפרדים עבור כל לוחית S lysed. ערכת הפריימר הראשונה מגבירה שבר של 500 bp של הגן התת-יחידה הקטן 18S rDNA (SSU); oECA1271 = פריימר קדימה TACAATGGAAGGCAGCAGGC, oECA1272 = פריימר הפוך CCTCTGACTTTCGTTCTTGATTAA 12. PCR זה משמש לבדיקת איכות ה- DNA של התבנית. PCR מגביר את אזור SSU עבור כמעט כל מיני נמטודות. אם ה- PCR של SSU אינו מצליח להגביר, תוצאה זו מצביעה על כך שאיכות התזה ירודה ויש לחזור על תמוגה זו עבור לוחית S זו. ערכת הפריימר השנייה מגבירה קטע של 2,000 bp של אזור החלל הפנימי המתומלל בין הגנים rDNA של 5.8S ו- 28S (ITS2); oECA1687 = פריימר קדמי CTGCGTTACTTACCACGAATTGCARAC, oECA202 = primer הפוך GCGGTATTTGCTACTCAYYAMGATCTGC3. מוצר ה- PCR ITS2 הוא סנגר ברצף והרצף משמש לזיהוי נמטודות בסוג Caenorhabditis לרמת המין לפי דמיון רצף.
- השתמש בכלי הסינון בגיליון genotyping כדי להציג רק את תוויות ה- S שישמשו עבור PCR.
- עדכן את pcr_plate_number ואת העמודות pcr_well בגיליון הגנוטיפינג. כדי למנוע השפלה של חומר תמוגה, SSU ו- ITS2 PCR מופעלים בו-זמנית.
- השתמש באותו pcr_plate_number עבור ITS2 ו- SSU PCR למרות שאלו תגובות נפרדות בלוחות נפרדים. הם יכובדו עם תוויות 'SSU' או 'ITS2'.
- הקצה pcr_plate_number לשמונה או פחות צינורות רצועה (שפופרת רצועה אחת בכל שורה של לוחית PCR של 96 בארות, המסודרת בסדר עולה, למשל, מספר צינור הרצועה הנמוך ביותר למעלה). לאחר מכן הקצה pcr_plate_well לכל תווית S בצינורות הרצועה.
הערה: צינורות הרצועה מסודרים בסדר עולה, כאשר מספר צינור הרצועה הנמוך ביותר מוקצה לשורה A והמספר הגבוה ביותר בשורה H. מיקום 1 של כל צינורות הרצועה מוקצה לעמודה 1. לכן, צינור רצועה מספר 1, עמדה 1 יוקצה לוחית PCR מספר 1, גם A01. - סמן תווית של לוחות PCR 96-well כדי להתאים את הדוגמאות שישמשו עבור PCR. תייג כל צלחת PCR במידע הבא: שם פרוייקט, סוג PCR, מספר לוחית PCR ותאריך של PCR (לדוגמה, 2020FebruaryAustralia_SSU_1_20200304). כמו כן, סמן את הצלחת עם מספרי שפופרת החשפנות שייטענו בכל שורה.
- הסר את חומר תמוגה מהמקפיא -80 °C (80 °F) ולהפשיר את צינורות הרצועה המכילים את חומר תמוגה על קרח. בזמן שחומר הזיזה מפשיר, הכינו את ITS2 ו- SSU מאסטר מתערבבים בצינורות נפרדים על קרח. מתכוני SSU ו- ITS2 PCR נמצאים בטבלה משלימה 2.
הערה: הכן 100 תגובות של תערובת מאסטר PCR עבור כל צלחת 96-well כדי לאפשר שגיאת צנרת. השתמש בחרוטית של 15 מ"ל או 50 מ"ל כדי להכיל את התערובת הראשית אם יש להשתמש באמצעי אחסון גדולים. - מערבבים את האב בעדינות עד שטאק מופץ לאורך כל התערובת. לאחר ערבוב, aliquot 38 μL של תערובת מאסטר לבארות המתאימות של לוחות PCR על קרח. השתמשו בשוקת V-bottom סטרילית לשימוש חד-פעמי ובפיפטה רב-ערוצית של 12 בארות כדי להעביר את התערובת הראשית ללוחות ה-PCR.
- לסובב את צינורות רצועת תמוגה מופשרים כדי להסיר חומר תמוגה מן הכובעים. הסר בזהירות את המכסים של כל צינורות הרצועה כי יהיה טעון לתוך צלחת PCR הראשון. השתמש פיפטה רב ערוצית בנפח נמוך (או 12-well או 8-well) כדי להוסיף 2 μL של lysate לבאר המתאימה בצלחת PCR. בעדינות פיפטה lysate למעלה ולמטה פעם אחת לפני הסרת 2 μL.
הערה: בדוק את העצות כדי לוודא שהן מכילות את תמוגה לפני ההעברה. זכור לשנות עצות בין שורות או עמודות. - לכסות את צלחת PCR עם רדיד דבק PCR ולהשתמש רולר כדי ליצור חותם הדוק. לאחר רדיד הכסף מוחל, לסובב לזמן קצר את לוחות PCR בצנטריפוגה. שמור את הצלחת על קרח עד שהוא מוכן לרוץ בתרמוציקלר.
- הפעל את PCRs עם תוכנית thermocycler המתאימה. עיין בטבלה משלימה 2 לקבלת הפרטים של תוכניות SSU ו- ITS2 PCR.
- חזור על שלבים 8.4- 8.8 עד להפעלת כל מחשבי ה- PCR.
- בזמן שתגובות ה-PCR פועלות, יוצקים ג'ל 100 מ"ל 1.5% אגרוז. כל ג'ל יחזיק דגימות או צלחת PCR אחת.
- הוסף 1.5 גרם של agarose לבקבוק 500 מ"ל, ולאחר מכן להוסיף 100 מ"ל של 1x מאגר TAE (טבלה משלימה 3) ומערבול לערבב. מיקרוגל כדי להמיס ולקרר את הג'ל.
- לאחר שהפתרון מקורר, מוסיפים 5 μL של 10 מ"ג / מ"ל פתרון ברומיד אתידיום ומערבבים כדי לשלב. יוצקים את התמיסה למגש יציקה עם ארבעה מסרקים של 25 בארות, כך שהג'ל יכול להכיל 96 דוגמאות בתוספת סולם לכל שורה בג'ל.
הערה: אתידיום ברומיד הוא מוטגן רב עוצמה. בעת טיפול באתידיום ברומיד, השתמש בחלוק מעבדה, כפפות עמידות בפני כימיקלים ומשקפי בטיחות כימיים.
- רגע לפני שה-PCR מסתיים, הוסיפו צבע טעינה פי 6 לשוקת חד פעמית והשתמשו בפיפטה רב-ערוצית כדי להוסיף 2 μL של צבע טעינה פי 6 לכל באר של צלחת PCR חדשה בת 96 באר. צלחת זו תשמש לטעינת הדגימות לתוך הג'ל. הכינו מספיק מהצלחות האלה כדי להכיל את כל הדגימות.
- כאשר PCRs נעשים, להסיר את לוחות PCR לזמן קצר צנטריפוגה אותם ב 300 x g עבור 15 s ב RT. לאחסן את לוחות PCR על קרח עד מוצרי PCR ניתן להיגמר על ג'ל.
- כדי להריץ את המוצרים על ג'ל, השתמש בפיפטה רב-ערוצית של 12 בארות כדי להוסיף 5 μL של כל דגימה לבאר המתאימה של צלחת 96-well המכילה 2 μL של צבע טעינה 6x.
- ואז לטעון 6 μL של תערובת זו לתוך כל באר של ג'ל יצוק לאחרונה. טען 6 μL של 1 KB בתוספת סולם לתוך הבאר הראשונה של כל שורה של הג'ל.
הערה: כדי למלא את הבארות של הג'ל, ייתכן שיהיה צורך לשלב שורה A ושורה B מצלחת PCR בשורה הראשונה של הג'ל. כדי למנוע בלבול, הקלט את gel_number ואת gel_position בגיליון genotyping עבור כל דגימת PCR.
- מניחים מכסה נייר כסף חדש על ה-PCR הנותר בצלחת ומאחסנים אותם בטמפרטורה של 4 מעלות צלזיוס. מוצרי תגובה אלה ישמשו לרצף בשלב 9.
- הפעל את מוצרי PCR על הג'ל ב 120 V במשך 20 דקות. דמיינו את הג'ל והרשמו אילו תוויות S מניבות מוצרי IT2 ו/או SSU PCR בעמודות 'pcr_product_its2' ו'prc_product_ssu' של גיליון הגנוטיפינג. סמן את נוכחותה של להקה עם '1'; סמן '0' ללא להקה.
9. זיהוי נמטודות עם רצף סנגר ורצף BLAST
הערה: סעיף זה מספק הוראות לרצף את האמפליקונים של ITS2 מתוויות S, יישור רצפים אלה למסד הנתונים של המרכז הלאומי למידע ביוטכנולוגיה (NCBI) באמצעות אלגוריתם BLAST, וניתוח תוצאות BLAST כדי לזהות את הנמטודות בלוחות S.
- עבור כל דגימה שהיא ITS2-חיובית, השתמש במוצר PCR ITS2 הנותר עבור ריצוף סנגר באמצעות הפריימר הקדמי oECA306 (CACTTTTTCAAGCAAGCAACCCGAC). סדר שקבצי הפלט של הרצף יקושרו בקלות לתווית S על-ידי הקלטת העמודות 'sequencing_plate' ו'sequencing_well' של כל תווית S בגיליון הגנוטיפינג.
- השג את קבצי הפלט .seq עבור כל תווית S מפלטפורמת הרצף. סדר את קבצי ה- .seq עבור פרוייקט בספריה בודדת עם קבצי .seq עבור כל אצווה של רצף הממוקמת בספריות משנה.
- פתח את כלי ממשק שורת הפקודה ונווט אל הספריה העליונה המכילה את קבצי ה- .seq על-ידי הזנת הפקודה: תקליטור . אם הוא עדיין לא קיים, צור FASTA ממוזג עבור כל קבצי ה- .seq על-ידי הזנת הפקודה הבאה: עבור dir in */; do cd $dir; עבור קובץ ב- *.seq; האם הד ">"$file; חתול $file; נעשה >>.. /all_seqs.fa; cd ..; סיימתי.
הערה: קוד זה ייצור קובץ FASTA ממוזג בשם 'all_seqs.fa' מכל קבצי ה- .seq בספריית הפרוייקט. ניתן להשתמש בקובץ זה בכלי הנוקלאוטיד BLAST המקוון של NCBI כדי ליישר במהירות את רצף ITS2 של כל תווית S למסד הנתונים של הרצף של NCBI. - בדפדפן אינטרנט, נווט אל אתר האינטרנט של NBCI BLAST20 ולחץ על לחצן בחר קובץ . בחר את הקובץ all_seqs.fa שנוצר זה עתה, ולאחר מכן לחץ על הלחצן רצפים דומים במקצת (BLASTn). לחץ על לחצן BLAST בתחתית הדף כדי להתחיל בחיפוש BLAST.
- עדכן את גיליון הגנוטיפינג עם תוצאות BLAST עבור כל תווית S. השתמש בכלי הסינון כדי להקל על עדכון גיליון google genotyping. לחץ על נתונים > צור מסנן כדי להוסיף לחצן סינון לכל כותרת עמודה. סנן את עמודת sequencing_plate כדי לבחור את לוחות הרצף שיש לעדכן בתוצאות BLAST.
- השתמש בתפריט הנפתח בדף התוצאות של NCBI BLAST כדי לבדוק את התוצאות עבור כל רצף ITS2 של לוחית S (איור 6).
- בדוק אם אין להיטי BLAST. מזהה רצף בקידומת הנפתחת עם * אין להיטי פיצוץ. עבור תוויות S אלה, הזן 'ללא פגיעה <תאריך נוכחי>' בעמודה manual_blast_notes של גליון genotyping.
- בדוק אם יש זן חדש אפשרי של קנורהבדיטיס . לחץ על הקישור בפגיעה העליונה כדי להציג את היישור באופן חזותי (איור 6). אם הפגיעה העיקרית היא (1) מין של Caenorhabditis , (2) היישור מכיל יותר מחמישה אי-התאמות במרכז הרצף, ו-(3) כיסוי השאילתה גדול מ-50%, תוצאה זו מצביעה על כך שהבידוד עשוי להיות מין חדש של קאנורהבדיטיס (איור 7). עבור לוחות S אלה להיכנס, המינים של הלהיט BLAST העליון בעמודה 'species_id', להזין 1 בעמודה 'possible_new_caeno_sp', ו 'אפשרי חדש Caeno sp.' לתוך העמודה 'manual_blast_notes' יחד עם אחוז זהות, (למשל, "Caeno חדש אפשרי sp. 89% זהות").
- עבור רצפי S-plate כי BLAST למין Caenorhabditis , הזן את הסוג המלא ואת שם המין של הלהיט BLAST העליון בעמודה 'species_id'. לדוגמה, 'אלגנים קנורהבדיטיס'.
- עבור רצפי S-plate כי BLAST למין שאינו Caenorhabditis , הזן רק את הסוג של להיט BLAST העליון ואחריו 'sp.' בעמודה 'species_id'. משמעות הסימון היא שהבידוד הוא מין לא ידוע בתוך הסוג בעל השם. לדוגמה, 'אושיוס sp.'.
הערה: לא ניתן להשתמש ברצף ITS2 כדי לזהות באופן אמין מבודדים לרמת המין מחוץ לסוג Caenorhabditis 3,13. - הזן 1 ב'עמודת make_strain_name ' של גיליון הגנוטיפינג אם 'species_id' = 'Caenorhabditis elegans', 'Caenorhabditis briggsae', או 'Caenorhabditis tropicalis', או 'possible_new_caeno_sp' = 1.
- שם הזנים עם שמות ייחודיים בעקבות מוסכמות המינוח Caenorhabditis , כלומר, ייעוד מעבדה ייחודי המורכב 2-3 אותיות רישיות ואחריו מספר עבור כל זן ייחודי23. הזן את שמות הזנים בעמודה 'strain_name'.
- לאחר זנים נקראים, הם יכולים להיות cryopreserved באמצעות פרוטוקולים הוקמה 24.
10. עיבוד נתוני האיסוף עם חבילת נקודת המשען הקלה ב- R
הערה: שלב זה מתאר כיצד לקשר את נתוני האיסוף (תוויות C) ואת נתוני בידוד הנמטודות (תוויות S) יחד באמצעות חבילת EasyFulcrum R. התוכנה מכילה פונקציות שיצטרפו עוד יותר לנתוני נקודת המשען עם נתוני הגנוטיפינג מגיליון הגנוטיפינג כך שזהויות מינים של תוויות S ושמות זנים מאורגנים במסגרת נתונים אחת.
- צור ספריה חדשה בשם פרוייקט האוסף. סדר את מבנה התיקיות בתוך הספריה כך שיתאים לדרישות המתוארות בחבילת R easyFulcrum15.
- נווט אל אתר האינטרנט של נקודת המשען והיכנס. יצא את נתוני הפרוייקט הגולמי ממסד הנתונים של נקודת המשען באמצעות כלי ייצוא הנתונים של אתר Fulcrum משמאל ובחר את תיבות הסימון הבאות: פרויקט, כלול תמונות, כולל נתוני GPS, דגימת שדה ובידוד.
הערה: נתוני נקודת המשען עבור הפרוייקט ייוצאו כחמישה קבצי ערך המופרדים באמצעות פסיקים (.csv). נתוני הפרוייקט המלאים יצטרפו יחד למסגרת נתונים אחת באמצעות חבילת נקודת המשען הקלה ב- R. - העבר את חמשת קבצי .csv שיוצאו מ- Fulcrum לספריית הפרוייקט שנוצרה בשלב 10.1 כפי שהונחה ב- vignette נקודת המשען הקלה21.
- פתח הפעלת Rstudio והתקן חבילת נקודת משען קלה ב- R על-ידי הזנת הפקודות הבאות במסוף R 'install.packages("devtools")" ו- 'devtools::install_github("AndersenLab/easyfulcrum")'.
- פתח סקריפט R חדש ובצע את ההוראות ב- vignette נקודת המשען הקלה כדי לעבד את נתוני האוסף21.
תוצאות
פרוטוקול זה שימש לאיסוף נמטודות Caenorhabditis ממקומות מרובים, כולל הוואי וקליפורניה. שיעור ההצלחה בבידוד עבור נמטודות Caenorhabditis משתנה בהתאם למיקום האיסוף, האקלים, חוויית הדגימה וסוגי המצע שנדגמו. הפרוטוקול שימש כדי לדגום בהרחבה את איי הוואי, שם נערכו תשעה פרויקטי איסוף במשך שנים ועונות רבות. שיעורי ההצלחה בבידוד עבור מינים עצמיים Caenorhabditis הם כמעט זהים עבור C. briggsae (162 מתוך 4,506 דגימות, 3.6%) ו C. elegans (163 מתוך 4,506 דגימות, 3.6%), ונמוך בהרבה עבור C. tropicalis (26 מתוך 4,506 דגימות, 0.58%)8. כל אחד ממיני העצמאים מועשר על מצעים נרקבים של פירות ופרחים ביחס לשאר קטגוריות המצע. מדגם פירות נרקבים ומצעי פרחים אם החוקר מנסה למקסם את שיעור ההצלחה במקום לאפיין העדפות מצע. עם זאת, שיעור ההצלחה משתנה בהתאם לאיכות המצע שנבחר. לדוגמה, בקרב מצעים של פירות ופרחים, מצעים יבשים מדי, רטובים או טריים לא יניבו ככל הנראה נמטודות של Caenorhabditis .
המדרגיות של פרוטוקול אוסף זה ניכרת ממספר האוספים שצמד חוקרים יחיד יכול לאסוף מהטבע. לדוגמה, באוקטובר 2018, זוג חוקרים שהשתמשו בפרוטוקול איסוף זה הצליחו לאסוף בסך הכל יותר מ-1,000 דגימות ב-7 ימים ממספר מיקומים בשני איי הוואי. צוות שדה זה שלח את הדגימות בן לילה למעבדה, שם צוות של שמונה חוקרים בודד מעל 2,000 נמטודות מהדגימות כשהגיעו. יתרון מרכזי של פרוטוקול זה הוא שהוא ממזער את העלות הכרוכה בדגימה במקומות מרוחקים על ידי הפחתת הציוד וכוח האדם הנדרשים בשטח. באמצעות פרוטוקול זה, צוות שדה קטן יכול להתמקד בדגימה בעוד צוות הבידוד יכול לעבד את הדגימות במוסד הביתי שלהם באמצעות ציוד שביר וכבד כמו ניתוח מיקרוסקופים וצלחות אגר לבידוד נמטודות. יתר על כן, יישום יישום איסוף הנתונים הנייד מאפשר לכל נתוני השדה המשויכים לדגימות להיות מקושרים ישירות לתווית C, המאפשרת לצוות הבידוד לעבוד בנפרד מצוות השטח בעת עיבוד דוגמאות.
חוקרים המשתמשים בפרוטוקול איסוף זה חייבים לשקול את המאמץ הנדרש כדי לבודד נמטודות לפני פרויקט איסוף. שלבי הבידוד והזיהוי מגבילים את הקצב, וצוות איסוף קטן יכול להציף במהירות מבודדים בדגימות. יתר על כן, מרחב המעבדה הנדרש לעיבוד אוספים רבים עלול להפריע למחקר המתמשך (איור 3). בנוסף, כמה נמטודות מבודדות דורשות מאמץ נוסף לגנוטיפ. לדוגמה, כ-2% מהמבודדים אינם מצליחים להגברה עם פריימר ה-SSU PCR שנקבע לאחר ניסיון התזה הראשון ויש להוסיף אותם מחדש כדי לוודא שחומר התזה מתאים להגברה באמצעות ערכת הפריימרים ITS2 (איור 8). יתר על כן, כ -3% מהמבודדים אינם מצליחים לייצר רצפים איכותיים לאחר סיבוב ראשוני של רצף סנגר. עבור מבודדים אלה, סיבוב נוסף של תמוגה, ITS2 PCR, ורצף סנגר נדרש לעתים קרובות, אשר יכול להגדיל את זמן המסירה עבור צוות הבידוד. חשוב לציין, זהות הרצף לבדה אינה ראיה מספקת כדי להצדיק מין חדש של קנורהבדיטיס (איור 7). כדי להצדיק כראוי העלאת מבודד כמין חדש Caenorhabditis , יש לעשות מאמץ נוסף כדי לבצע ניסויי הזדווגות ולהקים דגימה מוקלדת13. תיאור מורפולוגי רשמי של הדגימה המוקלדת עדיף גם הוא אך לא נדרש3. יחד, שיקולים אלה מציעים כי חוקרים המאמצים פרוטוקול איסוף זה ייהנו מבדיקות ניסיון של שלבי הבידוד והזיהוי כדי להבטיח הקצאת משאבים כראוי לפני תחילת פרויקט איסוף. חשוב לציין, אפילו פרויקטי איסוף קטנים יכולים להפיק תועלת מפרוטוקול זה מכיוון שהתהליך ניתן לשחזור מאוד, וניתן לבקר בקלות את הנתונים למטרות בקרת איכות בקבוצות מעבדה שונות.

איור 1: דוגמאות מצע. (א) פרי נרקב אידיאלי מוצג במרכז התמונה (1), הפרי כמעט בלתי ניתן לזיהוי. פירות רקובים פחות מוצגים בקרבת מקום; הימנעו מדגימת פירות טריים שנפלו (2). (B) פרח מפורק באופן אידיאלי מוצג בחלק העליון (3). הימנעו מדגימת פרחים טריים שנפלו (4). (ג) המלטה עלים כהים מתחת לשכבה העליונה של עלים יבשים היא אידיאלית בעת דגימה עבור עצמית Caenorhabditis נמטודות (5). יש להימנע מדגימת המלטה של עלים יבשים (6). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2: יישום נייד דגימת שדה נמטודות. (A) המסך הראשוני לאחר פתיחת יישום דגימת שדה נמטודה במכשיר אפל בפולקרום. החץ האדום בפינה השמאלית התחתונה מצביע על לחצן + המשמש ליצירת רשומת אוסף חדשה. (ב) דוגמה לרשומת אוסף חדשה המוצגת במכשיר Apple. החץ האדום מצביע על השדה 'פרוייקט' בחלק העליון של מסך רשומת האוסף. הקפד לבחור את הפרוייקט הנכון בעת דגימה בשדה. שדה הפרוייקט יהיה ברירת המחדל לפרוייקט האחרון בו נעשה שימוש בעת יצירת רשומות אוסף עוקבות. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: תיקי איסוף ולוחות איסוף מאורגנים לפני ציפוי דגימות. איור זה מציג את הדגימות בשקיות איסוף עם תווית C משמאל. על כל תיק קולקציה יש צלחת תואמת עם תווית C בגודל 10 ס"מ. מימין לוחות איסוף בגודל 10 ס"מ המכילים חומר מדגם לאחר שהועבר משקיות האיסוף. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4: לוחית איסוף (לוחית C) עם דגימה שהועברה כראוי. צלחת C בגודל 10 ס"מ עם פירות נרקבים המונחים על קצה מדשאת החיידקים. תווית C מחוברת למכסה הצלחת. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
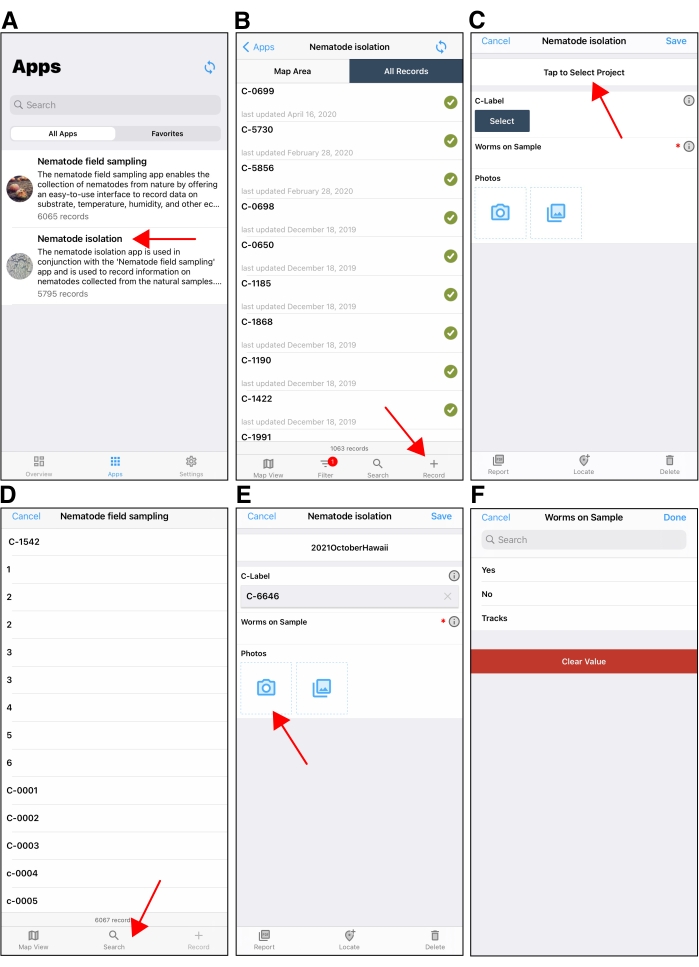
איור 5: יישום נייד בידוד Nematode. (א) מסך בחירת היישומים ביישום הנייד של נקודת המשען. החץ האדום מצביע על יישום בידוד נמטודות . (ב) המסך ההתחלתי לאחר פתיחת היישום Nematode Isolation במכשיר Apple ב-Fulcrum. החץ האדום בפינה השמאלית התחתונה מצביע על לחצן + המשמש ליצירת רשומת בידוד חדשה. (ג) דוגמה לרשומת בידוד חדשה המוצגת במכשיר Apple. החץ האדום מצביע על השדה 'פרוייקט' בחלק העליון של מסך רשומת הבידוד. הקפד לבחור את הפרוייקט הנכון בעת בידוד. שדה הפרוייקט יהיה ברירת המחדל לפרוייקט האחרון המשמש בעת יצירת רשומות בידוד עוקבות. (D) לאחר הקשה על השדה בחר תחת תווית C, משתמשים יקישו על לחצן החיפוש (חץ אדום) כדי למצוא את תווית C שממנה הם מבודדים נמטודות. (ה) לאחר בחירת התווית C, המשתמשים יצלמו את לוחית C באמצעות מצלמת ההתקן. (ו) משתמשים מזינים אם יש נמטודות על לוחית C או לא. תוויות S מתווספות לרשומת הבידוד אם יש נמטודות שיש לבודד. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
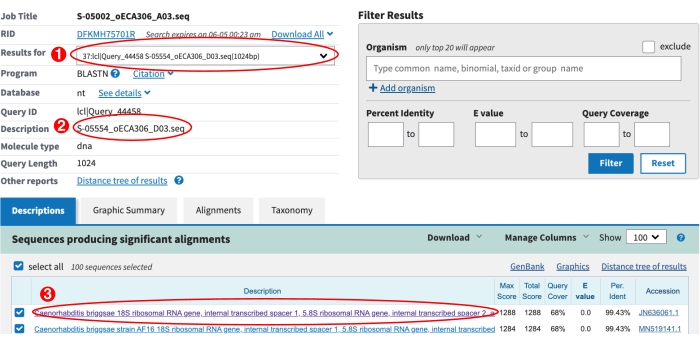
איור 6: דף התוצאות של NCBI BLAST. (1) התפריט הנפתח המשמש להצגת תוצאות BLAST עבור כל הרצפים. (2) תיאור הרצף הנוכחי שנבחר מהרשימה הנפתחת. במקרה זה מוצגות התוצאות עבור S-label S-05554. (3) הלהיט העליון BLAST עבור S-05554 מוצג. הטקסט הסגול מציין את הקישור כדי להציג יישור חזותי זה כבר לחץ. אנא הקפד לבדוק את היישורים על ידי עין כדי לזהות מינים חדשים אפשריים Caenorhabditis , ראה שלב 9.8 לעיל. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 7: דוגמאות להדמיה של יישור NCBI BLAST. (A) דוגמה לרצף שאילתת ITS2 של מבודד מיושר לרצף נושאים של C. kamaaina . (1) אחוז הזהות של היישור (89%), שהוא נמוך עבור להיט BLAST העליון. (2) חוסר התאמה בין השאילתה לרצף הנושאים (ז עד A). (3) פער של ארבעה זוגות בסיסים ברצף הנושאים שנעשה על ידי אלגוריתם היישור; רווחים בשאילתה או בנושא מצביעים על יישור לקוי. (4) אזור כללי במרכז היישור עם אי-התאמות ופערים רבים. אזור כמו זה מצביע על כך שרצף השאילתות עשוי להגיע ממין חדש של קאנורהבדיטיס . מוצגת דוגמה ליישור בפועל של מין חדש, C. oiwi, שהתגלה בשנת 2017. (ב) דוגמה ליישור טוב בין רצף שאילתת ITS2 של מבודד לבין רצף נושאים. (5) אחוז הזהות של היישור (99%), מה שאומר בדרך כלל שרצף השאילתה מגיע מניתוק של אותו מין כמו הנושא. (6) אזור מרכזי של היישור עם זהות מושלמת. אזור כמו זה מצביע על כך שהשאילתה מבודדת היא ככל הנראה אותו מין כמו הנושא. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
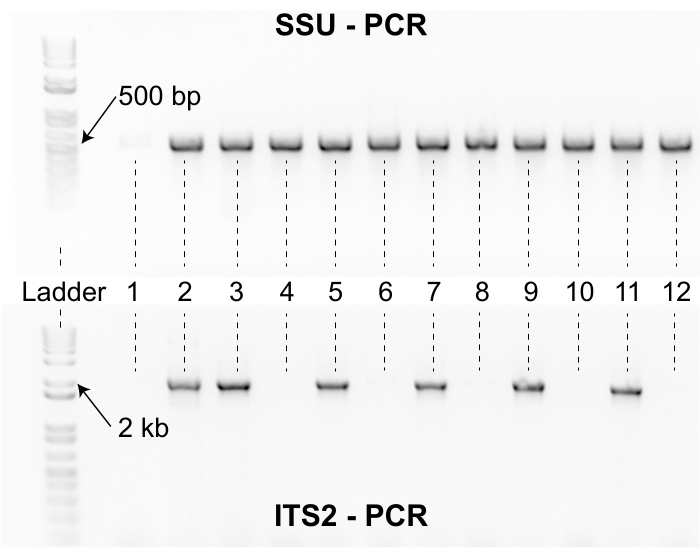
איור 8: SSU ומוצרי PCR של ITS2. הג'ל העליון מציג מוצרי PCR שנוצרו עם פריימר SSU להגדיר עבור 12 דגימות מייצגות. סולם דנ"א כלול בצד שמאל כהתייחסות. מוצרי SSU PCR עבור נמטודות Caenorhabditis הם כ 500 bp אורך. דגימות 2-12 מוגבר עם ערכת פריימר SSU אבל מדגם אחד לא. היעדר אמפליקון SSU 500 bp עבור מדגם אחד עולה כי חומר תמוגה היה באיכות ירודה ואת המדגם חייב להיות מחדש lysed. הג'ל התחתון מציג מוצרי PCR שנוצרו עם ערכת הפריימר ITS2 לאותן 12 דוגמאות המוצגות בג'ל העליון. הסולם והדגימות נמצאים באותה כיוון עבור שני הג'לים. שש מתוך 12 הדגימות לא הגבירו עם סט הפריימר של ITS2. הדגימות עם להקות SSU ו- ITS2 הן סנגר רצף ומזוהה על ידי דמיון רצף באמצעות אלגוריתם NCBI BLAST. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
קובץ משלים 1: תוויות C. קובץ PDF המכיל 2500 תוויות C ייחודיות. נא לחץ כאן כדי להוריד קובץ זה.
קובץ משלים 2: תוויות S. קובץ PDF המכיל 5,000 תוויות S ייחודיות. נא לחץ כאן כדי להוריד קובץ זה.
טבלה משלימה 1: חומרי שדה. רשימת אריזה של חומרים המשמשים בתחום לדגימת נמטודות. לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 2: מתכוני PCR ותנאי תרמוציקלר. טבלה של מתכוני PCR ותנאי thermocycler עבור ITS2 ו- SSU PCR. אנא לחץ כאן כדי להוריד טבלה זו.
טבלה משלימה 3: מתכוני מאגר אלקטרופורזה. מתכון לתמיסת חומצה אתילנדימינטטאצטטית (EDTA) של 0.5 M pH 8.0 ופתרון המאגר TRIS-אצטט-EDTA (TAE). לחץ כאן כדי להוריד טבלה זו.
Discussion
פרוטוקול זה מכיל שלבים קריטיים שיש לבצע בזהירות. לדוגמה, חשוב שצוותי השטח והבידוד יקפידו לבחור את פרויקט האיסוף הנכון ביישום לפני איסוף דגימות מהשטח או בידוד נמטודות מהדגימות במעבדה. במקרה שנבחר פרוייקט איסוף שגוי, רשומות הנתונים השגויות יתוקנו בצורה הטובה ביותר במסד הנתונים של נקודת המשען באמצעות כלי עריכת הרשומות באופן מקוון. תהליך זה יכול להיות מייגע עבור רשומות רבות שאינן במקומן. עם זאת, מסד הנתונים שומר את כל השינויים ברשומות כך שניתן יהיה לבצע ביקורת מלאה על רשומות איסוף ובידוד. הצעדים הקריטיים האחרים בפרוטוקול זה כוללים טיפול בדגימות מהשטח והנמטודות המבודדות מדגימות אלה. כדי להבטיח נמטודות Caenorhabditis לשרוד את הדגימה ואת שלבי המשלוח, הטמפרטורה של דגימות צריך להישמר בין 4 °C (60 °F) ו 25 °C (65 °F). טמפרטורות מעל 25 °C (60 °F) יכול לגרום סטריליות ב C. elegans14. ודא שדגימות מועברות מתיקי איסוף ללוחות איסוף תוך חמישה ימים במידת האפשר כדי למזער את האובדן לנמטודות. לאחר נמטודות מבודדות, זה קריטי כי הם genotyped ו cryopreserved לפני שהם מתים. קשה למצוא נמטודות חיות על S-צלחות כי הם יותר משבועיים עד שלושה שבועות כי זיהום פטרייתי וחיידקי יכול להפוך את S-לוחות עוינים.
ניתן לשנות פרוטוקול זה בקלות כדי להתאים לסוגים שונים של נתונים שחוקרים עשויים לרצות לאסוף בזמן שהם בשטח. לדוגמה, קל להתאים אישית את היישום 'דגימת שדה Nematode' עם שדות הזנת נתונים חדשים באמצעות ממשק המשתמש ב- GUI המקוון של Fulcrum לעריכת יישומים. יתר על כן, חבילת ניתוח הנתונים, easyFulcrum, יכולה להכיל עריכות אלה בעת עיבוד הנתונים החדשים15. שינוי נוסף שמשתמשים עשויים למצוא מושך הוא להשתמש בשיטת דגימה שונה בתחום. במקום לדגום מצעים נפרדים, החוקרים עשויים לרצות לדגום אזורים גדולים יותר המכילים סוגי מצעים מרובים. דגימות גדולות אלה מעובדות בצורה הטובה ביותר במעבדה באמצעות משפך Baermann או שיטות חילוץ מגש13. חשוב לציין, השימוש בתוויות C ותוויות S עדיין חל על טכניקות אלה ולכן תואמים ליישומים ניידים.
המגבלות העיקריות של פרוטוקול זה מתייחסות לזמן הטיפול בנמטודות לפני הבידוד במעבדה. ראשית, זמן הפיגור בין איסוף מדגם לבידוד נמטודות אינו מאפשר לתעד את השלבים ההתפתחותיים של נמטודות על מדגם נתון בזמן האיסוף. שנית, התדירות של הזכרים וההתפרצויות בטבע הן שאלות אבולוציוניות מרכזיות להעצמת נמטודות Caenorhabditis10. שיטה זו אינה מתאימה לטיפול בשאלות אלה מכיוון שסביר להניח שנמטודות עברו דורות רבים לפני הבידוד. בידוד מאוחר פירושו שראיות ישירות לתדירות הגברית בטבע הן בלתי אפשריות. יתר על כן, העיכוב הרב-דורי במהלך שלבי הגנוטיפינג פירושו שראיות גנומיות של outcrossing (הטרוזיגוסיטיות) יישחקו לפני שניתן יהיה לרצף זן נמטודות. כדי לזהות הטרוזיגוסיות בטבע, הצאצאים המיוצרים על ידי נמטודה מבודדת ישירות מהטבע משמשים לרצף2. מגבלה פוטנציאלית נוספת של פרוטוקול זה היא כי הוא מוטה כלפי זיהוי של Selfing Caenorhabditis. הסיבה לכך היא נמטודות מבודדות של מינים עצמיים יש סיכוי גדול יותר להתרבות מאשר outcrossers חובה, אשר יתרבו רק אם נקבה מופרית מבודדת.
שיטת איסוף זו מבוססת על פרוטוקולי איסוף קיימים13,14. ההתקדמות העיקרית של טכניקה זו היא השימוש בטכנולוגיה ניידת ותוכנה מותאמת אישית כדי להקל על הארגון של כמויות עצומות של נתונים אקולוגיים ומולקולריים הקשורים לפרויקטי איסוף בקנה מידה גדול. הנתונים האקולוגיים שנוצרו באמצעות פרוטוקול איסוף זה יכולים לשמש כדי לענות על שאלות יוצאות דופן עבור אוכלוסיות טבעיות של מינים Caenorhabditis. לדוגמה, נתונים שנוצרו בשיטה זו שימשו לגילוי העדפות נישה עבור המינים ברחבי איי הוואי. יתר על כן, על ידי ריצוף הגנום של נמטודות קריופ שמורות, חוקרים יכולים לחקור כיצד דפוסים של וריאציה גנטית מתואמים עם נתונים אקולוגיים. מחקר מסוג זה יכול לחשוף חתימות של הסתגלות מקומית באוכלוסיות Caenorhabditis ולספק תובנות חשובות על הרלוונטיות של וריאציה גנטית בהקשרים טבעיים8. כדי לקבל הבנה תפקודית של גנים רבים ב נמטודות Caenorhabditis, מחקרים אקולוגיים נדרשים ככל הנראה11. אפילו עבור C. elegans חלק גדול של גנים חסר ביאורים פונקציונליים, למרות שזה היה החיה הרב תאית הראשונה להיות רצף ואחד החיות נחקרו ביסודיות ביותר על פני כדור הארץ. פרוטוקול אוסף זה פותח כדי לעזור לטפל בפער ידע זה על ידי הקלה על האוסף של נמטודות Caenorhabditis בר וחקר האקולוגיה שלהם ואת המגוון הגנטי הטבעי.
Disclosures
המחברים אינם מדווחים על ניגודי עניינים.
Acknowledgements
מחקר זה נתמך על ידי קרנות סטארט-אפ מאוניברסיטת נורת'ווסטרן ופרס הקריירה של הקרן הלאומית למדע (IOS-1751035), שניהם הוענקו ל- E.C.A.
Materials
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
References
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved