Method Article
Kendinden Çıkan Caenorhabditis Nematodlarının Ekolojik Araştırmalarını Gerçekleştirmek için Yüksek Oranda Ölçeklenebilir Bir Yaklaşım
Bu Makalede
Özet
Bu protokol, kendi kendine Caenorhabditis nematodlarının büyük ölçekli ekolojik araştırmalarını gerçekleştirmek için kullanılabilir. Bu yöntemin birincil avantajı, doğadan toplanan nematodlarla ilişkili ekolojik ve moleküler verilerin verimli bir şekilde düzenlenmesi ve analiz edilmesidir.
Özet
Caenorhabditis elegans , biyolojideki en önemli model organizmalardan biridir, ancak son zamanlarda araştırmacılar doğal ekolojisine odaklanmışlardır. C. elegans hakkındaki bilgilerin doğal bağlamındaki göreceli seyrekliği, doğadaki küçük nematodun tanımlanmasında yer alan zorluklardan kaynaklanmaktadır. Bu zorluklara rağmen, C. elegans'ın ekolojisine giderek daha fazla odaklanmak, laboratuvar dışındaki yaşamı hakkında çok sayıda yeni bilgiye neden olmuştur. Doğada C. elegans için yoğunlaşan araştırmalar, birçok yeni Caenorhabditis türünün keşfine katkıda bulundu ve konjenerik nematodların, çürüyen bitki materyali ile ilişkili mikrobiyal çiçeklerle beslendikleri vahşi doğada sıklıkla birlikte yaşadıklarını ortaya koydu. Yeni türlerin tanımlanması, erkeklerin androdioecious çiftleşme sisteminin ve kendi kendini dölleyen hermafroditlerin Caenorhabditis'te bağımsız olarak üç kez geliştiğini de ortaya koymuştur. Diğer iki kendini-bilen tür, C. briggsae ve C. tropicalis, C. elegans'ın deneysel avantajlarını paylaşır ve kendi kendine döllenme de dahil olmak üzere önemli özelliklerin mekanik temeline ilişkin karşılaştırmalı çalışmalar yapılmasını sağlamıştır. Bu ilerlemelere rağmen, bu önemli türlerin ekolojisi ve doğal çeşitliliği hakkında öğrenilecek çok şey var. Örneğin, genlerinin çoğu için hala işlevsel bilgiden yoksunuz, bu da yalnızca doğal ekolojilerinin anlaşılmasıyla elde edilebilir. Caenorhabditis nematodlarının ekolojik araştırmalarını kolaylaştırmak için, nematodları vahşi doğadan toplamak için oldukça ölçeklenebilir bir yöntem geliştirdik. Yöntemimiz, araştırmacıların vahşi doğadan nematodları toplama, ilişkili ekolojik verileri kaydetme ve moleküler barkodları kullanarak vahşi nematodları tanımlama yeteneklerini geliştirmek için mobil veri toplama platformlarından, bulut tabanlı veritabanlarından ve R yazılım ortamından yararlanır.
Giriş
Son yirmi yıl, Caenorhabditis nematodlarının ekolojisine artan bir ilgi getirmiştir. Bu çalışmalardan, serbest yaşayan Caenorhabditis türlerinin hem ılıman hem de tropikal bölgelerdeki geçici mikro habitatlardan izole edilebildiğini, burada bazen de sempatry'de ayrışan bitki materyali ile ilişkili mikrobiyal çiçeklerle beslendiklerini biliyoruz1,2,3,4,5,6,7,8 . Ayrıca, cinste kendi kendine döllenmenin yakınsak evriminin üç kez gerçekleştiğini ve kendiliğinden, C. briggsae, C. elegans ve C. tropicalis9,10 için baskın üreme şekli olduğunu öğrendik. Bu benciller arasında C. elegans, Dünya'da en çok çalışılan hayvanlardan biridir ve araştırmacılar tarafından biyolojide kritik ilerlemeler yapmak için kullanılmıştır. Daha da önemlisi, diğer kendikendine özgü Caenorhabditis türleri, C. elegans'ın deneysel avantajlarının çoğunu paylaşır ve cinste karşılaştırmalı çalışmaları hızla ilerletmektedir. Bununla birlikte, bu nematodların vahşi doğadaki gizemli doğası, ekolojilerini ve doğal çeşitliliğini incelemeyi zorlaştırır, bu da genlerinin biyolojik işlevlerini ve evrimin türler arasındaki genetik çeşitliliği nasıl şekillendirdiğini anlamak için kritik öneme sahiptir10,11.
Vahşi doğada Caenorhabditis nematodlarının ekolojisini incelemenin en büyük zorluğu küçük boyutlarıdır; yetişkin nematodlar genellikle 1 mm uzunluğunda veya daha azdır. Bu zorluk, araştırmacıların vahşi doğadaki substratları örneklemelerini ve vahşi doğada hayvanları gözlemleme yeteneği olmadan ilgilenilen nematodları laboratuvardaki substratlardan ayırmaya çalışmalarını gerektirir. Eğitimli uzmanlar bile, kendi kendine hareket eden Caenorhabditis nematodlarını mikroskop altında diğer serbest yaşayan nematodlardan ayırt etmeyi zor bulduklarından, nematodlar tipik olarak substrattan çıkarılır, izole edilir ve yerleşik moleküler barkodlar kullanılarak dizi kimliği ile tanımlanmadan önce çoğalmaya bırakılır3,12,13,14 . Her bir nematodu bu şekilde işlemek için gereken zaman ve çaba örgütsel bir zorluk teşkil etmektedir, çünkü araştırmacılar laboratuvarda izole edilen her nematodun kimliğini tam substrata ve sahada örneklenen ilgili ekolojik verilere kadar izleyebilmelidir. Burada, sahadan kendi kendine gelen Caenorhabditis nematodlarını verimli bir şekilde toplamak ve tanımlamak ve bu izolatları ilişkili mekansal ve ekolojik verileriyle yüksek ölçekte sadık bir şekilde bağlamak için adım adım bir süreç açıklıyoruz.
Bu toplama yöntemi, mobil veri toplama platformlarını, bulut tabanlı veritabanlarını ve R yazılım ortamını kullanarak ekolojik araştırmaların ölçeğini ve doğruluğunu artırır. Fulcrum, çoğu mobil cihazla çalışan ve kullanıcıların konum tabanlı verileri toplamak ve düzenlemek için özel uygulamalar oluşturmasına olanak tanıyan özelleştirilebilir bir veri toplama platformudur (https://www.fulcrumapp.com). Bu protokol, sahadan mekansal olarak açık ekolojik verileri düzenlemek ve bu verileri laboratuvarda izole edilen nematodların kimliğiyle doğru bir şekilde ilişkilendirmek için özelleştirilmiş veri toplama uygulamalarının nasıl kullanılacağına dair ayrıntılı talimatlar sağlar. Protokol ayrıca, yerleşik moleküler barkodları kullanarak kendi kendine Caenorhabditis nematodlarının nasıl verimli bir şekilde tanımlanacağını da açıklar. Bu yöntemlerden elde edilen veriler, doğal Caenorhabditis popülasyonlarının ekolojisini ve genetik çeşitliliğini keşfetmek için beraberindeki R yazılım paketi easyFulcrum15 ile basit ve tekrarlanabilir bir şekilde işlenebilir.
Protokol
1. Koleksiyon hazırlama
- Caenorhabditis nematodlarını araştırmak için bir yer belirleyin.
NOT: Çoğu ılıman bölgede, C. elegans ve C. briggsae tarım alanları veya kırsal ve kentsel bahçeler gibi insanla ilişkili habitatlardan kolayca izole edilebilir1. Subtropikal ve tropikal bölgelerde, C. briggsae, C. elegans ve C. tropicalis, yukarıda listelenen insanla ilişkili habitatlarda, bazen birbirine yakın yerlerde bulunabilir. Bununla birlikte, C. elegans tropik habitatlardaki diğer türlerden daha serin ve daha kuru habitatları tercih ediyor gibi görünmektedir7,8. Türlerin her biri, insanlarla ilişkili olmayan vahşi habitatlardan da izole edilebilir, ancak bu habitatlar daha az sıklıkla örneklenir. - Mobil veri toplama uygulamalarıyla toplama ve yalıtım verilerini düzenlemek için bir Dayanak noktası projesi oluşturun.
- Ücretsiz eğitim sözleşmesini kullanarak Fulcrum ile çevrimiçi olarak bir hesap oluşturun16. APP EKLE düğmesine17 tıklayarak Nematod Alan Örnekleme uygulamasını bir Fulcrum hesabına ekleyin.
- Uygulama EKLE düğmesine18 tıklayarak Nematod İzolasyon uygulamasını bir hesaba ekleyin.
NOT: Bir konuma yapılan her seyahatin, 2020FebruaryAustralia gibi 'YearMonthLocation' adlandırma kuralı kullanılarak toplama projesi olarak düzenlenmesi önerilir.
- Koleksiyon projesine erişim izni vermek için kullanıcıları Fulcrum hesabına ekleyin. Her kullanıcının projeye katılmak için Fulcrum mobil uygulamasını indirdiğinden emin olun.
- Mobil uygulama ile koleksiyonları (C etiketleri) ve nematod izolasyonlarını (S etiketleri) izlemek için bir dizi QR kodu etiketi yazdırın. C etiketlerini fermuarlı plastik torbalara takın, etiketli torbaları 25'li gruplar halinde yuvarlayın ve paketleme için lastik bir bantla sarın. Laboratuvarda kullanılmak üzere S-etiket setini saklayın.
NOT: Bu protokol boyunca, koleksiyonlar (sahadan alınan alt tabakalar) torbalarda veya plakalarda bulunur ve C etiketleriyle etiketlenir. İzole nematodlar S-etiketleri ile etiketlenmiştir. C etiketleri benzersiz koleksiyonları tanımlamak için kullanılır ve S-etiketleri benzersiz nematod izolatlarını tanımlamak için kullanılır. Bu iki etiket türü, belirli bir koleksiyon (C-etiketi) ile Fulcrum veritabanındaki bu koleksiyondan yalıtılmış nematodlar (S-etiketleri) arasındaki bağlantıyı kurmak için kullanılır. Bir koleksiyon projesi için C etiketleri olarak iki kat daha fazla S-etiketi yazdırın, çünkü koleksiyon başına ortalama iki nematod izole edilir. Gerekirse daha sonra daha fazla S-etiketi yazdırılabilir. Ekte 2.500 benzersiz C etiketi (Ek Dosya 1) ve 5.000 benzersiz S etiketi (Ek Dosya 2) verilmiştir. - Koleksiyonlar için 10 cm NGMA plakaları ve nematodları izole etmek için 3,5 cm NGMA plakaları hazırlayın. Koleksiyon başına bir adet 10 cm'lik plaka ve en az iki adet 3,5 cm'lik plaka yapın21. Bu plakalar, belirlenmiş protokolleri takiben Escherichia coli suşu OP50 ile tohumlanır. Kullanmadan önce plakaları 4 ° C'de en fazla 1 ay saklayın.
2. Alan koleksiyonu
NOT: Caenorhabditis nematodları çoğunlukla meyveler, fındıklar, tohumlar, baklalar, çiçekler, saplar, bitkisel çöpler ve kompostlar dahil olmak üzere çürüyen bitkisel maddelerden izole edilir1,5,6,8. En iyi substratlar çürümüş ve meyve veya çiçek olarak neredeyse tanınamaz; Çok kuru veya ıslak olan yüzeylerden kaçının (Şekil 1). Substratlar, çiftler halinde çalışarak sahadan en verimli şekilde toplanır. Temassız kızılötesi termometreye sahip olan kişi, toplama için bir substrat seçecek ve numuneyi toplarken, ortakları toplama verilerini kaydetmek için Fulcrum'daki Nematod Alan Örneklemesi uygulamasını kullanacaktır. Kollektör çifti, istenen sayıda numune toplanana kadar bu işlemi tekrarlayacaktır. Saha çalışması için gerekli malzemelerin listesi (Ek Tablo 1)'de bulunmaktadır.
- Fulcrum mobil uygulamasını açın, açılır menüden Nematod Field Sampling öğesini seçin. Projede yeni bir kayıt başlatmak için + tuşuna basın (Şekil 2A). Alt tabakanın fotoğrafını çekin.
- Adım 1.2'de yapılan doğru toplama projesini seçmek için üst ortadaki kutuya tıklayın (Şekil 2B). Koleksiyon kaydının altındaki C etiketi alanına dokunun ve istem göründüğünde Tara'yı seçin. Mobil cihaz kamerasını kullanarak toplama çantasındaki barkodu tarayın , ardından ekranın sağ üst köşesindeki Bitti'ye dokunun.
- Alt tabaka alanına dokunun ve açılır menüden bir alt tabaka türü seçin. Alt Tabaka Notları alanına dokunup notları el ile girerek alt tabaka hakkında notlar ekleyin.
- Açılır menüden bir manzara seçin. Örnekleme alanını en iyi temsil eden manzarayı seçin.
- Bir gökyüzü görünümü seçin. Gökyüzü görünümünü seçerken, örnekleme alanındaki gökyüzü görünürlüğünü açıklayın (örneğin; Ağaçlardan veya diğer yapılardan engellenmiş görünümler olmadan tam gökyüzü görünümü = tam).
- Temassız termometreyi kullanarak substratın yüzey sıcaklığını ölçün ve substrat sıcaklık alanındaki değeri kaydedin.
NOT: Sıcaklığı kaydederken temassız termometreyi alt tabakadan en fazla 14 inç uzakta tutun. - El tipi cihaz ile ortam sıcaklığını ve nemini ölçün ve bu verileri uygun alanlara kaydedin.
NOT: Ortam sıcaklığı ve nem cihazının Beklemede olmadığını kontrol edin. Ölçü birimi, düğme serbest bırakıldığında değişecektir. Düzensiz okumaları önlemek için cihazı dış cebinde tutun. - Ekranın sol üst köşesindeki Kaydet'e dokunarak kaydı Dayanak Noktası'na kaydedin.
- Substratı almak için bir "eldiven" olarak kullanmak üzere toplama torbasını ters çevirerek çubuklar veya diğer sert parçalar olmadan substratın yaklaşık bir çorba kaşığı toplayın, ardından torbayı kapatın. Numune özellikle nemliyse torbaya bir kağıt havlu koyun.
NOT: Sıcak iklimlerde, koleksiyonları serin tutmak için torbaları soğutucu paketlere sahip yumuşak soğutuculara yerleştirin. - Tüm numuneler gün boyunca toplandıktan sonra, toplama ekipmanını temizleyin, pilleri problardan çıkarın, pilleri şarj edin, dondurucu paketlerini yeniden dondurun. Nematod Field Sampling uygulamasının sol üst köşesindeki Sync düğmesine dokunarak Fulcrum toplama verilerini senkronize edin.
NOT: Yüklemeler güçlü bir hücresel bağlantı olmadan birkaç dakika sürebilir, bu nedenle WiFi erişimini beklemek en iyisi olabilir. Veriler mobil cihazlarda kalır ve bulutla senkronize edilir. - Numuneleri bir gecede nakliye kutusuna yerleştirerek bir ev kurumuna gönderin. Yüklerin taşındığı günlerde paketler göndererek numunelerin 11 °C'nin altındaki veya 25 °C'nin üzerindeki sıcaklıklara maruz kalma süresini en aza indirin.
NOT: Çoğu nakliye tesisi, uzak yerlerde hafta sonları bir gecede kargo göndermez.
3. Laboratuvarda saha koleksiyonlarının kaplanması
NOT: Bu bölümde, numunelerin etiketli toplama torbalarından etiketli plakalara aktarımının nasıl organize edileceği açıklanmaktadır. Numuneler bir gecede yapılan bir gönderiden veya doğrudan sahadan gelebilir.
- Koleksiyonların gönderisini alın ve kırık torbalar veya diğer hasar kanıtları için kontrol edin. Torbalar kırılırsa, malzemeyi atın ve kırılmamış toplama torbalarını% 70 etanol ile temizleyin; Etanol içeren torbadaki C etiketinden kaçının, çünkü etiketin rengini değiştirir ve okunmasını zorlaştırır.
- Her fermuarlı torba için, torbanın üzerindeki C etiketini not edin ve OP50 bakterileriyle lekelenmiş 10 cm'lik bir plakanın kapağına eşleşen bir C etiketi takın.
NOT: Etiketli 10 cm'lik plakalar, protokolün geri kalanı için 'C-plakaları' olarak adlandırılır. Numuneleri organize etmenin en kolay yolu, toplama torbalarını üstte eşleşen C-plakası bulunan bir laboratuvar tezgahına yerleştirmektir (Şekil 3). - Her koleksiyon için, temiz bir plastik kaşık kullanarak toplama torbasından C plakasına yaklaşık bir çorba kaşığı numune aktarın. Numuneyi bakteri çiminin etrafına hilal veya halka şeklinde ekleyin, bakteri çimini tamamen örtmeyin (Şekil 4).
NOT: Yemek kaşığı, kullanılmadığı zamanlarda %95 etanollük bir beherin içine koyarak temiz tutun. Ek numuneleri aktarmadan önce çorba kaşığı kurutmak için bir kağıt havlu kullanın. - Koleksiyonların toplama torbalarından C plakalarına aktarıldığı zamanı kaydedin ve bölüm 4'teki nematodları izole etmeye çalışmadan önce C plakalarını oda sıcaklığında (RT) en az 24 saat tutun.
4. Nematodların koleksiyonlardan izole edilmesi
- Mobil cihazda Fulcrum uygulamasını açın ve uygulama menüsünden Nematod İzolasyonu'nu seçin (Şekil 5A). Sağ alttaki + simgesine dokunarak yeni bir yalıtım kaydı yapın (Şekil 5B).
- Yeni yalıtım kaydı ekranında, üst ortadaki kutuda görüntülenen proje adını işaretleyerek doğru toplama projesini onaylayın. Yanlış proje görüntülenirse, doğru projeye geçmek için proje adına dokunun (Şekil 5C).
- Nematodların izole edildiği örnekle ilişkili C etiketini bulmak için C-label alanının altındaki Seç düğmesine dokunun (Şekil 5D). Ara simgesine dokunun, ardından C plakasındaki C etiketli QR kodunu cihaz kamerasıyla taramak için Tara simgesine dokunun. QR kodu tarandıktan sonra, C etiketi alanında bir C etiketi kaydı görünür.
- Cihaz kamerasını açmak için Fotoğraflar alanındaki Kamera simgesine dokunun ve QR kodu görünür durumdayken C plakasındaki numunenin fotoğrafını çekmek için kullanın (Şekil 5E). İzolasyon ekranına dönmek için Bitti'ye dokunun.
NOT: Bu yalıtım kaydı fotoğrafları, alt tabakanın belirli özniteliklerini daha sonra keşfetmek için kullanılabilir. - C-plakasındaki nematodları aramak için diseksiyon mikroskobu kullanın. Numune üzerindeki nematodların varlığını kaydetmek için Worms on Sample alanına dokunun (Şekil 5F). C plakasında nematodlar varsa Evet'e , nematod yoksa Hayır'a dokunun.
- Yalnızca nematod izleri varsa İzler'e dokunun. Nematodlar yoksa, C plakasını parafilm haline getirin ve biyolojik tehlike kutusuna atın.
NOT: C plakasını biyolojik tehlike atık kutusunun üzerine ters çevirin ve numune alınan tüm substratları yerinden çıkarmak için plakanın arkasına hafifçe dokunun. Bu adım, C plakasındaki substratın altında bulunabilecek nematodları bulmayı ve izole etmeyi kolaylaştırır. - Nematodlar varsa, C plakasından beş adede kadar nematodu izole edin. Bir nematodu izole etmek için, bir platin tel toplama kullanarak bir nematodu C plakasından bir S plakasına aktarın. Mümkünse sağlıklı, gravid yetişkinleri izole edin. Bununla birlikte, yetişkinler bulunmazsa diğer aşamaları izole edin.
NOT: İzolasyondan sonra, en fazla beş S-plakasının her birinde tek bir nematod bulunacaktır. Bu S-plakalarını aynı C-plakasından izole nematodlarla birlikte, Dayanak Noktasına girene kadar diğer S-plakalarından uzakta düzgün bir yığın halinde birlikte organize edilmiş halde tutun. - Bu yalıtım için kullanılan S plakalarını girmek için S etiketli Plakalar alanına dokunun. Sağ alttaki + işaretine dokunun. S-etiketine dokunun ve ardından cihaz kamerasını açmak için Tara'ya tıklayın. S plakasındaki S-label QR kodunu taramak için cihaz kamerasını kullanın.
NOT: S-label kodunun plaka üzerindeki kodla eşleştiğinden emin olun. Eşleşirse, Bitti'ye dokunun. Eşleşmezse , İptal'e dokunun ve eşleşene kadar yeniden tarayın, ardından Bitti'ye tıklayın. Bazen yakındaki plakaların QR kodları yanlışlıkla taranır. - Her bir S-plakasını girdikten sonra, girişi sağ üstteki Kaydet düğmesiyle kaydedin. Giriş kaydedilmezse kaybolur. C plakasından izole edilen tüm nematodlar girilene kadar gerekirse daha fazla S etiketli plaka eklemek için sağ alttaki + işaretine dokunun. İzolasyon kaydı için tüm S etiketli plakaları ekledikten sonra, izolasyon kaydı ekranına geri dönmek için sol üstteki < düğmesine dokunun.
NOT: Hatalar çözülemediği için bir yalıtım kaydını iptal etmek için sol üstteki İptal'e tıklayın. Bu adım, kaydın kaydedilmeden atılıp atılamayacağını soran bir iletişim kutusu açar. İsterseniz, Evet, At'a tıklayın. - İzolasyon kaydında tüm bilgiler doğru şekilde eklendikten sonra sağ üstteki Kaydet düğmesine dokunun. Daha sonra S-plakalarını izole nematodlarla parafilm haline getirin ve bunları nematodlarla S-plakalarını tutmak için belirlenmiş bir alana koyun.
- C plakasını parafilm haline getirin ve biyolojik tehlike kutusuna atın. Tüm verileri Fulcrum'a yüklemek için Senkronize Et simgesine dokunun.
- Tüm S plakalarını alfasayısal düzende sıralayın, sonra S plakalarını karton kutulara yerleştirin. S plakalarının kapak tarafı aşağı ve parafilme alındığından emin olun. Dört adede kadar S plakasını kutuda tek bir konuma yerleştirin ve karton kutuyu proje adı, tarih, saat ve benzersiz bir kutu numarasıyla etiketleyin.
- Etiketli kutuları RT'de saklayın. Bu izolatlar proliferasyon açısından 48 saatte ve gerekirse tekrar 168 saatte kontrol edilecektir.
5. Fulcrum'dan S-plakalarının dışa aktarılması
NOT: Bu bölümde, yalıtım işleminde kullanılan S etiketlerinin Fulcrum proje veritabanından nasıl dışa aktarılacağı ayrıntılı olarak açıklanmaktadır. Bu S-etiketleri, bölüm 6-9'da sekans kimliği ile tanımlanırken çoğalan izodişi çizgileri izlemek için kullanılacaktır.
- Fulcrum web sitesinde oturum açın ve Nematod İzolasyonu uygulamasını seçin. Ekranın sol tarafındaki Dışa Aktarıcı'ya tıklayın. İstediğiniz projeyi seçmek için tıklatın ve Nematod İzolasyonu kutusunu işaretleyin. '.zip' dosyasını içeren bir nematode_isolation_s_labeled_plates.csv dosyası indirmek için İleri'yi tıklayın.
- 'nematode_isolation_s_labeled_plates.csv' dosyasını açın ve artan düzende 'S-etiketi' sütununa göre sıralayın (en küçük S-etiketi üstte olacaktır). Tüm S etiketlerini seçin ve e-tablodan kopyalayın.
- Bir web tarayıcısı kullanarak vahşi soyat genotipleme şablonu google sayfasına (wild_isolate_genotyping_template) gidin19.
- Genotyping Template (Genotipleme Şablonu) sekmesini sağ tıklayıp Yeni E-tabloya Kopyala seçeneğini belirleyerek bu google sayfasının bir kopyasını oluşturun. Yeni google sayfasını görüntülemek için E-Tabloyu Aç'ı seçin.
- Bu yeni sayfaya, dayanak noktası proje adının ardından 'wild_isolate_genotyping' (ör. "2020FebruaryAustralia_wild_isolate_genotyping) ile ad verin.
NOT: Bu sayfa, protokolün geri kalanında 'genotipleme sayfası' olarak adlandırılır.
- 'nematode_isolation_s_labeled_plates.csv' 's_label' sütunundan kopyalanan S etiketlerini, 's_label' başlıklı genotipleme sayfası sütununa yapıştırın. '1'ler için 's_label_repeat_error' sütununu kontrol edin. Bu sütundaki '1' değeri, S-etiketinin genotipleme sayfasında bir yerde çoğaltıldığı anlamına gelir. Çoğaltmalar keşfedilirse, ilerlemeden önce bunları araştırın ve düzeltin.
- Tüm S etiketleri için genotipleme sayfası 'isolation_box_number' sütununu doldurun.
6. S plakalarında çoğalma olup olmadığını kontrol edin
- İzolasyondan 48 saat sonra S plakalarında çoğalan hayvanları kontrol edin (zamanlamayı yönlendirmek için 4.11 adımındaki kutudaki son izolasyonun tarihini ve saatini kullanın).
NOT: Çoğalan nematodlar, S plakasındaki yavrularla karakterize edilir. - Bir S-plakası çoğalıyorsa, genotipleme sayfasındaki proliferation_48 sütununa '1' girin, sonra S-plakasını '48 saat çoğalma, kutu 1' etiketli bir kutuya taşıyın. Bir çoğaltma kutusuna maksimum 88 S plakası yerleştirin, ardından '48 saat çoğalma, kutu 2' etiketli yeni bir kutu doldurmaya başlayın. S etiketlerinin 48 saatlik çoğalma kutularında alfasayısal sırayla düzenlendiğinden emin olun.
NOT: Çoğalmayan S plakalarını atmayın; Bu plakalar izolasyon sonrası saat 168'de tekrar kontrol edilecektir. İsterseniz, bu S plakalarını '48 saat çoğalmaz, X kutusu' etiketli kutularda sayısal sırayla birleştirin, ancak yeni kutuda 168 saat denetiminin gerçekleşmesi gerektiğinde kaydetmeyi unutmayın. - 48 saatte çoğalan tüm S-etiketlerini belirledikten sonra, 48 saatte çoğalan S-plakaları için bölüm 7'ye geçin.
- İzolasyon sonrası 48 saatte çoğalmayan S plakalarını izolasyon sonrası 168 saatte tekrar kontrol edin.
- Bir S-plakası şimdi çoğalıyorsa, genotipleme sayfasındaki proliferation_168 sütununa '1' girin ve ardından S-plakasını '168 saat çoğalma, kutu 1' etiketli bir kutuya taşıyın.
- Bir çoğaltma kutusuna maksimum 88 S plakası yerleştirin, ardından '168 saat çoğalma, kutu 2' etiketli yeni bir kutu doldurmaya başlayın. S etiketlerini 168 saatlik çoğalma kutularında alfasayısal sırada düzenlediğinizden emin olun.
- 168 saat sonra çoğalmayan S plakalarını atın. 168 saatte çoğalan S-plakaları için bölüm 7'ye geçin.
7. İzodişi çizgilerin lizisi
NOT: Bu adım, çoğaltma kutularındaki S plakaları için lizis çalışma sayfalarının yazdırılmasına yardımcı olmak üzere google sayfalardaki veri filtresi aracını kullanır. Lizis çalışma sayfalarının amacı, personele tezgahtaki lizis şeridi tüplerinde S-etiketleri için doğru pozisyonları sağlamaktır.
- İstediğiniz proje için genotipleme sayfasını açın ve Cmd+A yazarak tüm hücreleri seçin. Her sütun başlığına bir filtre düğmesi eklemek için Veri > Filtre Oluştur'a tıklayın. Yalnızca genotiplendirilecek S-plakalarını görüntülemek için Filtre düğmelerini kullanın. Örneğin, 48 saatte çoğalan tüm S plakaları lize edilecekse: 'proliferation_48' sütunundaki Filtre düğmesine tıklayın ve '1'i seçin.
- Genotipleme google sayfası filtrelendikten sonra, çalışma sayfasına yazdırılacak S-etiketleri olduklarından emin olmak için görüntülenen S etiketlerinin listesini gözden geçirin.
- Genotyping google sayfasının 'strip_tube_number' sütununa, her 11 satırda bir benzersiz bir sayı girin.
- Bir projenin şerit tüp numaralarını 1'den başlayarak ve hiçbir zaman çoğaltılmamış olarak ardışık sırayla girin. "strip_tube_position", her şerit tüp numarası için 2 ile 12 arasında girin.
NOT: Lizis için 12 tüplü şerit tüpler kullanın. İlk pozisyon (strip_tube_position 1) kontrol olacaktır, ancak kontroller lizis çalışma sayfalarına eklenmez (yalnızca strip_tube_positions eklenir, 2-12). Lizis sırasında, pozitif kontrol suşu 'N2', pozitif bir kontrol olarak her çift numaralı şerit tüpünün 1. konumuna eklenecektir. Her tek numaralı şerit tüpünün 1. konumuna negatif kontrol olarak solucan eklenmez.
- Genotipleme google sayfasını daha fazla filtreleyerek yalnızca S-etiketlerini lize edilecek bir çoğalma kutusuna ekleyin, ardından 's_label' ile 'lysis_notes' sütunlarını seçin. Lize edilecek her çoğalma kutusu için bir lizis çalışma sayfası yazdırın.
- Print (Yazdır) alanındaki açılır menüye tıklayın ve Selected Cells (Seçili Hücreler) öğesini seçin. Sağ üstteki İleri'ye tıklayın, ardından çoğaltma kutusunun lizis çalışma sayfasını yazdırmak için iletişim kutusunu kullanın.
- Her çoğaltma kutusu için bir lysis çalışma sayfası yazdırmak üzere 7.3-7.5 arasındaki adımları yineleyin.
NOT: Her proliferasyon kutusu, sekiz adet 12 delikli şerit tüpüne karşılık gelen 88 adede kadar S plakası tutar. - Lize edilecek tüm numuneler için 12 delikli şerit tüpler hazırlayın. Bir şerit tüpünü, lizis çalışma sayfasında atanmış benzersiz bir 'strip_tube_number' ile etiketleyin. Bu etiket, ayrıldıklarında karışıklığı önlemek için kapak şeridine ve şerit tüpüne yazılmalıdır. EVEN şerit tüpleri, 1. konumda pozitif bir kontrole (N2 solucanları) sahiptir. ODD şerit tüpleri, 1. konumda negatif bir kontrole (solucan yok) sahiptir.
- Tüm numuneler için yeterli lizis tamponu (100 mM KCl, 20 mM Tris pH 8.2, 5 mM MgCl2, %0.9 IGEPAL, %0.9 Ara 20, 0.4 mg/mL'lik son konsantrasyona proteinaz K eklenmiş %0.02 jelatin) oluşturun ve pipet hatası için %5 ekstra ilave ekleyin. Gerektiği gibi ölçeklendirin.
NOT: Lizis tamponu, proteinaz K hariç tüm bileşenlerin birleştirilmesi ve -20 °C'de 10-50 mL alikotlarda dondurulmasıyla en iyi şekilde hazırlanır. Alikotları çözün ve kullanmadan önce 4 ° C'de tutun; Kullanmadan hemen önce, proteinaz K ekleyin ve iyice karıştırın. Çalışırken lizis tamponunu buz üzerinde tutun. - Basılı lizis çalışma sayfasını kılavuz olarak kullanarak belirli bir şerit tüp için S plakalarını düzenleyin.
- Bir şerit tüpün kapağını açın ve tekrarlanan bir pipetleyici ile her kapağa 8 μL lizis tamponu ekleyin. Lizis tamponunu bir seferde bir kapak şeridine ekleyin, çünkü lizis tamponu RT'de bırakılırsa ve açıkta bırakılırsa buharlaşır. Kaynak plakalardan (pozitif kontroller için S-plakası veya N2 stok plakası) 3-5 hayvanı lizis çalışma sayfasında belirtilen uygun kapak konumlarına seçin. Lizis çalışma sayfasının lysis_notes bölümünde lizise 5'ten az solucan eklenmiş herhangi bir S plakası için notları kaydedin.
- Nematodları şerit tüpünün her bir konumuna yükledikten sonra, kapak şeridini tekrar şerit tüpüne yerleştirin. İşaretli kapağı (konum 1) işaretli tüple (konum 1) eşleştirin. Kapatıldıktan sonra, nematodlar tüpün dibinde olana kadar şerit tüpünü kısa bir süre santrifüj edin.
- Şeridi tamamen donana kadar (en az 10 dakika) -80 °C dondurucuya yerleştirin. Tüm şeritlerde lizis için nematodlar eklenene kadar 7,9 ile 7,11 arasındaki adımları tekrarlayın. Tüp şeritlerini sayısal sırayla düzenleyin.
- Şerit tüp setlerini çıkarın ve lizis programını bir termosikletörde çalıştırın: 60 ° C'de 1 saat, 95 ° C'de 15 dakika, 12 ° C'de tutun. Lizis programı tamamlandığında, numuneleri RT'de 15 s için 300 x g'da döndürün ve lizatları 1 haftaya kadar -80 ° C'de saklayın.
- 96 delikli plaka tutucuları kullanarak tüp şeritlerini sayısal sırayla düzenleyin ve çoğalma kutusu numarası, şerit tüp numarası aralığı, tarih ve araştırmacının baş harflerini içeren bir etiket ekleyin. 'lysis_date' ve 'lysis_notes' genotipleme sayfası sütunlarını lizis çalışma sayfasındaki bilgilerle güncelleyin.
8. SSU ve ITS2 dizilerinin PCR'si
NOT: Bu bölümde, her lize S-plakası için iki ayrı PCR'nin nasıl gerçekleştirileceğine ilişkin talimatlar sağlanacaktır. İlk astar seti, 18S rDNA küçük alt birim geninin (SSU) 500-bp fragmanını güçlendirir; oECA1271 = ileri astar TACAATGGAAGGCAGCAGGC, oECA1272 = ters primer CCTCTGACTTTCGTTCTTGATTAA 12. Bu PCR, şablon DNA'nın kalitesini kontrol etmek için kullanılır. PCR, neredeyse tüm nematod türleri için SSU bölgesini güçlendirir. SSU PCR yükseltemezse, bu sonuç lizis kalitesinin düşük olduğunu ve lizisin bu S-plakası için tekrarlanması gerektiğini düşündürmektedir. İkinci astar seti, 5.8S ve 28S rDNA genleri (ITS2) arasındaki dahili transkribe edilmiş ara parça bölgesinin 2.000-bp fragmanını güçlendirir; oECA1687 = ileri astar CTGCGTTACTTACCACGAATTGCARAC, oECA202 = ters primer GCGGTATTTGCTACTACCAYYAMGATCTGC3. ITS2 PCR ürünü Sanger'ın sıralandığı ve sekans, Caenorhabditis cinsindeki nematodları dizi benzerliği ile tür seviyesine tanımlamak için kullanılır.
- Genotipleme sayfasındaki filtreleme aracını kullanarak yalnızca PCR için kullanılacak S-etiketlerini görüntüleyin.
- pcr_plate_number güncelleştirin ve genotipleme sayfasındaki sütunları pcr_well. Lizis malzemesinin bozulmasını önlemek için, SSU ve ITS2 PCR'ler aynı anda çalıştırılır.
- ITS2 ve SSU PCR'ler için ayrı plakalarda ayrı reaksiyonlar olsa bile aynı pcr_plate_number kullanın. 'SSU' veya 'ITS2' etiketleriyle ayırt edilecekler.
- Sekiz veya daha az şerit tüpe bir pcr_plate_number atayın (96 delikli PCR plakasının sırası başına bir şerit tüpü, artan düzende düzenlenmiş, örneğin, üstteki en düşük şerit tüp numarası). Ardından, şerit tüplerindeki her bir S-etiketine bir pcr_plate_well atayın.
NOT: Şerit tüpler, A satırına atanan en düşük şerit tüp numarası ve H satırındaki en yüksek sayı ile artan düzende düzenlenmiştir. Bu nedenle, 1 numaralı şerit tüpü, pozisyon 1, PCR plaka numarası 1, kuyu A01'e atanacaktır. - PCR için kullanılacak numuneleri yerleştirmek için 96 delikli PCR plakalarını etiketleyin. Her PCR plakasını aşağıdaki bilgilerle etiketleyin: proje adı, PCR tipi, PCR plaka numarası ve PCR tarihi (örneğin, 2020FebruaryAustralia_SSU_1_20200304). Ayrıca, plakayı her sıraya yüklenecek şerit tüp numaralarıyla etiketleyin.
- Lizis malzemesini -80 °C dondurucudan çıkarın ve lizis malzemesini içeren şerit tüpleri buz üzerinde çözün. Lizis malzemesi çözülürken, ITS2 ve SSU master karışımlarını buz üzerinde ayrı tüplerde hazırlayın. SSU ve ITS2 PCR tarifleri Ek Tablo 2'de bulunur.
NOT: Pipetleme hatasına izin vermek için her 96 delikli plaka için 100 reaksiyonluk PCR ana karışımı hazırlayın. Büyük hacimler kullanılacaksa ana karışımı tutmak için 15 mL veya 50 mL konik kullanın. - Vortex ana karışımı, Taq karışım boyunca dağıtılana kadar nazikçe karıştırın. Karıştırıldıktan sonra, ana maddenin aliquot 38 μL'si buz üzerindeki PCR plakalarının uygun kuyucuklarına karışır. Ana karışımı PCR plakalarına aktarmak için steril tek kullanımlık v tabanlı oluklar ve 12 delikli çok kanallı pipet kullanın.
- Lizis malzemesini kapaklardan çıkarmak için çözülmüş lizis şeridi tüplerini aşağı doğru döndürün. İlk PCR plakasına yüklenecek tüm şerit tüplerin kapaklarını dikkatlice çıkarın. PCR plakasındaki uygun kuyucuğa 2 μL lizat eklemek için düşük hacimli çok kanallı pipet (12 delikli veya 8 delikli) kullanın. 2 μL'yi çıkarmadan önce lizatı bir kez yukarı ve aşağı yavaşça pipetleyin.
NOT: Aktarımdan önce lizis içerdiklerinden emin olmak için ipuçlarını kontrol edin. İpuçlarını satırlar veya sütunlar arasında değiştirmeyi unutmayın. - PCR plakasını PCR yapışkan folyo ile örtün ve sıkı bir sızdırmazlık oluşturmak için bir rulo kullanın. Folyo uygulandıktan sonra, PCR plakalarını bir santrifüjde kısaca döndürün. Termosiklette çalışmaya hazır olana kadar plakayı buz üzerinde tutun.
- PCR'leri uygun termosikler programı ile çalıştırın. SSU ve ITS2 PCR programlarının ayrıntıları için Ek Tablo 2'ye bakın.
- Tüm PCR'ler çalıştırılana kadar 8.4- 8.8 arasındaki adımları yineleyin.
- PCR reaksiyonları devam ederken, 100 mL% 1.5 agaroz jeli dökün. Her jel numuneleri veya tek bir PCR plakasını tutacaktır.
- 500 mL'lik bir şişeye 1,5 g agaroz ekleyin, ardından 100 mL 1x TAE tamponu ekleyin (Ek Tablo 3) ve karıştırmak için girdap yapın. Jeli çözmek ve soğutmak için mikrodalga.
- Çözelti soğutulduktan sonra, 5 μL 10 mg / mL ethidyum bromür çözeltisi ekleyin ve birleştirmek için karıştırın. Çözeltiyi dört adet 25 delikli taraklı bir döküm tepsisine dökün, böylece jel 96 numune ve jeldeki her sıra için bir merdiven barındırabilir.
NOT: Ethidium bromür güçlü bir mutajendir. Ethidium bromür kullanırken, bir laboratuvar önlüğü, kimyasallara dayanıklı eldivenler ve kimyasal güvenlik gözlükleri kullanın.
- PCR bitmeden hemen önce, tek kullanımlık bir oluğa 6x yükleme boyası ekleyin ve yeni 96 delikli PCR plakasının her bir kuyucuğuna 2 μL 6x yükleme boyası eklemek için çok kanallı bir pipet kullanın. Bu plaka, numuneleri jele yüklemek için kullanılacaktır. Tüm örnekleri barındırmak için bu plakalardan yeterince yapın.
- PCR'ler tamamlandığında, PCR plakalarını çıkarın ve RT'de 15 s için 300 x g'de kısaca santrifüj yapın. PCR plakalarını PCR ürünleri bir jel üzerinde tükenene kadar buz üzerinde saklayın.
- Ürünleri bir jel üzerinde çalıştırmak için, 2 μL 6x yükleme boyası içeren 96 delikli bir plakanın uygun kuyucuğuna her numunenin 5 μL'sini eklemek için 12 delikli çok kanallı bir pipet kullanın.
- Daha sonra bu karışımın 6 μL'sini yakın zamanda dökülmüş bir jelin her bir kuyucuğuna yükleyin. Jelin her sırasının ilk kuyucuğuna 6 μL 1 KB artı merdiven yükleyin.
NOT: Jelin kuyucuklarını doldurmak için, jelin ilk sırasındaki PCR plakasından A ve B Sırasının serpiştirilmesi gerekebilir. Karışıklığı önlemek için, gel_number ve gel_position her PCR örneği için genotipleme sayfasına kaydedin.
- Plakalarda kalan PCR'ye yeni bir folyo kapağı yerleştirin ve bunları 4 ° C'de saklayın. Bu reaksiyon ürünleri, adım 9'da sıralama için kullanılacaktır.
- PCR ürünlerini jel üzerinde 120 V'ta 20 dakika boyunca çalıştırın. Jeli görüntüleyin ve genotipleme sayfasının 'pcr_product_its2' ve 'prc_product_ssu' sütunlarına hangi S-etiketlerinin ITS2 ve / veya SSU PCR ürünleri verdiğini kaydedin. Bir grubun varlığını '1' ile işaretleyin; bant yok için '0' işaretini işaretleyin.
9. Nematodların Sanger dizilimi ve dizi PATLAMASI ile tanımlanması
NOT: Bu bölümde, ITS2 amplikonlarının S-etiketlerinden sıralanması, BLAST algoritmasının kullanılarak bu dizilerin Ulusal Biyoteknoloji Bilgi Merkezi (NCBI) veritabanına hizalanması ve S-plakalarındaki nematodları tanımlamak için BLAST sonuçlarının ayrıştırılması için talimatlar sağlanmaktadır.
- ITS2 pozitif olan her numune için, ileri astar oECA306 (CACTTTCAAGCAACCCGAC) kullanarak Sanger dizilimi için kalan ITS2 PCR ürününü kullanın. Genotipleme sayfasına her S-etiketinin 'sequencing_plate' ve 'sequencing_well' sütunlarını kaydederek sıralama çıktı dosyalarının bir S-etiketine kolayca bağlanmasını sağlayın.
- Sıralama platformundan her S-etiketi için .seq çıktı dosyalarını edinin. Bir projenin .seq dosyalarını, alt dizinlerde bulunan her sıralama toplu işlemi için .seq dosyalarıyla tek bir dizinde düzenleyin.
- Komut satırı arabirim aracını açın ve şu komutu girerek .seq dosyalarını içeren üst dizine gidin: cd . Zaten mevcut değilse, aşağıdaki komutu girerek tüm .seq dosyaları için birleştirilmiş bir FASTA oluşturun: dir in */; do cd $dir; for file in *.seq; do echo ">"$file; cat $file; done >>.. /all_seqs.fa; cd ..; bitti.
Not: Bu kod, proje dizinindeki tüm .seq dosyalarından 'all_seqs.fa' adlı birleştirilmiş bir FASTA dosyası oluşturur. Bu dosya, NCBI çevrimiçi nükleotid BLAST aracında, her bir S-etiketinin ITS2 dizisini NCBI'nin dizi veritabanına hızlı bir şekilde hizalamak için kullanılabilir. - Bir web tarayıcısında, NBCI BLAST websitesine20 gidin ve Dosya Seç düğmesine tıklayın. Yeni oluşturulan all_seqs.fa dosyasını seçin, ardından Biraz Benzer Diziler (BLASTn) düğmesine tıklayın. BLAST aramasına başlamak için sayfanın altındaki BLAST düğmesine tıklayın.
- Genotipleme sayfasını her S-etiketi için BLAST sonuçlarıyla güncelleştirin. Genotyping google sayfasını güncellemeyi kolaylaştırmak için filtre aracını kullanın. Her sütun başlığına bir filtre düğmesi eklemek için Veri > Filtre Oluştur'a tıklayın. BLAST sonuçlarıyla güncelleştirilecek sıralama plakalarını seçmek için sequencing_plate sütununu filtreleyin.
- Her bir S-plate ITS2 dizisinin sonuçlarını kontrol etmek için NCBI BLAST sonuçları sayfasındaki açılır menüyü kullanın (Şekil 6).
- BLAST isabeti olup olmadığını kontrol edin. * önekli açılır menüdeki bir dizi kimliğinin patlama isabeti yoktur. Bu S etiketleri için, genotipleme sayfasının < sütununa 'isabet yok >geçerli tarih manual_blast_notes' girin.
- Olası yeni bir Caenorhabditis türü olup olmadığını kontrol edin. Hizalamayı görselleştirmek için üst vuruştaki bağlantıya tıklayın (Şekil 6). En iyi isabet (1) bir Caenorhabditis türüyse, (2) hizalama dizinin merkezinde beşten fazla uyumsuzluk içeriyorsa ve (3) sorgu kapsamı %50'den büyükse, bu sonuç izolatın yeni bir Caenorhabditis türü olabileceğini düşündürmektedir (Şekil 7). Bu S-plakaları için, 'species_id' sütununda en üstteki BLAST isabetinin türleri, 'possible_new_caeno_sp sütununa' 1 ve 'olası yeni Caeno sp.' ile birlikte yüzde kimliği ile birlikte 'manual_blast_notes' sütununa girin (örneğin, 'olası yeni Caeno sp. %89 kimlik').
- Bir Caenorhabditis türüne PATLATAN S-plakası dizileri için, 'species_id' sütununa en üstteki BLAST isabetinin tam cinsini ve tür adını girin. Örneğin, 'Caenorhabditis elegans'.
- Caenorhabditis olmayan bir türe PATLAMA yapan S-plakası dizileri için, yalnızca üst BLAST vuruşunun cinsini ve ardından 'species_id' sütununa 'sp.' girin. Bu gösterim, izolatın adlandırılmış cins içinde bilinmeyen bir tür olduğu anlamına gelir. Örneğin, 'Oscheius sp.'.
NOT: ITS2 dizisi, Caenorhabditis cinsinin dışındaki tür düzeyindeki izolatları güvenilir bir şekilde tanımlamak için kullanılamaz3,13. - Genotipleme sayfasının 'make_strain_name sütununa' 1 girin, eğer 'species_id' = 'Caenorhabditis elegans', 'Caenorhabditis briggsae' veya 'Caenorhabditis tropicalis', VEYA 'possible_new_caeno_sp' = 1.
- Suşları Caenorhabditis isimlendirme kurallarına uygun benzersiz adlarla adlandırın, yani 2-3 büyük harften oluşan benzersiz bir laboratuvar tanımı ve ardından her benzersiz suş için bir sayı23. "strain_name" sütununa gerinim adlarını girin.
- Suşlar adlandırıldıktan sonra, belirlenmiş protokoller 24 kullanılarak kriyokorunabilirler.
10. Toplama verilerinin R'deki easyFulcrum paketi ile işlenmesi
NOT: Bu adımda, easyFulcrum R paketini kullanarak toplama verilerinin (C-etiketleri) ve nematod yalıtım verilerinin (S-etiketleri) birbirine nasıl bağlanacağı açıklanmaktadır. Yazılım, genotipleme sayfasındaki genotipleme verileriyle Fulcrum verilerini daha da birleştirecek işlevler içerir, böylece S-label tür kimlikleri ve suş adları tek bir veri çerçevesinde düzenlenir.
- Koleksiyon projesi için adlandırılmış yeni bir dizin oluşturun. Dizindeki klasör yapısını, easyFulcrum15 R paketinde açıklanan gereksinimlerle eşleşecek şekilde düzenleyin.
- Fulcrum web sitesine gidin ve oturum açın. Soldaki Dayanak noktası web sitesinin veri dışa aktarma aracını kullanarak ve aşağıdaki onay kutularını seçerek ham proje verilerini Dayanak noktası veritabanından dışa aktarın: proje, fotoğraf ekleme, GPS verilerini dahil etme, alan örneklemesi ve yalıtım.
NOT: Projenin Dayanak noktası verileri, virgülle ayrılmış beş değer (.csv) dosyası olarak dışa aktarılır. Tüm proje verileri, R'deki easyFulcrum paketi kullanılarak tek bir veri çerçevesinde birleştirilecektir. - Fulcrum'dan dışa aktarılan beş .csv dosyasını, easyFulcrum vinyetinde21 açıklandığı gibi adım 10.1'de oluşturulan proje dizinine taşıyın.
- Bir Rstudio oturumu açın ve R konsoluna 'install.packages("devtools")" ve "devtools::install_github("AndersenLab/easyfulcrum") komutlarını girerek easyfulcrum paketini R'ye yükleyin.
- Yeni bir R betiği açın ve koleksiyon verilerini işlemek için easyfulcrum vinyetindeki yönergeleri izleyin21.
Sonuçlar
Bu protokol, Hawaii ve Kaliforniya da dahil olmak üzere birçok yerden Caenorhabditis nematodlarını toplamak için kullanılmıştır. Caenorhabditis nematodları için izolasyon başarı oranı, toplama yerine, iklime, örnekleme deneyimine ve örneklenen substrat tiplerine göre değişir. Protokol, dokuz toplama projesinin birkaç yıl ve mevsim boyunca yürütüldüğü Hawaii Adaları'nı kapsamlı bir şekilde örneklemek için kullanılmıştır. Caenorhabditis türlerinin kendi kendine izolasyon başarı oranları C. briggsae (4.506 numunenin 162'si, % 3.6) ve C. elegans (4.506 numunenin 163'ü,% 3.6) için neredeyse aynıdır ve C. tropicalis (4.506 numunenin 26'sı,% 0.58)8 için çok daha düşüktür. Kendi kendine çalışan türlerin her biri, diğer substrat kategorilerine göre çürüyen meyve ve çiçek substratları üzerinde zenginleştirilmiştir. Araştırmacı substrat tercihlerini karakterize etmek yerine başarı oranını en üst düzeye çıkarmaya çalışıyorsa, çürüyen meyve ve çiçek substratlarını örnekleyin. Bununla birlikte, başarı oranı, seçilen substratın kalitesine göre değişir. Örneğin, meyve ve çiçek substratları arasında, çok kuru, ıslak veya taze olan substratlar muhtemelen Caenorhabditis nematodları vermeyecektir.
Bu toplama protokolünün ölçeklenebilirliği, tek bir araştırmacı çiftinin vahşi doğadan toplayabileceği koleksiyon sayısından açıkça anlaşılmaktadır. Örneğin, Ekim 2018'de, bu toplama protokolünü kullanan bir çift araştırmacı, iki Hawaii Adası'ndaki birden fazla yerden 7 gün içinde toplam 1.000'den fazla örnek toplayabildi. Bu saha ekibi, örnekleri bir gecede laboratuvara gönderdi ve sekiz araştırmacıdan oluşan bir ekip, geldiklerinde örneklerden 2.000'den fazla nematodu izole etti. Bu protokolün önemli bir avantajı, sahada ihtiyaç duyulan ekipman ve personeli azaltarak uzak yerlerde örnekleme ile ilişkili maliyeti en aza indirmesidir. Bu protokolü kullanarak, küçük bir saha ekibi örneklemeye odaklanabilirken, izolasyon ekibi numuneleri kendi kurumlarında nematodları izole etmek için mikroskopları ve agar plakalarını parçalamak gibi kırılgan ve ağır ekipmanlar kullanarak işleyebilir. Ayrıca, mobil veri toplama uygulamasının uygulanması, numunelerle ilişkili tüm saha verilerinin doğrudan C-etiketine bağlanmasına izin verir, bu da yalıtım ekibinin numuneleri işlerken saha ekibinden bağımsız olarak çalışmasını sağlar.
Bu toplama protokolünü kullanan araştırmacılar, bir toplama projesinden önce nematodları izole etmek için gereken çabayı göz önünde bulundurmalıdır. İzolasyon ve tanımlama adımları hız sınırlayıcıdır ve küçük bir toplama ekibi izolatörleri numunelerle hızlı bir şekilde boğabilir. Ayrıca, birçok koleksiyonu işlemek için gereken laboratuvar alanı, devam eden araştırmalara müdahale edebilir (Şekil 3). Ek olarak, bazı izole nematodlar genotip için ek çaba gerektirir. Örneğin, izolatların yaklaşık% 2'si ilk lizis girişiminden sonra SSU PCR astar seti ile amplifikasyonda başarısız olur ve lizis malzemesinin ITS2 astar seti ile amplifikasyon için uygun olduğundan emin olmak için yeniden lize edilmelidir (Şekil 8). Ayrıca, izolatların yaklaşık% 3'ü, Sanger dizilemesinin ilk turundan sonra kaliteli diziler üretememektedir. Bu izolatlar için, başka bir lizis turu, ITS2 PCR ve Sanger dizilimi sıklıkla gereklidir, bu da izolasyon ekibi için teslim süresini artırabilir. Daha da önemlisi, dizi kimliği tek başına yeni bir Caenorhabditis türünü haklı çıkarmak için yeterli kanıt değildir (Şekil 7). Bir izolatın yeni bir Caenorhabditis türü olarak yetiştirilmesini uygun şekilde haklı çıkarmak için, çiftleşme deneyleri yapmak ve daktilo edilmiş bir örnek oluşturmak için ek çaba gösterilmelidir13. Yazılan numunenin resmi bir morfolojik açıklaması da tercih edilir, ancak gerekli değildir3. Birlikte, bu hususlar, bu toplama protokolünü benimseyen araştırmacıların, bir toplama projesi başlamadan önce kaynakların uygun şekilde tahsis edilmesini sağlamak için izolasyon ve tanımlama adımlarının deneme testlerinden yararlanacağını göstermektedir. Daha da önemlisi, küçük toplama projeleri bile bu protokolden yararlanabilir, çünkü süreç yüksek oranda tekrarlanabilir ve veriler laboratuvar grupları arasında kalite kontrol amacıyla kolayca denetlenebilir.

Şekil 1: Substrat örnekleri. (A) İdeal çürüyen meyve görüntünün ortasında gösterilmiştir (1), meyve neredeyse tanınamaz. Yakınlarda daha az çürümüş meyve gösterilmiştir; taze düşmüş meyveleri örneklemekten kaçının (2). (B) İdeal olarak ayrışmış bir çiçek en üstte gösterilir (3). Taze düşmüş çiçekleri örneklemekten kaçının (4). (C) Kuru yaprakların üst tabakasının altındaki koyu yaprak çöpü, Caenorhabditis nematodlarının kendi kendine örneklenmesi için örnekleme yaparken idealdir (5). Kuru yaprak çöpü örneklemekten kaçının (6). Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 2: Nematod Field Sampling mobil uygulaması . (A) Fulcrum'daki bir Apple cihazında Nematod Field Sampling uygulamasını açtıktan sonraki ilk ekran. Sağ alttaki kırmızı ok, yeni bir koleksiyon kaydı oluşturmak için kullanılan + düğmesine işaret eder. (B) Bir Apple aygıtında gösterilen yeni bir koleksiyon kaydı örneği. Kırmızı ok, koleksiyon kaydı ekranının üst kısmındaki 'Proje' alanını işaret eder. Sahada örnekleme yaparken doğru projeyi seçtiğinizden emin olun. Proje alanı, varsayılan olarak sonraki koleksiyon kayıtları oluşturulurken kullanılan son projeye ayarlanır. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 3: Numunelerin kaplanmasından önce düzenlenen toplama torbaları ve toplama plakaları. Bu şekilde, soldaki C etiketli toplama torbalarındaki örnekler gösterilmektedir. Her toplama çantasının üstünde eşleşen C etiketli 10 cm'lik bir plaka bulunur. Sağ tarafta, toplama torbalarından transfer edildikten sonra örnek materyal içeren 10 cm'lik toplama plakaları bulunmaktadır. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 4: Düzgün bir şekilde aktarılmış bir numuneye sahip bir toplama plakası (C-plakası). Bakteri çimlerinin kenarına yerleştirilmiş ayrışan meyveli 10 cm'lik bir C plakası. C-etiketi plaka kapağına tutturulmuştur. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
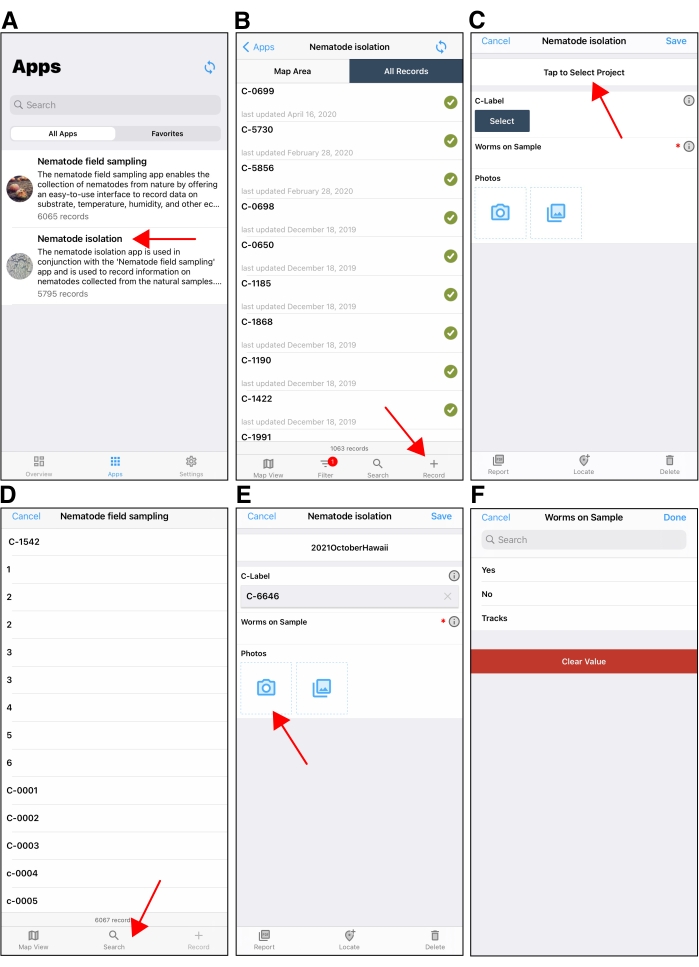
Şekil 5: Nematod izolasyon mobil uygulaması . (A) Fulcrum mobil uygulamasındaki uygulama seçim ekranı. Kırmızı ok, Nematod İzolasyonu uygulamasına işaret eder. (B) Fulcrum'daki bir Apple aygıtında Nematod İzolasyonu uygulamasını açtıktan sonraki ilk ekran. Sağ alttaki kırmızı ok, yeni bir yalıtım kaydı oluşturmak için kullanılan + düğmesine işaret eder. (C) Bir Apple aygıtında gösterilen yeni bir yalıtım kaydı örneği. Kırmızı ok, yalıtım kaydı ekranının üst kısmındaki 'Proje' alanını işaret eder. İzolasyon sırasında doğru projeyi seçtiğinizden emin olun. Proje alanı, varsayılan olarak sonraki yalıtım kayıtları oluşturulurken kullanılan son projeye ayarlanır. (D) C-etiketi altındaki Seç alanına dokunduktan sonra, kullanıcılar nematodları izole ettikleri C etiketini bulmak için arama düğmesine (kırmızı ok) dokunacaklardır. (E) C-etiketi seçildikten sonra, kullanıcılar cihaz kamerasını kullanarak C-plakasını fotoğraflayacaklardır. (F) Kullanıcılar daha sonra C plakasında nematod olup olmadığını girerler. İzole edilecek nematodlar varsa izolasyon kaydına s-etiketleri eklenir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
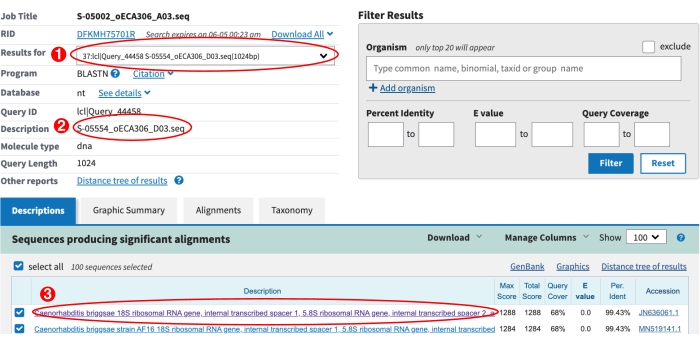
Şekil 6: NCBI BLAST sonuçları sayfası . (1) Tüm dizilerin BLAST sonuçlarını görüntülemek için kullanılan açılır menü. (2) Açılır menüden seçilen geçerli dizinin açıklaması. Bu durumda S-label S-05554 için sonuçlar gösterilir. (3) S-05554 için en iyi BLAST vuruşu gösterilir. Mor metin, bu hizalamanın tıklandığını görselleştirmek için bağlantıyı gösterir. Lütfen olası yeni Caenorhabditis türlerini tanımlamak için hizalamaları gözle incelediğinizden emin olun, yukarıdaki adım 9.8'e bakın. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.

Şekil 7: NCBI BLAST hizalama görselleştirme örnekleri . (A) C. kamaaina konu dizisine hizalanmış bir yalıtımın ITS2 sorgu dizisine bir örnek. (1) Hizalamanın yüzde kimliği (% 89), en iyi BLAST isabeti için düşüktür. (2) Sorgu ve konu dizisi (G'den A'ya) arasındaki uyumsuzluk. (3) Hizalama algoritması tarafından yapılan konu dizisindeki dört baz çifti boşluğu; sorgudaki veya konudaki boşluklar zayıf hizalamayı gösterir. (4) Hizalamanın merkezinde, birçok uyumsuzluk ve boşluk bulunan genelleştirilmiş bir bölge. Bunun gibi bir bölge, sorgu dizisinin yeni bir Caenorhabditis türünden gelebileceğini düşündürmektedir. Gösterilen, 2017 yılında keşfedilen yeni bir tür olan C. oiwi'nin gerçek bir hizalama örneğidir. (B) Bir yalıtımın ITS2 sorgu dizisi ile bir konu dizisi arasındaki iyi hizalamanın bir örneği. (5) Hizalamanın yüzde kimliği (% 99), bu genellikle sorgu dizisinin özneyle aynı türün bir izolatından geldiği anlamına gelir. (6) Mükemmel kimlikle uyumun merkezi bir bölgesi. Bunun gibi bir bölge, sorgu yalıtımının muhtemelen özneyle aynı tür olduğunu göstermektedir. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
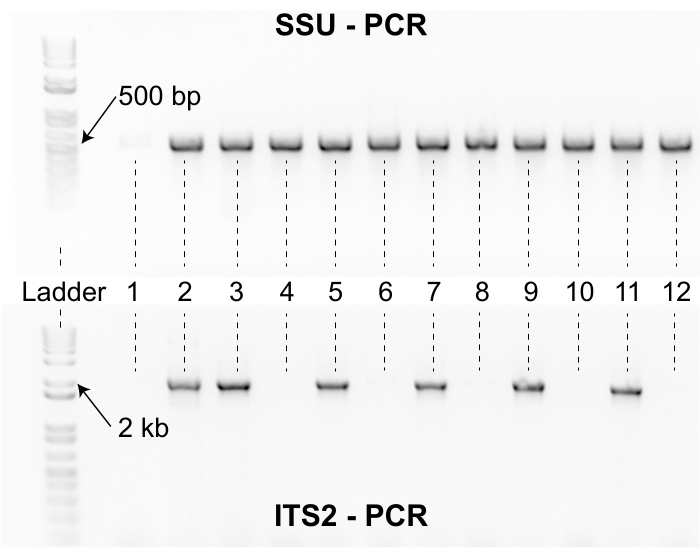
Şekil 8: SSU ve ITS2 PCR ürünleri. Üst jel, 12 temsili numune için SSU astar seti ile üretilen PCR ürünlerini gösterir. Sol tarafta referans olarak bir DNA merdiveni bulunur. Caenorhabditis nematodları için SSU PCR ürünleri yaklaşık 500 bp uzunluğundadır. Örnekler 2-12, SSU astar seti ile güçlendirildi, ancak örnek bir tane yapmadı. Birinci numune için 500 bp SSU amplikonunun olmaması, lizis malzemesinin kalitesiz olduğunu ve numunenin yeniden lize edilmesi gerektiğini düşündürmektedir. Alt jel, üst jelde gösterilen aynı 12 Numune için ITS2 astar seti ile üretilen PCR ürünlerini gösterir. Merdiven ve numuneler her iki jel için de aynı yöndedir. 12 numuneden altısı ITS2 astar seti ile yükselmedi. SSU ve ITS2 bantlarına sahip örnekler Sanger'da sıralanır ve NCBI BLAST algoritması kullanılarak dizi benzerliği ile tanımlanır. Bu şeklin daha büyük bir versiyonunu görüntülemek için lütfen buraya tıklayın.
Ek Dosya 1: C-etiketleri. 2500 benzersiz C etiketi içeren bir PDF dosyası. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Dosya 2: S-etiketleri. 5000 benzersiz S-etiketi içeren bir PDF dosyası. Bu Dosyayı indirmek için lütfen tıklayınız.
Ek Tablo 1: Saha Materyalleri. Nematodları örneklemek için sahada kullanılan malzemelerin bir ambalaj listesi. Bu tabloyu indirmek için lütfen tıklayınız.
Ek Tablo 2: PCR tarifleri ve termosikler koşulları. ITS2 ve SSU PCR'ler için PCR tarifleri ve termosikler koşulları tablosu. Bu tabloyu indirmek için lütfen buraya tıklayın.
Ek Tablo 3: Elektroforez tampon tarifleri. 0,5 M pH 8,0 Etilendiamintetraasetik asit çözeltisi (EDTA) ve TRIS-asetat-EDTA (TAE) tampon çözeltisi için bir reçete. Bu tabloyu indirmek için lütfen tıklayınız.
Tartışmalar
Bu protokol, dikkatle yürütülmesi gereken kritik adımları içerir. Örneğin, saha ve izolasyon ekiplerinin, sahadan numune toplamadan veya laboratuvardaki numunelerden nematodları izole etmeden önce uygulamada doğru toplama projesini seçmeye özen göstermeleri önemlidir. Yanlış toplama projesinin seçilmesi durumunda, hatalı veri kayıtları çevrimiçi kayıt düzenleme araçları kullanılarak Dayanak noktası veritabanında en iyi şekilde düzeltilir. Bu süreç, yanlış yerleştirilmiş birçok kayıt için sıkıcı olabilir. Ancak, veritabanı kayıtlarda yapılan değişiklikleri saklar, böylece toplama ve yalıtım kayıtlarının tam olarak denetlenmesi mümkün olur. Bu protokoldeki diğer kritik adımlar, sahadan alınan örneklerin ve bu örneklerden izole edilen nematodların işlenmesini içerir. Caenorhabditis nematodlarının numune alma ve sevkiyat adımlarında hayatta kalmasını sağlamak için, numunelerin sıcaklığı 4 °C ile 25 °C arasında tutulmalıdır. 25 °C'nin üzerindeki sıcaklıklar C. elegans14'te steriliteye neden olabilir. Nematodların kaybını en aza indirmek için numunelerin toplama torbalarından toplama plakalarına mümkün olduğunca beş gün içinde aktarıldığından emin olun. Nematodlar izole edildikten sonra, yok olmadan önce genotiplendirilmeleri ve kriyokorunmuş olmaları çok önemlidir. İki ila üç haftalıktan daha eski olan S plakalarında canlı nematodlar bulmak zordur, çünkü mantar ve bakteriyel kontaminasyon S plakalarını kabul edilemez hale getirebilir.
Bu protokol, araştırmacıların sahadayken toplamak isteyebilecekleri farklı veri türlerini barındıracak şekilde kolayca değiştirilebilir. Örneğin, uygulama düzenleme için Fulcrum'un çevrimiçi GUI'sini kullanarak 'Nematod alan örnekleme' uygulamasını yeni veri giriş alanlarıyla özelleştirmek kolaydır. Ayrıca, easyFulcrum adlı veri analiz paketi, yeni verileri işlerken bu düzenlemelere uyum sağlayabilir15. Kullanıcıların çekici bulabileceği bir diğer değişiklik, alanda farklı bir örnekleme yöntemi kullanmaktır. Ayrık substratları örneklemek yerine, araştırmacılar birden fazla substrat tipi içeren daha geniş alanları örneklemek isteyebilirler. Bu daha büyük numuneler laboratuvarda en iyi şekilde Baermann hunisi veya tepsi ekstraksiyon yöntemleri kullanılarak işlenir13. Daha da önemlisi, C-etiketlerinin ve S-etiketlerinin kullanımı bu teknikler için hala geçerlidir ve bu nedenle mobil uygulamalarla uyumludur.
Bu protokolün birincil sınırlamaları, laboratuvarda izolasyondan önce nematodların kullanım süresi ile ilgilidir. İlk olarak, numune toplama ve nematod izolasyonu arasındaki gecikme süresi, toplama sırasında nematodların gelişim aşamalarını belirli bir numuneye kaydetmeyi imkansız kılar. İkincisi, erkeklerin sıklığı ve doğadaki geçiş, Caenorhabditis nematodlarının kendini geliştirmesi için anahtar evrimsel sorulardır10. Bu yöntem bu soruları ele almak için uygun değildir, çünkü nematodların izolasyondan önce birden fazla nesilden geçmiş olması muhtemeldir. Gecikmiş izolasyon, doğadaki erkek sıklığının doğrudan kanıtının imkansız olduğu anlamına gelir. Ayrıca, genotipleme adımları sırasındaki çok kuşaklı gecikme, bir nematod suşu dizilenmeden önce genomik geçiş kanıtının (heterozigotluk) aşınacağı anlamına gelir. Doğadaki heterozigotluğu tanımlamak için, doğrudan doğadan izole edilmiş bir nematod tarafından üretilen yavrular dizileme için kullanılır2. Bu protokolün bir diğer potansiyel sınırlaması, kendi kendine Caenorhabditis'in tanımlanmasına yönelik önyargılı olmasıdır. Bunun nedeni, kendi kendine çalışan türlerin izole nematodlarının, zorunlu geçitlerden daha fazla çoğalma şansına sahip olmasıdır; bu, yalnızca döllenmiş bir dişi izole edildiğinde çoğalacaktır.
Bu toplama yöntemi, varolan toplama protokollerini temel alır13,14. Bu tekniğin en büyük ilerlemesi, büyük ölçekli toplama projeleriyle ilişkili çok miktarda ekolojik ve moleküler verinin organizasyonunu kolaylaştırmak için mobil teknolojinin ve özelleştirilmiş yazılımın kullanılmasıdır. Bu toplama protokolü kullanılarak üretilen ekolojik veriler, Caenorhabditis türlerinin doğal popülasyonları için olağanüstü soruları ele almak için kullanılabilir. Örneğin, bu yöntemle üretilen veriler, Hawaii Adaları'ndaki türlerin niş tercihlerini keşfetmek için kullanılmıştır. Dahası, kriyokorunmuş nematodların genomlarını sıralayarak, araştırmacılar genetik varyasyon kalıplarının ekolojik verilerle nasıl ilişkili olduğunu araştırabilirler. Bu tür araştırmalar, Caenorhabditis popülasyonlarındaki yerel adaptasyonun imzalarını ortaya çıkarabilir ve doğal bağlamlardaki genetik çeşitliliğin önemi hakkında önemli bilgiler sağlayabilir8. Caenorhabditis nematodlarındaki birçok genin işlevsel bir şekilde anlaşılması için, ekolojik çalışmalara ihtiyaç vardır11. C. elegans için bile, genlerin büyük bir kısmı, dizilenen ilk çok hücreli hayvan ve Dünya'daki en iyi çalışılmış hayvanlardan biri olmasına rağmen, işlevsel ek açıklamalardan yoksundur. Bu toplama protokolü, vahşi Caenorhabditis nematodlarının toplanmasını ve ekolojilerinin ve doğal genetik çeşitliliğinin incelenmesini kolaylaştırarak bu bilgi boşluğunun giderilmesine yardımcı olmak için geliştirilmiştir.
Açıklamalar
Yazarlar herhangi bir çıkar çatışması bildirmemektedir.
Teşekkürler
Bu araştırma, Northwestern Üniversitesi'nden başlangıç fonları ve her ikisi de E.C.A.'ya verilen Ulusal Bilim Vakfı KARİYER Ödülü (IOS-1751035) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
Referanslar
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır