Method Article
自己 カエノールハブディティス 線虫の生態学的調査を行うための高度にスケーラブルなアプローチ
要約
このプロトコルは、自己発症 性Caenorhabditis 線虫の大規模な生態学的調査を実行するために使用することができる。この方法の主な利点は、自然から収集された線虫に関連する生態学的および分子的データの効率的な組織化および分析である。
要約
Caenorhabditis elegans は生物学の主要なモデル生物の1つですが、研究者はごく最近になってその自然生態学に焦点を当てています。C . elegans に関する情報の自然な文脈における相対的な希薄さは、自然界の小さな線虫の同定に伴う課題から来ている。これらの課題にもかかわらず、 C. elegans の生態学への関心の高まりは、実験室の外でのその生活に関する豊富な新しい情報を引き起こしました。自然界における C. elegans の探索の激化は、多くの新しい Caenorhabditis 種の発見に貢献し、同属線虫が野生で頻繁に共存し、腐敗した植物材料に関連する微生物の花を食べることを明らかにした。新種の同定はまた、男性と自己受精雌雄同体のアンドロディオエキアス交配系が カエノラブディティス内で独立して3回進化したことを明らかにした。他の2つの自己受容種、 C. briggsae とC. tropicalisは、 C. elegans の実験的利点を共有し、自己受精を含む重要な形質の機構的基礎への比較研究を可能にした。これらの進歩にもかかわらず、これらの重要な種の生態学と自然の多様性については、まだ多くのことが学ばれています。例えば、私たちはまだ彼らの遺伝子の多くについて機能的な情報を欠いており、それは彼らの自然生態学の理解を通してのみ達成されるかもしれません。自己発生 性Caenorhabditis 線虫の生態学的研究を促進するために、我々は野生から線虫を収集するための非常にスケーラブルな方法を開発しました。我々の方法は、モバイルデータ収集プラットフォーム、クラウドベースのデータベース、およびRソフトウェア環境を利用して、野生から線虫を収集し、関連する生態学的データを記録し、分子バーコードを使用して野生の線虫を同定する研究者の能力を強化する。
概要
過去20年間、Caenorhabditis線虫の生態学への関心が高まっています。これらの研究から、自由生活のカエノルハブディティス種は、温帯地域と熱帯地域の両方の一時的なマイクロ生息地から単離することができ、植物材料の分解に関連する微生物の花を、時にはシンパトリーで摂食することができることが分かっています1,2,3,4,5,6,7,8.また、この属では自己受精の収斂進化が3回起こっており、C. briggsae、C. elegans、およびC. tropicalisの自己生産が支配的な生殖様式であることもわかっています9,10。これらの自殖業者の中で、C. elegansは地球上で最も広く研究されている動物の1つであり、生物学の重要な進歩を遂げるために研究者によって使用されてきました。重要なことに、他の自己性Caenorhabditis種は、C. elegansの実験的利点の多くを共有しており、この属における比較研究を急速に進めている。しかし、野生におけるこれらの線虫の不可解な性質は、その生態と自然の多様性を研究することを困難にし、それは彼らの遺伝子の生物学的機能と進化が種間の遺伝的多様性を形作った方法を理解するために重要です10,11。
野生のCaenorhabditis線虫の自殖の生態学を研究するための最大の課題は、その小さなサイズです。成虫線虫は、しばしば長さが1mm以下である。この課題は、研究者が野生の基質をサンプリングし、野生の動物を観察する能力なしに実験室で目的の線虫を基質から分離しようとすることを必要とする。訓練を受けた専門家でさえ、顕微鏡下で自己化カエノラブディティス線虫を他の自由生活線虫と区別することは困難であると考えているため、線虫は通常、確立された分子バーコードを使用して配列同一性によって同定される前に基質から除去され、単離され、増殖するために放置される3,12,13,14.この方法で各線虫を処理するために必要な時間と労力は、研究者が実験室で単離された各線虫の同一性を、現場でサンプリングされた正確な基質および関連する生態学的データまでさかのぼって追跡できなければならないため、組織的な課題を提示する。ここでは、フィールドから自己組織化Caenorhabditis線虫を効率的に収集して同定し、これらの分離株を関連する空間的および生態学的データと忠実に大規模にリンクするための段階的なプロセスについて説明します。
この収集方法は、モバイルデータ収集プラットフォーム、クラウドベースのデータベース、およびRソフトウェア環境を使用して、生態学的調査の規模と精度を向上させます。Fulcrum はカスタマイズ可能なデータ収集プラットフォームであり、ほとんどのモバイル デバイスで動作し、ユーザーは位置ベースのデータを収集および整理するためのカスタム アプリケーションを構築できます (https://www.fulcrumapp.com)。このプロトコルは、カスタマイズされたデータ収集アプリケーションを使用して、現場からの空間的に明示的な生態学的データを整理し、それらのデータを実験室で単離された線虫のアイデンティティと正確にリンクさせる方法に関する詳細な指示を提供する。このプロトコルはまた、確立された分子バーコードを使用して、自己化Caenorhabditis線虫を効率的に同定する方法も説明する。これらの方法からのデータは、付属のRソフトウェアパッケージeasyFulcrum15で簡単かつ再現性よく処理して、天然のカエノラブディティス集団の生態学的および遺伝的多様性を探索することができます。
プロトコル
1. コレクションの準備
- Caenorhabditis線虫を調査する場所を特定する。
注:ほとんどの温帯地域では、C. elegansとC. briggsaeは、農業畑や農村部や都市部の庭園などの人間関連の生息地から簡単に隔離できます1。亜熱帯および熱帯地域では、C. briggsae、C. elegans、およびC. tropicalisはすべて、上記の人間関連の生息地で、時には互いに近接して見つけることができます。しかし、C. elegansは熱帯生息地の他の種よりも涼しく乾燥した生息地を好むようです7,8。各種は、人間と関連していない野生の生息地から隔離することもできますが、これらの生息地はそれほど頻繁にサンプリングされません。 - Fulcrum プロジェクトを作成して、モバイル・データ収集アプリケーションを使用して収集および分離データを編成します。
- 無料の教育契約を使用して、Fulcrumのアカウントをオンラインで作成します16。 線虫フィールドサンプリング アプリケーションをフルクラムアカウントに追加するには、[ APPを追加 ]ボタン17をクリックします。
- 線虫分離アプリケーションをアカウントに追加するには、[APPを追加]ボタン18をクリックします。
注: ある場所への各旅行は、命名規則 'YearMonthLocation' (例: 2020FebruaryAustralia) を使用してコレクション プロジェクトとして編成することをお勧めします。
- ユーザーを Fulcrum アカウントに追加して、コレクション プロジェクトへのアクセス権を付与します。各ユーザーがプロジェクトに参加するために Fulcrum モバイル・アプリケーションをダウンロードしていることを確認します。
- 一連のQRコードラベルを印刷して、モバイルアプリケーションでコレクション(Cラベル)と線虫分離(Sラベル)を追跡します。Cラベルをジップロック式のビニール袋に取り付け、ラベルの付いた袋を25個のグループに丸め、梱包用のゴムバンドで包みます。実験室で使用するためにSラベルのセットを保管してください。
注:このプロトコル全体を通して、コレクション(現場からの基板)はバッグまたはプレートに含まれ、Cラベルでラベル付けされています。単離された線虫はS標識で標識されている。Cラベルは固有のコレクションを識別するために使用され、Sラベルは一意の線虫分離株を識別するために使用されます。これら 2 種類のラベルは、Fulcrum データベース内の特定のコレクション (C ラベル) と、そのコレクションから分離された線虫 (S ラベル) との間の接続を行うために使用されます。コレクション プロジェクトの C ラベルとして 2 倍の数の S ラベルを印刷するのは、コレクションごとに平均して 2 つの線虫が分離されるためです。必要に応じて、後でより多くの S ラベルを印刷できます。2,500のユニークなCラベル(補足ファイル1)と5,000のユニークなSラベル(補足ファイル2)が補足で提供されています。 - 採取用に10cmのNGMAプレートを、線虫を単離するために3.5cmのNGMAプレートを準備する。10cmのプレートを1枚と3.5cmのプレートを2枚以上作成します21。これらのプレートに、確立されたプロトコールに従って 大腸菌 株OP50を播種する。使用前にプレートを4°Cで1ヶ月以内に保管してください。
2. フィールド収集
注:Caenorhabditis線虫は、果物、ナッツ、種子、さや、花、茎、植物性ゴミ、堆肥1,5,6,8を含む腐った植物材料から最も頻繁に単離されます。最高の基質は腐っていて、果物や花としてほとんど認識できません。基板が乾燥しすぎたり濡れたりしないようにします(図1)。基板は、ペアで作業することによって現場から最も効率的に収集されます。非接触赤外線温度計を持つ個人は、収集用の基板を選択してサンプルを収集し、パートナーはFulcrumの線虫フィールドサンプリングアプリケーションを使用して収集データを記録します。コレクタのペアは、必要な数のサンプルが収集されるまでこのプロセスを繰り返します。フィールドワークに必要な資料のリストは、(補足表1)にあります。
- Fulcrum モバイル アプリを開き、ドロップダウン メニューから [ 線虫フィールド サンプリング] を選択します。 + を押して、プロジェクトで新しいレコードを開始します (図 2A)。基板の写真を撮ります。
- 上部中央のボックスをクリックして、手順1.2で作成した正しいコレクションプロジェクトを選択します(図2B)。コレクションレコードの下部にある Cラベル フィールドをタップし、プロンプトが表示されたら[スキャン]を選択します。モバイルデバイスのカメラを使用してコレクションバッグのバーコード をスキャンし 、画面の右上にある[ 完了 ]をタップします。
- 基板 フィールドを タップし、ドロップダウンメニューから基板タイプを選択します。基板に関するメモを追加するには、「基板 メモ」(Substrate Notes) フィールドをタップし、手動でメモを入力します。
- ドロップダウンメニューからランドスケープを選択します。サンプリング サイトを最もよく表すランドスケープを選択します。
- スカイビューを選択します。スカイビューを選択するときは、サンプリングサイトでの空の可視性を記述します(たとえば、木や他の構造物からの遮るもののないフルスカイビュー=フル)。
- 非接触温度計を用いて基板の表面温度を測定し、その値を基板温度フィールドに記録する。
メモ: 温度を記録している間は、非接触温度計を基板から 14 インチ以内に離してください。 - ハンドヘルドデバイスで周囲温度と湿度を測定し、これらのデータを適切なフィールドに記録します。
メモ: 周囲温度および湿度デバイスが 保留になっていないことを確認します。ボタンを離すと測定単位が変わります。不規則な読み取りを避けるために、デバイスを外側のポケットに入れてください。 - 画面の左上にある [ 保存] をタップして、レコードを支点に保存します。
- スティックやその他の硬い部分なしで大さじ1杯の基板を、収集袋を反転させて基板を拾うための「手袋」として使用し、袋を密封します。サンプルが特に湿っている場合は、ペーパータオルを袋に入れます。
注:暑い気候では、コレクションを涼しく保つために、より涼しいパック付きの柔らかいクーラーにバッグを置きます。 - その日のすべてのサンプルが収集されたら、収集機器を清掃し、プローブからバッテリーを取り出し、バッテリーを充電し、冷凍庫パックを再凍結します。線 虫フィールドサンプリング アプリケーションの左上にある同期ボタンをタップして、支点コレクションデータを同期します。
注:アップロードには、強力な携帯電話接続がないと数分かかる場合があるため、WiFiアクセスを待つことをお勧めします。データはモバイルデバイスに残り、クラウドに同期されます。 - サンプルを夜間配送箱に入れて、ホームインスティテューションに出荷します。貨物が輸送される日にパッケージを出荷することにより、サンプルが11°C未満または25°Cを超える温度にさらされる時間を最小限に抑えます。
注:ほとんどの配送施設では、週末に遠隔地で一晩貨物を出荷することはありません。
3. 実験室でのめっき外フィールドコレクション
注: このセクションでは、ラベル付き収集バッグからラベル付きプレートへのサンプルの移送を整理する方法について説明します。サンプルは、一晩の出荷から、またはフィールドから直接到着することができます。
- コレクションの貨物を受け取り、壊れたバッグやその他の損傷の証拠がないか検査します。袋が壊れている場合は、材料を捨て、壊れていない収集袋を70%エタノールで清掃してください。エタノールを含む袋のCラベルは、ラベルを変色させ、読みにくくするため、避けてください。
- 各ジップロックバッグについて、バッグのCラベルを書き留め、OP50細菌が付着した10cmプレートの蓋に一致するCラベルを貼り付けます。
注: ラベルの付いた 10 cm プレートは、プロトコルの残りの部分では「C プレート」と呼ばれます。サンプルを整理する最も簡単な方法は、収集バッグをラボベンチに置き、その上に一致するCプレートを置くことです(図3)。 - 各コレクションについて、きれいなプラスチックスプーンを使用して、コレクションバッグから約1杯のサンプルをCプレートに移します。細菌芝生の周囲にサンプルを三日月またはリング状に添加し、細菌芝生を完全に覆わないでください(図4)。
注:使用していないときは、大さじを95%エタノールのビーカーに入れて清潔に保ちます。追加のサンプルを移す前に、ペーパータオルを使用して大さじを乾かします。 - コレクションが収集バッグからCプレートに移された時間を記録し、セクション4で線虫の単離を試みる前に、Cプレートを室温(RT)に少なくとも24時間保持する。
4.コレクションからの線虫の分離
- モバイルデバイスで Fulcrum アプリケーションを開き、アプリケーションメニューから [線虫の分離] を選択します(図5A)。右下の+アイコンをタップして、新しい分離記録を作成します(図5B)。
- 新しい分離記録画面で、上部中央のボックスに表示されているプロジェクト名にチェックを入れて、正しい収集プロジェクトを確認します。間違ったプロジェクトが表示された場合は、プロジェクト名をタップして正しいプロジェクトに切り替えます(図5C)。
- Cラベルフィールドの下にあるSelectボタンをタップして、線虫が単離されているサンプルに関連付けられているCラベルを見つけます(図5D)。検索アイコンをタップしてから、スキャンアイコンをタップして、デバイスのカメラでCプレート上のCラベルQRコードをスキャンします。QR コードがスキャンされると、C ラベル レコードが C ラベル フィールドに表示されます。
- [写真]フィールドの[カメラ]アイコンをタップしてデバイスのカメラを開き、それを使用してQRコードが表示されたCプレート上のサンプルの写真を撮ります(図5E)。[完了]をタップして[分離]画面に戻ります。
注:これらの分離記録写真は、後で基板の特定の属性を調べるために使用できます。 - 解剖顕微鏡を使用して、Cプレート上の線虫を探します。[サンプル上のワーム]フィールドをタップして、 サンプル上の 線虫の存在を記録します(図5F)。線虫がCプレート上に存在する場合は[ はい ]をタップし、線虫が存在しない場合は [いいえ ]をタップします。
- 線虫のトラックのみが存在する場合は、 トラック をタップします。線虫が存在しない場合は、Cプレートをパラフィルムし、バイオハザードビンに処分します。
メモ:Cプレートをバイオハザード廃棄物箱の上に反転させ、プレートの背面を軽く叩いて、サンプリングしたすべての基板を外します。このステップにより、Cプレート上の基質の下にある線虫を見つけて単離することが容易になる。 - 線虫が存在する場合は、Cプレートから最大5つの線虫を単離する。線虫を単離するには、白金線ピックを使用して、1つの線虫をCプレートからSプレートに移す。可能であれば、健康でグラビッドな大人を隔離してください。ただし、成虫が見つからない場合は、他の段階を分離してください。
注:単離後、最大5つのSプレートにそれぞれ1つの線虫が1つずつあります。同じCプレートから分離された線虫を持つこれらのSプレートは、支点に入るまで他のSプレートから離れたきれいなスタックにまとめてください。 - 「 S ラベルの付いたプレート」 フィールドをタップして、この分離に使用する S プレートを入力します。右下の + をタップします。 Sラベル をタップし、[ スキャン ]をクリックしてデバイスのカメラを開きます。デバイスのカメラを使用して、S プレート上の S ラベル QR コードをスキャンします。
メモ: S ラベルのコードがプレートのコードと一致していることを確認します。一致する場合は、[ 完了]をタップします。そうでない場合は、[ キャンセル ]をタップして一致するまで再スキャンし、[ 完了]をクリックします。近くのプレートのQRコードが誤ってスキャンされることがあります。 - 各Sプレートを入力したら、右上の保存ボタンでエントリ を保存し ます。エントリは、保存されていない場合は失われます。右下の + をタップして、Cプレートから分離されたすべての線虫が入力されるまで、必要に応じてSラベルプレートを追加します。隔離記録にすべてのSラベル付きプレートを追加したら、左上の < ボタンをタップして隔離記録画面に戻ります。
メモ: 間違いを解決できないために分離レコードをキャンセルするには、左上の [ キャンセル] をクリックします。この手順では、保存せずにレコードを破棄できるかどうかを尋ねるダイアログが開きます。必要に応じて、[ はい、破棄]をクリックします。 - 分離レコードにすべての情報が正しく追加されたら、右上の [保存] ボタンをタップします。次に、孤立した線虫でSプレートをパラフィルムし、線虫でSプレートを保持するように指定された領域にそれらを脇に置きます。
- Cプレートをパラフィルム化し、バイオハザードビンに捨てます。 同期 アイコンをタップして、すべてのデータをフルクラムにアップロードします。
- すべてのSプレートを英数字順にソートし、Sプレートを段ボール箱に入れます。Sプレートが蓋側を下にしてパラフィルムされていることを確認してください。箱の中の1つの位置に最大4枚のSプレートを積み重ね、段ボール箱にプロジェクト名、日付、時刻、および一意の箱番号のラベルを付けます。
- ラベルの付いたボックスをRTに保管します。これらの分離株は、48時間で増殖し、必要に応じて168時間で再び増殖するかどうかチェックされます。
5. 支点からのSプレートの輸出
注: このセクションでは、分離プロセスで使用される S ラベルを Fulcrum プロジェクト データベースからエクスポートする方法について説明します。これらのS標識は、セクション6〜9の配列同一性によって同定されている間、増殖する等女性ラインを追跡するために使用される。
- Fulcrum Web サイトにサインインし、 線虫分離 アプリケーションを選択します。画面の左側から [エクスポーター ]をクリックします。目的のプロジェクトをクリックして選択し、[ 線虫の分離] チェックボックスをオンにします。「 次へ 」をクリックして、「.zip」ファイルを含むnematode_isolation_s_labeled_plates.csvファイルをダウンロードします。
- 「nematode_isolation_s_labeled_plates.csv」ファイルを開き、「Sラベル」列で昇順にソートします(最小のSラベルが一番上に表示されます)。すべての S ラベルを選択し、スプレッドシートからコピーします。
- ウェブブラウザを使用して、野生の分離ジェノタイピングテンプレートGoogleシート(wild_isolate_genotyping_template)に移動します19。
- このGoogleシートのコピーを作成するには、[ ジェノタイピングテンプレート ]タブを右クリックし、[ 新しいスプレッドシートにコピー] オプションを選択します。[ スプレッドシートを開く] を選択して、新しい Google スプレッドシートを表示します。
- この新しいシートには、支点プロジェクト名の後に 'wild_isolate_genotyping' を付けます (例: '2020FebruaryAustralia_wild_isolate_genotyping')。
注: このシートは、プロトコルの残りの部分で「ジェノタイピング シート」と呼ばれます。
- 「nematode_isolation_s_labeled_plates.csv」の「s_label」列からコピーしたSラベルを「s_label」というタイトルの遺伝子型シート列に貼り付けます。「s_label_repeat_error」列で「1」を確認してください。この列の値 '1' は、S ラベルがジェノタイピング シートのどこかに複製されていることを意味します。重複が見つかった場合は、先に進む前に調査して修正してください。
- すべてのSラベルのジェノタイピングシート「isolation_box_number」列に記入してください。
6. Sプレート上の拡散を確認する
- 隔離後48時間でSプレート上で動物が増殖していないか確認します(タイミングをガイドするには、ステップ4.11のボックス上の最後の隔離の日時を使用します)。
注:増殖する線虫は、Sプレート上の子孫によって特徴付けられる。 - Sプレートが増殖している場合は、遺伝子型決定シートのproliferation_48列に「1」と入力し、Sプレートを「48時間増殖、ボックス1」というラベルの付いたボックスに移動します。最大88枚のSプレートを増殖ボックスに入れ、「48時間増殖、ボックス2」というラベルの付いた新しいボックスの充填を開始します。S ラベルが 48 時間の拡散ボックスで英数字順に整理されていることを確認します。
注:増殖しないSプレートを処分しないでください。これらのプレートは、絶縁後168時間で再度チェックされます。必要に応じて、これらの S プレートを「48 時間非増殖、ボックス X」というラベルの付いたボックスに番号順に統合しますが、新しいボックスで 168 時間のチェックをいつ行う必要があるかを記録することを忘れないでください。 - 48時間で増殖するすべてのSラベルを特定した後、48時間で増殖するSプレートのセクション7に進みます。
- 単離後48時間で増殖しなかったSプレートを、単離後168時間で再度確認してください。
- 現在、S プレートが増殖している場合は、ジェノタイピング シートの proliferation_168 列に「1」と入力し、S プレートを「168 時間増殖、ボックス 1」というラベルの付いたボックスに移動します。
- 最大88枚のSプレートを増殖ボックスに入れ、「168時間増殖、ボックス2」というラベルの付いた新しいボックスの充填を開始します。Sラベルは必ず英数字順に168時間拡散ボックスに整理してください。
- 168時間後に増殖しないSプレートを捨てる。168時間で増殖するSプレートのセクション7に進みます。
7.等女性線の溶解
注:この手順では、Googleスプレッドシートのデータフィルタツールを使用して、拡散ボックスのSプレートの溶解ワークシートを印刷します。溶解ワークシートの目的は、ベンチの溶解ストリップチューブ内のSラベルの正しい位置を人員に提供することです。
- 目的のプロジェクトのジェノタイピングシートを開き、 Cmd + Aと入力してすべてのセルを選択します。[ データ] > [フィルターの作成] をクリックして、各列ヘッダーにフィルター ボタンを追加します。 [フィルター] ボタンを使用して、遺伝子型決定される S プレートのみを表示します。たとえば、48時間で増殖するすべてのSプレートを溶解する場合、「proliferation_48」列の[フィルタ]ボタンをクリックし、[1]を選択します。
- 遺伝子型決定 Google シートがフィルター処理されたら、表示された S ラベルのリストを確認して、ワークシートに印刷する S ラベルであることを確認します。
- ジェノタイピング Google シートの [strip_tube_number] 列に、11 行ごとに一意の番号を入力します。
- プロジェクトのストリップチューブ番号を 1 から始まる連続した順序で入力し、重複することはありません。「strip_tube_position」に、ストリップチューブ番号ごとに2~12と入力します。
注:溶解には12チューブストリップチューブを使用してください。最初の位置 (strip_tube_position 1) は管理対象になりますが、管理は溶解ワークシートに追加されません (strip_tube_positionsのみが追加されます (2 ~ 12)。溶解時には、陽性対照株'N2'が陽性対照として偶数番号帯状管毎の位置1に添加されることになる。ネガティブコントロールとして、奇数番号のストリップチューブのすべての位置1にワームは追加されません。
- 遺伝子型決定のGoogleシートをさらにフィルタリングして、溶解する1つの拡散ボックスにSラベルのみを含め、列「s_label」から「lysis_notes」を選択します。溶解する増殖ボックスごとに溶解ワークシートを印刷します。
- [印刷] フィールドのドロップダウン メニューをクリックし、[選択したセル] を選択します。右上の[次へ]をクリックし、ダイアログを使用して拡散ボックスの溶解ワークシートを印刷します。
- 手順 7.3 ~ 7.5 を繰り返して、増殖ボックスごとに溶解ワークシートを印刷します。
注:各増殖ボックスには最大88枚のSプレートが収納でき、これは8本の12ウェルストリップチューブに対応します。 - 溶解するすべてのサンプル用に12ウェルストリップチューブを準備します。ストリップチューブに、溶解ワークシートに割り当てられた一意の「strip_tube_number」でラベルを付けます。このラベルは、キャップストリップとストリップチューブが分離されている場合の混乱を避けるために、キャップストリップに書き込む必要があります。偶数ストリップチューブは、位置1にポジティブコントロール(N2ワーム)を有する。ODDストリップチューブは、位置1にネガティブコントロール(ワームなし)を有する。
- すべてのサンプルに対して十分な溶解バッファー (100 mM KCl、20 mM Tris pH 8.2、5 mM MgCl2、0.9% IGEPAL、0.9% Tween 20、0.02% ゼラチン、プロテイナーゼ K を終濃度 0.4 mg/mL に添加) を作り、ピペットエラーのために 5% 余分に加えます。必要に応じてスケーリングします。
注:溶解バッファーは、プロテイナーゼKを除くすべての成分を組み合わせて、-20°Cで10〜50mLのアリコートで凍結することによって最もよく調製されます。 アリコートを解凍し、使用前に4°Cに保ちます。使用直前にプロテイナーゼKを加え、よく混ぜる。作業中は溶解バッファーを氷の上に保管してください。 - 印刷された溶解ワークシートをガイドとして使用して、特定のストリップチューブ用のSプレートを順番に配置します。
- ストリップチューブ1本をキャップを外し、繰り返しピペッターで各キャップに8μLの溶解バッファーを追加します。溶解バッファーを一度に 1 つのキャップストリップに加えるのは、RT に放置して覆い隠すと溶解バッファーが蒸発するためです。ソースプレート(陽性対照用のSプレートまたはN2ストックプレート)から3〜5匹の動物を、溶解ワークシートに示された適切なキャップ位置に選びます。溶解ワークシートのlysis_notesのセクションに、溶解にピックされたワームが5つ未満のSプレートのメモを記録します。
- 線虫をストリップチューブの各位置にロードした後、キャップストリップをストリップチューブに戻す。マークされたキャップ(位置1)とマークされたチューブ(位置1)を一致させます。蓋をしたら、線虫がチューブの底になるまでストリップチューブを短時間遠心分離します。
- ストリップを-80°Cの冷凍庫に入れ、完全に凍結するまで(少なくとも10分間)置く。すべてのストリップに溶解のために線虫が追加されるまで、手順7.9〜7.11を繰り返します。チューブストリップを番号順に整理します。
- ストリップチューブのセットを取り出し、サーモサイクラーで溶解プログラムを実行します:60°Cで1時間、95°Cで15分、12°Cで保持します。 溶解プログラムが完了したら、サンプルを 300 x g で RT で 15 秒間スピンダウンし、溶解液を -80 °C で最大 1 週間保存します。
- 96ウェルプレートホルダーを使用してチューブストリップを番号順に整理し、増殖ボックス番号、ストリップチューブ番号の範囲、日付、および研究者のイニシャルを含むラベルを含めます。遺伝子型決定シートの列「lysis_date」と「lysis_notes」を溶解ワークシートからの情報で更新します。
8. SSU および ITS2 配列の PCR
注: このセクションでは、溶解した S プレートごとに 2 つの個別の PCR を実行する方法について説明します。第1のプライマーセットは、18S rDNAスモールサブユニット遺伝子(SSU)の500-bp断片を増幅する;oECA1271 = フォワードプライマー TACAATGGAAGGCAGCAGGC, oECA1272 = リバースプライマー CCTCTGACTTTCGTTCTTGATTAA 12.このPCRは、鋳型DNAの品質をチェックするために使用されます。PCRは、ほぼすべての線虫種のSSU領域を増幅します。SSU PCRが増幅に失敗した場合、この結果は、溶解の質が悪く、このSプレートに対して溶解を繰り返す必要があることを示唆している。第2のプライマーセットは、5.8Sおよび28S rDNA遺伝子間の内部転写スペーサー領域の2,000-bp断片を増幅する(ITS2);oECA1687 = フォワードプライマー CTGCGTTACTTACCACGAATTGCARAC, oECA202 = リバースプライマー GCGGTATTTGCTACTACCAYYAMGATCTGC3.ITS2 PCR産物はサンガー配列決定されており、その配列は、配列類似性によって種レベルにカエノラブディティス属の線虫を同定するために使用される。
- ジェノタイピングシートのフィルタリングツールを使用して、PCRに使用するSラベルのみを表示します。
- pcr_plate_numberを更新し、ジェノタイピングシートの列をpcr_wellします。溶解材料の分解を防ぐために、SSU および ITS2 PCR が同時に実行されます。
- ITS2 PCR と SSU PCR は、別々のプレートでの別々の反応であっても、同じpcr_plate_numberを使用します。それらは「SSU」または「ITS2」ラベルで区別されます。
- pcr_plate_numberを8本以下のストリップチューブ(96ウェルPCRプレートの1列につき1本のストリップチューブ、昇順で配置、例えば、一番上に最も低いストリップチューブ番号)に割り当てます。次に、ストリップチューブ内の各Sラベルにpcr_plate_wellを割り当てます。
メモ: ストリップチューブは昇順で配置され、最も低いストリップチューブ番号が行 A に、最も高い番号が行 H に割り当てられます。すべてのストリップチューブの位置 1 が列 1 に割り当てられます。したがって、ストリップチューブ番号1、位置1はPCRプレート番号1、ウェルA01に割り当てられることになる。 - PCR に使用するサンプルを収容するために、96 ウェル PCR プレートにラベルを付けます。各PCRプレートに、プロジェクト名、PCRタイプ、PCRプレート番号、PCRの日付(2020FebruaryAustralia_SSU_1_20200304など)の情報をラベル付けします。また、プレートには、各行にロードされるストリップチューブ番号のラベルを付けます。
- -80°Cの冷凍庫から溶解物質を取り出し、溶解物質を含むストリップチューブを氷上で解凍する。溶解材料が融解している間に、ITS2およびSSUマスターミックスを氷上の別々のチューブで調製する。SSU および ITS2 PCR レシピは、 補足表 2 に記載されています。
メモ: ピペッティングエラーを許容するために、各 96 ウェルプレートに対して PCR マスターミックスを 100 反応準備します。大容量を使用する場合は、15 mL または 50 mL の円錐形を使用してマスターミックスを保持します。 - Taqがミックス全体に分散するまで、マスターミックスを穏やかに渦巻きます。混合したら、マスターミックス38 μLを氷上のPCRプレートの適切なウェルにアリコートします。滅菌シングルユース v-bottom トラフと 12 ウェルマルチチャンネルピペットを使用して、マスターミックスを PCR プレートに移します。
- 解凍した溶解ストリップチューブをスピンダウンして、キャップから溶解材料を除去します。最初の PCR プレートにロードされるすべてのストリップチューブの蓋を慎重に取り外します。少量のマルチチャンネルピペット(12 ウェルまたは 8 ウェル)を使用して、PCR プレートの適切なウェルに 2 μL の溶解液を追加します。2μLを除去する前に、溶解液を1回上下に軽くピペットでピペットする。
メモ: ヒントをチェックして、転送前に溶解が含まれていることを確認してください。行または列の間でヒントを変更することを忘れないでください。 - PCR プレートを PCR 接着箔で覆い、ローラーを使用して密閉を作成します。ホイルを塗布した後、遠心分離機でPCRプレートを短時間スピンダウンします。サーモサイクラーで作動する準備ができるまで、プレートを氷の上に置きます。
- 適切なサーモサイクラープログラムでPCRを実行します。SSU および ITS2 PCR プログラムの詳細については、 補足表 2 を参照してください。
- すべてのPCRが実行されるまで、手順8.4~8.8を繰り返します。
- PCR反応が実行されている間、100 mLの1.5%アガロースゲルを注ぎます。各ゲルは、サンプルまたは単一のPCRプレートを保持する。
- 1.5 g のアガロースを 500 mL フラスコに加え、次いで 100 mL の 1x TAE バッファー (補足表 3) を加え、旋回して混合します。電子レンジでゲルを溶解・冷却する。
- 溶液が冷却されたら、5 μLの10 mg/mL臭化エチジウム溶液を加え、混合して混ぜ合わせる。ゲルが96サンプルとゲル内の各行のためのはしごを収容できるように、4つの25ウェルコームを備えたキャスティングトレイに溶液を注ぎます。
注:臭化エチジウムは強力な変異原です。臭化エチジウムを取り扱う場合は、白衣、耐薬品性手袋、化学安全ゴーグルを使用してください。
- PCR が終了する直前に、使い捨てトラフに 6 倍のローディング色素を追加し、マルチチャンネルピペットを使用して、新しい 96 ウェル PCR プレートの各ウェルに 2 μL の 6 倍ローディング色素を追加します。このプレートは、サンプルをゲルにロードするために使用されます。すべてのサンプルを収容するのに十分なこれらのプレートを作ります。
- PCR が完了したら、PCR プレートを取り出し、RT で 300 x g で 15 秒間短時間遠心分離します。PCR 産物がゲル上で使い果たされるまで、PCR プレートを氷上に保管します。
- 製品をゲル上に流すには、12 ウェルのマルチチャンネルピペットを使用して、2 μL の 6x ローディング色素を含む 96 ウェルプレートの適切なウェルに各サンプルを 5 μL 加えます。
- 次いで、この混合物の6μLを最近鋳造されたゲルの各ウェルにロードする。6 μL の 1 KB プラスラダーをゲルの各列の最初のウェルにロードします。
注: ゲルのウェルを充填するには、ゲルの最初の行の PCR プレートから行 A と行 B を散在させる必要がある場合があります。混乱を避けるために、各PCRサンプルのgel_numberとgel_positionをジェノタイピングシートに記録します。
- プレート内の残りのPCRに新しいホイル蓋を置き、4°Cで保存します。 これらの反応生成物は、ステップ9における配列決定に使用されるであろう。
- PCR 産物をゲル上で 120 V で 20 分間実行します。ゲルを画像化し、どのSラベルがITS2および/またはSSU PCR産物をジェノタイピングシートの「pcr_product_its2」および「prc_product_ssu」列に記録するかを記録する。バンドの存在を「1」でマークする。バンドなしの場合は '0' をマークします。
9. サンガーシーケンシングとシークエンスBLASTによる線虫の同定
注:このセクションでは、SラベルからITS2アンプリコンを配列決定し、BLASTアルゴリズムを使用してこれらの配列を国立バイオテクノロジー情報センター(NCBI)データベースにアライメントし、BLAST結果を解析してSプレート上の線虫を同定する手順を提供します。
- ITS2陽性である各サンプルについて、フォワードプライマーoECA306(CACTTTCAAGCAACCCGAC)を用いたサンガーシークエンシングのために残りのITS2 PCR産物を使用する。各Sラベルの「sequencing_plate」列と「sequencing_well」列をジェノタイピングシートに記録することで、シーケンシング出力ファイルをSラベルに簡単にリンクできるように手配します。
- 各 S ラベルの .seq 出力ファイルをシーケンス プラットフォームから取得します。プロジェクトの .seq ファイルを 1 つのディレクトリに配置し、サブディレクトリにあるシーケンスの各バッチの .seq ファイルを配置します。
- コマンド行インターフェース・ツールを開き、コマンド cd を入力して、.seq ファイルが入っている最上位ディレクトリーに移動します。まだ存在しない場合は、次のコマンドを入力して、すべての .seq ファイルに対してマージされた FASTA を作成します。* / のディレクトリに対して、cd $dirを実行する、*.seq 内のファイルに対して、">" をエコーする$file; cat $file;done >>../all_seqs.fa;cd ..;完了しました。
注: このコードは、プロジェクト ディレクトリ内のすべての .seq ファイルから 'all_seqs.fa' という名前のマージされた FASTA ファイルを作成します。このファイルは、NCBIオンラインヌクレオチドBLASTツールで使用して、各S標識のITS2配列をNCBIの配列データベースに迅速にアラインメントすることができます。 - ウェブブラウザで、NBCI BLASTウェブサイト20に移動し、[ ファイルを選択] ボタンをクリックします。作成したばかりのall_seqs.faファイルを選択し、 やや類似したシーケンス(BLASTn)ボタンをクリックします。ページの下部にある BLASTボタンをクリックして、 BLAST検索を開始します。
- 各SラベルのBLAST結果でジェノタイピングシートを更新します。フィルターツールを使用して、ジェノタイピングGoogleシートの更新を容易にします。[ データ] > [フィルターの作成] をクリックして、各列ヘッダーにフィルター ボタンを追加します。sequencing_plate列をフィルタリングして、BLAST結果で更新するシーケンシングプレートを選択します。
- NCBI BLAST結果ページのドロップダウンメニューを使用して、各SプレートITS2シーケンスの結果を確認します(図6)。
- BLASTがヒットしていないかどうかを確認します。*という接頭辞が付いたドロップダウンのシーケンスIDには、爆発ヒットはありません。これらの S ラベルについては、ジェノタイピング シートの<列に「ヒットなし>現在の日付manual_blast_notes」と入力します。
- 新しいカエノラブディティスの可能性のある種を確認してください。一番上のヒットのリンクをクリックして、アライメントを視覚化します(図6)。トップヒットが(1)カエノラブディティス種であり、(2)アラインメントが配列の中心に5つ以上のミスマッチを含み、(3)クエリカバレッジが50%を超える場合、この結果は、分離株が新しいカエノハブディティス種である可能性があることを示唆している(図7)。これらのSプレートが入ると、一番上のBLASTの種が「species_id」列にヒットし、「possible_new_caeno_sp列」に1を入力し、「可能な新しいCaeno sp.」を「manual_blast_notes」列に「可能な新しいCaeno sp.」と同一性の割合とともに入力します(例:「可能な新しいCaeno sp. 89%の同一性」)。
- Caenorhabditis種にBLASTするSプレート配列の場合、「species_id」列にBLASTがヒットした上位の属名と種名を入力します。たとえば、「Caenorhabditis elegans」などです。
- 非カエノラブディティス種にBLASTするSプレート配列の場合、「species_id」列にBLASTヒットの上位の属のみを入力し、その後に「sp.」と入力します。この表記は、分離株が命名された属内の未知の種であることを意味する。たとえば、'Oscheius sp.' などです。
注:ITS2配列は、Caenorhabditis属の外側の種レベルまでの分離株を確実に同定するために使用することはできません3,13。 - 'make_strain_name' = 'Caenorhabditis elegans'、'Caenorhabditis briggsae'、または 'Caenorhabditis tropicalis'、または 'species_id' = 1 の場合、ジェノタイピングシートの 'possible_new_caeno_sp列' に 1 を入力します。
- Caenorhabditisの命名規則に従って、株に一意の名前を付ける、すなわち、2〜3個の大文字とそれに続く各固有の株の数字からなる一意の実験室指定23。「strain_name」列にひずみ名を入力します。
- 株に名前を付けた後、確立されたプロトコル 24を使用して凍結保存することができます。
10. R の easyFulcrum パッケージでコレクション データを処理する
注: この手順では、easyFulcrum R パッケージを使用して、コレクション データ (C ラベル) と線虫分離データ (S ラベル) をリンクする方法について説明します。このソフトウェアには、Fulcrum データをジェノタイピングシートのジェノタイピングデータとさらに結合する機能が含まれているため、S ラベル種のアイデンティティと株名が 1 つのデータ フレームに編成されます。
- コレクションプロジェクトの名前の新しいディレクトリを作成します。ディレクトリ内のフォルダー構造は、R パッケージ easyFulcrum15 で説明されている要件に合わせて配置します。
- フルクラム Web サイトに移動し、サインインします。左側の Fulcrum Web サイトのデータエクスポートツールを使用して、フルクラムデータベースから生のプロジェクトデータをエクスポートし、プロジェクト、写真を含める、GPS データを含める、フィールドサンプリング、および分離のチェックボックスをオンにします。
注: プロジェクトの支点データは、5 つのコンマ区切り値 (.csv) ファイルとしてエクスポートされます。完全なプロジェクトデータは、RのeasyFulcrumパッケージを使用して単一のデータフレームに結合されます。 - Fulcrum からエクスポートされた 5 つの.csvファイルを、easyFulcrum ビネット 21 の指示に従って、手順 10.1 で作成したプロジェクトディレクトリに移動します。
- Rstudio セッションを開き、R コンソール 'install.packages("devtools")' と 'devtools::install_github("AndersenLab/easyfulcrum")' で次のコマンドを入力して、Rに easyfulcrum パッケージをインストールします。
- 新しい R スクリプトを開き、イージーフルクラム ビネットの指示に従ってコレクション データを処理します21。
結果
このプロトコルは、ハワイやカリフォルニアを含む複数の場所からCaenorhabditis線虫を収集するために使用されています。Caenorhabditis線虫の単離成功率は、採取場所、気候、サンプリング経験、およびサンプリングされた基質タイプによって異なります。このプロトコルは、ハワイ諸島を広範囲にサンプリングするために使用されており、そこでは9つの収集プロジェクトが複数の年と季節にわたって実施されています。自己発症性Caenorhabditis種の単離成功率は、C. briggsae (4,506サンプル中162サンプル、3.6%)とC. elegans(4,506サンプル中163サンプル、3.6%)でほぼ同じであり、C. tropicalis(4,506サンプル中26サンプル、0.58%)ではるかに低い8。自己所有種の各々は、他の基質カテゴリーと比較して腐敗した果実および花の基質上で濃縮される。研究者が基質の嗜好を特徴付けるのではなく、成功率を最大化しようとしている場合、腐った果物および花の基質をサンプリングする。ただし、成功率は、選択した基板の品質によって異なります。例えば、果物および花の基質のうち、乾燥しすぎたり、濡れたり、新鮮すぎたりする基質は、カエノラブディティス線虫を産出しない可能性が高い。
この収集プロトコルのスケーラビリティは、1組の研究者が野生から収集できるコレクションの数から明らかです。例えば、2018年10月、この収集プロトコルを使用した研究者のペアは、2つのハワイ諸島の複数の場所から7日間で合計1,000以上のサンプルを収集することができました。このフィールドチームはサンプルを一晩で実験室に出荷し、そこで8人の研究者のチームが到着時にサンプルから2,000以上の線虫を単離しました。このプロトコルの主な利点は、現場で必要な機器と人員を削減することで、遠隔地でのサンプリングに関連するコストを最小限に抑えることです。このプロトコルを使用すると、小規模なフィールドチームはサンプリングに集中でき、隔離チームは、線虫を単離するための解剖顕微鏡や寒天プレートなどの壊れやすく重い機器を使用して、自宅の施設でサンプルを処理できます。さらに、モバイルデータ収集アプリケーションの実装により、サンプルに関連付けられたすべてのフィールドデータをCラベルに直接リンクできるため、分離チームはサンプルの処理中にフィールドチームから独立して作業できます。
この収集プロトコルを使用する研究者は、収集プロジェクトの前に線虫を単離するために必要な努力を考慮する必要があります。絶縁と同定の手順はレート制限があり、小規模な収集チームはアイソレータをサンプルで迅速に圧倒することができます。さらに、多くのコレクションを処理するために必要な実験室スペースは、進行中の研究を妨げる可能性があります(図3)。さらに、いくつかの単離された線虫は、遺伝子型決定に追加の努力を必要とする。例えば、分離株の約2%は、最初の溶解試行後にSSU PCRプライマーセットで増幅できず、溶解材料がITS2プライマーセットによる増幅に適していることを確認するために再溶解する必要があります(図8)。さらに、分離株の約3%は、サンガーシーケンシングの最初のラウンド後に高品質の配列を生成できません。これらの分離株では、別の溶解ラウンド、ITS2 PCR、およびサンガーシーケンシングがしばしば必要となり、分離チームの引き渡し時間が長くなる可能性があります。重要なことに、配列同一性だけでは、新しい カエノラブディティス 種を正当化するのに十分な証拠ではない(図7)。分離株を新しい カエノラブディティス 種として飼育することを適切に正当化するためには、交配実験を行い、型指定された標本を確立するために追加の努力がなされなければならない13。型指定された標本の正式な形態学的記述も好ましいが、必須ではない3。これらの考慮事項は、この収集プロトコルを採用している研究者が、収集プロジェクトを開始する前にリソースが適切に割り当てられていることを確認するために、分離および識別手順の試験テストの恩恵を受けることを示唆しています。重要なのは、小規模な収集プロジェクトでも、プロセスの再現性が高く、ラボグループ全体の品質管理目的でデータを簡単に監査できるため、このプロトコルの恩恵を受けることができます。

(A)理想的な腐朽した果実が画像(1)の中央に示されているが、果実はほとんど認識できない。腐敗した果物が近くに表示されます。落ちたばかりの果物のサンプリングは避けてください(2)。(B)理想的に分解された花が上部に示されている(3)。落ちたばかりの花のサンプリングは避けてください(4)。(C)乾燥した葉の最上層の下の暗い葉のゴミは、Caenorhabditis線虫を自家採りにサンプリングするときに理想的です(5)。乾燥した葉のゴミのサンプリングは避けてください(6)。この図の拡大版を表示するには、ここをクリックしてください。

図 2: 線虫フィールドサンプリングモバイルアプリケーション。(A) フルクラムのAppleデバイスで線虫フィールドサンプリングアプリケーションを開いた後の初期画面。右下の赤い矢印は、新しいコレクション レコードの作成に使用する [+] ボタンを指しています。(B) Apple デバイスに表示される新しいコレクションレコードの例。赤い矢印は、コレクションレコード画面の上部にある「プロジェクト」フィールドを指しています。現場でサンプリングするときは、必ず正しいプロジェクトを選択してください。project フィールドは、後続のコレクションレコードを作成するときに最後に使用されたプロジェクトにデフォルト設定されます。この図の拡大版を表示するには、ここをクリックしてください。

図3:サンプルをメッキアウトする前に整理された収集バッグと収集プレート。 この図は、左側の C ラベルの付いた収集バッグのサンプルを示しています。各コレクションバッグには、その上に一致するCラベルの10cmプレートがあります。右側には、収集バッグから移した後のサンプル材料を含む10cmの収集プレートがあります。 この図の拡大版を表示するには、ここをクリックしてください。

図4:適切に転写されたサンプルを含む収集プレート(Cプレート)。 分解する果実が入った10cmのCプレートを細菌芝生の端に置いた。Cラベルはプレート蓋に取り付けられています。 この図の拡大版を表示するには、ここをクリックしてください。
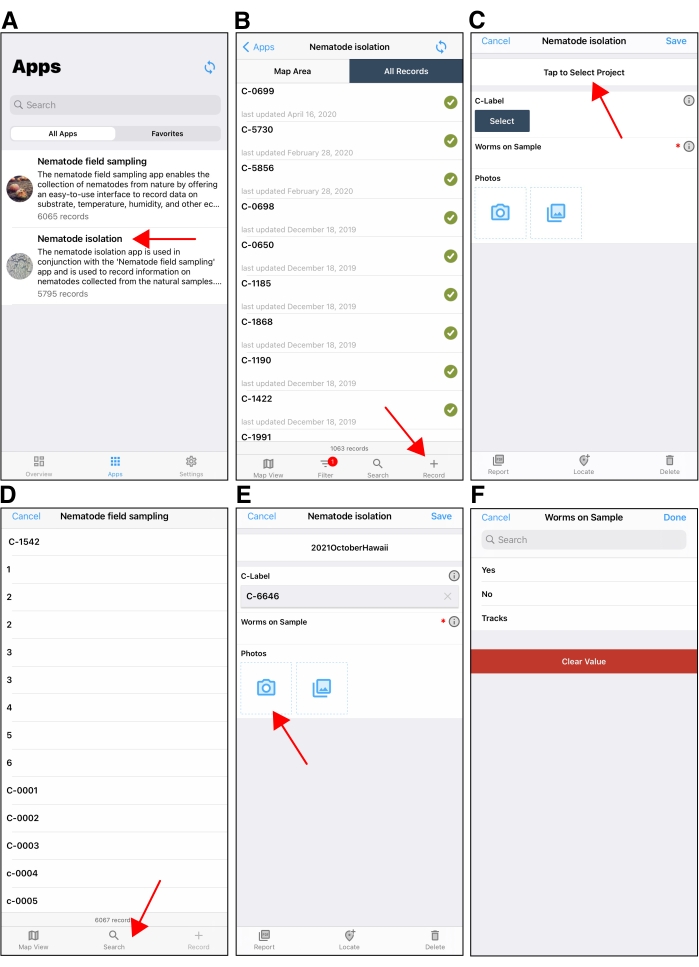
図5:線虫分離モバイルアプリケーション。 (A)Fulcrumモバイルアプリケーションのアプリケーション選択画面。赤い矢印は線 虫分離 アプリケーションを指しています。(B) Fulcrum の Apple デバイスで 線虫分離 アプリケーションを開いた後の初期画面。右下の赤い矢印は、新しい分離レコードの作成に使用する [+ ] ボタンを指しています。(C) Apple デバイスに表示される新しい分離レコードの例。赤い矢印は、分離記録画面の上部にある「プロジェクト」フィールドを指しています。分離するときは、必ず正しいプロジェクトを選択してください。project フィールドは、後続の分離レコードを作成するときに最後に使用されたプロジェクトに既定で使用されます。(D) Cラベルの下の [選択] フィールドをタップすると、ユーザーは検索ボタン(赤い矢印)をタップして、線虫を隔離しているCラベルを見つけます。(E)Cラベルを選択した後、ユーザーはデバイスのカメラを使用してCプレートを撮影します。(F)その後、ユーザーはCプレート上に線虫があるかどうかを入力します。分離する線虫がある場合、S ラベルが単離レコードに追加されます。 この図の拡大版を表示するには、ここをクリックしてください。
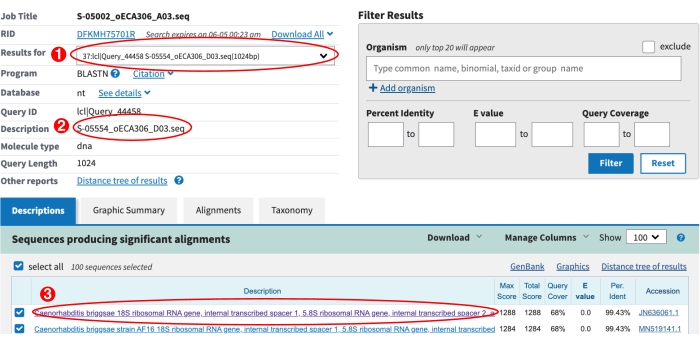
図 6: NCBI BLASTの結果ページ。 (1)すべてのシーケンスのBLAST結果を表示するために使用するドロップダウンメニュー。(2) ドロップダウンから選択した現在のシーケンスの説明。この場合、SラベルS-05554の結果が表示されます。(3)S-05554の一番上の爆風ヒットが示されています。紫色のテキストは、この配置を視覚化するためのリンクがクリックされたことを示します。可能性のある新しい Caenorhabditis 種を特定するために、目でアライメントを検査してください、上記のステップ9.8を参照してください。 この図の拡大版を表示するには、ここをクリックしてください。

図7:NCBI BLASTアラインメントの可視化例。 (A) C. kamaaina サブジェクト配列にアラインメントされた分離株のITS2クエリ配列の例。(1) アライメントの同一性パーセント (89%)で、BLASTヒット数が多いほど低い。(2) クエリとサブジェクト シーケンス (G から A) の間の不一致。(3)前記アラインメントアルゴリズムにより作製された前記被験体配列における4塩基対ギャップと、クエリまたはサブジェクトのギャップは、アライメントが不十分であることを示します。(4)多くのミスマッチやギャップのあるアライメントの中心にある一般化された領域。このような領域は、クエリシーケンスが新しい Caenorhabditis 種から来ている可能性があることを示唆しています。2017年に発見された新種 C. oiwiの実際のアライメント例が示されています。(B)分離株のITS2クエリ配列と対象配列との間の良好なアラインメントの例。(5)アラインメントの同一性パーセント(99%)は、通常、クエリ配列が対象と同じ種の単離物に由来することを意味する。(6)完全な同一性を有する整列の中心領域。このような領域は、クエリ分離がサブジェクトと同じ種である可能性が高いことを示唆しています。 この図の拡大版を表示するには、ここをクリックしてください。
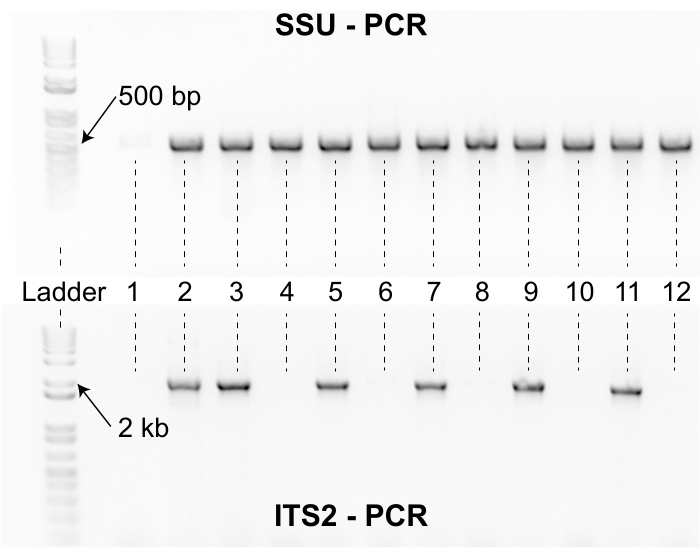
図 8: SSU および ITS2 PCR 産物 上のゲルは、12の代表的なサンプルに対するSSUプライマーセットを用いて生成されたPCR産物を示す。DNAはしごが参考として左側に含まれています。 カエノラブディティス 線虫のSSU PCR産物の長さは約500bpです。サンプル2〜12はSSUプライマーセットで増幅したが、サンプル1はそうしなかった。サンプル1に500bp SSUアンプリコンがないことは、溶解材料の品質が悪く、サンプルを再溶解しなければならないことを示唆しています。下のゲルは、上のゲルに示したのと同じ12サンプルのITS2プライマーセットで生成されたPCR産物を示す。はしごおよびサンプルは、両方のゲルに対して同じ配向にある。12サンプルのうち6サンプルはITS2プライマーセットで増幅しなかった。SSU および ITS2 バンドを持つサンプルはサンガー配列決定され、NCBI BLAST アルゴリズムを使用して配列類似性によって同定されます。 この図の拡大版を表示するには、ここをクリックしてください。
補足ファイル 1: C ラベル。 2500 個の一意の C ラベルを含む PDF ファイル。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 2: S ラベル。 5000 個の一意の S ラベルを含む PDF ファイル。 このファイルをダウンロードするには、ここをクリックしてください。
補足表 1: フィールド資料 線虫をサンプリングするために現場で使用される材料のパッキングリスト。 この表をダウンロードするには、ここをクリックしてください。
補足表 2: PCR レシピおよびサーモサイクラー条件。 ITS2 および SSU PCR の PCR レシピとサーモサイクラー条件の表です。 この表をダウンロードするには、ここをクリックしてください。
補足表3:電気泳動バッファーレシピ。 0.5 M pH 8.0 エチレンジアミン四酢酸溶液 (EDTA) および TRIS-酢酸-EDTA (TAE) 緩衝液のレシピ。 この表をダウンロードするには、ここをクリックしてください。
ディスカッション
このプロトコルには、注意して実行する必要がある重要な手順が含まれています。たとえば、フィールドチームと分離チームは、フィールドからサンプルを収集したり、実験室のサンプルから線虫を単離したりする前に、アプリケーションで正しい収集プロジェクトを選択するように注意することが重要です。間違った収集プロジェクトが選択された場合、誤ったデータレコードは、オンラインでレコード編集ツールを使用してフルクラムデータベースで修正するのが最適です。このプロセスは、多くの誤って配置されたレコードにとって面倒な作業になる可能性があります。ただし、データベースはレコードに対する変更を保持するため、収集レコードと分離レコードの完全な監査が可能になります。このプロトコルの他の重要なステップには、フィールドからのサンプルと、それらのサンプルから単離された線虫の処理が含まれます。Caenorhabditis線虫がサンプリングおよび出荷ステップで生き残ることを保証するために、サンプルの温度は4°C〜25°Cの間で維持されるべきである。 25°Cを超える温度は、C. elegans14に無菌を引き起こす可能性があります。線虫への損失を最小限に抑えるために、可能な限り5日以内にサンプルを収集バッグから収集プレートに移すようにしてください。線虫が単離された後、それらが滅びる前に遺伝子型決定され、凍結保存されることが重要である。真菌や細菌の汚染がSプレートを不快にさせる可能性があるため、生後2〜3週間以上のSプレート上に生きた線虫を見つけることは困難です。
このプロトコルは、研究者が現場にいる間に収集したいさまざまな種類のデータに対応するために簡単に変更できます。たとえば、「線虫フィールドサンプリング」アプリケーションは、アプリケーション編集用のフルクラムのオンラインGUIを使用して、新しいデータ入力フィールドで簡単にカスタマイズできます。さらに、データ分析パッケージ easyFulcrumは、新しいデータを処理するときにこれらの編集に対応できます15。ユーザーが魅力的に感じるかもしれない別の修正は、現場で異なるサンプリング方法を使用することです。研究者は、離散的な基板をサンプリングするのではなく、複数の基板タイプを含むより大きな領域をサンプリングしたいと考えるかもしれません。これらのより大きなサンプルは、Baermann漏斗またはトレイ抽出方法を使用して実験室で最もよく処理されます13。重要なことに、CラベルとSラベルの使用は、これらの技術に依然として適用され、したがってモバイルアプリケーションと互換性があります。
このプロトコルの主な制限は、実験室での単離前の線虫の取り扱い時間に関連する。第一に、試料採取と線虫単離との間のラグタイムは、採取時に所与の試料上の線虫の発生段階を記録することを不可能にする。第二に、自然界における雄の頻度と交配頻度は 、カエノラブディティス 線虫を自殖させるための重要な進化上の疑問です10。この方法は、線虫が単離される前に何世代も経っている可能性が高いため、これらの質問に対処するのにはあまり適していません。遅延分離は、本質的に男性の頻度の直接的な証拠が不可能であることを意味します。さらに、ジェノタイピングステップ中の多世代にわたる遅延は、線虫株を配列決定する前に、アウトクロス(ヘテロ接合性)のゲノム証拠が侵食されることを意味する。自然界におけるヘテロ接合性を同定するために、自然界から直接単離された線虫によって産生される子孫をシーケンシングに使用する2。このプロトコルの別の潜在的な制限は、自己発症 性Caenorhabditisの同定に偏っていることである。これは、自走種の孤立した線虫は、受精した雌が単離された場合にのみ増殖する義務的なアウトクロッサーよりも増殖する可能性が高いためである。
この収集方法は、既存の収集プロトコル13,14に基づいています。この技術の主な進歩は、大規模な収集プロジェクトに関連する膨大な量の生態学的および分子的データの組織化を容易にするために、モバイル技術とカスタマイズされたソフトウェアの使用である。この収集プロトコルを使用して生成された生態学的データは、Caenorhabditis種の自然集団に対する未解決の問題に対処するために使用することができる。例えば、この方法で生成されたデータは、ハワイ諸島全体の種のニッチな好みを発見するために使用されてきました。さらに、凍結保存された線虫のゲノムを配列決定することにより、研究者は遺伝的変異のパターンが生態学的データとどのように相関しているかを調査することができます。この種の研究は、カエノラブディティス集団における局所適応の兆候を明らかにし、自然の文脈における遺伝的変異の関連性に関する重要な洞察を提供することができます8。Caenorhabditis線虫の多くの遺伝子の機能的理解を得るためには、生態学的研究が必要になる可能性が高い11。C. elegansでさえ、配列決定された最初の多細胞動物であり、地球上で最も徹底的に研究された動物の1つであるにもかかわらず、遺伝子の大部分は機能的注釈を欠いている。この収集プロトコルは、野生のカエノラブディティス線虫の収集とその生態と自然の遺伝的多様性の研究を促進することによって、この知識のギャップに対処するために開発されました。
開示事項
著者らは利益相反を報告していない。
謝辞
この研究は、ノースウェスタン大学からのスタートアップ資金と、米国国立科学財団のキャリア賞(IOS-1751035)によって支援され、どちらもE.C.A.に授与されました。
資料
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
参考文献
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved