Method Article
一种高度可扩展的方法,用于对自发 性Caenorhabditis 线虫进行生态调查
摘要
该协议可用于对自生 的Caenorhabditis 线虫进行大规模的生态调查。该方法的主要优点是有效地组织和分析与从自然界收集的线虫相关的生态和分子数据。
摘要
秀丽隐杆线虫 是生物学中的主要模式生物之一,但直到最近,研究人员才开始关注其自然生态学。关于 秀丽隐杆线虫 在其自然环境中的信息相对稀少,这来自于识别自然界中小线虫所涉及的挑战。尽管面临这些挑战,但对 秀丽隐杆线虫 生态学的日益关注已经引起了关于其实验室外生活的大量新信息。在自然界中对 秀丽隐杆线虫 的强化寻找有助于发现许多新的 Caenorhabditis 物种,并揭示了同源线虫经常在野外共存,在那里它们以与腐烂的植物材料相关的微生物水华为食。新物种的鉴定还表明,雄性和自育雌雄同体的雄激素交配系统在 Caenorhabditis中独立进化了三次。另外两个自给自足的物种, C. briggsae 和 C. tropicalis,具有 秀丽隐杆线虫 的实验优势,并且能够对重要性状的机制基础进行比较研究,包括自我受精。尽管取得了这些进展,但这些重要物种的生态和自然多样性仍有待了解。例如,我们仍然缺乏它们许多基因的功能信息,这可能只有通过了解它们的自然生态才能获得。为了促进自发 性Caenorhabditis 线虫的生态学研究,我们开发了一种高度可扩展的方法从野外收集线虫。我们的方法利用移动数据收集平台,基于云的数据库和R软件环境来增强研究人员从野外收集线虫,记录相关生态数据以及使用分子条形码识别野生线虫的能力。
引言
在过去的二十年中,人们对线虫的生态学越来越感兴趣。从这些研究中,我们知道自由生活的Caenorhabditis物种可以从温带和热带地区的短暂微生境中分离出来,在那里它们以与分解植物材料相关的微生物水华为食,有时在sympatry1,2,3,4,5,6,7,8.我们还了解到,该属已经发生了三次自我受精的趋同进化,并且自发是C. briggsae,C. elegans和C. tropicalis的主要繁殖模式9,10。在这些自我者中,秀丽隐杆线虫是地球上研究最广泛的动物之一,已被研究人员用于在生物学方面取得关键进展。重要的是,其他自生的Caenorhabditis物种具有许多秀丽隐杆线虫的实验优势,并且正在迅速推进该属的比较研究。然而,这些线虫在野外的神秘性质使得研究其生态学和自然多样性变得困难,这对于了解其基因的生物学功能以及进化塑造物种遗传多样性的方式至关重要10,11。
研究野外自生 Caenorhabditis 线虫生态学的最大挑战是它们的小尺寸;成虫线虫的长度通常为1毫米或更小。这一挑战要求研究人员从野外采集底物样本,并试图在实验室中将感兴趣的线虫与底物分离,而无需在野外观察动物。因为即使是训练有素的专家也很难在显微镜下将自生 的Caenorhabditis 线虫与其他自由生活的线虫区分开来,因此线虫通常从底物中取出,分离出来,并在使用已建立的分子条形码通过序列标识进行鉴定之前进行增殖3,12,13,14.以这种方式处理每条线虫所需的时间和精力构成了组织上的挑战,因为研究人员必须能够将实验室中分离出的每条线虫的身份追溯到确切的底物和在现场采样的相关生态数据。在这里,我们描述了一个循序渐进的过程,以有效地从现场收集和识别自发 的Caenorhabditis 线虫,并在高尺度上忠实地将这些分离物与其相关的空间和生态数据联系起来。
这种收集方法通过使用移动数据收集平台、基于云的数据库和R软件环境来提高生态调查的规模和准确性。Fulcrum是一个可定制的数据收集平台,可与大多数移动设备配合使用,并允许用户构建自定义应用程序来收集和组织基于位置的数据(https://www.fulcrumapp.com)。该协议提供了有关如何使用定制数据收集应用程序来组织来自现场的空间显式生态数据的详细说明,并将这些数据与实验室中分离的线虫的身份准确联系起来。该协议还解释了如何使用已建立的分子条形码有效地识别自我的Caenorhabditis线虫。来自这些方法的数据可以使用随附的R软件包easyFulcrum15进行简单且可重复的处理,以探索天然Caenorhabditis种群的生态学和遗传多样性。
研究方案
1. 收集准备
- 确定一个位置来调查 Caenorhabditis 线虫。
注意:在大多数温带地区, 秀丽隐杆线虫 和 布里格塞氏体 可以很容易地与农业田地或农村和城市花园等人类相关的栖息地隔离1。在亚热带和热带地区, C. briggsae, C. elegans和 C. tropicalis 都可以在上面列出的与人类相关的栖息地中找到,有时彼此非常接近。然而, 秀丽隐杆线虫 似乎更喜欢更凉爽、更干燥的栖息地,而不是热带栖息地中的其他物种7,8。每个物种也可以从与人类无关的野生栖息地中分离出来,但这些栖息地的采样频率较低。 - 创建支点项目,以使用移动数据收集应用程序组织收集和隔离数据。
- 使用免费教育协议在线创建支点帐户16。通过单击"添加应用程序"按钮17,将线虫现场采样应用程序添加到支点帐户。
- 通过单击"添加应用"按钮18将线虫隔离应用程序添加到帐户。
注意:建议使用命名约定"YearMonthLocation"(例如,2020年2月澳大利亚)将每次前往某个地点的旅行组织为其收集项目。
- 将用户添加到支点帐户以授予他们对集合项目的访问权限。确保每个用户下载支点移动应用程序以参与项目。
- 打印一组QR码标签,以使用移动应用程序跟踪集合(C标签)和线虫隔离(S标签)。将C标签贴在拉链锁塑料袋上,将贴有标签的塑料袋卷成25个,并用橡皮筋包裹以进行包装。保留一套S标签,以便在实验室中使用。
注意:在整个协议中,集合(来自现场的基质)包含在袋子或板上,并用C标签标记。分离的线虫用S标签标记。C标签用于识别唯一集合,S标签用于识别唯一线虫分离物。这两种类型的标签用于在特定集合(C标签)与支点数据库中与该集合隔离的线虫(S标签)之间建立连接。对于一个集合项目,将两倍于 S 标签的数量打印为 C 标签,因为平均每个集合隔离两个线虫。如果需要,以后可以打印更多S标签。补充文件中提供了 2,500 个唯一的 C 标签(补充文件 1)和 5,000 个唯一的 S 标签(补充文件 2)。 - 准备10厘米NGMA板用于收集和3.5厘米NGMA板用于分离线虫。每个收藏制作一个 10 厘米的盘子和至少两个 3.5 厘米的盘子21。这些板按照既定的方案接种 大肠杆菌 菌株OP50。使用前将板在4°C下储存不超过1个月。
2. 现场收集
注意: 线 虫最常从腐烂的蔬菜材料中分离出来,包括水果,坚果,种子,豆荚,花,茎,植物凋落物和堆肥1,5,6,8。最好的基质是腐烂的,几乎无法识别为水果或花朵;避免使用太干或太湿的基材(图1)。通过成对工作,从现场收集基质最有效。使用非接触式红外测温仪的个人将选择用于收集的基板并收集样品,而其伴侣则使用支点中的线虫现场采样应用程序来记录收集数据。一对收集器将重复此过程,直到收集到所需数量的样品。实地工作所需材料清单见(补充表1)。
- 打开支点移动应用程序,从下拉菜单中选择 线虫场采样。按 + 在项目中开始一个新记录(图 2A)。拍摄基材的照片。
- 单击顶部中心的框以选择在步骤1.2中进行的正确收集项目(图2B)。点击集合记录底部的 C 标签 字段,然后在出现提示时选择"扫描"。使用移动设备摄像头 扫描 收集袋上的条形码,然后点击屏幕右上角的 完成 。
- 点击 "基板" 字段,然后从下拉菜单中选择基板类型。通过点击"基底注释"字段并手动输入注释来添加有关 基材 的注释。
- 从下拉菜单中选择一个景观。选择最能代表采样地点的景观。
- 选择天空视图。选择天空视图时,请描述采样点的天空能见度(例如,没有树木或其他结构阻挡视图的全天空视图 =完整)。
- 使用非接触式温度计测量基板的表面温度,并在基板温度场中记录该值。
注:在记录温度时,将非接触式温度计保持在离基板不超过14英寸的地方。 - 使用手持设备测量环境温度和湿度,并将这些数据记录在正确的字段中。
注:检查环境温度和湿度设备是否未处于 保持状态。当按钮被释放时,测量单位将更改。将设备放在外部口袋中,以避免读数不规则。 - 通过点击屏幕左上角的 "保存",将记录保存 在支点中。
- 通过倒置收集袋将其用作"手套"来收集大约一汤匙的基材,没有棍子或其他硬块,然后密封袋子。如果样品特别潮湿,请在袋子里放一张纸巾。
注意:在炎热的气候下,将袋子放在带有较冷包装的软冷却器中,以保持收藏品凉爽。 - 收集当天的所有样品后,清洁收集设备,从探头中取出电池,为电池充电,重新冷冻冷冻冰箱包装。通过点击 线虫现场采样 应用程序左上角的同步按钮来同步支点收集数据。
注意:如果没有强大的蜂窝网络连接,上传可能需要几分钟时间,因此最好等待WiFi访问。数据将保留在移动设备上,并将同步到云。 - 通过将样品放入隔夜运输箱中,将样品运送到家庭机构。在运输货物的日子里,通过运输包裹,最大限度地减少样品暴露在低于11°C或高于25°C的温度下的时间。
注意:大多数运输设施不会在周末在偏远地区过夜运输货物。
3. 在实验室中电镀现场收集
注意:本节详细介绍了如何将样品从贴标签的收集袋转移到贴标签的板。样品可能来自隔夜发货或直接从现场发货。
- 接收收货件,并检查破损的袋子或其他损坏的证据。如果袋子破损,丢弃材料并用70%乙醇清洁未破碎的收集袋;避免在装有乙醇的袋子上贴上C标签,因为它会使标签变色并使其难以阅读。
- 对于每个拉链锁袋,请注意袋子上的C标签,并将匹配的C标签贴在标有OP50细菌的10厘米板的盖子上。
注意:在协议的其余部分,标记的10厘米板被称为"C板"。组织样品的最简单方法是将收集袋放在实验室工作台上,顶部有匹配的C板(图3)。 - 对于每次收集,使用干净的塑料勺将大约一汤匙样品从收集袋转移到C板。将样品以新月形或环形添加到细菌草坪周围,不要完全覆盖细菌草坪(图4)。
注意:在不使用时,将汤匙放入95%乙醇的烧杯中,以保持汤匙清洁。在转移其他样品之前,使用纸巾干燥汤匙。 - 记录收集物从收集袋转移到C板的时间,并将C板在室温(RT)下保持至少24小时,然后再尝试在第4部分中分离线虫。
4. 从集合中分离线虫
- 在移动设备上打开 支点 应用程序,然后从应用程序菜单中选择 线虫隔离 (图5A)。通过点击右下角的 + 图标创建新的隔离记录(图 5B)。
- 在新的隔离记录屏幕中,通过选中顶部中心框中显示的项目名称来确认正确的集合项目。如果显示错误的项目,请点击项目名称以切换到正确的项目(图5C)。
- 点击C标签字段下的"选择"按钮,找到与从中分离线虫的样品相关的C标签(图5D)。点击搜索图标,然后点击扫描图标,使用设备摄像头扫描C板上的C标签QR码。扫描QR码后,C标签记录将显示在C标签字段中。
- 点击"照片"字段中的"相机"图标以打开设备相机,并使用它来拍摄C板上样品的照片,并显示QR码(图5E)。点击完成以返回到隔离屏幕。
注:这些隔离记录照片可用于在以后探索基板的特定属性。 - 使用解剖显微镜在C板上寻找线虫。点击 样品上的蠕虫 字段以记录样品上线虫的存在(图5F)。 如果C 板上存在线虫,请点击"是",如果没有线虫,则点击" 否 "。
- 如果只有线虫 踪迹 存在,请点击轨道。如果没有线虫存在,请对C板进行parafilm并将其丢弃在生物危害箱中。
注意:将C板倒置在生物危害废物箱上,轻轻敲击板的背面以移开所有取样的基质。这一步使得更容易找到和分离可以在C板上基底下的线虫。 - 如果存在线虫,从C板中分离出最多五条线虫。要隔离线虫,请使用铂线镐将一个线虫从C板转移到S板。如果可能的话,隔离健康的、有妊娠的成年人。但是,如果未发现成人,则隔离其他阶段。
注意:隔离后,每个S板上最多有五个线虫。将这些S板与来自同一C板的孤立线虫一起组织在一起,整齐地堆叠在一起,远离其他S板,直到它们进入支点。 - 点击 "S 标记的板" 字段以输入用于此隔离的 S 板。点击右下角的 + 。点击 S标签, 然后单击 扫描 以打开设备相机。使用设备摄像头扫描 S 板上的 S 标签二维码。
注:确保 S 标签代码与印版上的代码匹配。如果匹配,请点击 完成。如果没有,请点击 "取消" 并重新扫描,直到匹配,然后单击" 完成"。有时附近印版的QR码会被意外扫描。 - 输入每个 S 板后,使用右上角的"保存"按钮 保存 条目。如果未保存,该条目将丢失。如有必要,点击右下角的 + 以添加更多S标记的板,直到输入与C板分离的所有线虫。为隔离记录添加所有S标记的板后,点击左上角 的< 按钮返回隔离记录屏幕。
注意:要取消隔离记录,因为错误无法解决,请单击左上角的 "取消" 。此步骤将打开一个对话框,询问是否可以在不保存的情况下丢弃记录。如果需要,请单击" 是,放弃"。 - 点击右上角的" 保存 "按钮,一旦隔离记录正确添加了所有信息。然后用孤立的线虫对S板进行parafilm,并将它们放在指定用于容纳带有线虫的S板的区域。
- 对C板进行Parafilm并将其丢弃在生物危害箱中。点击 同步 图标将所有数据上传到支点。
- 将所有S板按字母数字顺序分类,然后将S板放入纸板箱中。确保 S 板的盖子朝下并铺有胶片。最多将四个S板堆叠到盒子的一个位置,并在纸板箱上标有项目名称,日期,时间和唯一的箱号。
- 将贴有标签的盒子存放在 RT 处。这些分离株将在48小时检查增殖情况,如有必要,将在168小时再次检查。
5. 从支点出口S板
注意:本节详细介绍了如何从支点项目数据库导出隔离过程中使用的 S 标签。这些S标签将用于跟踪增殖的异性系,同时通过第6-9节中的序列标识来识别它们。
- 登录到支点网站,然后选择 线虫隔离 应用程序。点击 导出器 从屏幕左侧。单击以选择所需的项目,然后选中 线虫隔离 框。单击" 下一步 "下载包含"nematode_isolation_s_labeled_plates.csv"文件的.zip文件。
- 打开"nematode_isolation_s_labeled_plates.csv"文件,然后按"S标签"列按升序排序(最小的S标签将位于顶部)。选择所有 S 标签,然后从电子表格中复制它们。
- 使用网络浏览器导航到野生分离基因分型模板谷歌表(wild_isolate_genotyping_template)19。
- 通过右键单击" 基因分型模板 "选项卡,然后选择" 复制到新电子表格 "选项来制作此Google工作表的副本。选择 打开电子表格 以查看新的Google工作表。
- 用支点项目名称命名这个新工作表,后跟"wild_isolate_genotyping",例如,"2020FebruaryAustralia_wild_isolate_genotyping"。
注意:该表在方案的其余部分被称为"基因分型表"。
- 将从"nematode_isolation_s_labeled_plates.csv""s_label"列复制的S标签粘贴到标题为"s_label"的基因分型表列中。检查"s_label_repeat_error"列中的"1"。此列中的值为"1"表示 S 标签在基因分型表上的某个位置重复。如果发现重复项,请在继续操作之前进行调查并更正它们。
- 填写所有 S 标签的基因分型表"isolation_box_number"列。
6. 检查S型板上的增殖情况
- 隔离后48小时检查S-plate上的增殖动物(使用步骤4.11中盒上最后一次隔离的日期和时间来指导时间)。
注意:增殖线虫的特征是S-板上的后代。 - 如果S板增殖,请在基因分型表的proliferation_48列中输入"1",然后将S板移动到标有"48 h增殖,框1"的框中。在增殖箱中最多放置88个S板,然后开始填充标有"48小时增殖,箱2"的新箱。确保S标签在48小时扩散盒中按字母数字顺序组织。
注意:不要处理不扩散的S板;这些板将在隔离后168小时再次检查。如果需要,将这些S板按数字顺序合并到标有"48小时不扩散,框X"的框中,但请记住记录何时必须在新盒子上进行168小时检查。 - 在48小时鉴定出所有增殖的S-标签后,继续进入第7部分,用于在48小时增殖的S-板。
- 在隔离后168小时再次检查隔离后48小时未增殖的S板。
- 如果S板现在正在增殖,请在基因分型表上proliferation_168列中输入"1",然后将S板移动到标有"168 h增殖,框1"的框中。
- 在增殖箱中最多放置88个S板,然后开始填充标有"168小时增殖,箱2"的新箱。确保在168小时扩散框中按字母数字顺序组织S标签。
- 丢弃168小时后没有增殖的S板。转到第7部分,用于在168小时增殖的S板。
7. 异戊二线的裂解
注意:此步骤将使用Google表格中的数据过滤工具来帮助打印扩散框中S板的裂解工作表。裂解工作表的目的是为工作人员提供工作台上裂解试管中S标签的正确位置。
- 打开所需项目的基因分型表,然后通过键入 Cmd+A 选择所有单元格。单击" 数据>创建筛选器" ,将筛选器按钮添加到每个列标题。使用" 过滤器 "按钮仅显示将要进行基因分型的 S 板。例如,如果要裂解所有在48小时时增殖的S板:单击"proliferation_48"列中的"过滤器"按钮,然后选择"1"。
- 过滤基因分型Google工作表后,请查看显示的S标签列表,以确保它们是要打印在工作表上的S标签。
- 在基因分型谷歌工作表的"strip_tube_number"列中,每11行输入一个唯一的数字。
- 按从 1 开始的连续顺序输入项目的条带管编号,并且从不重复。在"strip_tube_position"中,为每个条形管编号输入 2 到 12。
注意:使用12管条带管进行裂解。第一个位置(strip_tube_position 1)将是控件,但控件不会添加到裂解工作表中(仅添加strip_tube_positions,2-12)。在裂解时,阳性对照菌株"N2"将作为阳性对照添加到每个偶数条带管的位置1中。没有蠕虫被添加到每个奇数号条带管的位置1作为阴性对照。
- 进一步过滤基因分型谷歌工作表,在一个要裂解的扩散框中仅包含S标签,然后选择"s_label"到"lysis_notes"列。为每个要裂解的增殖箱打印裂解工作表。
- 单击 打印字段中的下 拉菜单,然后选择 所选单元格。单击右上角的" 下一步 ",然后使用对话框打印扩散框的裂解工作表。
- 重复步骤7.3-7.5,为每个增殖箱打印裂解工作表。
注:每个增殖箱最多可容纳 88 个 S 板,相当于 8 个 12 孔条形管。 - 为所有将要裂解的样品准备12孔条带管。使用裂解工作表中指定的唯一"strip_tube_number"标记条带管。该标签必须写在盖条和带管上,以避免在分开时混淆。EVEN条形管在位置1处具有阳性对照(N2蠕虫)。ODD条形管在位置1处具有阴性对照(无蠕虫)。
- 为所有样品补充足够的裂解缓冲液(100 mM KCl,20 mM Tris pH 8.2,5 mM MgCl2,0.9%IGEPAL,0.9%吐温20,0.02%明胶,加入蛋白酶K至终浓度0.4mg / mL),并额外添加5%用于移液器错误。根据需要进行缩放。
注意:裂解缓冲液最好通过结合除蛋白酶K以外的所有成分并在-20°C下冷冻在10-50mL等分试样中制备。 解冻等分试样,并在使用前保持在4°C;使用前立即加入蛋白酶K并充分混合。工作时将裂解缓冲液保持在冰上。 - 使用打印的裂解工作表作为指南,按顺序排列特定条带管的S板。
- 取出一个条形管,并用重复移液器向每个盖子加入8μL裂解缓冲液。一次将裂解缓冲液添加到一个盖子条中,因为如果将裂解缓冲液留在室温下并未盖开,裂解缓冲液将蒸发。从源板(S-板或N2储备板,用于阳性对照)中选取3-5只动物到裂解工作表上指示的适当盖位置。在裂解工作表的lysis_notes部分中记录任何S板的注释,其中选择的蠕虫少于5个。
- 将线虫加载到条带管的每个位置后,将帽条放回条带上。将标记的盖子(位置 1)与标记的管(位置 1)相匹配。封盖后,短暂离心条带管,直到线虫位于管的底部。
- 将条带放入-80°C冰箱中,直到完全冷冻(至少10分钟)。重复步骤7.9至7.11,直到所有条带都添加了线虫进行裂解。按数字顺序组织管条。
- 取出成套的条带管并在热循环器中运行裂解程序:在60°C下1小时,在95°C下15分钟,在12°C下保持。 裂解程序完成后,在室温下以300× g 离心样品15秒,并将裂解物在-80°C下储存长达1周。
- 使用96孔板架按数字顺序组织试管条,并包括带有增殖盒编号,条带管编号范围,日期和研究人员首字母缩写的标签。使用裂解工作表中的信息更新基因分型表列"lysis_date"和"lysis_notes"。
8. SSU和ITS2序列的PCR
注:本节将说明如何对每个裂解的S板进行两个单独的PCR。第一个引物集扩增18S rDNA小亚基基因(SSU)的500-bp片段;oECA1271 = 前向引物 TACAATGGAAGGCAGCAGGC, oECA1272 = 反向引物 CCTCTGACTTTCGTTCTTGATTAA 12.该PCR用于检查模板DNA的质量。PCR 扩增了几乎所有线虫物种的 SSU 区域。如果SSU PCR无法扩增,则该结果表明裂解质量较差,必须对该S板重复裂解。第二个引物集扩增5.8S和28S rDNA基因(ITS2)之间内部转录间隔区的2,000-bp片段;oECA1687 = 正向引物 CTGCGTTACTTACCACGAATTGCARAC, oECA202 = 反向引物 GCGGTATTTGCTACTACCAYYAMGATCTGC3.ITS2 PCR产物是Sanger测序的,该序列用于通过序列相似性将Caenorhabditis属中的线虫鉴定到物种水平。
- 使用基因分型表中的过滤工具仅查看用于 PCR 的 S 标记。
- 更新pcr_plate_number,并在基因分型表中pcr_well列。为了防止裂解材料的降解,SSU和ITS2 PCR同时运行。
- 对 ITS2 和 SSU PCR 使用相同的pcr_plate_number,即使它们是在单独的板中单独反应。它们将使用"SSU"或"ITS2"标签进行区分。
- 为八个或更少的条带管分配一个pcr_plate_number(每排96孔PCR板一个条带管,按升序排列,例如,顶部的最低条带管数)。然后为条带管中的每个S标签分配一个pcr_plate_well。
注:带钢管按升序排列,最低的带钢管编号分配给A行,H行中编号最高。因此,将1号带钢管、位置1分配给PCR板号1、孔A01。 - 标记 96 孔 PCR 板,以容纳将用于 PCR 的样品。用以下信息标记每个PCR板:项目名称、PCR类型、PCR板编号和PCR日期(例如,2020FebruaryAustralia_SSU_1_20200304)。此外,用将加载到每行中的条带管编号标记板。
- 从-80°C冰箱中取出裂解材料,并在冰上解冻含有裂解材料的条带管。当裂解材料解冻时,在冰上的单独管中制备ITS2和SSU预混液。SSU和ITS2 PCR配方可在 补充表2中找到。
注意:为每个96孔板准备100个PCR预混液反应,以允许移液错误。如果要使用大容量,请使用15 mL或50 mL锥形来保持预混液。 - 轻轻涡旋预混料,直到Taq分布在整个混合物中。混合后,将38μL预混液等分到PCR板的冰上的适当孔中。使用无菌一次性V型底槽和12孔多通道移液器将预混液转移到PCR板中。
- 向下旋转解冻的裂解条管,以从盖子上除去裂解材料。小心地取出所有将装入第一个PCR板的条形管的盖子。使用低容量多通道移液器(12 孔或 8 孔)将 2 μL 裂解物加入 PCR 板中的相应孔中。在取出2μL之前,轻轻地上下移液裂解物一次。
注意:在转移之前,请检查吸头以确保它们含有裂解。请记住在行或列之间更改提示。 - 用PCR粘合箔覆盖PCR板,并使用滚筒形成紧密密封。涂上箔片后,在离心机中短暂地旋转PCR板。将盘子放在冰上,直到它准备好在热循环器中运行。
- 使用适当的热循环仪程序运行PCR。有关 SSU 和 ITS2 PCR 程序的详细信息,请参阅 补充表 2。
- 重复步骤8.4-8.8,直到运行所有PCR。
- 在PCR反应进行时,倒入100 mL 1.5%琼脂糖凝胶。每个凝胶将容纳样品或单个PCR板。
- 将1.5克琼脂糖加入500 mL烧瓶中,然后加入100 mL 1x TAE缓冲液(补充表3)并旋转混合。微波溶解并冷却凝胶。
- 溶液冷却后,加入5μL10mg / mL溴化乙锭溶液并混合以混合。将溶液倒入带有四个25孔梳子的浇注盘中,以便凝胶可容纳96个样品以及凝胶中每排的分子量标准品。
注:溴化乙锭是一种强效诱变剂。处理溴化乙锭时,请使用实验室外套、耐化学腐蚀手套和化学安全护目镜。
- 在PCR完成之前,将6x上样染料加入一次性槽中,并使用多通道移液管向新的96孔PCR板的每个孔中加入2μL6x上样染料。该板将用于将样品上样到凝胶中。制作足够的这些板以容纳所有样品。
- PCR完成后,取出PCR板,并在室温下以300× g 短暂离心15秒。
- 要在凝胶上运行产物,请使用12孔多通道移液器将每个样品的5μL加入到含有2μL6x上样染料的96孔板的适当孔中。
- 然后将6μL该混合物上样到最近铸造的凝胶的每个孔中。将6μL的1 KB加梯形品加载到每排凝胶的第一孔中。
注意:为了填充凝胶的孔,可能需要将PCR板的A行和B行散布在凝胶的第一行中。为避免混淆,请将gel_number和gel_position记录在基因分型表中,用于每个PCR样品。
- 在板中剩余的PCR上放置一个新的铝箔盖,并将其储存在4°C。 这些反应产物将用于步骤9中的测序。
- 将PCR产物在凝胶上以120 V运行20分钟。对凝胶进行成像,并在基因分型表的"pcr_product_its2"和"prc_product_ssu"列中记录哪些S标签产生ITS2和/或SSU PCR产物。用"1"标记乐队的存在;标记"0"表示无频带。
9. 使用 Sanger 测序和序列 BLAST 鉴定线虫
注意:本节提供了从S标签对ITS2扩增子进行测序的说明,使用BLAST算法将这些序列与国家生物技术信息中心(NCBI)数据库对齐,并解析BLAST结果以识别S板上的线虫。
- 对于每个 ITS2 阳性的样品,使用剩余的 ITS2 PCR 产物,使用前向引物 oECA306 (CACTTTCAAGCAACCCGAC) 进行 Sanger 测序。通过在基因分型表中记录每个S标签的"sequencing_plate"和"sequencing_well"列,安排将测序输出文件轻松链接到S标签。
- 从排序平台获取每个 S 标签的 .seq 输出文件。将项目的 .seq 文件排列在单个目录中,并将子目录中每批序列的 .seq 文件排列在一个目录中。
- 打开命令行界面工具,然后通过输入命令: cd 导航到包含 .seq 文件的顶部目录。如果尚不存在,请通过输入以下命令为所有 .seq 文件创建一个合并的 FASTA: 对于 */中的 dir;执行 cd $dir;对于 *.seq 中的文件;执行回显">"$file; cat $file;>>.。/all_seqs.fa;cd ..;完成。
注意:此代码将从项目目录中的所有 .seq 文件创建一个名为"all_seqs.fa"的合并 FASTA 文件。该文件可用于NCBI在线核苷酸BLAST工具,以快速将每个S标记的ITS2序列与NCBI的序列数据库对齐。 - 在 Web 浏览器中,导航到 NBCI BLAST 网站20,然后单击" 选择文件" 按钮。选择刚刚创建的all_seqs.fa 文件,然后单击按钮 "有些相似的序列(BLASTn)"。单击页面底部的 "BLAST "按钮以开始 BLAST 搜索。
- 使用每个 S 标签的 BLAST 结果更新基因分型表。使用过滤工具使更新基因分型谷歌工作表更容易。单击" 数据>创建筛选器, 将筛选器按钮添加到每个列标题。过滤sequencing_plate列以选择要使用BLAST结果更新的测序板。
- 使用NCBI BLAST结果页面上的下拉菜单检查每个S-板ITS2序列的结果(图6)。
- 检查是否没有 BLAST 命中。下拉列表中以 * 为前缀的序列 ID 没有爆炸命中。对于这些 S 标签,请在基因分型表的manual_blast_notes列中输入"无命中<当前日期>"。
- 检查可能的新 Caenorhabditis 物种。单击顶部点击的链接以可视化对齐方式(图 6)。如果命中最多的是(1) Caenorhabditis 物种,(2)对齐在序列中心包含五个以上的不匹配,以及(3)查询覆盖率大于50%,则此结果表明分离物可能是新的 Caenorhabditis 物种(图7)。对于这些S板进入,顶部BLAST的物种在"species_id"列中命中,在"possible_new_caeno_sp列"中输入1,在"manual_blast_notes"列中输入"可能的新Caeno sp."以及百分比身份(例如,"可能的新Caeno sp.89%身份")。
- 对于 BLAST 到 Caenorhabditis 物种的 S 板序列,请在"species_id"列中输入顶部 BLAST 命中的完整属和物种名称。例如,"秀丽隐杆线虫"。
- 对于 BLAST 到非 Caenorhabditis 物种的 S 板序列,在"species_id"列中仅输入顶部 BLAST 命中的属,后跟"sp."。这种符号意味着分离物是命名属内的未知物种。例如, "Oscheius sp."。
注意:ITS2 序列不能用于可靠地鉴定分离株到 Caenorhabditis 属以外的物种水平3,13。 - 如果"species_id"="秀丽隐杆线虫"、"秀丽隐杆线虫炎"、"热带卡诺钩端菌炎"或"热带卡诺钩端炎",则在基因分型表的"make_strain_name列"中输入1,或"possible_new_caeno_sp"= 1。
- 按照 Caenorhabditis 命名惯例使用唯一名称命名菌株,即由2-3个大写字母组成的唯一实验室名称,后跟每个唯一菌株的数字23。在"strain_name"列中输入菌株名称。
- 在菌株命名后,可以使用已建立的方案 24对其进行冷冻保存。
10. 使用 R 中的 easyFulcrum 包处理收集数据
注意:此步骤介绍如何使用 easyFulcrum R 包将收集数据(C 标签)和线虫隔离数据(S 标签)链接在一起。该软件包含的功能将进一步将支点数据与基因分型表中的基因分型数据连接起来,以便将S标签物种身份和菌株名称组织在单个数据框中。
- 创建一个以集合项目命名的新目录。在目录中排列文件夹结构,以匹配 R 包 easyFulcrum15 中描述的要求。
- 导航到支点网站并登录。使用左侧 Fulcrum 网站的数据导出工具,并选中以下复选框,从 Fulcrum 数据库导出原始项目数据:项目、包括照片、包括 GPS 数据、现场采样和隔离。
注意:项目的支点数据将导出为五个逗号分隔值(.csv)文件。完整的项目数据将使用 R 中的 easyFulcrum 包组合成一个数据帧。 - 按照 easyFulcrum vignette21 中的说明,将从 Fulcrum 导出的五个.csv文件移动到步骤 10.1 中创建的项目目录中。
- 打开一个 Rstudio 会话,并在 R 控制台 'install.packages("devtools")' 和 'devtools::install_github("AndersenLab/easyfulcrum") 中输入以下命令,在 R 中安装 easyfulcrum 软件包。
- 打开一个新的 R 脚本,并按照 easyfulcrum 小插图中的说明处理集合数据21。
结果
该协议已被用于从多个地点(包括夏威夷和加利福尼亚州)收集 Caenorhabditis 线虫。 线虫的 分离成功率因采集地点、气候、采样经验和采样的底物类型而异。该协议已被用于对夏威夷群岛进行广泛采样,其中九个收集项目已经进行了多年和季节。自发 性Caenorhabditis 物种的分离成功率对于 C. briggsae (4,506个样本中的162个,3.6%)和 秀丽隐杆线虫 (4,506个样本中的163个,3.6%)几乎相同,而 C. c. tropicalis (4,506个样本中的26个,0.58%)的分离成功率要低得多8。相对于其他基质类别,每个自给自足物种都富含腐烂的果实和花基质。如果研究人员试图最大限度地提高成功率而不是表征底物偏好,则对腐烂的水果和花基质进行采样。然而,成功率随所选基板的质量而变化。例如,在水果和花卉基质中,那些太干燥,潮湿或新鲜的基质可能不会产生 Caenorhabditis 线虫。
这种收集协议的可扩展性从一对研究人员可以从野外收集的集合数量中显而易见。例如,在2018年10月,一对使用这种收集方案的研究人员能够在7天内从夏威夷两个岛屿的多个地点收集总共超过1,000个样本。这个现场团队在一夜之间将样本运送到实验室,在那里,一个由八名研究人员组成的团队从样本中分离出2000多种线虫。该协议的一个关键优点是,它通过减少现场所需的设备和人员,最大限度地降低了与远程位置采样相关的成本。使用该协议,小型现场团队可以专注于采样,而隔离团队可以使用易碎和重型设备(如解剖显微镜和琼脂平板)在其家庭机构处理样本,以分离线虫。此外,移动数据收集应用程序的实现允许与样品相关的所有现场数据直接链接到C标签,这使得隔离团队能够在处理样品时独立于现场团队工作。
使用此收集协议的研究人员必须考虑在收集项目之前分离线虫所需的努力。隔离和鉴定步骤具有速率限制,小型收集团队可以快速用样品淹没隔离器。此外,处理许多馆藏所需的实验室空间可能会干扰正在进行的研究(图3)。此外,一些分离的线虫需要额外的努力来进行基因分型。例如,在第一次裂解尝试后,大约2%的分离物无法用SSU PCR引物组扩增,必须重新裂解以确保裂解材料适合使用ITS2引物组扩增(图8)。此外,大约3%的分离株在第一轮Sanger测序后无法产生高质量的序列。对于这些分离株,通常需要另一轮裂解,ITS2 PCR和Sanger测序,这可能会增加分离团队的处理时间。重要的是,序列鉴定本身并不足以证明新的 Caenorhabditis 物种的合理性(图7)。为了正确地证明将分离株作为新的 Caenorhabditis 物种饲养是合理的,必须做出额外的努力来进行交配实验并建立类型标本13。打字标本的正式形态学描述也是首选,但不是必需的3。总之,这些考虑因素表明,采用这种收集方案的研究人员将受益于隔离和鉴定步骤的试验测试,以确保在收集项目开始之前正确分配资源。重要的是,即使是小型收集项目也可以从该协议中受益,因为该过程具有高度可重复性,并且可以轻松审核数据以用于实验室组的质量控制目的。

图1:基质示例 (A)理想的腐烂果实显示在图像(1)的中心,该果实几乎无法识别。附近显示腐烂的水果较少;避免对新鲜落下的水果进行取样(2)。(B)理想分解的花朵显示在顶部(3)。避免对新落的花朵进行采样(4)。(C)干燥叶片顶层下的深色落叶是自生 自生线虫采样 的理想选择(5)。避免取样干燥的落叶(6)。 请点击此处查看此图的放大版本。

图 2:线虫场采样移动应用程序。 (A) 在 Fulcrum 的 Apple 设备上打开 线虫场采样 应用程序后的初始屏幕。右下角的红色箭头指向用于创建新收藏记录的 + 按钮。(B) Apple 设备上显示的新收藏记录的示例。红色箭头指向收藏记录屏幕顶部的"项目"字段。在现场采样时,请务必选择正确的项目。项目字段将默认为创建后续收集记录时使用的最后一个项目。 请点击此处查看此图的放大版本。

图3:在电镀样品之前整理的收集袋和收集板。 此图显示了左侧 C 标记收集袋中的样品。每个收集袋的顶部都有一个匹配的C标签10厘米板。右边是10厘米的收集板,其中包含从收集袋中转移后的样品材料。 请点击此处查看此图的放大版本。

图4:具有正确转移样品的收集板(C板)。 将带有腐烂果实的10厘米C板放置在细菌草坪的边缘。C标签贴在板盖上。 请点击此处查看此图的放大版本。
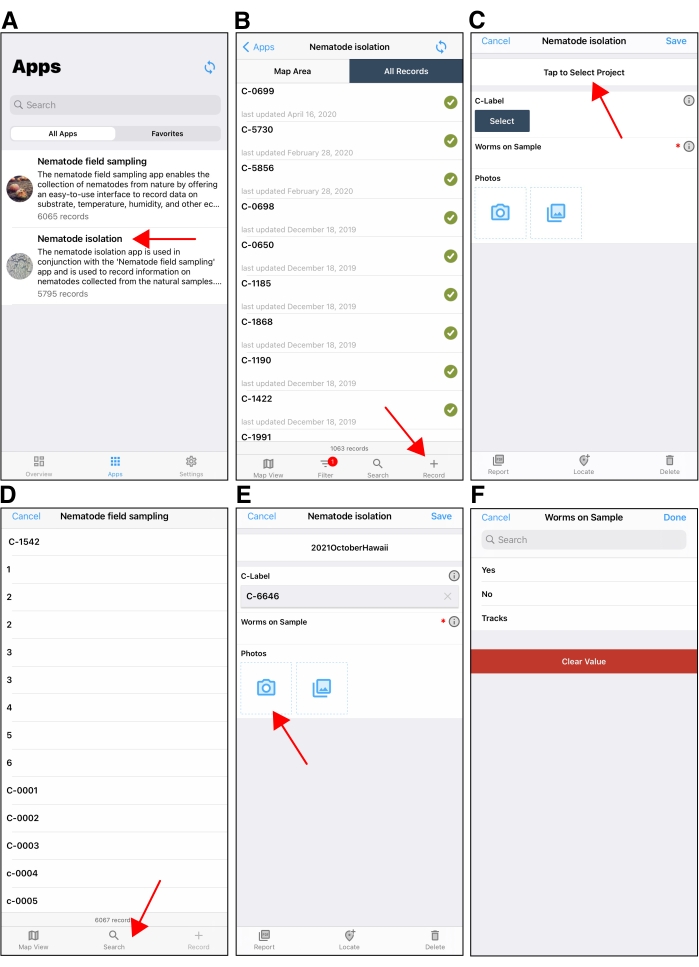
图 5:线虫隔离移动应用程序。 (A) 支点移动应用程序中的应用程序选择屏幕。红色箭头指向 线虫隔离 应用程序。(B) 在 Fulcrum 中的 Apple 设备上打开 线虫隔离 应用程序后的初始屏幕。右下角的红色箭头指向用于创建新隔离记录的 + 按钮。(C) Apple 设备上显示的新隔离记录的示例。红色箭头指向隔离记录屏幕顶部的"项目"字段。隔离时,请务必选择正确的项目。项目字段将默认为创建后续隔离记录时使用的最后一个项目。(D)点击C标签下的 "选择" 字段后,用户将点击搜索按钮(红色箭头)以找到他们从中分离线虫的C标签。(E)选择C标签后,用户将使用设备摄像头拍摄C板。(F)用户然后输入C板上是否有线虫。如果有要隔离的线虫,则将 S 标签添加到隔离记录中。 请点击此处查看此图的放大版本。
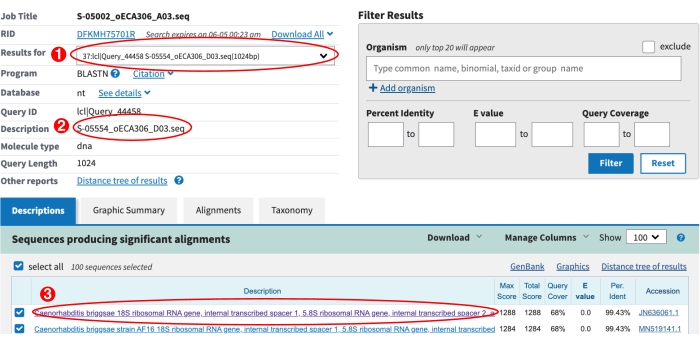
图 6:NCBI BLAST 结果页面。 (1) 用于查看所有序列的 BLAST 结果的下拉菜单。(2) 从下拉列表中选择的当前序列的描述。在本例中,将显示 S 标签 S-05554 的结果。(3) 显示 S-05554 的顶级 BLAST 命中。紫色文本表示已单击用于可视化此对齐方式的链接。请务必用眼睛检查对齐方式,以确定可能的新 Caenorhabditis 物种,请参阅上面的步骤9.8。 请点击此处查看此图的放大版本。

图 7:NCBI BLAST 对齐可视化示例。 (A) 分离株的 ITS2 查询序列与 C. kamaaina 受试者序列对齐的示例。(1) 对齐的百分比标识 (89%),对于顶部 BLAST 命中而言,这是低的。(2) 查询和主题序列(G 到 A)之间的不匹配。(3)通过比对算法在主体序列中形成的四个碱基对间隙;查询或主题中的间隙表示对齐不良。(4)对齐中心的广义区域,有许多不匹配和间隙。像这样的区域表明,查询序列可能来自一个新的 Caenorhabditis 物种。图中显示了2017年发现的新物种 C. oiwi的实际对齐示例。(B) 隔离的 ITS2 查询序列与主题序列之间良好对齐的示例。(5)比对的百分比(99%),这通常意味着查询序列来自与受试者相同物种的分离物。(6)与完美身份对齐的中心区域。像这样的区域表明,查询隔离物可能与受试者是同一物种。 请点击此处查看此图的放大版本。
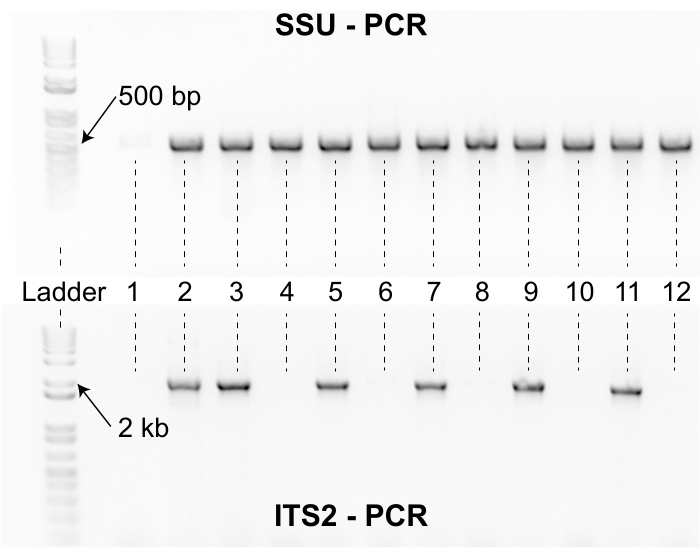
图 8:SSU 和 ITS2 PCR 产物。 顶部凝胶显示使用SSU引物组生成的PCR产物,用于12个代表性样品。DNA分子量标准包含在左侧作为参考。用于线虫尾状动脉 炎的 SSU PCR产物长度约为500 bp。样品2-12用SSU引物组扩增,但样品1没有。样品1缺乏500 bp SSU扩增子表明裂解材料质量差,必须重新裂解样品。底部凝胶显示使用ITS2引物组生成的PCR产物,用于顶部凝胶中所示的相同12个样品。两种凝胶的分子量标准品和样品处于相同的方向。12款样本中有6款未用ITS2引物组扩增。具有SSU和ITS2波段的样本采用Sanger测序,并使用NCBI BLAST算法通过序列相似性进行识别。 请点击此处查看此图的放大版本。
补充文件1:C标签。 包含 2500 个唯一 C 标签的 PDF 文件。 请点击此处下载此文件。
补充文件 2:S 标签。 包含 5000 个唯一 S 标签的 PDF 文件。 请点击此处下载此文件。
补充表1:现场材料。 现场用于对线虫进行采样的材料的包装清单。 请点击此处下载此表格。
补充表2:PCR配方和热循环器条件。 ITS2 和 SSU PCR 的 PCR 配方和热循环器条件 表。请单击此处下载此表。
补充表3:电泳缓冲液配方。 0.5 M pH 8.0乙二胺四乙酸溶液(EDTA)和TRIS-醋酸-EDTA(TAE)缓冲溶液的配方。 请点击此处下载此表格。
讨论
此协议包含必须谨慎执行的关键步骤。例如,在从现场收集样本或从实验室的样本中分离线虫之前,现场和隔离团队必须谨慎地在应用程序中选择正确的收集项目。如果选择了错误的收集项目,则最好使用在线记录编辑工具在支点数据库中更正错误的数据记录。对于许多放错位置的记录,此过程可能很繁琐。但是,数据库会保留对记录所做的任何更改,以便可以对收集和隔离记录进行完整的审核。该协议中的其他关键步骤涉及处理来自现场的样品以及从这些样品中分离出的线虫。为确保 线虫食 莲属在取样和装运步骤中存活,样品的温度应保持在4°C至25°C之间。 温度高于25°C会导致 秀丽隐杆线虫无菌14。确保尽可能在五天内将样品从收集袋转移到收集板,以尽量减少线虫的损失。在分离线虫后,在它们死亡之前对它们进行基因分型和冷冻保存至关重要。很难在超过两到三周龄的S板上找到活的线虫,因为真菌和细菌污染会使S板不适宜居住。
该协议可以轻松修改,以适应研究人员在现场可能想要收集的不同类型的数据。例如,使用Fulcrum的在线GUI进行应用程序编辑,使用新的数据输入字段可以轻松自定义"线虫场采样"应用程序。此外,数据分析包 easyFulcrum 可以在处理新数据时适应这些编辑15。用户可能发现吸引人的另一个修改是在现场使用不同的采样方法。研究人员可能希望对包含多种基板类型的较大区域进行采样,而不是对离散底物进行采样。这些较大的样品最好在实验室中使用 Baermann 漏斗或托盘提取方法进行处理13。重要的是,C标签和S标签的使用仍然适用于这些技术,因此与移动应用程序兼容。
该协议的主要局限性与在实验室中分离之前线虫的处理时间有关。首先,样本采集和线虫分离之间的滞后时间使得在采集时无法记录给定样本上线虫的发育阶段。其次,雄性和异族杂交的频率是自给自发 的Caenorhabditis 线虫的关键进化问题10。这种方法不太适合解决这些问题,因为线虫在隔离之前可能已经经历了多代。延迟隔离意味着不可能直接证明男性在自然界中的频率。此外,基因分型步骤中的多代延迟意味着在对线虫菌株进行测序之前,异型杂交(杂合性)的基因组证据将被侵蚀。为了鉴定自然界中的杂合性,使用从自然界直接分离的线虫产生的后代进行测序2。该协议的另一个潜在局限性是它偏向于识别自发性 Caenorhabditis。这是因为自生物种的孤立线虫比专性异型线虫具有更大的增殖机会,而专性异型动物只有在隔离受精的雌性时才会增殖。
此收集方法基于现有的收集协议13,14。该技术的主要进步是使用移动技术和定制软件来促进与大规模收集项目相关的大量生态和分子数据的组织。使用该收集协议生成的生态数据可用于解决 Caenorhabditis 物种自然种群的悬而未决的问题。例如,使用此方法生成的数据已用于发现夏威夷群岛上物种的生态位偏好。此外,通过对冷冻保存的线虫的基因组进行测序,研究人员可以研究遗传变异模式如何与生态数据相关联。此类研究可以揭示 Caenorhabditis 种群中局部适应的特征,并为自然环境中遗传变异的相关性提供重要见解8。为了获得对 线 虫内膜炎许多基因的功能性理解,可能需要进行生态学研究11。即使对于 秀丽隐杆线虫 来说,很大一部分基因也缺乏功能注释,尽管它是第一个被测序的多细胞动物,也是地球上研究最彻底的动物之一。该收集方案的开发旨在通过促进野生 Caenorhabditis 线虫的收集以及对其生态学和自然遗传多样性的研究来帮助解决这一知识差距。
披露声明
作者报告没有利益冲突。
致谢
这项研究得到了西北大学的启动资金和国家科学基金会职业奖(IOS-1751035)的支持,两者都授予E.C.A.
材料
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
参考文献
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。