Method Article
Un enfoque altamente escalable para realizar estudios ecológicos de los nematodos de Caenorhabditis Selfing
En este artículo
Resumen
Este protocolo se puede utilizar para realizar estudios ecológicos a gran escala de los nematodos Caenorhabditis . La principal ventaja de este método es la organización y el análisis eficientes de los datos ecológicos y moleculares asociados con los nematodos recogidos de la naturaleza.
Resumen
Caenorhabditis elegans es uno de los principales organismos modelo en biología, pero solo recientemente los investigadores se han centrado en su ecología natural. La relativa escasez de información sobre C. elegans en su contexto natural proviene de los desafíos involucrados en la identificación del pequeño nematodo en la naturaleza. A pesar de estos desafíos, un enfoque cada vez mayor en la ecología de C. elegans ha causado una gran cantidad de nueva información sobre su vida fuera del laboratorio. La búsqueda intensificada de C. elegans en la naturaleza ha contribuido al descubrimiento de muchas nuevas especies de Caenorhabditis y reveló que los nematodos congenéricos cohabitan con frecuencia en la naturaleza, donde se alimentan de floraciones microbianas asociadas con material vegetal en descomposición. La identificación de nuevas especies también ha revelado que el sistema de apareamiento androdioico de machos y hermafroditas autofertilizantes ha evolucionado tres veces de forma independiente dentro de Caenorhabditis. Las otras dos especies de selfing, C. briggsae y C. tropicalis, comparten las ventajas experimentales de C. elegans y han permitido estudios comparativos sobre la base mecanicista de rasgos importantes, incluida la autofertilización. A pesar de estos avances, queda mucho por aprender sobre la ecología y la diversidad natural de estas importantes especies. Por ejemplo, todavía carecemos de información funcional para muchos de sus genes, que solo podría lograrse a través de una comprensión de su ecología natural. Para facilitar la investigación ecológica de los nematodos Caenorhabditis , desarrollamos un método altamente escalable para recolectar nematodos de la naturaleza. Nuestro método hace uso de plataformas móviles de recopilación de datos, bases de datos basadas en la nube y el entorno de software R para mejorar la capacidad de los investigadores para recolectar nematodos de la naturaleza, registrar datos ecológicos asociados e identificar nematodos silvestres utilizando códigos de barras moleculares.
Introducción
Las últimas dos décadas han traído un mayor interés en la ecología de los nematodos Caenorhabditis. A partir de estos estudios, sabemos que las especies de Caenorhabditis de vida libre pueden aislarse de microhábitats efímeros tanto en regiones templadas como tropicales, donde se alimentan de floraciones microbianas asociadas con material vegetal en descomposición, a veces en simpatía1,2,3,4,5,6,7,8 . También hemos aprendido que la evolución convergente de la autofertilización ha ocurrido en el género tres veces, y el auto-envenenamiento es el modo dominante de reproducción para C. briggsae, C. elegans y C. tropicalis9,10. Entre estos selfers, C. elegans es uno de los animales más ampliamente estudiados en la Tierra y ha sido utilizado por los investigadores para hacer avances críticos en biología. Es importante destacar que las otras especies de Caenorhabditis comparten muchas de las ventajas experimentales de C. elegans y están avanzando rápidamente en los estudios comparativos en el género. Sin embargo, la naturaleza críptica de estos nematodos en la naturaleza dificulta el estudio de su ecología y diversidad natural, lo cual es fundamental para comprender las funciones biológicas de sus genes y las formas en que la evolución ha dado forma a la diversidad genética entre las especies10,11.
El mayor desafío para estudiar la ecología de los nematodos Caenorhabditis en la naturaleza es su pequeño tamaño; los nematodos adultos son a menudo de 1 mm de longitud o menos. Este desafío requiere que los investigadores tomen muestras de sustratos de la naturaleza e intenten separar los nematodos de interés de los sustratos en el laboratorio sin la capacidad de observar animales en la naturaleza. Debido a que incluso a los expertos capacitados les resulta difícil discriminar a los nematodos caenorhabditis de otros nematodos de vida libre bajo el microscopio, los nematodos generalmente se eliminan del sustrato, se aíslan y se dejan proliferar antes de que se identifiquen por identidad de secuencia utilizando códigos de barras moleculares establecidos3,12,13,14 . El tiempo y el esfuerzo necesarios para procesar cada nematodo de esta manera presentan un desafío organizativo, ya que los investigadores deben poder rastrear la identidad de cada nematodo aislado en el laboratorio hasta el sustrato exacto y los datos ecológicos asociados muestreados en el campo. Aquí, describimos un proceso paso a paso para recopilar e identificar eficientemente los nematodos Caenorhabditis del campo y vincular fielmente estos aislamientos con sus datos espaciales y ecológicos asociados a gran escala.
Este método de recolección aumenta la escala y la precisión de los estudios ecológicos mediante el uso de plataformas móviles de recopilación de datos, bases de datos basadas en la nube y el entorno de software R. Fulcrum es una plataforma de recopilación de datos personalizable que funciona con la mayoría de los dispositivos móviles y permite a los usuarios crear aplicaciones personalizadas para recopilar y organizar datos basados en la ubicación (https://www.fulcrumapp.com). Este protocolo proporciona instrucciones detalladas sobre cómo utilizar aplicaciones personalizadas de recopilación de datos para organizar datos ecológicos espacialmente explícitos del campo y vincular con precisión esos datos con la identidad de los nematodos aislados en el laboratorio. El protocolo también explica cómo identificar eficientemente los nematodos Caenorhabditis que se autogestionan utilizando códigos de barras moleculares establecidos. Los datos de estos métodos se pueden procesar de manera simple y reproducible con el paquete de software R que lo acompaña easyFulcrum15 para explorar la ecología y la diversidad genética de las poblaciones naturales de Caenorhabditis.
Protocolo
1. Preparación de la colección
- Identifique un lugar para inspeccionar los nematodos Caenorhabditis .
NOTA: En la mayoría de las regiones templadas, C. elegans y C. briggsae se pueden aislar fácilmente de hábitats asociados con el hombre, como campos agrícolas o jardines rurales y urbanos1. En las regiones subtropicales y tropicales, C. briggsae, C. elegans y C. tropicalis se pueden encontrar en los hábitats asociados con el hombre enumerados anteriormente, a veces muy cerca uno del otro. Sin embargo, C. elegans parece preferir hábitats más fríos y secos que las otras especies en hábitats tropicales7,8. Cada una de las especies también se puede aislar de hábitats silvestres que no están asociados con los humanos, pero estos hábitats se muestrean con menos frecuencia. - Cree un proyecto Fulcrum para organizar la recopilación y el aislamiento de datos con las aplicaciones móviles de recopilación de datos.
- Cree una cuenta con Fulcrum en línea utilizando un acuerdo educativo sin costo16. Agregue la aplicación Nematode Field Sampling a una cuenta Fulcrum haciendo clic en el botón ADD APP17.
- Agregue la aplicación Nematode Isolation a una cuenta haciendo clic en el botón ADD APP18.
NOTA: Se recomienda que cada viaje a un lugar se organice como su proyecto de recolección utilizando la convención de nomenclatura 'YearMonthLocation', por ejemplo, 2020FebruaryAustralia.
- Agregue usuarios a la cuenta de Fulcrum para concederles acceso al proyecto de colección. Asegúrese de que cada usuario descargue la aplicación móvil Fulcrum para participar en el proyecto.
- Imprima un conjunto de etiquetas con código QR para realizar un seguimiento de las colecciones (etiquetas C) y los aislamientos de nematodos (etiquetas S) con la aplicación móvil. Coloque las etiquetas C para bloquear con cremallera las bolsas de plástico, enrolle las bolsas etiquetadas en grupos de 25 y envuélvalas con una banda elástica para empacar. Mantenga el conjunto de etiquetas S para su uso en el laboratorio.
NOTA: A lo largo de este protocolo, las colecciones (sustratos del campo) están contenidas en bolsas o en placas y están etiquetadas con etiquetas C. Los nematodos aislados están etiquetados con etiquetas S. Las etiquetas C se utilizan para identificar colecciones únicas, y las etiquetas S se utilizan para identificar aislados de nematodos únicos. Estos dos tipos de etiquetas se utilizan para establecer la conexión entre una colección en particular (etiqueta C) y los nematodos aislados de esa colección (etiquetas S) en la base de datos Fulcrum. Imprima el doble del número de etiquetas S que de etiquetas C para un proyecto de colección porque, en promedio, se aíslan dos nematodos por colección. Más tarde se pueden imprimir más etiquetas S si es necesario. En el suplemento se proporcionan 2,500 etiquetas C únicas (Archivo Suplementario 1) y 5,000 etiquetas S únicas (Archivo Suplementario 2). - Preparar placas NGMA de 10 cm para colecciones y placas NGMA de 3,5 cm para aislar nematodos. Haga una placa de 10 cm y al menos dos placas de 3,5 cm por colección21. Estas placas se siembran con Escherichia coli cepa OP50 siguiendo los protocolos establecidos. Guarde las placas antes de usarlas a 4 °C durante no más de 1 mes.
2. Colección de campo
NOTA: Los nematodos caenorhabditis se aíslan con mayor frecuencia del material vegetal en descomposición, incluidas frutas, nueces, semillas, vainas, flores, tallos, basura vegetal y compost1,5,6,8. Los mejores sustratos son podridos y casi irreconocibles como frutos o flores; evitar sustratos demasiado secos o húmedos (Figura 1). Los sustratos se recogen de manera más eficiente del campo trabajando en pares. El individuo con el termómetro infrarrojo sin contacto seleccionará un sustrato para la recolección y recogerá la muestra, mientras que su compañero utiliza la aplicación Nematode Field Sampling en Fulcrum para registrar los datos de recolección. El par de colectores repetirá este proceso hasta que se recoja el número deseado de muestras. La lista de materiales requeridos para el trabajo de campo se encuentra en (Tabla suplementaria 1).
- Abra la aplicación móvil Fulcrum, seleccione Muestreo de campo de nematodos en el menú desplegable. Presione + para iniciar un nuevo registro en el proyecto (Figura 2A). Tome una foto del sustrato.
- Haga clic en el cuadro en la parte superior central para seleccionar el proyecto de colección correcto realizado en el paso 1.2 (Figura 2B). Toque el campo C-label en la parte inferior del registro de colección y elija Escanear cuando aparezca el mensaje. Escanee el código de barras en la bolsa de recolección con la cámara del dispositivo móvil, luego toque Listo en la parte superior derecha de la pantalla.
- Toque el campo Sustrato y seleccione un tipo de sustrato en el menú desplegable. Agregue notas sobre el sustrato tocando el campo Notas de sustrato e ingresando notas manualmente.
- Elija un paisaje en el menú desplegable. Elija el paisaje que mejor represente el sitio de muestreo.
- Elija una vista del cielo. Al elegir la vista del cielo, describa la visibilidad del cielo en el sitio de muestreo (por ejemplo, una vista del cielo completo sin vistas obstruidas desde los árboles u otras estructuras = lleno).
- Mida la temperatura superficial del sustrato utilizando el termómetro sin contacto y registre el valor en el campo de temperatura del sustrato.
NOTA: Sostenga el termómetro sin contacto a no más de 14 pulgadas del sustrato mientras registra la temperatura. - Mida la temperatura ambiente y la humedad con el dispositivo de mano y registre estos datos en los campos adecuados.
NOTA: Compruebe que el dispositivo de temperatura ambiente y humedad no esté en espera. La unidad de medida cambiará cuando se suelte el botón. Mantenga el dispositivo en un bolsillo exterior para evitar lecturas irregulares. - Guarde el registro en Fulcrum tocando Guardar en la parte superior izquierda de la pantalla.
- Recoge alrededor de una cucharada del sustrato sin palos u otras piezas duras invirtiendo la bolsa de recolección para usarla como un "guante" para recoger el sustrato, luego sella la bolsa. Coloque una toalla de papel en la bolsa si la muestra está particularmente húmeda.
NOTA: En climas cálidos, coloque las bolsas en refrigeradores suaves con paquetes más fríos para mantener las colecciones frescas. - Después de que se hayan recolectado todas las muestras del día, limpie el equipo de recolección, saque las baterías de las sondas, recargue las baterías, vuelva a congelar los paquetes de congelación. Sincronice los datos de recopilación de Fulcrum tocando el botón Sincronizar en la parte superior izquierda de la aplicación Nematode Field Sampling .
NOTA: Las cargas pueden tardar varios minutos sin una conexión celular fuerte, por lo que podría ser mejor esperar el acceso WiFi. Los datos permanecerán en dispositivos móviles y se sincronizarán con la nube. - Envíe las muestras a una institución de origen colocándolas en una caja de envío durante la noche. Minimice el tiempo que las muestras están expuestas a temperaturas inferiores a 11 ° C o superiores a 25 ° C mediante el envío de paquetes en los días en que se transporta la carga.
NOTA: La mayoría de las instalaciones de envío no envían carga durante la noche los fines de semana en lugares remotos.
3. Emplatado de colecciones de campo en el laboratorio
NOTA: Esta sección detalla cómo organizar la transferencia de muestras de las bolsas de recolección etiquetadas a las placas etiquetadas. Las muestras pueden llegar de un envío nocturno o directamente desde el campo.
- Reciba el envío de colecciones e inspeccione en busca de bolsas rotas u otra evidencia de daños. Si las bolsas están rotas, deseche el material y limpie las bolsas de recolección intactas con etanol al 70%; evite la etiqueta C en la bolsa con el etanol, ya que decolorará la etiqueta y dificultará su lectura.
- Para cada bolsa con cierre hermético, anote la etiqueta C en la bolsa y coloque una etiqueta C a juego en la tapa de una placa de 10 cm manchada con bacterias OP50.
NOTA: Las placas de 10 cm etiquetadas se denominan "placas C" para el resto del protocolo. La forma más fácil de organizar las muestras es colocar las bolsas de recolección en un banco de laboratorio con la placa C correspondiente en la parte superior (Figura 3). - Para cada recolección, transfiera aproximadamente una cucharada de muestra de la bolsa de recolección a la placa C usando una cuchara de plástico limpia. Agregue la muestra alrededor del césped bacteriano en forma de media luna o anillo, no cubra completamente el césped bacteriano (Figura 4).
NOTA: Mantenga la cucharada limpia colocándola en un vaso de precipitados de etanol al 95% cuando no esté en uso. Use una toalla de papel para secar la cucharada antes de transferir muestras adicionales. - Registre el tiempo en que las colecciones se transfirieron de las bolsas de recolección a las placas C y mantenga las placas C a temperatura ambiente (RT) durante al menos 24 h antes de intentar aislar los nematodos en la sección 4.
4. Aislar nematodos de las colecciones
- Abra la aplicación Fulcrum en el dispositivo móvil y elija Aislamiento de nematodos en el menú de la aplicación (Figura 5A). Realice un nuevo registro de aislamiento tocando el icono + en la parte inferior derecha (Figura 5B).
- En la nueva pantalla de registro de aislamiento, confirme el proyecto de recopilación correcto marcando el nombre del proyecto que se muestra en el cuadro en la parte superior central. Si se muestra el proyecto incorrecto, pulse el nombre del proyecto para cambiar al proyecto correcto (Figura 5C).
- Toque el botón Seleccionar debajo del campo etiqueta C para encontrar la etiqueta C asociada con la muestra de la que se están aislando los nematodos (Figura 5D). Toque el ícono buscar , luego toque el ícono Escanear para escanear el código QR de la etiqueta C en la placa C con la cámara del dispositivo. Una vez que se escanea el código QR, aparecerá un registro de etiqueta C en el campo etiqueta C .
- Toque el icono Cámara en el campo Fotos para abrir la cámara del dispositivo y úsela para tomar una foto de la muestra en la placa C con el código QR visible (Figura 5E). Toque en Listo para volver a la pantalla Aislamiento.
NOTA: Estas fotos de registro de aislamiento se pueden utilizar para explorar atributos específicos del sustrato en un momento posterior. - Use un microscopio de disección para buscar nematodos en la placa C. Toque el campo Gusanos en la muestra para registrar la presencia de nematodos en la muestra (Figura 5F). Toque Sí si hay nematodos presentes en la placa C y toque No si no hay nematodos presentes.
- Toque en Pistas si solo hay pistas de nematodos presentes. Si no hay nematodos presentes, parafilme la placa C y deséchela en un contenedor de riesgo biológico.
NOTA: Invierta la placa C sobre el contenedor de residuos de riesgo biológico y golpee suavemente la parte posterior de la placa para desalojar todos los sustratos muestreados. Este paso hace que sea más fácil encontrar y aislar nematodos que pueden estar debajo del sustrato en la placa C. - Si hay nematodos presentes, aísle hasta cinco nematodos de la placa C. Para aislar un nematodo, transfiera un nematodo de la placa C a una placa S usando una púa de alambre de platino. Aísle a los adultos sanos y grávidos si es posible. Sin embargo, aísle otras etapas si no se encuentran adultos.
NOTA: Después del aislamiento, hasta cinco placas S tendrán cada una un solo nematodo en ellas. Mantenga estas placas S con nematodos aislados de la misma placa C organizados juntos en una pila ordenada lejos de otras placas S hasta que se introduzcan en Fulcrum. - Toque el campo Placas con etiqueta S para ingresar las placas S utilizadas para este aislamiento. Toca el + en la parte inferior derecha. Toque la etiqueta S y luego haga clic en Escanear para abrir la cámara del dispositivo. Utilice la cámara del dispositivo para escanear el código QR de la etiqueta S en la placa S.
NOTA: Asegúrese de que el código de la etiqueta S coincida con el código de la placa. Si coincide, toca Listo. Si no es así, toque Cancelar y vuelva a escanear hasta que coincida, luego haga clic en Listo. A veces, los códigos QR de las placas cercanas se escanean accidentalmente. - Después de ingresar cada placa S, guarde la entrada con el botón Guardar en la parte superior derecha. La entrada se perderá si no se guarda. Toque el + en la parte inferior derecha para agregar más placas con etiqueta S si es necesario hasta que se ingresen todos los nematodos aislados de la placa C. Después de agregar todas las placas con etiqueta S para el registro de aislamiento, toque el botón < en la parte superior izquierda para volver a la pantalla de registro de aislamiento.
NOTA: Para cancelar un registro de aislamiento porque los errores no se pueden resolver, haga clic en Cancelar en la parte superior izquierda. Este paso abrirá un cuadro de diálogo que preguntará si el registro se puede descartar sin guardar. Si lo desea, haga clic en Sí, Descartar. - Toque el botón Guardar en la parte superior derecha una vez que el registro de aislamiento tenga toda la información agregada correctamente. Luego parafilme las placas S con nematodos aislados y déjelos a un lado en un área designada para sostener placas S con nematodos.
- Parafilme la placa C y deséchela en el contenedor de riesgos biológicos. Toque el icono Sincronizar para cargar todos los datos en Fulcrum.
- Ordene todas las placas S en orden alfanumérico, luego coloque las placas S en cajas de cartón. Asegúrese de que las placas S estén del lado de la tapa hacia abajo y parafilmadas. Apile hasta cuatro placas S en una posición en la caja y etiquete la caja de cartón con el nombre del proyecto, la fecha, la hora y un número de caja único.
- Guarde las cajas etiquetadas en RT. Estos aislamientos se verificarán para detectar la proliferación a las 48 h y nuevamente a las 168 h si es necesario.
5. Exportación de placas S desde Fulcrum
Nota : en esta sección se detalla cómo exportar etiquetas S utilizadas en el proceso de aislamiento desde la base de datos del proyecto Fulcrum. Estas etiquetas S se utilizarán para rastrear la proliferación de líneas isofemale mientras se identifican por identidad de secuencia en las secciones 6-9.
- Inicie sesión en el sitio web de Fulcrum y seleccione la aplicación Nematode Isolation . Haga clic en Exportador en el lado izquierdo de la pantalla. Haga clic para seleccionar el proyecto deseado y marque la casilla Aislamiento de nematodos . Haga clic en Siguiente para descargar un archivo de .zip que contenga el archivo 'nematode_isolation_s_labeled_plates.csv'.
- Abra el archivo 'nematode_isolation_s_labeled_plates.csv' y ordénelo por la columna 'S-label' en orden ascendente (la etiqueta S más pequeña estará en la parte superior). Seleccione todas las etiquetas S y cópielas de la hoja de cálculo.
- Navegue a la plantilla de genotipado aislado salvaje hoja de google (wild_isolate_genotyping_template) utilizando un navegador web19.
- Haga una copia de esta hoja de Google haciendo clic con el botón derecho en la pestaña Plantilla de genotipado y luego seleccionando la opción Copiar a nueva hoja de cálculo . Selecciona Abrir hoja de cálculo para ver la nueva hoja de cálculo de Google.
- Asigne a esta nueva hoja el nombre del proyecto fulcro seguido de 'wild_isolate_genotyping', por ejemplo, '2020FebruaryAustralia_wild_isolate_genotyping'.
NOTA: Esta hoja se conoce como la "hoja de genotipado" en el resto del protocolo.
- Pegue las etiquetas S copiadas de la columna 'nematode_isolation_s_labeled_plates.csv' 's_label' en la columna de la hoja de genotipado titulada 's_label'. Marque la columna 's_label_repeat_error' para '1's. Un valor de '1' en esta columna significa que la etiqueta S está duplicada en algún lugar de la hoja de genotipado. Si se descubren duplicaciones, investigue y corríjalas antes de seguir adelante.
- Rellene la columna 'isolation_box_number' de la hoja de genotipado para todas las etiquetas S.
6. Compruebe si hay proliferación en las placas S
- Compruebe si hay animales que proliferen en las placas S 48 h después del aislamiento (utilice la fecha y hora del último aislamiento en la casilla del paso 4.11 para guiar el tiempo).
NOTA: Los nematodos en proliferación se caracterizan por descendencia en la placa S. - Si una placa S está proliferando, ingrese '1' en la columna proliferation_48 en la hoja de genotipado, luego mueva la placa S a una caja etiquetada 'Proliferación de 48 h, caja 1'. Coloque un máximo de 88 placas S en una caja de proliferación, luego comience a llenar una nueva caja etiquetada como 'Proliferación de 48 h, caja 2'. Asegúrese de que las etiquetas S estén organizadas en orden alfanumérico en las cajas de proliferación de 48 h.
NOTA: No deseche las placas S que no proliferan; estas placas se revisarán de nuevo a las 168 h después del aislamiento. Si lo desea, consolide estas placas S en orden numérico en casillas etiquetadas como '48 h sin proliferación, casilla X', pero recuerde registrar cuándo debe producirse la comprobación de 168 h en la nueva casilla. - Después de identificar todas las etiquetas S en proliferación a las 48 h, pase a la sección 7 para las placas S con proliferación a las 48 h.
- Compruebe las placas S que no proliferaban a las 48 h después del aislamiento de nuevo a las 168 h después del aislamiento.
- Si una placa S está proliferando ahora, ingrese '1' en la columna proliferation_168 en la hoja de genotipado y luego mueva la placa S a una caja etiquetada como 'Proliferación de 168 h, caja 1'.
- Coloque un máximo de 88 placas S en una caja de proliferación, luego comience a llenar una nueva caja etiquetada como 'Proliferación de 168 h, caja 2'. Asegúrese de organizar las etiquetas S en orden alfanumérico en las cajas de proliferación de 168 h.
- Deseche las placas S que no tengan proliferación después de 168 h. Pase a la sección 7 para placas S con proliferación a 168 h.
7. Lisis de líneas isofemales
NOTA: Este paso utilizará la herramienta de filtro de datos en las hojas de cálculo de Google para ayudar a imprimir hojas de cálculo de lisis para las placas S en los cuadros de proliferación. El propósito de las hojas de trabajo de lisis es proporcionar al personal las posiciones correctas para las etiquetas S en los tubos de tira de lisis en el banco.
- Abra la hoja de genotipado para el proyecto deseado y seleccione todas las celdas escribiendo Cmd+A. Haga clic en Datos > Crear un filtro para agregar un botón de filtro a cada encabezado de columna. Utilice los botones Filtro para mostrar sólo las placas S que se genotiparán. Por ejemplo, si se van a lisar todas las placas S con proliferación a las 48 h: Haga clic en el botón Filtro en la columna 'proliferation_48' y seleccione '1'.
- Una vez que se haya filtrado la hoja de google de genotipado, revise la lista de etiquetas S que se muestran para asegurarse de que son las etiquetas S que se imprimirán en la hoja de cálculo.
- En la columna 'strip_tube_number' de la hoja de google de genotipado, introduce un número único cada 11 filas.
- Introduzca los números de tubo de tira para un proyecto en orden sucesivo a partir de 1 y nunca duplicados. En el 'strip_tube_position', ingrese del 2 al 12 para cada número de tubo de tira.
NOTA: Use tubos de tira de 12 tubos para la lisis. La primera posición (strip_tube_position 1) será control, pero los controles no se agregan a las hojas de cálculo de lisis (solo se agregan los strip_tube_positions, 2-12). En el momento de la lisis, la cepa de control positivo 'N2' se agregará a la posición 1 de cada tubo de tira par numerado como control positivo. No se agregan gusanos a la posición 1 de cada tubo de tira impar como control negativo.
- Filtre la hoja de Google de genotipado para incluir solo las etiquetas S en un cuadro de proliferación que se van a lisar, luego seleccione las columnas 's_label' a través de 'lysis_notes'. Imprima una hoja de cálculo de lisis para cada caja de proliferación que se va a lisar.
- Haga clic en el menú desplegable del campo Imprimir y seleccione Celdas seleccionadas. Haga clic en Siguiente en la parte superior derecha, luego use el diálogo para imprimir la hoja de cálculo de lisis para el cuadro de proliferación.
- Repita los pasos 7.3 a 7.5 para imprimir una hoja de cálculo de lisis para cada caja de proliferación.
NOTA: Cada caja de proliferación contiene hasta 88 placas S, lo que corresponde con ocho tubos de tira de 12 pocillos. - Prepare tubos de tira de 12 pocillos para todas las muestras que se lisarán. Etiquete un tubo de tira con un "strip_tube_number" único asignado en la hoja de trabajo de lisis. Esta etiqueta debe estar escrita en la tira de la tapa y en el tubo de la tira para evitar confusiones si están separadas. Los tubos de tira EVEN tienen un control positivo (gusanos N2) en la posición 1. Los tubos de tira ODD tienen un control negativo (sin gusanos) en la posición 1.
- Prepare suficiente tampón de lisis (100 mM KCl, 20 mM Tris pH 8.2, 5 mM MgCl2, 0.9% IGEPAL, 0.9% Tween 20, 0.02% gelatina con proteinasa K agregada a una concentración final de 0.4 mg / ml) para todas las muestras y agregue un 5% adicional para el error de pipeta. Escala según sea necesario.
NOTA: El tampón de lisis se prepara mejor combinando todos los ingredientes excepto la proteinasa K y la congelación en alícuotas de 10-50 ml a -20 °C. Descongelar las alícuotas y conservar a 4 °C antes de su uso; inmediatamente antes de usar, agregue proteinasa K y mezcle bien. Mantenga el tampón de lisis en hielo mientras trabaja. - Organice las placas S para un tubo de tira en particular en orden utilizando la hoja de trabajo de lisis impresa como guía.
- Desencapar un tubo de tira y añadir 8 μL de tampón de lisis a cada tapón con un pipettor de repetición. Agregue el tampón de lisis a una tira de tapas a la vez porque el tampón de lisis se evaporará si se deja en RT y se descubre. Elija de 3 a 5 animales de las placas de origen (placa S o placa de stock N2 para controles positivos) en las posiciones de tapa apropiadas indicadas en la hoja de trabajo de lisis. Registre notas para cualquier placa S con menos de 5 gusanos seleccionados para la lisis en la sección lysis_notes de la hoja de trabajo de lisis.
- Después de cargar los nematodos en cada posición del tubo de la tira, coloque la tira de la tapa de nuevo en el tubo de la tira. Haga coincidir la tapa marcada (posición 1) con el tubo marcado (posición 1). Una vez tapado, centrífuga el tubo de la tira brevemente hasta que los nematodos estén en la parte inferior del tubo.
- Coloque la tira en el congelador de -80 °C hasta que esté completamente congelada (al menos 10 min). Repita los pasos 7.9 a 7.11 hasta que todas las tiras tengan nematodos agregados para la lisis. Organice las tiras de tubo en orden numérico.
- Retire los juegos de tubos de tira y ejecute el programa de lisis en un termociclador: 1 h a 60 ° C, 15 min a 95 ° C, mantenga a 12 ° C. Cuando termine el programa de lisis, gire las muestras a 300 x g durante 15 s en RT y guarde los lisados a -80 ° C durante un máximo de 1 semana.
- Organice las tiras de tubo en orden numérico utilizando soportes de placa de 96 pocillos e incluya una etiqueta con un número de caja de proliferación, un rango de número de tubo de tira, una fecha y las iniciales del investigador. Actualice las columnas de la hoja de genotipado 'lysis_date' y 'lysis_notes' con información de la hoja de cálculo de lisis.
8. PCR de secuencias SSU e ITS2
NOTA: Esta sección proporcionará instrucciones sobre cómo realizar dos PCR separadas para cada placa S lisada. El primer conjunto de cebadores amplifica un fragmento de 500 pb del gen de subunidad pequeña (SSU) 18S rDNA; oECA1271 = imprimación hacia adelante TACAATGGAAGGCAGCAGGCGC, oECA1272 = imprimación inversa CCTCTGACTTTCGTTCTTGATTAA 12. Esta PCR se utiliza para comprobar la calidad del ADN de la plantilla. La PCR amplifica la región SSU para casi todas las especies de nematodos. Si la PCR SSU no se amplifica, este resultado sugiere que la calidad de la lisis es pobre y la lisis debe repetirse para esta placa S. El segundo conjunto de cebadores amplifica un fragmento de 2.000 pb de la región espaciadora transcrita interna entre los genes 5.8S y 28S rDNA (ITS2); oECA1687 = imprimación hacia adelante CTGCGTTACTTACCACGAATTGCARAC, oECA202 = imprimación inversa GCGGTATTTGCTACTACCAYYAMGATCTGC3. El producto ITS2 PCR está secuenciado por Sanger y la secuencia se utiliza para identificar nematodos en el género Caenorhabditis a nivel de especie por similitud de secuencia.
- Utilice la herramienta de filtrado de la hoja de genotipado para ver solo las etiquetas S que se utilizarán para la PCR.
- Actualice las columnas pcr_plate_number y pcr_well en la hoja de genotipado. Para evitar la degradación del material de lisis, las PCR SSU e ITS2 se ejecutan al mismo tiempo.
- Utilice la misma pcr_plate_number para las PCR ITS2 y SSU, aunque se trate de reacciones separadas en placas separadas. Se distinguirán con las etiquetas 'SSU' o 'ITS2'.
- Asigne un pcr_plate_number a ocho o menos tubos de tira (un tubo de tira por fila de la placa de PCR de 96 pocillos, dispuesto en orden ascendente, por ejemplo, el número de tubo de tira más bajo en la parte superior). A continuación, asigne un pcr_plate_well a cada etiqueta S en los tubos de tira.
NOTA: Los tubos de tira están dispuestos en orden ascendente, con el número de tubo de tira más bajo asignado a la fila A y el número más alto en la fila H. La posición 1 de todos los tubos de tira se asigna a la columna 1. Por lo tanto, el tubo de tira número 1, posición 1 se asignará a la placa de PCR número 1, pozo A01. - Etiquete las placas de PCR de 96 pocillos para acomodar las muestras que se utilizarán para la PCR. Etiquete cada placa de PCR con la siguiente información: nombre del proyecto, tipo de PCR, número de placa de PCR y fecha de PCR (por ejemplo, 2020FebruaryAustralia_SSU_1_20200304). Además, etiquete la placa con los números de tubo de tira que se cargarán en cada fila.
- Retire el material de lisis del congelador de -80 °C y descongele los tubos de tira que contienen el material de lisis en hielo. Mientras el material de lisis se descongela, prepare mezclas maestras ITS2 y SSU en tubos separados sobre hielo. Las recetas de SSU e ITS2 PCR se encuentran en la Tabla Suplementaria 2.
NOTA: Prepare 100 reacciones de mezcla maestra de PCR para cada placa de 96 pocillos para permitir un error de pipeteo. Use un cónico de 15 ml o 50 ml para mantener la mezcla maestra si se van a utilizar grandes volúmenes. - Vórtice la mezcla maestra suavemente hasta que Taq se distribuya por toda la mezcla. Una vez mezclado, alícuota 38 μL de la mezcla maestra a los pocillos apropiados de las placas PCR sobre hielo. Use canales estériles de fondo en V de un solo uso y una pipeta multicanal de 12 pocillos para transferir la mezcla maestra a las placas de PCR.
- Gire hacia abajo los tubos de la tira de lisis descongelada para eliminar el material de lisis de las tapas. Retire con cuidado las tapas de todos los tubos de tira que se cargarán en la primera placa de PCR. Use una pipeta multicanal de bajo volumen (ya sea de 12 pocillos o de 8 pocillos) para agregar 2 μL de lisado al pozo apropiado en la placa de PCR. Pipetee suavemente el lisado hacia arriba y hacia abajo una vez antes de retirar los 2 μL.
NOTA: Compruebe las puntas para asegurarse de que contienen la lisis antes de la transferencia. Recuerde cambiar las puntas entre filas o columnas. - Cubra la placa de PCR con una lámina adhesiva de PCR y use un rodillo para crear un sello hermético. Después de aplicar la lámina, gire brevemente las placas de PCR en una centrífuga. Mantenga la placa sobre hielo hasta que esté lista para funcionar en el termociclador.
- Ejecute las PCR con el programa de termociclador apropiado. Consulte la Tabla suplementaria 2 para obtener los detalles de los programas de PCR SSU e ITS2.
- Repita los pasos 8.4 a 8.8 hasta que se ejecuten todas las PCR.
- Mientras las reacciones de PCR están funcionando, vierta un gel de agarosa al 1,5% de 100 ml. Cada gel contendrá muestras o una sola placa de PCR.
- Agregue 1.5 g de agarosa a un matraz de 500 ml, luego agregue 100 ml de 1 tampón TAE (Tabla suplementaria 3) y revuelva para mezclar. Microondas para disolver y enfriar el gel.
- Una vez que la solución se haya enfriado, agregue 5 μL de solución de bromuro de etidio de 10 mg / ml y mezcle para combinar. Vierta la solución en una bandeja de fundición con cuatro peines de 25 pocillos para que el gel pueda acomodar 96 muestras más una escalera para cada fila en el gel.
NOTA: El bromuro de etidio es un mutágeno potente. Cuando manipule bromuro de etidio, use una bata de laboratorio, guantes resistentes a productos químicos y gafas de seguridad química.
- Justo antes de que termine la PCR, agregue un tinte de carga 6x a un comedero desechable y use una pipeta multicanal para agregar 2 μL de tinte de carga 6x a cada pocillo de una nueva placa DE PCR de 96 pocillos. Esta placa se utilizará para cargar las muestras en el gel. Haga suficientes de estas placas para acomodar todas las muestras.
- Cuando terminen las PCR, retire las placas de PCR y centrívelas brevemente a 300 x g durante 15 s en RT. Guarde las placas de PCR en hielo hasta que los productos de PCR se puedan agotar en un gel.
- Para ejecutar los productos en un gel, use una pipeta multicanal de 12 pocillos para agregar 5 μL de cada muestra al pozo apropiado de una placa de 96 pocillos que contenga 2 μL de tinte de carga 6x.
- Luego cargue 6 μL de esta mezcla en cada pozo de un gel recién fundido. Cargue 6 μL de 1 KB más escalera en el primer pozo de cada fila del gel.
NOTA: Para llenar los pocillos del gel, puede ser necesario intercalar la fila A y la fila B de la placa de PCR en la primera fila del gel. Para evitar confusiones, registre el gel_number y gel_position en la hoja de genotipado para cada muestra de PCR.
- Coloque una nueva tapa de lámina en la PCR restante en la placa o placas y guárdelas a 4 °C. Estos productos de reacción se utilizarán para la secuenciación en el paso 9.
- Ejecute los productos de PCR en el gel a 120 V durante 20 minutos. Imagine el gel y el registro de qué etiquetas S producen productos ITS2 y/o SSU PCR en las columnas "pcr_product_its2" y "prc_product_ssu" de la hoja de genotipado. Marcar la presencia de una banda con un '1'; marca un '0' para ninguna banda.
9. Identificación de nematodos con secuenciación de Sanger y secuencia BLAST
NOTA: Esta sección proporciona instrucciones para secuenciar los amplicones ITS2 de las etiquetas S, alinear esas secuencias con la base de datos del Centro Nacional de Información Biotecnológica (NCBI) utilizando el algoritmo BLAST y analizar los resultados de BLAST para identificar los nematodos en las placas S.
- Para cada muestra que sea ITS2 positiva, utilice el producto ITS2 PCR restante para la secuenciación de Sanger utilizando el cebador delantero oECA306 (CACTTTCAAGCAACCCGAC). Haga arreglos para que los archivos de salida de secuenciación se vinculen fácilmente a una etiqueta S registrando las columnas 'sequencing_plate' y 'sequencing_well' de cada etiqueta S en la hoja de genotipado.
- Obtenga los archivos de salida .seq para cada etiqueta S de la plataforma de secuenciación. Organice los archivos .seq de un proyecto en un único directorio con archivos .seq para cada lote de secuenciación ubicado en subdirectorios.
- Abra la herramienta de interfaz de línea de comandos y navegue hasta el directorio superior que contiene los archivos .seq ingresando el comando: cd . Si aún no existe, cree un FASTA combinado para todos los archivos .seq ingresando el siguiente comando: for dir in */; do cd $dir; for file in *.seq; do echo ">"$file; cat $file; done >>.. /all_seqs.fa; cd..; hecho.
NOTA: Este código creará un archivo FASTA combinado denominado 'all_seqs.fa' a partir de todos los archivos .seq en el directorio del proyecto. Este archivo se puede utilizar en la herramienta BLAST de nucleótidos en línea de NCBI para alinear rápidamente la secuencia ITS2 de cada etiqueta S con la base de datos de secuencias de NCBI. - En un navegador web, navegue hasta el sitio web de NBCI BLAST20 y haga clic en el botón Elegir archivo . Seleccione el archivo all_seqs.fa que se acaba de crear, luego haga clic en el botón Secuencias algo similares (BLASTn). Haga clic en el botón BLAST en la parte inferior de la página para comenzar la búsqueda de BLAST.
- Actualice la hoja de genotipado con los resultados de BLAST para cada etiqueta S. Utilice la herramienta de filtro para facilitar la actualización de la hoja de google de genotipado. Haga clic en Datos > Crear un filtro para agregar un botón de filtro a cada encabezado de columna. Filtre la columna sequencing_plate para seleccionar las placas de secuenciación que se actualizarán con los resultados de BLAST.
- Utilice el menú desplegable de la página de resultados de NCBI BLAST para comprobar los resultados de cada secuencia ITS2 de placa S (Figura 6).
- Compruebe que no hay golpes BLAST. Un ID de secuencia en el menú desplegable con el prefijo * no tiene golpes de explosión. Para estas etiquetas S, introduzca «sin acierto » en la columna manual_blast_notes de la hoja de genotipado.
- Compruebe si hay una posible nueva especie de Caenorhabditis . Haga clic en el enlace en la parte superior para visualizar la alineación (Figura 6). Si el golpe superior es (1) una especie de Caenorhabditis , (2) la alineación contiene más de cinco desajustes en el centro de la secuencia, y (3) la cobertura de la consulta es mayor al 50%, este resultado sugiere que el aislado puede ser una nueva especie de Caenorhabditis (Figura 7). Para estas placas S, las especies de blast superior golpean en la columna 'species_id', ingresan un 1 en la 'columna possible_new_caeno_sp' y 'posible nuevo Caeno sp.' en la columna 'manual_blast_notes' junto con la identidad porcentual, (por ejemplo, 'posible nueva identidad Caeno sp. 89%').
- Para las secuencias de placa S que BLAST a una especie de Caenorhabditis , ingrese el género completo y el nombre de la especie del golpe BLAST superior en la columna 'species_id'. Por ejemplo, 'Caenorhabditis elegans'.
- Para las secuencias de placa S que BLAST a una especie que no es Caenorhabditis , ingrese solo el género del golpe BLAST superior seguido de 'sp.' en la columna 'species_id'. Esta notación significa que el aislado es una especie desconocida dentro del género nombrado. Por ejemplo, 'Oscheius sp.'.
NOTA: La secuencia ITS2 no se puede utilizar para identificar de forma fiable aislados a nivel de especie fuera del género Caenorhabditis3,13. - Introduzca 1 en la 'columna make_strain_name' de la hoja de genotipado si 'species_id' = 'Caenorhabditis elegans', 'Caenorhabditis briggsae' o 'Caenorhabditis tropicalis', O 'possible_new_caeno_sp' = 1.
- Nombra las cepas con nombres únicos siguiendo las convenciones de nomenclatura de Caenorhabditis , es decir, una designación de laboratorio única que consiste en 2-3 letras mayúsculas seguidas de un número para cada cepa única23. Introduzca los nombres de las cepas en la columna 'strain_name'.
- Después de nombrar las cepas, se pueden criopreservar utilizando los protocolos establecidos 24.
10. Procesamiento de los datos de recolección con el paquete easyFulcrum en R
Nota : este paso describe cómo vincular los datos de recopilación (etiquetas C) y los datos de aislamiento de nematodos (etiquetas S) juntos mediante el paquete easyFulcrum R. El software contiene funciones que unirán aún más los datos de Fulcrum con los datos de genotipado de la hoja de genotipado para que las identidades de especies de la etiqueta S y los nombres de las cepas se organicen en un solo marco de datos.
- Cree un nuevo directorio con el nombre del proyecto de colección. Organice la estructura de carpetas dentro del directorio para que coincida con los requisitos descritos en el paquete de R easyFulcrum15.
- Vaya al sitio web de Fulcrum e inicie sesión. Exporte los datos sin procesar del proyecto desde la base de datos de Fulcrum utilizando la herramienta de exportación de datos del sitio web de Fulcrum a la izquierda y seleccionando las siguientes casillas de verificación: proyecto, incluir fotos, incluir datos GPS, muestreo de campo y aislamiento.
NOTA: Los datos de Fulcrum para el proyecto se exportarán como cinco archivos de valores separados por comas (.csv). Los datos completos del proyecto se unirán en un solo marco de datos utilizando el paquete easyFulcrum en R. - Mueva los cinco archivos .csv exportados desde Fulcrum al directorio del proyecto creado en el paso 10.1 como se indica en la viñeta easyFulcrum21.
- Abra una sesión de Rstudio e instale el paquete easyfulcrum en R introduciendo los siguientes comandos en la consola de R 'install.packages("devtools")' y 'devtools::install_github("AndersenLab/easyfulcrum")'.
- Abra un nuevo script de R y siga las instrucciones de la viñeta easyfulcrum para procesar los datos de recopilación21.
Resultados
Este protocolo se ha utilizado para recolectar nematodos Caenorhabditis de múltiples ubicaciones, incluyendo Hawai y California. La tasa de éxito del aislamiento para los nematodos caenorhabditis varía según la ubicación de recolección, el clima, la experiencia de muestreo y los tipos de sustrato muestreados. El protocolo se ha utilizado para muestrear ampliamente las islas hawaianas, donde se han llevado a cabo nueve proyectos de recolección durante varios años y temporadas. Las tasas de éxito de aislamiento para las especies de Caenorhabditis son casi idénticas para C. briggsae (162 de 4.506 muestras, 3,6%) y C. elegans (163 de 4.506 muestras, 3,6%), y mucho más bajas para C. tropicalis (26 de 4.506 muestras, 0,58%)8. Cada una de las especies de selfing se enriquece con sustratos de frutas y flores en descomposición en relación con las otras categorías de sustratos. Muestree sustratos de frutas y flores podridas si el investigador está tratando de maximizar la tasa de éxito en lugar de caracterizar las preferencias de sustrato. Sin embargo, la tasa de éxito varía con la calidad del sustrato seleccionado. Por ejemplo, entre los sustratos de frutas y flores, aquellos sustratos que son demasiado secos, húmedos o frescos probablemente no producirán nematodos Caenorhabditis .
La escalabilidad de este protocolo de recolección es evidente por el número de colecciones que un solo par de investigadores puede recolectar de la naturaleza. Por ejemplo, en octubre de 2018, un par de investigadores que utilizaron este protocolo de recolección pudieron recolectar un total de más de 1,000 muestras en 7 días de múltiples ubicaciones en dos islas hawaianas. Este equipo de campo envió las muestras durante la noche al laboratorio, donde un equipo de ocho investigadores aisló más de 2.000 nematodos de las muestras a medida que llegaban. Una ventaja clave de este protocolo es que minimiza el costo asociado con el muestreo en ubicaciones remotas al reducir el equipo y el personal requerido en el campo. Usando este protocolo, un pequeño equipo de campo puede centrarse en el muestreo, mientras que el equipo de aislamiento puede procesar las muestras en su institución de origen utilizando equipos frágiles y pesados como microscopios de disección y placas de agar para aislar nematodos. Además, la implementación de la aplicación móvil de recopilación de datos permite que todos los datos de campo asociados con las muestras se vinculen directamente a la etiqueta C, lo que permite al equipo de aislamiento trabajar de forma independiente del equipo de campo mientras procesa las muestras.
Los investigadores que utilizan este protocolo de recolección deben considerar el esfuerzo que se requiere para aislar los nematodos antes de un proyecto de recolección. Los pasos de aislamiento e identificación limitan la velocidad, y un pequeño equipo de recolección puede abrumar rápidamente a los aisladores con muestras. Además, el espacio de laboratorio requerido para procesar muchas colecciones puede interferir con la investigación en curso (Figura 3). Además, algunos nematodos aislados requieren un esfuerzo adicional para genotipar. Por ejemplo, aproximadamente el 2% de los aislados no logran amplificarse con el conjunto de cebadores de PCR SSU después del primer intento de lisis y deben volver a lisarse para garantizar que el material de lisis sea adecuado para la amplificación con el conjunto de cebadores ITS2 (Figura 8). Además, aproximadamente el 3% de los aislados no producen secuencias de calidad después de una ronda inicial de secuenciación de Sanger. Para estos aislamientos, a menudo se requiere otra ronda de lisis, ITS2 PCR y secuenciación de Sanger, lo que puede aumentar el tiempo de entrega para el equipo de aislamiento. Es importante destacar que la identidad de secuencia por sí sola no es evidencia suficiente para justificar una nueva especie de Caenorhabditis (Figura 7). Para justificar adecuadamente la cría de un aislado como una nueva especie de Caenorhabditis, se debe hacer un esfuerzo adicional para realizar experimentos de apareamiento y establecer un espécimen mecanografiado13. También se prefiere una descripción morfológica formal del espécimen mecanografiado, pero no se requiere3. En conjunto, estas consideraciones sugieren que los investigadores que adopten este protocolo de recolección se beneficiarán de las pruebas de ensayo de los pasos de aislamiento e identificación para garantizar que los recursos se asignen adecuadamente antes de que comience un proyecto de recolección. Es importante destacar que incluso los pequeños proyectos de recolección pueden beneficiarse de este protocolo porque el proceso es altamente reproducible y los datos se pueden auditar fácilmente con fines de control de calidad en todos los grupos de laboratorio.

Figura 1: Ejemplos de sustrato. (A) Una fruta podrida ideal se muestra en el centro de la imagen (1), la fruta es casi irreconocible. Se muestra menos fruta podrida cerca; evitar tomar muestras de frutas recién caídas (2). (B) Una flor idealmente descompuesta se muestra en la parte superior (3). Evite probar flores recién caídas (4). (C) La hojarasca oscura debajo de la capa superior de hojas secas es ideal cuando se toma el muestreo para autogestionar nematodos Caenorhabditis (5). Evite tomar muestras de hojarasca seca (6). Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: La aplicación móvil Nematode Field Sampling. (A) La pantalla inicial después de abrir la aplicación Nematode Field Sampling en un dispositivo Apple en Fulcrum. La flecha roja en la parte inferior derecha apunta al botón + utilizado para crear un nuevo registro de colección. (B) Un ejemplo de un nuevo registro de colección que se muestra en un dispositivo Apple. La flecha roja apunta al campo 'Proyecto' en la parte superior de la pantalla de registro de colección. Asegúrese de seleccionar el proyecto correcto al muestrear en el campo. El campo de proyecto será de forma predeterminada el último proyecto utilizado al crear registros de recopilación posteriores. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Bolsas de recolección y placas de recolección organizadas antes de emplatar muestras. Esta figura muestra las muestras en bolsas de recolección con etiqueta C a la izquierda. Cada bolsa de colección tiene una placa de 10 cm con etiqueta C a juego en la parte superior. A la derecha hay placas de recolección de 10 cm que contienen material de muestra después de que se transfirió de las bolsas de recolección. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Una placa de recolección (placa C) con una muestra transferida adecuadamente. Una placa C de 10 cm con fruta en descomposición colocada en el borde del césped bacteriano. La etiqueta C está unida a la tapa de la placa. Haga clic aquí para ver una versión más grande de esta figura.
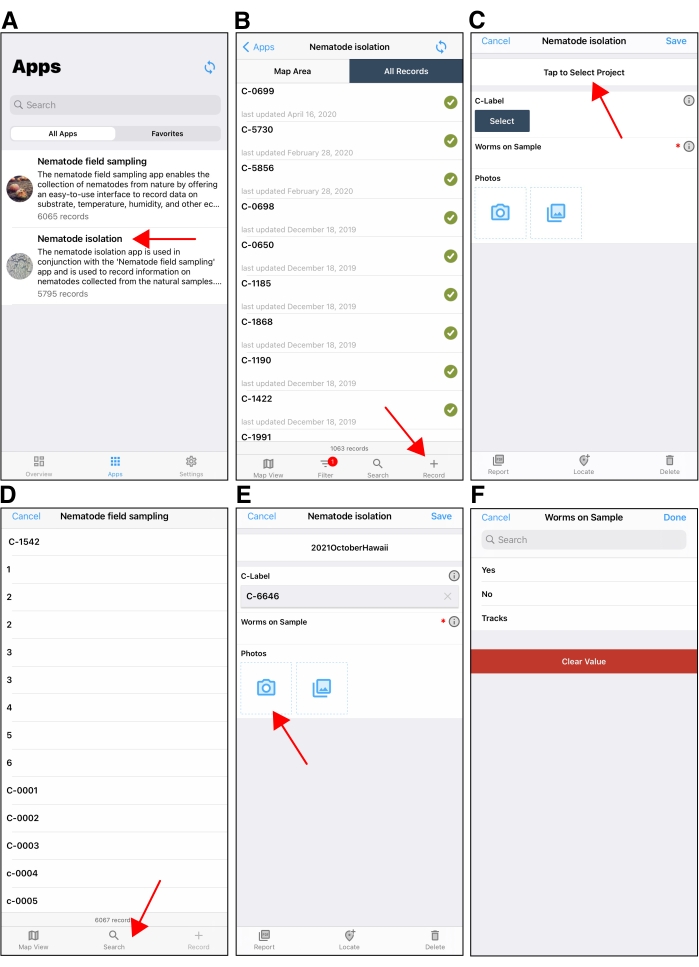
Figura 5: La aplicación móvil de aislamiento de nematodos. (A) La pantalla de selección de aplicaciones en la aplicación móvil Fulcrum. La flecha roja apunta a la aplicación Nematode Isolation . (B) La pantalla inicial después de abrir la aplicación Nematode Isolation en un dispositivo Apple en Fulcrum. La flecha roja en la parte inferior derecha apunta al botón + utilizado para crear un nuevo registro de aislamiento. (C) Un ejemplo de un nuevo registro de aislamiento que se muestra en un dispositivo Apple. La flecha roja apunta al campo 'Proyecto' en la parte superior de la pantalla de registro de aislamiento. Asegúrese de seleccionar el proyecto correcto al aislar. El campo de proyecto será de forma predeterminada el último proyecto utilizado al crear registros de aislamiento posteriores. (D) Después de tocar el campo Seleccionar debajo de la etiqueta C, los usuarios tocarán el botón de búsqueda (flecha roja) para encontrar la etiqueta C de la que están aislando los nematodos. (E) Después de seleccionar la etiqueta C, los usuarios fotografiarán la placa C utilizando la cámara del dispositivo. (F) Los usuarios luego ingresan si hay nematodos en la placa C o no. Las etiquetas S se agregan al registro de aislamiento si hay nematodos que se deben aislar. Haga clic aquí para ver una versión más grande de esta figura.
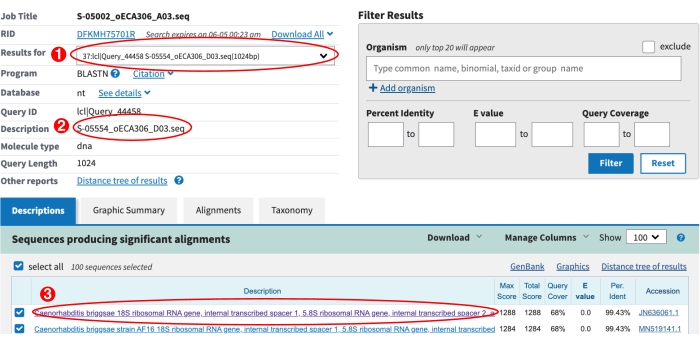
Figura 6: Página de resultados de NCBI BLAST. (1) El menú desplegable utilizado para ver los resultados de BLAST para todas las secuencias. (2) La descripción de la secuencia actual seleccionada en el menú desplegable. En este caso se muestran los resultados para la etiqueta S S-05554. (3) Se muestra el golpe BLAST superior para S-05554. El texto púrpura indica que se ha hecho clic en el enlace para visualizar esta alineación. Asegúrese de inspeccionar las alineaciones a ojo para identificar posibles nuevas especies de Caenorhabditis , consulte el paso 9.8 anterior. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Ejemplos de visualización de alineación de NCBI BLAST. (A) Un ejemplo de la secuencia de consulta ITS2 de un aislado alineada con una secuencia de sujeto de C. kamaaina . (1) La identidad porcentual de la alineación (89%), que es baja para un golpe BLAST superior. (2) Un desajuste entre la consulta y la secuencia de asunto (G a A). (3) Una brecha de cuatro pares de bases en la secuencia de sujetos hecha por el algoritmo de alineación; los huecos en la consulta o el asunto indican una alineación deficiente. (4) Una región generalizada en el centro de la alineación con muchos desajustes y brechas. Una región como esta sugiere que la secuencia de consulta podría provenir de una nueva especie de Caenorhabditis . Se muestra un ejemplo de alineación real de una nueva especie, C. oiwi, que se descubrió en 2017. (B) Un ejemplo de una buena alineación entre la secuencia de consulta ITS2 de un aislado y una secuencia de sujetos. (5) La identidad porcentual de la alineación (99%), lo que generalmente significa que la secuencia de consulta proviene de un aislado de la misma especie que el sujeto. (6) Una región central de la alineación con identidad perfecta. Una región como esta sugiere que el aislado de consulta es probablemente la misma especie que el sujeto. Haga clic aquí para ver una versión más grande de esta figura.
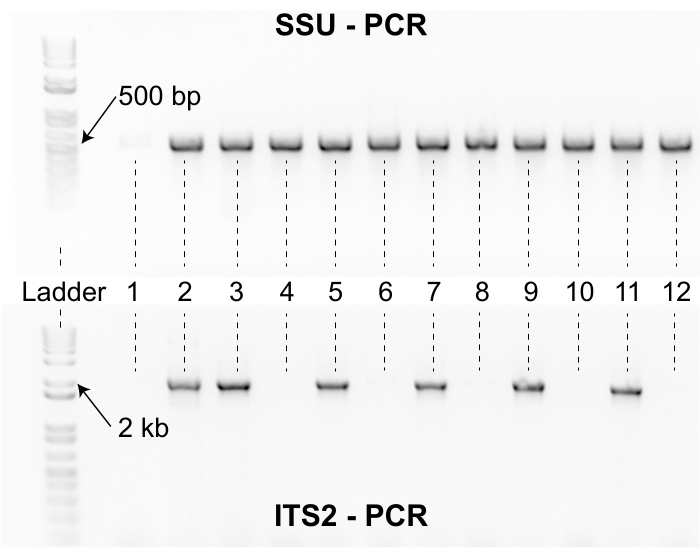
Figura 8: Productos de PCR SSU e ITS2. El gel superior muestra los productos de PCR generados con el juego de imprimación SSU para 12 muestras representativas. Una escalera de ADN se incluye a la izquierda como referencia. Los productos SSU PCR para nematodos Caenorhabditis tienen aproximadamente 500 pb de longitud. Las muestras 2-12 se amplificaron con el conjunto de cebadores SSU, pero la muestra uno no lo hizo. La ausencia de un amplicóN SSU de 500 pb para la muestra uno sugiere que el material de lisis era de mala calidad y la muestra debe volver a lisarse. El gel inferior muestra los productos de PCR generados con el conjunto de imprimación ITS2 para las mismas 12 muestras que se muestran en el gel superior. La escalera y las muestras están en la misma orientación para ambos geles. Seis de las 12 muestras no se amplificaron con el conjunto de cebadores ITS2. Las muestras con bandas SSU e ITS2 son secuenciadas por Sanger e identificadas por similitud de secuencia utilizando el algoritmo NCBI BLAST. Haga clic aquí para ver una versión más grande de esta figura.
Expediente suplementario 1: Etiquetas C. Un archivo PDF que contiene 2500 etiquetas C únicas. Haga clic aquí para descargar este archivo.
Expediente suplementario 2: Etiquetas S. Un archivo PDF que contiene 5000 etiquetas S únicas. Haga clic aquí para descargar este archivo.
Tabla suplementaria 1: Materiales de campo. Una lista de embalaje de los materiales utilizados en el campo para tomar muestras de nematodos. Haga clic aquí para descargar esta tabla.
Tabla suplementaria 2: Recetas de PCR y condiciones del termociclador. Tabla de recetas de PCR y condiciones del termociclador para las PCR ITS2 y SSU. Haga clic aquí para descargar esta Tabla.
Tabla suplementaria 3: Recetas tampón de electroforesis. Una receta para la solución de ácido etilendiaminotetraacético (EDTA) de 0,5 M pH 8,0 y la solución tampón TRIS-acetato-EDTA (TAE). Haga clic aquí para descargar esta tabla.
Discusión
Este protocolo contiene pasos críticos que deben ejecutarse con precaución. Por ejemplo, es importante que los equipos de campo y aislamiento tengan cuidado de seleccionar el proyecto de recolección correcto en la aplicación antes de recolectar muestras del campo o aislar nematodos de las muestras en el laboratorio. En el caso de que se seleccione el proyecto de recopilación incorrecto, los registros de datos errantes se corrigen mejor en la base de datos Fulcrum utilizando las herramientas de edición de registros en línea. Este proceso puede ser tedioso para muchos registros extraviados. Sin embargo, la base de datos conserva cualquier cambio en los registros para que sea posible una auditoría completa de los registros de recopilación y aislamiento. Los otros pasos críticos en este protocolo implican el manejo de muestras del campo y los nematodos aislados de esas muestras. Para garantizar que los nematodos caenorhabditis sobrevivan a las etapas de muestreo y envío, la temperatura de las muestras debe mantenerse entre 4 °C y 25 °C. Las temperaturas superiores a 25 °C pueden causar esterilidad en C. elegans14. Asegúrese de que las muestras se transfieran de las bolsas de recolección a las placas de recolección dentro de los cinco días siempre que sea posible para minimizar la pérdida de nematodos. Después de aislar los nematodos, es fundamental que sean genotipados y criopreservados antes de que perezcan. Es difícil encontrar nematodos vivos en las placas S que tienen más de dos o tres semanas de edad porque la contaminación fúngica y bacteriana puede hacer que las placas S sean inhóspitas.
Este protocolo se puede modificar fácilmente para acomodar diferentes tipos de datos que los investigadores pueden querer recopilar mientras están en el campo. Por ejemplo, es fácil personalizar la aplicación 'Nematode field sampling' con nuevos campos de entrada de datos utilizando la GUI en línea de Fulcrum para la edición de aplicaciones. Además, el paquete de análisis de datos, easyFulcrum, puede acomodar estas ediciones al procesar los nuevos datos15. Otra modificación que los usuarios pueden encontrar atractiva es usar un método de muestreo diferente en el campo. En lugar de muestrear sustratos discretos, los investigadores pueden desear muestrear áreas más grandes que contengan múltiples tipos de sustratos. Estas muestras más grandes se procesan mejor en el laboratorio utilizando los métodos de extracción de embudo o bandeja de Baermann13. Es importante destacar que el uso de etiquetas C y S todavía se aplican a estas técnicas y, por lo tanto, son compatibles con las aplicaciones móviles.
Las principales limitaciones de este protocolo se relacionan con el tiempo de manipulación de los nematodos antes del aislamiento en el laboratorio. En primer lugar, el tiempo de retraso entre la recolección de la muestra y el aislamiento de los nematodos hace que sea imposible registrar las etapas de desarrollo de los nematodos en una muestra determinada en el momento de la recolección. En segundo lugar, la frecuencia de los machos y el cruce en la naturaleza son cuestiones evolutivas clave para la autogestión de los nematodos Caenorhabditis10. Este método no es adecuado para abordar estas preguntas porque es probable que los nematodos hayan pasado por múltiples generaciones antes del aislamiento. El aislamiento tardío significa que la evidencia directa de la frecuencia masculina en la naturaleza es imposible. Además, el retraso multigeneracional durante los pasos de genotipado significa que la evidencia genómica de cruce (heterocigosidad) se erosionará antes de que se pueda secuenciar una cepa de nematodo. Para identificar la heterocigosidad en la naturaleza, se utilizan las crías producidas por un nematodo directamente aislado de la naturaleza para la secuenciación2. Otra limitación potencial de este protocolo es que está sesgado hacia la identificación de caenorhabditis autointencional. Esto se debe a que los nematodos aislados de las especies que se autoalifican tienen una mayor probabilidad de proliferar que los cruzados obligados, que solo proliferarán si se aísla una hembra fertilizada.
Este método de recolección se basa en los protocolos de recolección existentes13,14. El mayor avance de esta técnica es el uso de tecnología móvil y software personalizado para facilitar la organización de grandes cantidades de datos ecológicos y moleculares asociados con proyectos de recolección a gran escala. Los datos ecológicos generados utilizando este protocolo de recolección se pueden utilizar para abordar preguntas pendientes para las poblaciones naturales de especies de Caenorhabditis. Por ejemplo, los datos generados con este método se han utilizado para descubrir preferencias de nicho para la especie en las islas hawaianas. Además, al secuenciar los genomas de los nematodos criopreservados, los investigadores pueden investigar cómo los patrones de variación genética se correlacionan con los datos ecológicos. Investigaciones de este tipo pueden descubrir firmas de adaptación local en poblaciones de Caenorhabditis y proporcionar información importante sobre la relevancia de la variación genética en contextos naturales8. Para obtener una comprensión funcional de muchos genes en los nematodos caenorhabditis, es probable que se requieran estudios ecológicos11. Incluso para C. elegans una gran fracción de genes carece de anotaciones funcionales, a pesar de ser el primer animal multicelular en ser secuenciado y uno de los animales más estudiados en la Tierra. Este protocolo de recolección fue desarrollado para ayudar a abordar esta brecha de conocimiento al facilitar la recolección de nematodos silvestres de Caenorhabditis y el estudio de su ecología y diversidad genética natural.
Divulgaciones
Los autores informan que no hay conflictos de intereses.
Agradecimientos
Esta investigación fue apoyada por fondos de puesta en marcha de la Universidad Northwestern y un Premio CAREER de la Fundación Nacional de Ciencias (IOS-1751035), ambos otorgados a E.C.A.
Materiales
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
Referencias
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados