Method Article
Un approccio altamente scalabile per eseguire indagini ecologiche sui nematodi della caenorhabdite selfing
In questo articolo
Riepilogo
Questo protocollo può essere utilizzato per eseguire indagini ecologiche su larga scala dei nematodi Caenorhabditis selfing. Il vantaggio principale di questo metodo è l'organizzazione efficiente e l'analisi dei dati ecologici e molecolari associati ai nematodi raccolti dalla natura.
Abstract
Caenorhabditis elegans è uno dei principali organismi modello in biologia, ma solo di recente i ricercatori si sono concentrati sulla sua ecologia naturale. La relativa scarsità di informazioni su C. elegans nel suo contesto naturale deriva dalle sfide coinvolte nell'identificazione del piccolo nematode in natura. Nonostante queste sfide, una crescente attenzione all'ecologia di C. elegans ha causato una ricchezza di nuove informazioni riguardanti la sua vita al di fuori del laboratorio. L'intensificazione della ricerca di C. elegans in natura ha contribuito alla scoperta di molte nuove specie di Caenorhabditis e ha rivelato che i nematodi congenerici spesso convivono in natura, dove si nutrono di fioriture microbiche associate a materiale vegetale in decomposizione. L'identificazione di nuove specie ha anche rivelato che il sistema di accoppiamento androdioico dei maschi e degli ermafroditi autofertilizzanti si è evoluto tre volte indipendentemente all'interno di Caenorhabditis. Le altre due specie auto-antagoniste, C. briggsae e C. tropicalis, condividono i vantaggi sperimentali di C. elegans e hanno permesso studi comparativi sulle basi meccanicistiche di tratti importanti, tra cui l'autofecondazione. Nonostante questi progressi, resta ancora molto da imparare sull'ecologia e la diversità naturale di queste importanti specie. Ad esempio, ci mancano ancora informazioni funzionali per molti dei loro geni, che potrebbero essere raggiunti solo attraverso una comprensione della loro ecologia naturale. Per facilitare la ricerca ecologica dei nematodi Caenorhabditis selfing, abbiamo sviluppato un metodo altamente scalabile per raccogliere i nematodi dalla natura. Il nostro metodo utilizza piattaforme mobili di raccolta dati, database basati su cloud e l'ambiente software R per migliorare la capacità dei ricercatori di raccogliere nematodi dalla natura, registrare dati ecologici associati e identificare nematodi selvatici utilizzando codici a barre molecolari.
Introduzione
Gli ultimi due decenni hanno portato un crescente interesse per l'ecologia dei nematodi Caenorhabditis. Da questi studi, sappiamo che le specie di Caenorhabditis a vita libera possono essere isolate da micro-habitat effimeri sia nelle regioni temperate che tropicali, dove si nutrono di fioriture microbiche associate a materiale vegetale in decomposizione, a volte in simpatia1,2,3,4,5,6,7,8 . Abbiamo anche imparato che l'evoluzione convergente dell'autofecondazione si è verificata nel genere tre volte, e il selfing è la modalità dominante di riproduzione per C. briggsae, C. elegans e C. tropicalis9,10. Tra questi selfers, C. elegans è uno degli animali più studiati sulla Terra ed è stato utilizzato dai ricercatori per fare progressi critici in biologia. È importante sottolineare che le altre specie di Caenorhabditis condividono molti dei vantaggi sperimentali di C. elegans e stanno rapidamente avanzando studi comparativi nel genere. Tuttavia, la natura criptica di questi nematodi in natura rende difficile studiare la loro ecologia e diversità naturale, che è fondamentale per comprendere le funzioni biologiche dei loro geni e i modi in cui l'evoluzione ha modellato la diversità genetica tra le specie10,11.
La più grande sfida per studiare l'ecologia dei nematodi Caenorhabditis in natura è la loro piccola dimensione; i nematodi adulti sono spesso lunghi 1 mm o meno. Questa sfida richiede che i ricercatori campionino substrati selvatici e tentino di separare i nematodi di interesse dai substrati in laboratorio senza la possibilità di osservare gli animali in natura. Poiché anche gli esperti addestrati hanno difficoltà a discriminare i nematodi di Caenorhabditis autoalimentati da altri nematodi a vita libera al microscopio, i nematodi vengono in genere rimossi dal substrato, isolati e lasciati proliferare prima di essere identificati dall'identità di sequenza utilizzando codici a barre molecolari stabiliti3,12,13,14 . Il tempo e gli sforzi necessari per elaborare ogni nematode in questo modo rappresentano una sfida organizzativa, in quanto i ricercatori devono essere in grado di rintracciare l'identità di ciascun nematode isolato in laboratorio fino al substrato esatto e ai dati ecologici associati campionati sul campo. Qui, descriviamo un processo passo-passo per raccogliere e identificare in modo efficiente i nematodi caenorhabditis selfing dal campo e collegare fedelmente questi isolati con i loro dati spaziali ed ecologici associati su larga scala.
Questo metodo di raccolta aumenta la scala e l'accuratezza delle indagini ecologiche utilizzando piattaforme di raccolta dati mobili, database basati su cloud e l'ambiente software R. Fulcrum è una piattaforma di raccolta dati personalizzabile che funziona con la maggior parte dei dispositivi mobili e consente agli utenti di creare applicazioni personalizzate per raccogliere e organizzare dati basati sulla posizione (https://www.fulcrumapp.com). Questo protocollo fornisce istruzioni dettagliate su come utilizzare applicazioni di raccolta dati personalizzate per organizzare dati ecologici spazialmente espliciti dal campo e collegare accuratamente tali dati con l'identità dei nematodi isolati in laboratorio. Il protocollo spiega anche come identificare in modo efficiente i nematodi Caenorhabditis selfing utilizzando codici a barre molecolari stabiliti. I dati di questi metodi possono essere elaborati in modo semplice e riproducibile con il pacchetto software R di accompagnamento easyFulcrum15 per esplorare l'ecologia e la diversità genetica delle popolazioni naturali di Caenorhabditis.
Protocollo
1. Preparazione della raccolta
- Identificare una posizione per esaminare i nematodi Caenorhabditis .
NOTA: Nella maggior parte delle regioni temperate, C. elegans e C. briggsae possono essere facilmente isolati da habitat associati all'uomo come campi agricoli o giardini rurali e urbani1. Nelle regioni subtropicali e tropicali, C. briggsae, C. elegans e C. tropicalis si trovano tutti negli habitat associati all'uomo sopra elencati, a volte in prossimità l'uno dell'altro. Tuttavia, C. elegans sembra preferire habitat più freschi e secchi rispetto alle altre specie negli habitat tropicali7,8. Ciascuna delle specie può anche essere isolata da habitat selvatici che non sono associati all'uomo, ma questi habitat vengono campionati meno spesso. - Creare un progetto Fulcrum per organizzare la raccolta e l'isolamento dei dati con le applicazioni mobili di raccolta dati.
- Crea un account con Fulcrum online utilizzando un accordo educativo gratuito16. Aggiungi l'applicazione Nematode Field Sampling a un account Fulcrum facendo clic sul pulsante AGGIUNGI APP17.
- Aggiungi l'applicazione Nematode Isolation a un account facendo clic sul pulsante AGGIUNGI APP18.
NOTA: si consiglia che ogni viaggio in una località sia organizzato come progetto di raccolta utilizzando la convenzione di denominazione 'YearMonthLocation', ad esempio, 2020FebruaryAustralia.
- Aggiungere utenti all'account Fulcrum per concedere loro l'accesso al progetto di raccolta. Assicurarsi che ogni utente scarichi l'applicazione mobile Fulcrum per partecipare al progetto.
- Stampa una serie di etichette con codice QR per tenere traccia delle collezioni (etichette C) e degli isolamenti dei nematodi (etichette S) con l'applicazione mobile. Attaccare le etichette C ai sacchetti di plastica con chiusura lampo, arrotolare i sacchetti etichettati in gruppi di 25 e avvolgerli con un elastico per l'imballaggio. Conservare il set di etichette S per l'uso in laboratorio.
NOTA: In tutto questo protocollo, le collezioni (substrati dal campo) sono contenute in sacchetti o su piastre e sono etichettate con etichette C. I nematodi isolati sono etichettati con etichette S. Le etichette C vengono utilizzate per identificare collezioni uniche e le etichette S vengono utilizzate per identificare isolati di nematodi univoci. Questi due tipi di etichette vengono utilizzati per stabilire la connessione tra una particolare collezione (C-label) e i nematodi isolati da quella collezione (S-labels) nel database Fulcrum. Stampare il doppio del numero di etichette S come etichette C per un progetto di raccolta perché, in media, due nematodi sono isolati per collezione. Se necessario, è possibile stampare altre etichette S in un secondo momento. Nel supplemento sono fornite 2.500 etichette C univoche (File supplementare 1) e 5.000 etichette S uniche (File supplementare 2). - Preparare lastre NGMA da 10 cm per le raccolte e lastre NGMA da 3,5 cm per isolare i nematodi. Realizza un piatto da 10 cm e almeno due piatti da 3,5 cm per collezione21. Queste piastre sono seminate con Escherichia coli ceppo OP50 seguendo protocolli stabiliti. Conservare le piastre prima dell'uso a 4 °C per non più di 1 mese.
2. Raccolta dei campi
NOTA: i nematodi Caenorhabditis sono spesso isolati da materiale vegetale in decomposizione, tra cui frutta, noci, semi, baccelli, fiori, steli, lettiere vegetali e compost1,5,6,8. I migliori substrati sono marci e quasi irriconoscibili come frutti o fiori; evitare substrati troppo secchi o bagnati (Figura 1). I substrati vengono raccolti in modo più efficiente dal campo lavorando in coppia. L'individuo con il termometro a infrarossi senza contatto selezionerà un substrato per la raccolta e raccoglierà il campione mentre il suo partner utilizza l'applicazione Nematode Field Sampling in Fulcrum per registrare i dati di raccolta. La coppia di collezionisti ripeterà questo processo fino a quando non verrà raccolto il numero desiderato di campioni. L'elenco dei materiali necessari per il lavoro sul campo si trova in (Tabella supplementare 1).
- Apri l'app mobile Fulcrum, seleziona Nematode Field Sampling dal menu a discesa. Premere + per avviare un nuovo record nel progetto (Figura 2A). Scatta una foto del substrato.
- Fare clic sulla casella in alto al centro per selezionare il progetto di raccolta corretto realizzato nel passaggio 1.2 (Figura 2B). Tocca il campo C-label nella parte inferiore del record della raccolta e scegli Scansione quando viene visualizzato il prompt. Scansiona il codice a barre sul sacchetto di raccolta utilizzando la fotocamera del dispositivo mobile, quindi tocca Fine in alto a destra dello schermo.
- Toccare il campo Substrato e selezionare un tipo di substrato dal menu a discesa. Aggiungere note sul substrato toccando il campo Note substrato e immettendo manualmente le note.
- Scegli un paesaggio dal menu a discesa. Scegli il paesaggio che meglio rappresenta il sito di campionamento.
- Scegli una vista del cielo. Quando si sceglie la vista del cielo, descrivere la visibilità del cielo nel sito di campionamento (ad esempio, una vista a cielo intero senza viste ostruite da alberi o altre strutture = piena).
- Misurare la temperatura superficiale del substrato utilizzando il termometro senza contatto e registrare il valore nel campo della temperatura del substrato.
NOTA: tenere il termometro senza contatto a non più di 14 pollici dal substrato durante la registrazione della temperatura. - Misurare la temperatura e l'umidità ambiente con il dispositivo portatile e registrare questi dati nei campi appropriati.
NOTA: verificare che il dispositivo di temperatura e umidità ambiente non sia in attesa. L'unità di misura cambierà quando il pulsante viene rilasciato. Tenere il dispositivo in una tasca esterna per evitare letture irregolari. - Salva il record in Fulcrum toccando Salva in alto a sinistra dello schermo.
- Raccogli circa un cucchiaio del substrato senza bastoncini o altri pezzi duri invertendo il sacchetto di raccolta per usarlo come "guanto" per raccogliere il substrato, quindi sigilla il sacchetto. Metti un tovagliolo di carta nel sacchetto se il campione è particolarmente umido.
NOTA: Nei climi caldi, posizionare i sacchetti in soft cooler con confezioni di refrigeratori per mantenere fresche le collezioni. - Dopo che tutti i campioni sono stati raccolti per il giorno, pulire l'attrezzatura di raccolta, estrarre le batterie dalle sonde, ricaricare le batterie, ricongelare i pacchetti congelatori. Sincronizza i dati di raccolta di Fulcrum toccando il pulsante Sincronizza in alto a sinistra dell'applicazione Nematode Field Sampling .
NOTA: i caricamenti possono richiedere diversi minuti senza una forte connessione cellulare, quindi potrebbe essere meglio attendere l'accesso WiFi. I dati rimarranno sui dispositivi mobili e saranno sincronizzati con il cloud. - Spedisci i campioni a un istituto di origine mettendoli in una scatola di spedizione durante la notte. Ridurre al minimo il tempo in cui i campioni sono esposti a temperature inferiori a 11 °C o superiori a 25 °C spedendo i pacchi nei giorni in cui il carico viene trasportato.
NOTA: la maggior parte delle strutture di spedizione non spedisce merci durante la notte nei fine settimana in località remote.
3. Placcatura delle collezioni sul campo in laboratorio
NOTA: questa sezione spiega come organizzare il trasferimento dei campioni dai sacchetti di raccolta etichettati alle lastre etichettate. I campioni possono arrivare da una spedizione notturna o direttamente dal campo.
- Ricevi la spedizione delle collezioni e ispeziona i bagagli rotti o altre prove di danni. Se i sacchetti sono rotti, scartare il materiale e pulire i sacchetti di raccolta ininterrotti con il 70% di etanolo; evitare l'etichetta C sul sacchetto con l'etanolo in quanto scolorirà l'etichetta e renderà difficile la lettura.
- Per ogni sacchetto con chiusura a zip, annotare l'etichetta C sulla borsa e attaccare un'etichetta C corrispondente al coperchio di una piastra da 10 cm macchiata di batteri OP50.
NOTA: le piastre da 10 cm etichettate sono denominate "piastre C" per il resto del protocollo. Il modo più semplice per organizzare i campioni è posizionare i sacchetti di raccolta su un banco da laboratorio con la piastra C corrispondente in cima (Figura 3). - Per ogni raccolta, trasferire circa un cucchiaio di campione dal sacchetto di raccolta al C-plate usando un cucchiaio di plastica pulito. Aggiungere il campione intorno al prato batterico a forma di mezzaluna o anello, non coprire completamente il prato batterico (Figura 4).
NOTA: Mantenere pulito il cucchiaio mettendolo in un becher di etanolo al 95% quando non è in uso. Utilizzare un tovagliolo di carta per asciugare il cucchiaio prima di trasferire ulteriori campioni. - Registrare il tempo in cui le raccolte sono state trasferite dai sacchi di raccolta alle piastre C e mantenere le piastre C a temperatura ambiente (RT) per almeno 24 ore prima di tentare di isolare i nematodi nella sezione 4.
4. Isolare i nematodi dalle collezioni
- Aprire l'applicazione Fulcrum sul dispositivo mobile e scegliere Nematode Isolation dal menu dell'applicazione (Figura 5A). Crea un nuovo record di isolamento toccando l'icona + in basso a destra (Figura 5B).
- Nella nuova schermata del record di isolamento, confermare il progetto di raccolta corretto selezionando il nome del progetto visualizzato nella casella in alto al centro. Se viene visualizzato il progetto errato, toccare il nome del progetto per passare al progetto corretto (Figura 5C).
- Toccare il pulsante Seleziona sotto il campo C-label per trovare l'etichetta C associata al campione da cui vengono isolati i nematodi (Figura 5D). Tocca l'icona Cerca , quindi tocca l'icona Scansione per scansionare il codice QR dell'etichetta C sulla piastra C con la fotocamera del dispositivo. Una volta scansionato il codice QR, nel campo C-label verrà visualizzato un record C-label .
- Toccare l'icona Fotocamera nel campo Foto per aprire la fotocamera del dispositivo e utilizzarla per scattare una foto del campione sulla piastra C con il codice QR visibile (Figura 5E). Tocca Fine per tornare alla schermata Isolamento.
NOTA: queste foto dei record di isolamento possono essere utilizzate per esplorare attributi specifici del substrato in un secondo momento. - Utilizzare un microscopio di dissezione per cercare i nematodi sulla piastra C. Toccare il campo Worms on Sample per registrare la presenza di nematodi sul campione (Figura 5F). Toccare Sì se i nematodi sono presenti sulla piastra C e toccare No se non sono presenti nematodi.
- Tocca Tracce se sono presenti solo tracce di nematodi. Se non sono presenti nematodi, parafilmare la piastra C e smaltirla in un bidone a rischio biologico.
NOTA: capovolgere la piastra C sopra il cestino dei rifiuti a rischio biologico e picchiettare delicatamente il retro della piastra per rimuovere tutti i substrati campionati. Questo passaggio rende più facile trovare e isolare i nematodi che possono trovarsi sotto il substrato sulla piastra C. - Se sono presenti nematodi, isolare fino a cinque nematodi dalla piastra C. Per isolare un nematode, trasferire un nematode dalla piastra C a una piastra S utilizzando un plettro di filo di platino. Isolare adulti sani e gravidi, se possibile. Tuttavia, isolare altre fasi se gli adulti non vengono trovati.
NOTA: dopo l'isolamento, fino a cinque piastre S avranno ciascuna un singolo nematode su di esse. Tenere queste piastre S con nematodi isolati dalla stessa piastra C organizzate insieme in una pila ordinata lontano dalle altre piastre S fino a quando non vengono inserite in Fulcrum. - Toccare il campo Piastre con etichetta S per inserire le piastre S utilizzate per questo isolamento. Tocca il + in basso a destra. Toccare S-label e quindi fare clic su Scansione per aprire la fotocamera del dispositivo. Utilizzare la fotocamera del dispositivo per scansionare il codice QR S-label sulla piastra S.
NOTA: assicurarsi che il codice dell'etichetta S corrisponda al codice sulla piastra. Se corrisponde, tocca Fine. In caso contrario, tocca Annulla e ripeti la scansione fino a quando non corrisponde, quindi fai clic su Fine. A volte i codici QR delle piastre vicine vengono scansionati accidentalmente. - Dopo aver inserito ogni S-plate, salva la voce con il pulsante Salva in alto a destra. La voce andrà persa se non viene salvata. Toccare il + in basso a destra per aggiungere altre piastre con etichetta S, se necessario, fino a quando non vengono inseriti tutti i nematodi isolati dalla piastra C. Dopo aver aggiunto tutte le piastre con etichetta S per il record di isolamento, toccare il pulsante < in alto a sinistra per tornare alla schermata di registrazione dell'isolamento.
NOTA: per annullare un record di isolamento perché gli errori non possono essere risolti , fare clic su Annulla in alto a sinistra. Questo passaggio aprirà una finestra di dialogo che chiede se il record può essere eliminato senza salvare. Se lo si desidera, fare clic su Sì, Scarta. - Tocca il pulsante Salva in alto a destra una volta che il record di isolamento ha aggiunto tutte le informazioni correttamente. Quindi parafilmare le piastre S con nematodi isolati e metterle da parte in un'area designata per contenere placche S con nematodi.
- Parafilmare la piastra C e scartarla nel cestino a rischio biologico. Tocca l'icona Sincronizza per caricare tutti i dati su Fulcrum.
- Ordina tutte le piastre S in ordine alfanumerico, quindi posiziona le piastre S in scatole di cartone. Assicurarsi che le piastre S siano rivolte verso il basso e parafilmate. Impila fino a quattro piastre A S in un'unica posizione nella scatola ed etichetta la scatola di cartone con il nome del progetto, la data, l'ora e un numero di scatola univoco.
- Conservare le scatole etichettate su RT. Questi isolati saranno controllati per la proliferazione a 48 h e di nuovo a 168 h se necessario.
5. Esportazione di piastre S da Fulcrum
NOTA: in questa sezione viene descritto come esportare le etichette S utilizzate nel processo di isolamento dal database del progetto Fulcrum. Queste etichette S saranno utilizzate per tracciare le linee isofemine proliferanti mentre vengono identificate dall'identità della sequenza nelle sezioni 6-9.
- Accedi al sito Web Fulcrum e seleziona l'applicazione Nematode Isolation . Fai clic su Esporta dal lato sinistro dello schermo. Fare clic per selezionare il progetto desiderato e selezionare la casella Isolamento nematodi . Fare clic su Avanti per scaricare un file .zip contenente il file "nematode_isolation_s_labeled_plates.csv".
- Apri il file "nematode_isolation_s_labeled_plates.csv" e ordinalo in base alla colonna "S-label" in ordine crescente (l'etichetta S più piccola sarà in alto). Seleziona tutte le etichette S e copiale dal foglio di calcolo.
- Passa al modello di genotipizzazione isolato selvaggio foglio google (wild_isolate_genotyping_template) utilizzando un browser web19.
- Crea una copia di questo foglio Google facendo clic con il pulsante destro del mouse sulla scheda Modello di genotipizzazione , quindi selezionando l'opzione Copia in nuovo foglio di calcolo . Seleziona Apri foglio di calcolo per visualizzare il nuovo foglio Google.
- Assegna a questo nuovo foglio il nome del progetto fulcro seguito da "wild_isolate_genotyping", ad esempio "2020FebruaryAustralia_wild_isolate_genotyping".
NOTA: questo foglio è indicato come "foglio di genotipizzazione" in tutto il resto del protocollo.
- Incolla le etichette S copiate dalla colonna "nematode_isolation_s_labeled_plates.csv" "s_label" nella colonna del foglio di genotipizzazione intitolata "s_label". Seleziona la colonna "s_label_repeat_error" per "1". Un valore di '1' in questa colonna indica che l'etichetta S è duplicata da qualche parte sul foglio di genotipizzazione. Se vengono scoperte duplicazioni, esaminarle e correggerle prima di andare avanti.
- Compila la colonna "isolation_box_number" del foglio di genotipizzazione per tutte le etichette S.
6. Verificare la proliferazione sulle piastre S
- Verificare la presenza di animali proliferanti su piastre S 48 ore dopo l'isolamento (utilizzare la data e l'ora dell'ultimo isolamento sulla scatola dal passaggio 4.11 per guidare i tempi).
NOTA: i nematodi proliferanti sono caratterizzati da prole sulla piastra S. - Se una piastra S sta proliferando, inserisci "1" nella colonna proliferation_48 sul foglio di genotipizzazione, quindi sposta la piastra S in una casella etichettata "Proliferazione di 48 ore, casella 1". Posizionare un massimo di 88 piastre S in una scatola di proliferazione, quindi iniziare a riempire una nuova scatola con l'etichetta "48 h proliferation, box 2". Assicurarsi che le etichette S siano organizzate in ordine alfanumerico nelle caselle di proliferazione da 48 ore.
NOTA: Non smaltire le piastre S non proliferanti; queste piastre saranno nuovamente controllate a 168 ore dopo l'isolamento. Se lo si desidera, consolidare queste piastre S in ordine numerico in caselle etichettate "48 h non proliferanti, casella X", ma ricordarsi di registrare quando deve verificarsi il controllo di 168 ore sulla nuova casella. - Dopo aver identificato tutte le etichette S proliferanti a 48 h, passare alla sezione 7 per le piastre S con proliferazione a 48 h.
- Controllare le piastre S che non proliferavano a 48 ore dopo l'isolamento a 168 ore dopo l'isolamento.
- Se una piastra S sta proliferando, inserisci "1" nella colonna proliferation_168 sul foglio di genotipizzazione e quindi sposta la piastra S in una casella etichettata "Proliferazione di 168 ore, casella 1".
- Posiziona un massimo di 88 piastre S in una scatola di proliferazione, quindi inizia a riempire una nuova scatola con l'etichetta "Proliferazione di 168 ore, scatola 2". Assicurati di organizzare le etichette S in ordine alfanumerico nelle scatole di proliferazione da 168 ore.
- Scartare le piastre S che non hanno proliferazione dopo 168 ore. Passare alla sezione 7 per le piastre S con proliferazione a 168 h.
7. Lisi delle linee isofeme
NOTA: questo passaggio utilizzerà lo strumento di filtro dei dati nei fogli Google per aiutare a stampare i fogli di lavoro di lisi per le piastre S nelle caselle di proliferazione. Lo scopo dei fogli di lavoro di lisi è quello di fornire al personale le posizioni corrette per le etichette S nei tubi a strisce di lisi al banco.
- Aprire il foglio di genotipizzazione per il progetto desiderato e selezionare tutte le celle digitando Cmd+A. Fare clic su Data > Crea un filtro per aggiungere un pulsante filtro a ciascuna intestazione di colonna. Utilizzare i pulsanti Filtro per visualizzare solo le piastre S che verranno genotipizzate. Ad esempio, se tutte le piastre S con proliferazione a 48 h devono essere lisate: fare clic sul pulsante Filtro nella colonna "proliferation_48" e selezionare "1".
- Una volta filtrato il foglio google di genotipizzazione, rivedi l'elenco delle etichette S visualizzate per assicurarti che siano le etichette S da stampare sul foglio di lavoro.
- Nella colonna "strip_tube_number" del foglio google di genotipizzazione, inserisci un numero univoco ogni 11 righe.
- Immettere i numeri del tubo di striscia per un progetto in ordine successivo a partire da 1 e mai duplicati. Nella "strip_tube_position", inserisci da 2 a 12 per ogni numero di tubo di striscia.
NOTA: utilizzare tubi a strisce a 12 tubi per la lisi. La prima posizione (strip_tube_position 1) sarà il controllo, ma i controlli non vengono aggiunti ai fogli di lavoro di lisi (vengono aggiunti solo i strip_tube_positions, 2-12). Al momento della lisi, il ceppo di controllo positivo 'N2' verrà aggiunto alla posizione 1 di ogni tubo a strisce pari come controllo positivo. Nessun verme viene aggiunto alla posizione 1 di ogni tubo di striscia dispari come controllo negativo.
- Filtra ulteriormente il foglio google di genotipizzazione per includere solo le etichette S in una casella di proliferazione che devono essere lisate, quindi seleziona le colonne da "s_label" a "lysis_notes". Stampare un foglio di lavoro di lisi per ogni scatola di proliferazione da lisare.
- Fare clic sul menu a discesa nel campo Stampa e selezionare Celle selezionate. Fare clic su Avanti in alto a destra, quindi utilizzare la finestra di dialogo per stampare il foglio di lavoro di lisi per la casella di proliferazione.
- Ripetere i passaggi 7.3-7.5 per stampare un foglio di lavoro di lisi per ogni scatola di proliferazione.
NOTA: ogni scatola di proliferazione contiene fino a 88 piastre S, che corrispondono a otto tubi a strisce a 12 pozzetti. - Preparare tubi a 12 pozzetti per tutti i campioni che verranno lisati. Etichettare un tubo di striscia con un "strip_tube_number" univoco assegnato nel foglio di lavoro di lisi. Questa etichetta deve essere scritta sulla striscia del cappuccio e sul tubo della striscia per evitare confusione se sono separati. I tubi a striscia EVEN hanno un controllo positivo (vermi N2) in posizione 1. I tubi a striscia ODD hanno un controllo negativo (senza vermi) in posizione 1.
- Preparare un tampone di lisi sufficiente (100 mM KCl, 20 mM Tris pH 8,2, 5 mM MgCl2, 0,9% IGEPAL, 0,9% Tween 20, gelatina allo 0,02% con proteinasi K aggiunta a una concentrazione finale di 0,4 mg/mL) per tutti i campioni e aggiungere il 5% in più per errore della pipetta. Scalare secondo necessità.
NOTA: Il tampone di lisi si prepara al meglio combinando tutti gli ingredienti ad eccezione della proteinasi K e congelando in aliquote da 10-50 ml a -20 °C. Scongelare aliquote e mantenere a 4 °C prima dell'uso; immediatamente prima dell'uso, aggiungere la proteinasi K e mescolare accuratamente. Mantenere il tampone di lisi sul ghiaccio durante il lavoro. - Disporre le piastre S per un particolare tubo di striscia in ordine utilizzando il foglio di lavoro di lisi stampato come guida.
- Scomporre un tubo di striscia e aggiungere 8 μL di tampone di lisi a ciascun tappo con un pipettor ripetuto. Aggiungere il tampone di lisi a una striscia di tappi alla volta perché il tampone di lisi evaporerà se lasciato a RT e scoperto. Prelevare 3-5 animali dalle piastre di origine (piastra S o piastra di riserva N2 per controlli positivi) nelle posizioni appropriate del cappuccio indicate sul foglio di lavoro di lisi. Registrare le note per qualsiasi piastra S con meno di 5 vermi raccolti per la lisi nella sezione lysis_notes del foglio di lavoro di lisi.
- Dopo aver caricato i nematodi in ogni posizione del tubo della striscia, riposizionare la striscia del cappuccio sul tubo della striscia. Abbinare il cappuccio contrassegnato (posizione 1) con il tubo contrassegnato (posizione 1). Una volta tappato, centrifugare brevemente il tubo della striscia fino a quando i nematodi si trovano nella parte inferiore del tubo.
- Mettere la striscia nel congelatore a -80 °C fino a completa congelamento (almeno 10 min). Ripetere i passaggi da 7,9 a 7,11 fino a quando tutte le strisce non hanno nematodi aggiunti per la lisi. Organizzare le strisce tubolari in ordine numerico.
- Rimuovere i set di tubi a striscia ed eseguire il programma di lisi in un termociclatore: 1 h a 60 °C, 15 min a 95 °C, tenere a 12 °C. Al termine del programma di lisi, girare i campioni a 300 x g per 15 s a RT e conservare i lisati a -80 °C per un massimo di 1 settimana.
- Organizza le strisce di tubo in ordine numerico utilizzando i supporti per piastre a 96 pozzetti e includi un'etichetta con un numero di scatola di proliferazione, un intervallo di numeri di tubi di striscia, una data e le iniziali del ricercatore. Aggiornare le colonne del foglio di genotipizzazione 'lysis_date' e 'lysis_notes' con le informazioni del foglio di lavoro di lisi.
8. PCR di sequenze SSU e ITS2
NOTA: questa sezione fornirà istruzioni su come eseguire due PCR separate per ogni piastra S lisata. Il primo set di primer amplifica un frammento di 500 bp del gene delle piccole subunità (SSU) 18S rDNA); oECA1271 = primer forward TACAATGGAAGGCAGCAGGC, oECA1272 = primer inverso CCTCTGACTTTCGTTCTTGATTAA 12. Questa PCR viene utilizzata per verificare la qualità del DNA del modello. La PCR amplifica la regione SSU per quasi tutte le specie di nematodi. Se la SSU PCR non riesce ad amplificare, questo risultato suggerisce che la qualità della lisi è scarsa e la lisi deve essere ripetuta per questa piastra S. Il secondo set di primer amplifica un frammento di 2.000 bp della regione interna del distanziatore trascritto tra i geni rDNA 5.8S e 28S (ITS2); oECA1687 = primer in avanti CTGCGTTACTTACCACGAATTGCARAC, oECA202 = primer inverso GCGGTATTTGCTACTACCAYYAMGATCTGC3. Il prodotto ITS2 PCR è sequenziato sanger e la sequenza viene utilizzata per identificare i nematodi nel genere Caenorhabditis a livello di specie per somiglianza di sequenza.
- Utilizzare lo strumento di filtraggio nel foglio di genotipizzazione per visualizzare solo le etichette S da utilizzare per la PCR.
- Aggiornare le colonne pcr_plate_number e pcr_well nel foglio di genotipizzazione. Per prevenire la degradazione del materiale di lisi, le PCR SSU e ITS2 vengono eseguite contemporaneamente.
- Utilizzare lo stesso pcr_plate_number per le PCR ITS2 e SSU anche se si tratta di reazioni separate in piastre separate. Saranno distinti con etichette "SSU" o "ITS2".
- Assegnare un pcr_plate_number a otto o meno tubi strip (un tubo strip per fila della piastra PCR a 96 pozzetti, disposti in ordine crescente, ad esempio il numero più basso di strip tube in cima). Quindi assegnare un pcr_plate_well a ciascuna etichetta S nei tubi della striscia.
NOTA: i tubi strip sono disposti in ordine crescente, con il numero di strip tube più basso assegnato alla riga A e il numero più alto nella riga H. La posizione 1 di tutti i strip tube è assegnata alla colonna 1. Pertanto, il tubo di striscia numero 1, posizione 1 sarà assegnato alla targa PCR numero 1, bene A01. - Etichettare le piastre PCR a 96 pozzetti per ospitare i campioni che verranno utilizzati per la PCR. Etichettare ogni piastra PCR con le seguenti informazioni: nome del progetto, tipo di PCR, numero di targa PCR e data della PCR (ad esempio, 2020FebruaryAustralia_SSU_1_20200304). Inoltre, etichettare la piastra con i numeri del tubo di striscia che verranno caricati in ogni riga.
- Rimuovere il materiale di lisi dal congelatore a -80 °C e scongelare i tubi di striscia contenenti il materiale di lisi sul ghiaccio. Mentre il materiale di lisi si sta scongelando, preparare le miscele master ITS2 e SSU in tubi separati su ghiaccio. Le ricette SSU e ITS2 PCR si trovano nella Tabella supplementare 2.
NOTA: Preparare 100 reazioni di miscela master PCR per ogni piastra a 96 pozzetti per consentire l'errore di pipettaggio. Utilizzare un conico da 15 ml o 50 ml per contenere la miscela master se si devono utilizzare grandi volumi. - Ruotare delicatamente la miscela principale fino a quando Taq non è distribuito in tutto il mix. Una volta miscelato, aliquota 38 μL della miscela master nei pozzetti appropriati delle piastre PCR su ghiaccio. Utilizzare abbeveratoi sterili monouso con fondo a V e una pipetta multicanale a 12 pozzetti per trasferire la miscela master alle piastre PCR.
- Ruotare verso il basso i tubi della striscia di lisi scongelati per rimuovere il materiale di lisi dai tappi. Rimuovere con attenzione i coperchi di tutti i tubi di striscia che verranno caricati nella prima piastra PCR. Utilizzare una pipetta multicanale a basso volume (a 12 pozzetti o 8 pozzetti) per aggiungere 2 μL di lisato al pozzo appropriato nella piastra PCR. Pipettare delicatamente il lisato su e giù una volta prima di rimuovere i 2 μL.
NOTA: Controllare le punte per assicurarsi che contengano la lisi prima del trasferimento. Ricorda di cambiare i suggerimenti tra righe o colonne. - Coprire la piastra PCR con un foglio adesivo PCR e utilizzare un rullo per creare una tenuta ermetica. Dopo aver applicato il foglio, ruotare brevemente le piastre PCR in una centrifuga. Tenere la piastra sul ghiaccio fino a quando non è pronta per funzionare nel termociclatore.
- Eseguire le PCR con il programma termociclatore appropriato. Fare riferimento alla Tabella supplementare 2 per i dettagli dei programmi SSU e ITS2 PCR.
- Ripetere i passaggi da 8.4 a 8.8 fino a quando non vengono eseguite tutte le PCR.
- Mentre le reazioni PCR sono in esecuzione, versare un gel di agarosio all'1,5% da 100 ml. Ogni gel conterrà campioni o una singola piastra PCR.
- Aggiungere 1,5 g di agarosio in un matraccio da 500 mL, quindi aggiungere 100 mL di 1 tampone TAE (Tabella supplementare 3) e ruotare per mescolare. Microonde per sciogliere e raffreddare il gel.
- Una volta raffreddata la soluzione, aggiungere 5 μL di 10 mg/mL di soluzione di bromuro di etidio e mescolare per combinare. Versare la soluzione in un vassoio di colata con quattro pettini a 25 pozzetti in modo che il gel possa ospitare 96 campioni più una scala per ogni fila nel gel.
NOTA: Il bromuro di etidio è un potente mutageno. Quando si maneggia il bromuro di etidio, utilizzare un cappotto da laboratorio, guanti resistenti alle sostanze chimiche e occhiali di sicurezza chimica.
- Poco prima che la PCR sia terminata, aggiungere un colorante di carico 6x a un trogolo monouso e utilizzare una pipetta multicanale per aggiungere 2 μL di colorante di carico 6x a ciascun pozzetto di una nuova piastra PCR a 96 pozzetti. Questa piastra verrà utilizzata per caricare i campioni nel gel. Fai abbastanza di queste piastre per ospitare tutti i campioni.
- Al termine delle PCR, rimuovere le piastre PCR e centrifugarle brevemente a 300 x g per 15 s a RT. Conservare le piastre PCR su ghiaccio fino a quando i prodotti PCR possono essere esauriti su un gel.
- Per eseguire i prodotti su un gel, utilizzare una pipetta multicanale a 12 pozzetti per aggiungere 5 μL di ciascun campione al pozzetto appropriato di una piastra a 96 pozzetti contenente 2 μL di colorante di carico 6x.
- Quindi caricare 6 μL di questa miscela in ciascun pozzetto di un gel recentemente fuso. Caricare 6 μL di 1 KB più scala nel primo pozzetto di ogni fila del gel.
NOTA: Per riempire i pozzetti del gel, potrebbe essere necessario intervallare la riga A e la riga B dalla piastra PCR nella prima fila del gel. Per evitare confusione, registrare il gel_number e gel_position nel foglio di genotipizzazione per ciascun campione PCR.
- Posizionare un nuovo coperchio di alluminio sulla PCR rimanente nella piastra o nelle piastre e conservarle a 4 °C. Questi prodotti di reazione saranno utilizzati per il sequenziamento nella fase 9.
- Eseguire i prodotti PCR sul gel a 120 V per 20 minuti. Immagina il gel e registra quali etichette S producono prodotti ITS2 e/o SSU PCR nelle colonne "pcr_product_its2" e "prc_product_ssu" del foglio di genotipizzazione. Segna la presenza di una band con un '1'; contrassegnare uno '0' per nessuna band.
9. Identificazione dei nematodi con il sequenziamento Sanger e la sequenza BLAST
NOTA: questa sezione fornisce istruzioni per sequenziare gli ampliconi ITS2 dalle etichette S, allineare tali sequenze al database del National Center for Biotechnology Information (NCBI) utilizzando l'algoritmo BLAST e analizzare i risultati BLAST per identificare i nematodi sulle piastre S.
- Per ogni campione ITS2-positivo, utilizzare il restante prodotto ITS2 PCR per il sequenziamento Sanger utilizzando il primer forward oECA306 (CACTTTCAAGCAACCCGAC). Fai in modo che i file di output di sequenziamento siano facilmente collegati a un'etichetta S registrando le colonne "sequencing_plate" e "sequencing_well" di ciascuna etichetta S nel foglio di genotipizzazione.
- Ottenere i file di output .seq per ogni etichetta S dalla piattaforma di sequenziazione. Disporre i file con estensione seq per un progetto in un'unica directory con file con estensione seq per ogni batch di sequenziazione che si trova nelle sottodirectory.
- Aprire lo strumento di interfaccia della riga di comando e passare alla directory superiore contenente i file .seq immettendo il comando: cd . Se non esiste già, creare un FASTA unito per tutti i file .seq inserendo il seguente comando: for dir in */; do cd $dir; for file in *.seq; do echo ">"$file; cat $file; done >>.. /all_seqs.fa; cd ..; fatto.
NOTA: questo codice creerà un file FASTA unito denominato 'all_seqs.fa' da tutti i file .seq nella directory del progetto. Questo file può essere utilizzato nello strumento nucleotide blast online NCBI per allineare rapidamente la sequenza ITS2 di ciascuna etichetta S al database di sequenze ncbi. - In un browser Web, accedere al sito Web NBCI BLAST20 e fare clic sul pulsante Scegli file . Selezionare il file all_seqs.fa appena creato, quindi fare clic sul pulsante Sequenze leggermente simili (BLASTn). Fare clic sul pulsante BLAST nella parte inferiore della pagina per iniziare la ricerca BLAST.
- Aggiornare il foglio di genotipizzazione con i risultati BLAST per ogni S-label. Utilizza lo strumento filtro per semplificare l'aggiornamento del foglio google di genotipizzazione. Fare clic su Data > Crea un filtro per aggiungere un pulsante filtro a ciascuna intestazione di colonna. Filtrare la colonna sequencing_plate per selezionare le piastre di sequenziamento da aggiornare con i risultati BLAST.
- Utilizzare il menu a discesa nella pagina dei risultati NCBI BLAST per controllare i risultati per ogni sequenza ITS2 S-plate (Figura 6).
- Verificare la presenza di eventuali riscontri BLAST. Un ID sequenza nel menu a discesa con prefisso * non ha colpi di esplosione. Per queste etichette S, inserisci "no hit " nella colonna manual_blast_notes del foglio di genotipizzazione.
- Verificare la presenza di una possibile nuova specie di Caenorhabditis . Fare clic sul collegamento in alto per visualizzare l'allineamento (Figura 6). Se il top hit è (1) una specie di Caenorhabditis , (2) l'allineamento contiene più di cinque discrepanze al centro della sequenza e (3) la copertura della query è maggiore del 50%, questo risultato suggerisce che l'isolato potrebbe essere una nuova specie di Caenorhabditis (Figura 7). Per queste piastre S inserire, la specie del BLAST superiore colpito nella colonna 'species_id', inserire un 1 nella 'colonna possible_new_caeno_sp' e 'possibile nuovo Caeno sp.' nella colonna 'manual_blast_notes' insieme all'identità percentuale, (ad esempio, 'possibile nuova identità Caeno sp. 89%').
- Per le sequenze di piastre S che BLAST a una specie di Caenorhabditis , inserisci il genere completo e il nome della specie del BLAST superiore colpito nella colonna "species_id". Ad esempio, "Caenorhabditis elegans".
- Per le sequenze di piastre S che BLAST a una specie non Caenorhabditis , inserisci solo il genere del BLAST superiore colpito seguito da "sp." nella colonna "species_id". Questa notazione significa che l'isolato è una specie sconosciuta all'interno del genere denominato. Ad esempio, 'Oscheius sp.'.
NOTA: La sequenza ITS2 non può essere utilizzata per identificare in modo affidabile gli isolati a livello di specie al di fuori del genere Caenorhabditis3,13. - Immettere 1 nella colonna "make_strain_name" del foglio di genotipizzazione se "species_id" = "Caenorhabditis elegans", "Caenorhabditis briggsae" o "Caenorhabditis tropicalis", OPPURE "possible_new_caeno_sp" = 1.
- Denominare i ceppi con nomi univoci seguendo le convenzioni di nomenclatura di Caenorhabditis , ovvero una designazione di laboratorio unica composta da 2-3 lettere maiuscole seguite da un numero per ogni ceppo univoco23. Immettere i nomi dei ceppi nella colonna "strain_name".
- Dopo che i ceppi sono stati nominati, possono essere crioconservati utilizzando i protocolli stabiliti 24.
10. Elaborazione dei dati di raccolta con il pacchetto easyFulcrum in R
NOTA: questo passaggio descrive come collegare i dati di raccolta (etichette C) e i dati di isolamento dei nematodi (etichette S) utilizzando il pacchetto easyFulcrum R. Il software contiene funzioni che uniranno ulteriormente i dati Fulcrum con i dati di genotipizzazione dal foglio di genotipizzazione in modo che le identità delle specie S-label e i nomi dei ceppi siano organizzati in un unico frame di dati.
- Creare una nuova directory denominata per il progetto di raccolta. Disporre la struttura delle cartelle all'interno della directory in modo che corrisponda ai requisiti descritti nel pacchetto R easyFulcrum15.
- Vai al sito Web Fulcrum e accedi. Esporta i dati grezzi del progetto dal database Fulcrum utilizzando lo strumento di esportazione dei dati del sito Web Fulcrum a sinistra e selezionando le seguenti caselle di controllo: progetto, includi foto, includi dati GPS, campionamento del campo e isolamento.
NOTA: i dati Fulcrum per il progetto verranno esportati come cinque file con valori delimitati da virgole (.csv). I dati completi del progetto saranno uniti in un unico frame di dati utilizzando il pacchetto easyFulcrum in R. - Spostare i cinque file .csv esportati da Fulcrum nella directory del progetto creata nel passaggio 10.1 come indicato nella vignetta easyFulcrum21.
- Apri una sessione Rstudio e installa il pacchetto easyfulcrum in R inserendo i seguenti comandi nella console R 'install.packages("devtools")' e 'devtools::install_github("AndersenLab/easyfulcrum")'.
- Aprire un nuovo script R e seguire le istruzioni nella vignetta easyfulcrum per elaborare i dati di raccolta21.
Risultati
Questo protocollo è stato utilizzato per raccogliere i nematodi Caenorhabditis da più località, tra cui Hawaii e California. Il tasso di successo dell'isolamento per i nematodi Caenorhabditis varia in base alla posizione di raccolta, al clima, all'esperienza di campionamento e ai tipi di substrato campionati. Il protocollo è stato utilizzato per campionare ampiamente le isole Hawaii, dove nove progetti di raccolta sono stati condotti in più anni e stagioni. I tassi di successo dell'isolamento per le specie di Caenorhabditis auto-antagoniste sono quasi identici per C. briggsae (162 su 4.506 campioni, 3,6%) e C. elegans (163 su 4.506 campioni, 3,6%) e molto più bassi per C. tropicalis (26 su 4.506 campioni, 0,58%)8. Ciascuna delle specie di selfing è arricchita su substrati di frutta e fiori in decomposizione rispetto alle altre categorie di substrato. Campionare i substrati di frutta e fiori in decomposizione se il ricercatore sta tentando di massimizzare il tasso di successo piuttosto che caratterizzare le preferenze del substrato. Tuttavia, il tasso di successo varia in base alla qualità del substrato selezionato. Ad esempio, tra i substrati di frutta e fiori, quei substrati che sono troppo secchi, umidi o freschi probabilmente non produrranno nematodi caenorhabditis .
La scalabilità di questo protocollo di raccolta è evidente dal numero di collezioni che una singola coppia di ricercatori può raccogliere in natura. Ad esempio, nell'ottobre del 2018, una coppia di ricercatori che utilizzano questo protocollo di raccolta è stata in grado di raccogliere un totale di oltre 1.000 campioni in 7 giorni da più località su due isole hawaiane. Questo team sul campo ha spedito i campioni durante la notte al laboratorio, dove un team di otto ricercatori ha isolato oltre 2.000 nematodi dai campioni al loro arrivo. Un vantaggio chiave di questo protocollo è che riduce al minimo i costi associati al campionamento in località remote riducendo le attrezzature e il personale necessari sul campo. Utilizzando questo protocollo, un piccolo team sul campo può concentrarsi sul campionamento mentre il team di isolamento può elaborare i campioni presso la propria istituzione di origine utilizzando attrezzature fragili e pesanti come microscopi di dissezione e piastre di agar per isolare i nematodi. Inoltre, l'implementazione dell'applicazione mobile per la raccolta dei dati consente di collegare tutti i dati sul campo associati ai campioni direttamente all'etichetta C, il che consente al team di isolamento di lavorare indipendentemente dal team sul campo durante l'elaborazione dei campioni.
I ricercatori che utilizzano questo protocollo di raccolta devono considerare lo sforzo necessario per isolare i nematodi prima di un progetto di raccolta. Le fasi di isolamento e identificazione sono limitanti e un piccolo team di raccolta può rapidamente sovraccaricare gli isolatori con i campioni. Inoltre, lo spazio di laboratorio necessario per elaborare molte collezioni può interferire con la ricerca in corso (Figura 3). Inoltre, alcuni nematodi isolati richiedono uno sforzo aggiuntivo per il genotipo. Ad esempio, circa il 2% degli isolati non riesce ad amplificarsi con il set di primer SSU PCR dopo il primo tentativo di lisi e deve essere ri-lisato per garantire che il materiale di lisi sia adatto per l'amplificazione con il set di primer ITS2 (Figura 8). Inoltre, circa il 3% degli isolati non riesce a produrre sequenze di qualità dopo un ciclo iniziale di sequenziamento Sanger. Per questi isolati, è spesso necessario un altro ciclo di lisi, ITS2 PCR e sequenziamento Sanger, che può aumentare il tempo di consegna per il team di isolamento. È importante sottolineare che l'identità di sequenza da sola non è una prova sufficiente per giustificare una nuova specie di Caenorhabditis (Figura 7). Per giustificare adeguatamente l'allevamento di un isolato come nuova specie di Caenorhabditis, è necessario compiere ulteriori sforzi per eseguire esperimenti di accoppiamento e stabilire un campione tipizzato13. È anche preferibile, ma non richiesta, una descrizione morfologica formale del campione tipizzato3. Insieme, queste considerazioni suggeriscono che i ricercatori che adottano questo protocollo di raccolta trarranno beneficio dai test di prova delle fasi di isolamento e identificazione per garantire che le risorse siano allocate correttamente prima dell'inizio di un progetto di raccolta. È importante sottolineare che anche i piccoli progetti di raccolta possono trarre vantaggio da questo protocollo perché il processo è altamente riproducibile e i dati possono essere facilmente controllati a fini di controllo della qualità tra i gruppi di laboratorio.

Figura 1: Esempi di substrato. (A) Un frutto ideale in decomposizione è mostrato al centro dell'immagine (1), il frutto è quasi irriconoscibile. Meno frutta marcita è mostrata nelle vicinanze; evitare di assaggiare i frutti appena caduti (2). (B) Un fiore idealmente decomposto è mostrato in alto (3). Evitare di assaggiare fiori appena caduti (4). (C) La lettiera di foglie scure sotto lo strato superiore di foglie secche è ideale quando si campiona per auto-nematodi Caenorhabditis (5). Evitare di campionare la lettiera di foglie secche (6). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: L'applicazione mobile Nematode Field Sampling. (A) La schermata iniziale dopo l'apertura dell'applicazione Nematode Field Sampling su un dispositivo Apple in Fulcrum. La freccia rossa in basso a destra punta al pulsante + utilizzato per creare un nuovo record di raccolta. (B) Un esempio di un nuovo record di raccolta mostrato su un dispositivo Apple. La freccia rossa punta al campo "Progetto" nella parte superiore della schermata di registrazione della raccolta. Assicurarsi di selezionare il progetto corretto durante il campionamento sul campo. Per impostazione predefinita, il campo del progetto verrà utilizzato per l'ultimo progetto durante la creazione dei record di raccolta successivi. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Sacchetti di raccolta e piatti di raccolta organizzati prima della placcatura dei campioni. Questa figura mostra i campioni in sacchetti di raccolta con etichetta C a sinistra. Ogni borsa da collezione ha una targhetta da 10 cm con etichetta C abbinata. Sulla destra ci sono lastre di raccolta di 10 cm che contengono materiale campione dopo che è stato trasferito dai sacchetti di raccolta. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Una piastra di raccolta (piastra C) con un campione correttamente trasferito. Una piastra a C da 10 cm con frutta in decomposizione posta sul bordo del prato batterico. L'etichetta C è attaccata al coperchio della piastra. Fare clic qui per visualizzare una versione più grande di questa figura.
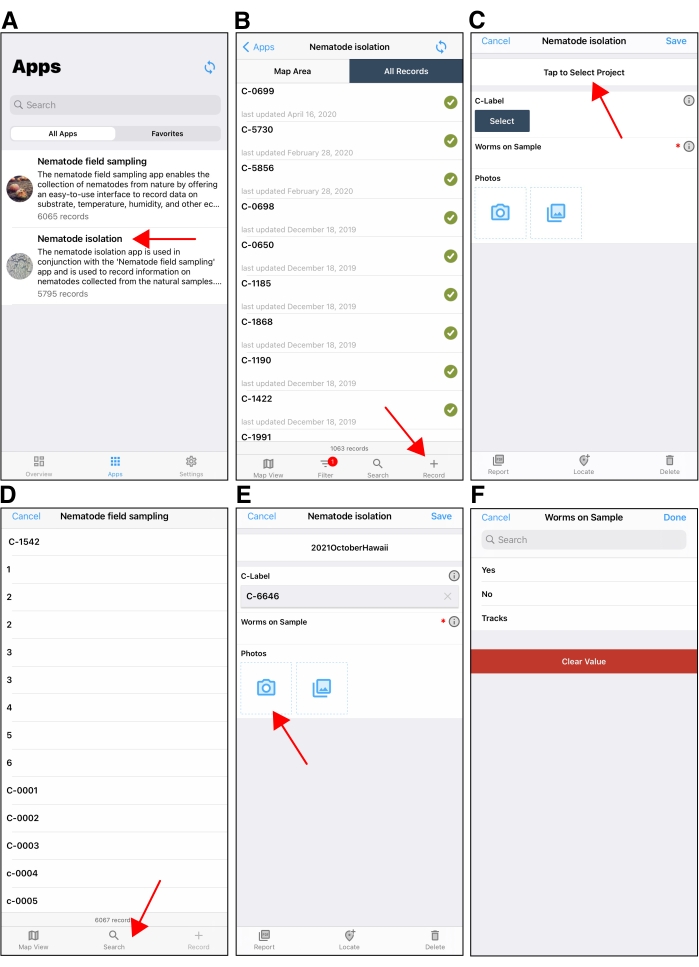
Figura 5: L'applicazione mobile per l'isolamento dei nematodi. (A) Schermata di selezione dell'applicazione nell'applicazione mobile Fulcrum. La freccia rossa punta all'applicazione Nematode Isolation . (B) La schermata iniziale dopo l'apertura dell'applicazione Nematode Isolation su un dispositivo Apple in Fulcrum. La freccia rossa in basso a destra punta al pulsante + utilizzato per creare un nuovo record di isolamento. (C) Un esempio di un nuovo record di isolamento mostrato su un dispositivo Apple. La freccia rossa punta al campo "Progetto" nella parte superiore della schermata del record di isolamento. Assicurarsi di selezionare il progetto corretto durante l'isolamento. Per impostazione predefinita, il campo del progetto verrà utilizzato per l'ultimo progetto durante la creazione di record di isolamento successivi. (D) Dopo aver toccato il campo Seleziona sotto C-label, gli utenti toccheranno il pulsante di ricerca (freccia rossa) per trovare l'etichetta C da cui stanno isolando i nematodi. (E) Dopo aver selezionato l'etichetta C, gli utenti fotograferanno la piastra C utilizzando la fotocamera del dispositivo. (F) Gli utenti quindi immettono se ci sono nematodi sulla piastra C o meno. Le etichette S vengono aggiunte al record di isolamento se ci sono nematodi da isolare. Fare clic qui per visualizzare una versione più grande di questa figura.
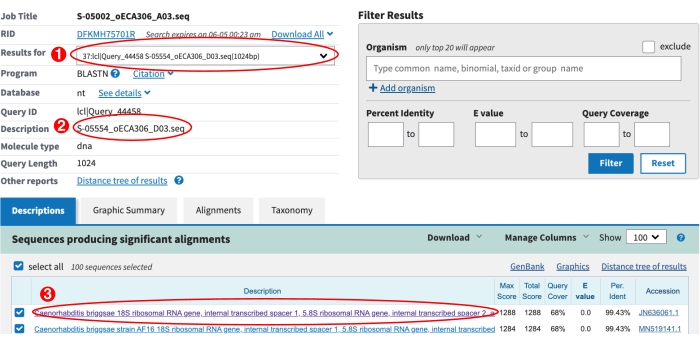
Figura 6: Pagina dei risultati di NCBI BLAST. (1) Il menu a discesa utilizzato per visualizzare i risultati BLAST per tutte le sequenze. (2) La descrizione della sequenza corrente selezionata dal menu a discesa. In questo caso vengono mostrati i risultati per S-label S-05554. (3) Viene mostrato il colpo blast più alto per S-05554. Il testo viola indica che il collegamento per visualizzare questo allineamento è stato cliccato. Assicurati di ispezionare gli allineamenti a occhio per identificare possibili nuove specie di Caenorhabditis , vedi il passaggio 9.8 sopra. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 7: Esempi di visualizzazione dell'allineamento NCBI BLAST. (A) Esempio di sequenza di query ITS2 di un isolato allineata a una sequenza di soggetti di C. kamaaina . (1) L'identità percentuale dell'allineamento (89%), che è bassa per un colpo BLAST superiore. (2) Una mancata corrispondenza tra la query e la sequenza dell'oggetto (da G ad A). (3) Un gap di quattro coppie di basi nella sequenza del soggetto fatto dall'algoritmo di allineamento; lacune nella query o nell'oggetto indicano uno scarso allineamento. (4) Una regione generalizzata al centro dell'allineamento con molti disallineamenti e lacune. Una regione come questa suggerisce che la sequenza di query potrebbe provenire da una nuova specie di Caenorhabditis . Viene mostrato un esempio di allineamento reale di una nuova specie, C. oiwi, scoperta nel 2017. (B) Un esempio di un buon allineamento tra la sequenza di query ITS2 di un isolato e una sequenza di soggetti. (5) L'identità percentuale dell'allineamento (99%), che di solito significa che la sequenza di query proviene da un isolato della stessa specie del soggetto. (6) Una regione centrale dell'allineamento con identità perfetta. Una regione come questa suggerisce che l'isolato di query è probabilmente la stessa specie del soggetto. Fare clic qui per visualizzare una versione più grande di questa figura.
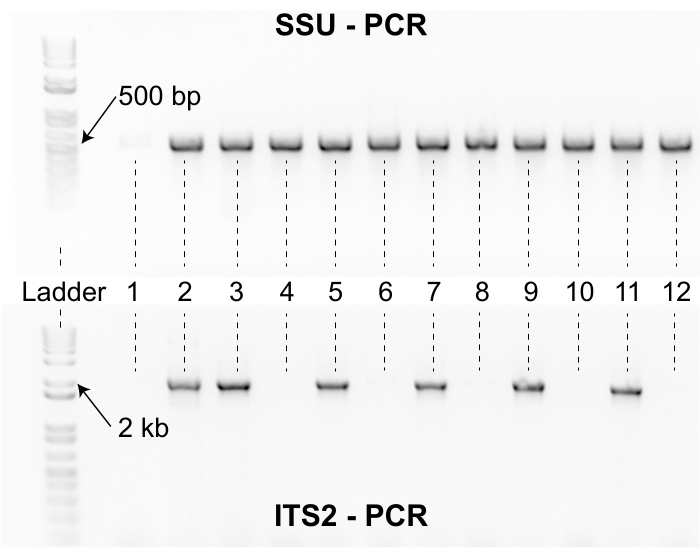
Figura 8: Prodotti SSU e ITS2 PCR. Il gel superiore mostra i prodotti PCR generati con il set di primer SSU per 12 campioni rappresentativi. Una scala del DNA è inclusa a sinistra come riferimento. I prodotti SSU PCR per i nematodi Caenorhabditis hanno una lunghezza di circa 500 bp. I campioni 2-12 amplificati con il set di primer SSU, ma il campione uno no. L'assenza di un amplicon SSU da 500 bp per il campione uno suggerisce che il materiale di lisi era di scarsa qualità e il campione deve essere ri-lisato. Il gel inferiore mostra i prodotti PCR generati con il set di primer ITS2 per gli stessi 12 campioni mostrati nel gel superiore. La scala e i campioni sono nello stesso orientamento per entrambi i gel. Sei dei 12 campioni non si sono amplificati con il set di primer ITS2. I campioni con bande SSU e ITS2 sono sequenziati da Sanger e identificati per somiglianza di sequenza utilizzando l'algoritmo NCBI BLAST. Fare clic qui per visualizzare una versione più grande di questa figura.
File supplementare 1: etichette C. Un file PDF contenente 2500 etichette C univoche. Fare clic qui per scaricare questo file.
File supplementare 2: S-etichette. Un file PDF contenente 5000 etichette S univoche. Fare clic qui per scaricare questo file.
Tabella supplementare 1: Materiali di campo. Una lista di imballaggio dei materiali utilizzati sul campo per campionare i nematodi. Fare clic qui per scaricare questa tabella.
Tabella supplementare 2: ricette PCR e condizioni del termociclatore. Una tabella delle ricette PCR e delle condizioni del termociclatore per le PCR ITS2 e SSU. Fare clic qui per scaricare questa tabella.
Tabella supplementare 3: Ricette tampone per elettroforesi. Una ricetta per 0,5 M pH 8,0 Soluzione di acido etilendiamminotetraacetico (EDTA) e la soluzione tampone TRIS-acetato-EDTA (TAE). Fare clic qui per scaricare questa tabella.
Discussione
Questo protocollo contiene passaggi critici che devono essere eseguiti con cautela. Ad esempio, è importante che i team sul campo e di isolamento siano attenti a selezionare il progetto di raccolta corretto nell'applicazione prima di raccogliere campioni dal campo o isolare i nematodi dai campioni in laboratorio. Nel caso in cui venga selezionato il progetto di raccolta errato, i record di dati errati vengono corretti al meglio nel database Fulcrum utilizzando gli strumenti di modifica dei record online. Questo processo può essere noioso per molti record fuori posto. Tuttavia, il database conserva tutte le modifiche apportate ai record in modo che sia possibile un controllo completo dei record di raccolta e isolamento. Gli altri passaggi critici in questo protocollo riguardano la gestione di campioni dal campo e i nematodi isolati da quei campioni. Per garantire che i nematodi Caenorhabditis sopravvivano alle fasi di campionamento e spedizione, la temperatura dei campioni deve essere mantenuta tra 4 °C e 25 °C. Temperature superiori a 25 °C possono causare sterilità in C. elegans14. Assicurarsi che i campioni vengano trasferiti dai sacchetti di raccolta alle piastre di raccolta entro cinque giorni, ove possibile, per ridurre al minimo la perdita di nematodi. Dopo che i nematodi sono stati isolati, è fondamentale che siano genotipizzati e crioconservati prima che periscano. È difficile trovare nematodi viventi su piastre S che hanno più di due o tre settimane perché la contaminazione fungina e batterica può rendere le piastre S inospitali.
Questo protocollo può essere facilmente modificato per adattarsi a diversi tipi di dati che i ricercatori potrebbero voler raccogliere mentre sono sul campo. Ad esempio, è facile personalizzare l'applicazione "Nematode field sampling" con nuovi campi di immissione dati utilizzando la GUI online di Fulcrum per l'editing delle applicazioni. Inoltre, il pacchetto di analisi dei dati, easyFulcrum, può ospitare queste modifiche durante l'elaborazione dei nuovi dati15. Un'altra modifica che gli utenti potrebbero trovare interessante è quella di utilizzare un metodo di campionamento diverso sul campo. Piuttosto che campionare substrati discreti, i ricercatori potrebbero voler campionare aree più grandi contenenti più tipi di substrato. Questi campioni più grandi vengono elaborati al meglio in laboratorio utilizzando i metodi di estrazione a imbuto o vassoio di Baermann13. È importante sottolineare che l'uso di etichette C e S si applica ancora a queste tecniche e sono quindi compatibili con le applicazioni mobili.
Le limitazioni primarie di questo protocollo riguardano il tempo di manipolazione dei nematodi prima dell'isolamento in laboratorio. In primo luogo, il tempo di ritardo tra la raccolta del campione e l'isolamento dei nematodi rende impossibile registrare le fasi di sviluppo dei nematodi su un determinato campione al momento della raccolta. In secondo luogo, la frequenza dei maschi e l'outcrossing in natura sono questioni evolutive chiave per i nematodi di Caenorhabditis autoalimentati10. Questo metodo non è adatto per affrontare queste domande perché è probabile che i nematodi abbiano attraversato più generazioni prima dell'isolamento. L'isolamento ritardato significa che la prova diretta della frequenza maschile in natura è impossibile. Inoltre, il ritardo multigenerazionale durante le fasi di genotipizzazione significa che l'evidenza genomica dell'outcrossing (eterozigosi) sarà erosa prima che un ceppo di nematode possa essere sequenziato. Per identificare l'eterozigosi in natura, la prole prodotta da un nematode direttamente isolato dalla natura viene utilizzata per il sequenziamento2. Un'altra potenziale limitazione di questo protocollo è che è prevenuto verso l'identificazione della Caenorhabditis selfing. Questo perché i nematodi isolati delle specie autoprodotte hanno maggiori possibilità di proliferare rispetto agli outcrosser obbligati, che proliferano solo se una femmina fecondata viene isolata.
Questo metodo di raccolta si basa sui protocolli di raccolta esistenti13,14. Il principale progresso di questa tecnica è l'uso della tecnologia mobile e del software personalizzato per facilitare l'organizzazione di grandi quantità di dati ecologici e molecolari associati a progetti di raccolta su larga scala. I dati ecologici generati utilizzando questo protocollo di raccolta possono essere utilizzati per affrontare le questioni in sospeso per le popolazioni naturali di specie Caenorhabditis. Ad esempio, i dati generati con questo metodo sono stati utilizzati per scoprire preferenze di nicchia per la specie attraverso le isole Hawaii. Inoltre, sequenziando i genomi dei nematodi crioconservati, i ricercatori possono studiare come i modelli di variazione genetica sono correlati con i dati ecologici. Ricerche di questo tipo possono scoprire le firme dell'adattamento locale nelle popolazioni di Caenorhabditis e fornire importanti informazioni sulla rilevanza della variazione genetica nei contesti naturali8. Per ottenere una comprensione funzionale di molti geni nei nematodi Caenorhabditis, sono probabilmente necessari studi ecologici11. Anche per C. elegans una grande frazione di geni manca di annotazioni funzionali, nonostante sia il primo animale multicellulare ad essere sequenziato e uno degli animali più accuratamente studiati sulla Terra. Questo protocollo di raccolta è stato sviluppato per aiutare a colmare questa lacuna di conoscenza facilitando la raccolta di nematodi selvatici caenorhabditis e lo studio della loro ecologia e diversità genetica naturale.
Divulgazioni
Gli autori non segnalano conflitti di interesse.
Riconoscimenti
Questa ricerca è stata sostenuta da fondi di start-up della Northwestern University e da un National Science Foundation CAREER Award (IOS-1751035), entrambi concessi a E.C.A.
Materiali
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
Riferimenti
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon