Method Article
Ein hochgradig skalierbarer Ansatz zur Durchführung ökologischer Untersuchungen von selbstsüchtigen Caenorhabditis-Nematoden
In diesem Artikel
Zusammenfassung
Dieses Protokoll kann verwendet werden, um groß angelegte ökologische Untersuchungen von sich selbst lebenden Caenorhabditis-Nematoden durchzuführen. Der Hauptvorteil dieser Methode ist die effiziente Organisation und Analyse von ökologischen und molekularen Daten, die mit den aus der Natur gesammelten Nematoden verbunden sind.
Zusammenfassung
Caenorhabditis elegans ist einer der wichtigsten Modellorganismen in der Biologie, aber erst in jüngster Zeit haben sich Forscher auf seine natürliche Ökologie konzentriert. Die relative Seltenheit der Informationen über C. elegans in seinem natürlichen Kontext ergibt sich aus den Herausforderungen, die mit der Identifizierung des kleinen Fadenwurs in der Natur verbunden sind. Trotz dieser Herausforderungen hat ein zunehmender Fokus auf die Ökologie von C. elegans eine Fülle neuer Informationen über sein Leben außerhalb des Labors verursacht. Die intensivierte Suche nach C. elegans in der Natur hat zur Entdeckung vieler neuer Caenorhabditis-Arten beigetragen und gezeigt, dass kongenerische Nematoden häufig in freier Wildbahn koexistieren, wo sie sich von mikrobiellen Blüten ernähren, die mit verrottendem Pflanzenmaterial assoziiert sind. Die Identifizierung neuer Arten hat auch gezeigt, dass sich das androdiözische Paarungssystem von Männchen und selbstbefruchtenden Hermaphroditen innerhalb der Caenorhabditis dreimal unabhängig voneinander entwickelt hat. Die anderen beiden selbstsüchtigen Arten, C. briggsae und C. tropicalis, teilen die experimentellen Vorteile von C. elegans und haben vergleichende Studien über die mechanistischen Grundlagen wichtiger Merkmale, einschließlich der Selbstbefruchtung, ermöglicht. Trotz dieser Fortschritte bleibt noch viel über die Ökologie und natürliche Vielfalt dieser wichtigen Arten zu lernen. Zum Beispiel fehlen uns immer noch funktionelle Informationen für viele ihrer Gene, die nur durch ein Verständnis ihrer natürlichen Ökologie erreicht werden können. Um die ökologische Erforschung von selbstsüchtigen Caenorhabditis-Nematoden zu erleichtern, haben wir eine hochskalierbare Methode entwickelt, um Nematoden aus der Wildnis zu sammeln. Unsere Methode nutzt mobile Datenerfassungsplattformen, Cloud-basierte Datenbanken und die R-Softwareumgebung, um die Fähigkeit der Forscher zu verbessern, Nematoden aus der Wildnis zu sammeln, damit verbundene ökologische Daten aufzuzeichnen und wilde Nematoden mithilfe molekularer Barcodes zu identifizieren.
Einleitung
Die letzten zwei Jahrzehnte haben ein erhöhtes Interesse an der Ökologie der Caenorhabditis-Nematoden mit sich gebracht. Aus diesen Studien wissen wir, dass die frei lebenden Caenorhabditis-Arten aus kurzlebigen Mikrohabitaten sowohl in gemäßigten als auch in tropischen Regionen isoliert werden können, wo sie sich von mikrobiellen Blüten ernähren, die mit sich zersetzendem Pflanzenmaterial verbunden sind, manchmal in Sympatry1,2,3,4,5,6,7,8 . Wir haben auch gelernt, dass eine konvergente Evolution der Selbstbefruchtung in der Gattung dreimal stattgefunden hat, und Selfing ist die vorherrschende Fortpflanzungsform für C. briggsae, C. elegans und C. tropicalis9,10. Unter diesen Selbstlern ist C. elegans eines der am meisten untersuchten Tiere auf der Erde und wurde von Forschern verwendet, um kritische Fortschritte in der Biologie zu erzielen. Wichtig ist, dass die anderen selbstsüchtigen Caenorhabditis-Arten viele der experimentellen Vorteile von C. elegans teilen und die vergleichenden Studien in der Gattung schnell vorantreiben. Die kryptische Natur dieser Nematoden in freier Wildbahn macht es jedoch schwierig, ihre Ökologie und natürliche Vielfalt zu untersuchen, was für das Verständnis der biologischen Funktionen ihrer Gene und der Art und Weise, wie die Evolution die genetische Vielfalt unter den Arten geprägt hat, von entscheidender Bedeutung ist10,11.
Die größte Herausforderung für das Studium der Ökologie der sich selbst lebenden Caenorhabditis-Nematoden in freier Wildbahn ist ihre geringe Größe; Erwachsene Nematoden sind oft 1 mm lang oder weniger. Diese Herausforderung erfordert, dass Forscher Substrate aus der Wildnis beproben und versuchen, Nematoden von Interesse von den Substraten im Labor zu trennen, ohne die Möglichkeit, Tiere in freier Wildbahn zu beobachten. Da selbst ausgebildete Experten Schwierigkeiten haben, selbstsüchtige Caenorhabditis-Nematoden von anderen frei lebenden Nematoden unter dem Mikroskop zu unterscheiden, werden Nematoden typischerweise aus dem Substrat entfernt, isoliert und vermehrt, bevor sie durch Sequenzidentität unter Verwendung etablierter molekularer Barcodes identifiziert werden3,12,13,14 . Der Zeit- und Arbeitsaufwand, der erforderlich ist, um jeden Nematoden auf diese Weise zu verarbeiten, stellt eine organisatorische Herausforderung dar, da die Forscher in der Lage sein müssen, die Identität jedes im Labor isolierten Nematoden auf das genaue Substrat und die damit verbundenen ökologischen Daten zurückzuführen, die im Feld beprobt wurden. Hier beschreiben wir einen schrittweisen Prozess, um selbstsüchtige Caenorhabditis-Nematoden aus dem Feld effizient zu sammeln und zu identifizieren und diese Isolate in hohem Maßstab mit ihren zugehörigen räumlichen und ökologischen Daten zu verknüpfen.
Diese Erfassungsmethode erhöht den Umfang und die Genauigkeit ökologischer Erhebungen durch die Verwendung mobiler Datenerfassungsplattformen, Cloud-basierter Datenbanken und der R-Softwareumgebung. Fulcrum ist eine anpassbare Datenerfassungsplattform, die mit den meisten mobilen Geräten funktioniert und es Benutzern ermöglicht, benutzerdefinierte Anwendungen zum Sammeln und Organisieren von standortbasierten Daten (https://www.fulcrumapp.com) zu erstellen. Dieses Protokoll enthält detaillierte Anweisungen zur Verwendung benutzerdefinierter Datenerfassungsanwendungen, um räumlich explizite ökologische Daten aus dem Feld zu organisieren und diese Daten genau mit der Identität der im Labor isolierten Nematoden zu verknüpfen. Das Protokoll erklärt auch, wie man selbstsüchtige Caenorhabditis-Nematoden mithilfe etablierter molekularer Barcodes effizient identifizieren kann. Die Daten aus diesen Methoden können mit dem beiliegenden R-Softwarepaket easyFulcrum15 einfach und reproduzierbar verarbeitet werden, um die Ökologie und genetische Vielfalt natürlicher Caenorhabditis-Populationen zu erforschen.
Protokoll
1. Sammlungsvorbereitung
- Identifizieren Sie einen Ort, um Caenorhabditis-Nematoden zu untersuchen.
HINWEIS: In den meisten gemäßigten Regionen können C. elegans und C. briggsae leicht von menschenassoziierten Lebensräumen wie landwirtschaftlichen Feldern oder ländlichen und städtischen Gärten isoliert werden1. In subtropischen und tropischen Regionen können C. briggsae, C. elegans und C. tropicalis alle in den oben aufgeführten humanassoziierten Lebensräumen gefunden werden, manchmal in unmittelbarer Nähe zueinander. C. elegans scheint jedoch kühlere, trockenere Lebensräume als die anderen Arten in tropischen Lebensräumen zu bevorzugen7,8. Jede der Arten kann auch aus wilden Lebensräumen isoliert werden, die nicht mit Menschen in Verbindung gebracht werden, aber diese Lebensräume werden seltener beprobt. - Erstellen Sie ein Fulcrum-Projekt, um die Erfassungs- und Isolationsdaten mit den mobilen Datenerfassungsanwendungen zu organisieren.
- Erstellen Sie online ein Konto bei Fulcrum mit einer kostenlosen Bildungsvereinbarung16. Fügen Sie die Nematode Field Sampling-Anwendung zu einem Fulcrum-Konto hinzu, indem Sie auf die Schaltfläche APP HINZUFÜGEN17 klicken.
- Fügen Sie die Nematode Isolation-Anwendung zu einem Konto hinzu, indem Sie auf die Schaltfläche APP HINZUFÜGEN18 klicken.
HINWEIS: Es wird empfohlen, dass jede Reise zu einem Ort als Sammlungsprojekt unter Verwendung der Namenskonvention "YearMonthLocation" organisiert wird, z. B. 2020FebruaryAustralia.
- Fügen Sie dem Fulcrum-Konto Benutzer hinzu, um ihnen Zugriff auf das Sammlungsprojekt zu gewähren. Stellen Sie sicher, dass jeder Benutzer die mobile Anwendung Fulcrum herunterlädt, um an dem Projekt teilzunehmen.
- Drucken Sie einen Satz QR-Code-Etiketten, um die Kollektionen (C-Etiketten) und die Nematodenisolationen (S-Etiketten) mit der mobilen Anwendung zu verfolgen. Befestigen Sie die C-Etiketten an Plastiktüten mit Reißverschluss, rollen Sie die beschrifteten Beutel in Gruppen von 25 Personen und wickeln Sie sie zum Verpacken mit einem Gummiband ein. Bewahren Sie den Satz S-Etiketten für den Einsatz im Labor auf.
HINWEIS: In diesem Protokoll sind die Sammlungen (Substrate aus dem Feld) in Beuteln oder auf Platten enthalten und mit C-Etiketten gekennzeichnet. Die isolierten Nematoden sind mit S-Etiketten gekennzeichnet. Die C-Etiketten werden verwendet, um eindeutige Sammlungen zu identifizieren, und die S-Etiketten werden verwendet, um eindeutige Nematodenisolate zu identifizieren. Diese beiden Arten von Labels werden verwendet, um die Verbindung zwischen einer bestimmten Sammlung (C-Label) und den von dieser Sammlung isolierten Nematoden (S-Labels) in der Fulcrum-Datenbank herzustellen. Drucken Sie doppelt so viele S-Etiketten wie C-Etiketten für ein Sammlungsprojekt, da im Durchschnitt zwei Nematoden pro Sammlung isoliert sind. Weitere S-Etiketten können bei Bedarf später gedruckt werden. 2.500 einzigartige C-Etiketten (Supplemental File 1) und 5.000 einzigartige S-Labels (Supplemental File 2) sind in der Ergänzung enthalten. - Bereiten Sie 10 cm NGMA-Platten für Sammlungen und 3,5 cm NGMA-Platten für die Isolierung von Nematoden vor. Machen Sie eine 10-cm-Platte und mindestens zwei 3,5-cm-Platten pro Sammlung21. Diese Platten sind mit dem Escherichia coli-Stamm OP50 nach festgelegten Protokollen ausgesät. Lagern Sie die Platten vor Gebrauch bei 4 °C für nicht mehr als 1 Monat.
2. Felderfassung
HINWEIS: Caenorhabditis-Nematoden werden am häufigsten aus verrottendem Pflanzenmaterial isoliert, einschließlich Früchten, Nüssen, Samen, Schoten, Blumen, Stängeln, Pflanzenstreu und Kompost1,5,6,8. Die besten Substrate sind faul und fast nicht als Früchte oder Blumen zu erkennen; Vermeiden Sie zu trockene oder nasse Substrate (Abbildung 1). Substrate werden am effizientesten aus dem Feld gesammelt, indem sie paarweise arbeiten. Die Person mit dem berührungslosen Infrarot-Thermometer wählt ein Substrat für die Entnahme aus und sammelt die Probe, während ihr Partner die Nematode Field Sampling-Anwendung in Fulcrum verwendet, um die Entnahmedaten aufzuzeichnen. Das Kollektorpaar wiederholt diesen Vorgang, bis die gewünschte Anzahl von Proben gesammelt ist. Die Liste der für die Feldarbeit erforderlichen Materialien finden Sie in (Ergänzende Tabelle 1).
- Öffnen Sie die Fulcrum Mobile App und wählen Sie Nematode Field Sampling aus dem Dropdown-Menü. Drücken Sie +, um einen neuen Datensatz im Projekt zu starten (Abbildung 2A). Machen Sie ein Foto des Substrats.
- Klicken Sie auf das Kästchen in der oberen Mitte, um das richtige Sammlungsprojekt auszuwählen, das in Schritt 1.2 erstellt wurde (Abbildung 2B). Tippen Sie auf das Feld C-Label am unteren Rand des Sammlungsdatensatzes und wählen Sie Scannen, wenn die Eingabeaufforderung angezeigt wird. Scannen Sie den Barcode auf der Abholtasche mit der Kamera des mobilen Geräts und tippen Sie dann oben rechts auf dem Bildschirm auf Fertig .
- Tippen Sie auf das Feld Substrat und wählen Sie einen Substrattyp aus dem Dropdown-Menü aus. Fügen Sie Notizen zum Substrat hinzu, indem Sie auf das Feld Substratnotizen tippen und Notizen manuell eingeben.
- Wählen Sie eine Landschaft aus dem Dropdown-Menü aus. Wählen Sie die Landschaft aus, die den Abtaststandort am besten darstellt.
- Wählen Sie eine Himmelsansicht. Beschreiben Sie bei der Auswahl der Himmelsansicht die Himmelssichtbarkeit an der Probenahmestelle (z. B. Eine Vollhimmelansicht ohne verdeckte Sicht von Bäumen oder anderen Strukturen = voll).
- Messen Sie die Oberflächentemperatur des Substrats mit dem berührungslosen Thermometer und notieren Sie den Wert im Substrattemperaturfeld.
HINWEIS: Halten Sie das berührungslose Thermometer nicht mehr als 14 Zoll vom Substrat entfernt, während Sie die Temperatur aufzeichnen. - Messen Sie die Umgebungstemperatur und Luftfeuchtigkeit mit dem Handgerät und zeichnen Sie diese Daten in den richtigen Feldern auf.
HINWEIS: Stellen Sie sicher, dass das Umgebungstemperatur- und Feuchtigkeitsgerät nicht in der Warteschleife ist. Die Maßeinheit ändert sich, wenn die Taste losgelassen wird. Bewahren Sie das Gerät in einer Außentasche auf, um unregelmäßige Messwerte zu vermeiden. - Speichern Sie den Datensatz in Fulcrum, indem Sie oben links auf dem Bildschirm auf Speichern tippen.
- Sammeln Sie etwa einen Esslöffel des Substrats ohne Stöcke oder andere harte Stücke, indem Sie den Auffangbeutel umdrehen, um ihn als "Handschuh" zu verwenden, um das Substrat aufzunehmen, und verschließen Sie dann den Beutel. Legen Sie ein Papiertuch in den Beutel, wenn die Probe besonders feucht ist.
HINWEIS: Legen Sie die Beutel in heißen Klimazonen in weiche Kühlboxen mit Kühlboxen, um die Sammlungen kühl zu halten. - Nachdem alle Proben für den Tag gesammelt wurden, reinigen Sie die Sammelausrüstung, nehmen Sie Batterien aus den Sonden, laden Sie die Batterien auf, frieren Sie die Gefrierbeutel wieder ein. Synchronisieren Sie die Fulcrum-Sammlungsdaten, indem Sie oben links in der Nematode Field Sampling-Anwendung auf die Schaltfläche Synchronisieren tippen.
HINWEIS: Die Uploads können ohne eine starke Mobilfunkverbindung mehrere Minuten dauern, daher ist es möglicherweise am besten, auf den WLAN-Zugang zu warten. Die Daten verbleiben auf mobilen Geräten und werden mit der Cloud synchronisiert. - Versenden Sie die Proben an eine Heimatinstitution, indem Sie sie über Nacht in einen Versandkarton legen. Minimieren Sie die Zeit, in der die Proben Temperaturen von weniger als 11 °C oder mehr als 25 °C ausgesetzt sind, indem Sie Pakete an Tagen versenden, an denen die Fracht transportiert wird.
HINWEIS: Die meisten Versandeinrichtungen versenden an Wochenenden an abgelegenen Orten keine Fracht über Nacht.
3. Beschichtung von Feldsammlungen im Labor
HINWEIS: In diesem Abschnitt wird beschrieben, wie Sie den Transfer von Proben aus etikettierten Sammelbeuteln auf etikettierte Platten organisieren. Die Proben können aus einer Nachtsendung oder direkt vom Feld kommen.
- Erhalten Sie die Sendung der Abholungen und untersuchen Sie auf zerbrochene Taschen oder andere Anzeichen von Schäden. Wenn die Beutel kaputt sind, entsorgen Sie das Material und reinigen Sie die ungebrochenen Sammelbeutel mit 70% Ethanol. Vermeiden Sie das C-Etikett auf dem Beutel mit dem Ethanol, da es das Etikett verfärbt und das Lesen erschwert.
- Notieren Sie sich für jeden Zip-Lock-Beutel das C-Etikett auf dem Beutel und befestigen Sie ein passendes C-Etikett am Deckel eines 10 cm großen Tellers, der mit OP50-Bakterien befleckt ist.
HINWEIS: Die beschrifteten 10-cm-Platten werden für den Rest des Protokolls als "C-Platten" bezeichnet. Der einfachste Weg, die Proben zu organisieren, besteht darin, die Entnahmebeutel auf einen Labortisch mit der passenden C-Platte oben zu legen (Abbildung 3). - Übertragen Sie für jede Sammlung etwa einen Esslöffel Probe aus dem Sammelbeutel mit einem sauberen Plastiklöffel auf die C-Platte. Fügen Sie die Probe um den Bakterienrasen in einer Halbmond- oder Ringform hinzu, bedecken Sie den Bakterienrasen nicht vollständig (Abbildung 4).
HINWEIS: Halten Sie den Esslöffel sauber, indem Sie ihn in ein Becherglas mit 95% Ethanol geben, wenn er nicht verwendet wird. Verwenden Sie ein Papiertuch, um den Esslöffel zu trocknen, bevor Sie zusätzliche Proben übertragen. - Notieren Sie die Zeit, zu der die Sammlungen von Sammelbeuteln auf C-Kennzeichen übertragen wurden, und bewahren Sie die C-Kennzeichen mindestens 24 Stunden lang bei Raumtemperatur (RT) auf, bevor Sie versuchen, Nematoden in Abschnitt 4 zu isolieren.
4. Isolierung von Nematoden aus Sammlungen
- Öffnen Sie die Anwendung Fulcrum auf dem mobilen Gerät und wählen Sie Nematode Isolation aus dem Anwendungsmenü (Abbildung 5A). Erstellen Sie einen neuen Isolationsdatensatz, indem Sie unten rechts auf das +-Symbol tippen (Abbildung 5B).
- Bestätigen Sie auf dem Bildschirm mit dem neuen Isolationsdatensatz das richtige Auflistungsprojekt, indem Sie den Projektnamen aktivieren, der im Feld oben in der Mitte angezeigt wird. Wenn das falsche Projekt angezeigt wird, tippen Sie auf den Projektnamen, um zum richtigen Projekt zu wechseln (Abbildung 5C).
- Tippen Sie auf die Schaltfläche Auswählen unter dem Feld C-Label, um das C-Label zu finden, das der Probe zugeordnet ist, aus der Nematoden isoliert werden (Abbildung 5D). Tippen Sie auf das Suchsymbol und dann auf das Symbol Scannen, um den C-Label-QR-Code auf dem C-Kennzeichen mit der Gerätekamera zu scannen. Sobald der QR-Code gescannt wurde, erscheint ein C-Label-Datensatz im Feld C-Label.
- Tippen Sie auf das Kamerasymbol im Feld Fotos , um die Gerätekamera zu öffnen und damit ein Foto der Probe auf der C-Platte mit sichtbarem QR-Code aufzunehmen (Abbildung 5E). Tippen Sie auf Fertig , um zum Isolationsbildschirm zurückzukehren.
HINWEIS: Diese Isolationsaufzeichnungsfotos können verwendet werden, um bestimmte Eigenschaften des Substrats zu einem späteren Zeitpunkt zu untersuchen. - Verwenden Sie ein Seziermikroskop, um nach Nematoden auf der C-Platte zu suchen. Tippen Sie auf das Feld "Würmer auf Probe", um das Vorhandensein von Nematoden in der Probe aufzuzeichnen (Abbildung 5F). Tippen Sie auf Ja, wenn Nematoden auf der C-Platte vorhanden sind, und tippen Sie auf Nein, wenn keine Nematoden vorhanden sind.
- Tippen Sie auf Tracks , wenn nur Nematodenspuren vorhanden sind. Wenn keine Nematoden vorhanden sind, verfilmen Sie die C-Platte und entsorgen Sie sie in einem Biohazard-Behälter.
HINWEIS: Drehen Sie die C-Platte über den Biohazard-Abfallbehälter um und klopfen Sie vorsichtig auf die Rückseite der Platte, um alle beprobten Substrate zu entfernen. Dieser Schritt erleichtert das Auffinden und Isolieren von Nematoden, die sich unter dem Substrat auf der C-Platte befinden können. - Wenn Nematoden vorhanden sind, isolieren Sie bis zu fünf Nematoden von der C-Platte. Um einen Nematoden zu isolieren, übertragen Sie einen Nematoden von der C-Platte auf eine S-Platte mit einem Platin-Drahtpicker. Isolieren Sie gesunde, gravide Erwachsene, wenn möglich. Isolieren Sie jedoch andere Stadien, wenn Erwachsene nicht gefunden werden.
HINWEIS: Nach der Isolierung haben bis zu fünf S-Platten jeweils einen einzigen Nematoden. Halten Sie diese S-Platte(n) mit isolierten Nematoden aus derselben C-Platte zusammen in einem ordentlichen Stapel von anderen S-Platten entfernt, bis sie in Fulcrum eingegeben werden. - Tippen Sie auf das Feld S-beschriftete Platten , um die für diese Isolierung verwendete(n) S-Platte(n) einzugeben. Tippen Sie auf das + unten rechts. Tippen Sie auf S-Label und dann auf Scannen , um die Gerätekamera zu öffnen. Verwenden Sie die Gerätekamera, um den S-Label-QR-Code auf der S-Platte zu scannen.
HINWEIS: Stellen Sie sicher, dass der S-Label-Code mit dem Code auf dem Schild übereinstimmt. Wenn es übereinstimmt, tippen Sie auf Fertig. Wenn dies nicht der Fall ist, tippen Sie auf Abbrechen und scannen Sie erneut, bis es übereinstimmt, und klicken Sie dann auf Fertig. Manchmal werden QR-Codes von Platten in der Nähe versehentlich gescannt. - Nachdem Sie jede S-Platte eingegeben haben, speichern Sie den Eintrag mit der Schaltfläche Speichern oben rechts. Der Eintrag geht verloren, wenn er nicht gespeichert wird. Tippen Sie auf das + unten rechts, um bei Bedarf weitere S-beschriftete Platten hinzuzufügen, bis alle von der C-Platte isolierten Nematoden eingegeben sind. Nachdem Sie alle mit S gekennzeichneten Platten für den Isolationsdatensatz hinzugefügt haben, tippen Sie auf die Schaltfläche < oben links, um zum Bildschirm des Isolationsdatensatzes zurückzukehren.
HINWEIS: Um einen Isolationsdatensatz abzubrechen, weil Fehler nicht behoben werden können, klicken Sie oben links auf Abbrechen . In diesem Schritt wird ein Dialogfeld geöffnet, in dem Sie gefragt werden, ob der Datensatz ohne Speichern verworfen werden kann. Klicken Sie bei Bedarf auf Ja, Verwerfen. - Tippen Sie oben rechts auf die Schaltfläche Speichern , sobald der Isolationsdatensatz alle Informationen korrekt hinzugefügt hat. Dann parafilmieren Sie die S-Platten mit isolierten Nematoden und legen Sie sie in einem Bereich beiseite, der für die Aufnahme von S-Platten mit Nematoden bestimmt ist.
- Parafilm der C-Platte und entsorgen Sie sie in der Biohazard-Tonne. Tippen Sie auf das Synchronisierungssymbol, um alle Daten auf Fulcrum hochzuladen.
- Sortieren Sie alle S-Kennzeichen in alphanumerischer Reihenfolge und legen Sie die S-Kennzeichen dann in Kartons. Stellen Sie sicher, dass die S-Kennzeichen mit der Deckelseite nach unten und paralysiert sind. Stapeln Sie bis zu vier S-Kennzeichen an einer Position im Karton und beschriften Sie den Karton mit dem Projektnamen, dem Datum, der Uhrzeit und einer eindeutigen Kartonnummer.
- Lagern Sie die beschrifteten Kartons bei RT. Diese Isolate werden bei 48 h und bei Bedarf erneut bei 168 h auf Proliferation überprüft.
5. Export von S-Kennzeichen aus Fulcrum
HINWEIS: In diesem Abschnitt wird beschrieben, wie S-Labels, die im Isolationsprozess verwendet werden, aus der Fulcrum-Projektdatenbank exportiert werden. Diese S-Labels werden verwendet, um proliferierende isoweibliche Linien zu verfolgen, während sie durch Sequenzidentität in den Abschnitten 6-9 identifiziert werden.
- Melden Sie sich auf der Fulcrum-Website an und wählen Sie die Anwendung Nematode Isolation aus. Klicken Sie auf der linken Seite des Bildschirms auf Exporter . Klicken Sie, um das gewünschte Projekt auszuwählen, und aktivieren Sie das Kontrollkästchen Nematodenisolation. Klicken Sie auf Weiter , um eine .zip Datei herunterzuladen, die die Datei "nematode_isolation_s_labeled_plates.csv" enthält.
- Öffnen Sie die Datei 'nematode_isolation_s_labeled_plates.csv' und sortieren Sie sie nach der Spalte 'S-label' in aufsteigender Reihenfolge (das kleinste S-Label befindet sich oben). Wählen Sie alle S-Etiketten aus und kopieren Sie sie aus der Tabelle.
- Navigieren Sie mit einem Webbrowser zur wilden isolierten Genotypisierungsvorlage Google Sheet (wild_isolate_genotyping_template).
- Erstellen Sie eine Kopie dieses Google-Blatts, indem Sie mit der rechten Maustaste auf die Registerkarte Genotyping Template klicken und dann die Option In neue Tabelle kopieren auswählen. Wählen Sie Tabelle öffnen , um die neue Google-Tabelle anzuzeigen.
- Benennen Sie dieses neue Blatt mit dem Drehpunktprojektnamen, gefolgt von "wild_isolate_genotyping", z. B. "2020FebruaryAustralia_wild_isolate_genotyping".
HINWEIS: Dieses Blatt wird im Rest des Protokolls als "Genotypisierungsblatt" bezeichnet.
- Fügen Sie die aus der Spalte 'nematode_isolation_s_labeled_plates.csv' 's_label' kopierten S-Etiketten in die Genotypisierungsblattspalte mit dem Titel 's_label' ein. Überprüfen Sie die Spalte "s_label_repeat_error" auf "1". Der Wert "1" in dieser Spalte bedeutet, dass das S-Label irgendwo auf dem Genotypisierungsblatt dupliziert wird. Wenn Duplikate entdeckt werden, untersuchen und korrigieren Sie sie, bevor Sie fortfahren.
- Füllen Sie das Genotypisierungsblatt "isolation_box_number" für alle S-Etiketten aus.
6. Auf S-Kennzeichen auf Proliferation prüfen
- Überprüfen Sie, ob sich die Tiere 48 Stunden nach der Isolierung auf den S-Kennzeichen vermehren (verwenden Sie das Datum und die Uhrzeit der letzten Isolierung auf der Box von Schritt 4.11 bis zum Leitzeitpunkt).
HINWEIS: Proliferierende Nematoden sind durch Nachkommen auf der S-Platte gekennzeichnet. - Wenn sich eine S-Platte ausbreitet, geben Sie "1" in die Spalte proliferation_48 auf dem Genotypisierungsblatt ein und bewegen Sie die S-Platte dann in ein Feld mit der Bezeichnung "48 h proliferation, box 1". Legen Sie maximal 88 S-Platten in eine Proliferationsbox und füllen Sie dann eine neue Box mit der Aufschrift "48 h proliferation, box 2". Stellen Sie sicher, dass die S-Etiketten in alphanumerischer Reihenfolge in den 48-Stunden-Proliferationsfeldern organisiert sind.
HINWEIS: Entsorgen Sie nicht die nicht wuchernden S-Kennzeichen; Diese Kennzeichen werden nach der Isolierung um 168 h erneut überprüft. Wenn gewünscht, konsolidieren Sie diese S-Kennzeichen in numerischer Reihenfolge in Kästchen mit der Bezeichnung "48 h nicht vermehrend, Feld X", aber denken Sie daran, aufzuzeichnen, wann die 168-h-Prüfung auf der neuen Box erfolgen muss. - Nachdem Sie alle proliferierenden S-Etiketten bei 48 h identifiziert haben, fahren Sie mit Abschnitt 7 für S-Kennzeichen mit Proliferation bei 48 h fort.
- Überprüfen Sie die S-Kennzeichen, die sich nach der Isolierung um 48 h nicht vermehrt haben, erneut bei 168 h nach der Isolierung.
- Wenn sich jetzt eine S-Platte ausbreitet, geben Sie "1" in die Spalte proliferation_168 auf dem Genotypisierungsblatt ein und verschieben Sie dann die S-Platte in ein Feld mit der Bezeichnung "168 h proliferation, box 1".
- Legen Sie maximal 88 S-Platten in eine Proliferationsbox und füllen Sie dann eine neue Box mit der Aufschrift "168 h proliferation, box 2". Achten Sie darauf, S-Labels in alphanumerischer Reihenfolge in den 168 h Proliferationsboxen zu organisieren.
- Entsorgen Sie die S-Kennzeichen, die nach 168 h keine Proliferation aufweisen. Fahren Sie mit Abschnitt 7 für S-Platten mit Proliferation bei 168 h fort.
7. Lyse von isoweiblichen Linien
HINWEIS: In diesem Schritt wird das Datenfilter-Tool in Google Sheets verwendet, um Lysis-Arbeitsblätter für die S-Platten in den Proliferationsboxen zu drucken. Der Zweck der Lyse-Arbeitsblätter besteht darin, dem Personal die richtigen Positionen für S-Etiketten in Lysestreifenröhrchen am Prüfstand zur Verfügung zu stellen.
- Öffnen Sie das Genotypisierungsblatt für das gewünschte Projekt, und markieren Sie alle Zellen, indem Sie Cmd+A eingeben. Klicken Sie auf Daten > Erstellen Sie einen Filter , um jeder Spaltenüberschrift eine Filterschaltfläche hinzuzufügen. Verwenden Sie die Filterschaltflächen , um nur die S-Kennzeichen anzuzeigen, die genotypisiert werden. Zum Beispiel, wenn alle S-Platten mit Proliferation bei 48 h lysiert werden sollen: Klicken Sie auf die Schaltfläche Filter in der Spalte "proliferation_48" und wählen Sie "1".
- Sobald das genotypisierende Google-Blatt gefiltert wurde, überprüfen Sie die Liste der angezeigten S-Etiketten, um sicherzustellen, dass es sich um die S-Etiketten handelt, die auf dem Arbeitsblatt gedruckt werden sollen.
- Geben Sie in der Spalte "strip_tube_number" des genotypisierenden Google-Tabellenblatts alle 11 Zeilen eine eindeutige Zahl ein.
- Geben Sie die Bandrohrnummern für ein Projekt in nachfolgender Reihenfolge ab 1 ein und duplizieren Sie sie niemals. Geben Sie im Feld "strip_tube_position" für jede Bandrohrnummer 2 bis 12 ein.
HINWEIS: Verwenden Sie 12-Rohr-Streifenrohre für die Lyse. Die erste Position (strip_tube_position 1) ist die Steuerung, aber die Steuerelemente werden nicht zu den Lysearbeitsblättern hinzugefügt (nur die strip_tube_positions werden hinzugefügt, 2-12). Zum Zeitpunkt der Lyse wird der Positivkontrollstamm 'N2' an Position 1 jedes geraden Streifenrohrs als Positivkontrolle hinzugefügt. An Position 1 jedes ungeraden Streifenrohrs werden keine Würmer als Negativkontrolle hinzugefügt.
- Filtern Sie das genotypisierende Google-Blatt weiter, um nur die S-Labels in einer Proliferationsbox aufzunehmen, die lysiert werden sollen, und wählen Sie dann die Spalten 's_label' bis 'lysis_notes' aus. Drucken Sie ein Lyse-Arbeitsblatt für jede Proliferationsbox, die lysiert werden soll.
- Klicken Sie auf das Dropdown-Menü im Feld Drucken und wählen Sie Ausgewählte Zellen. Klicken Sie oben rechts auf Weiter und drucken Sie dann im Dialog das Lysis-Arbeitsblatt für die Proliferationsbox.
- Wiederholen Sie die Schritte 7.3-7.5, um ein Lyse-Arbeitsblatt für jede Proliferationsbox zu drucken.
HINWEIS: Jede Proliferationsbox fasst bis zu 88 S-Platten, was acht 12-Well-Streifenrohren entspricht. - Bereiten Sie 12-Well-Streifenröhrchen für alle Proben vor, die lysiert werden. Beschriften Sie ein Streifenröhrchen mit einem eindeutigen "strip_tube_number", das im Lyse-Arbeitsblatt zugewiesen ist. Dieses Etikett muss auf den Kappenstreifen und das Streifenrohr geschrieben werden, um Verwechslungen zu vermeiden, wenn sie getrennt sind. Die EVEN-Streifenrohre haben eine Positivkontrolle (N2-Würmer) in Position 1. Die ODD-Streifenrohre haben eine Negativkontrolle (keine Würmer) in Position 1.
- Stellen Sie genügend Lysepuffer (100 mM KCl, 20 mM Tris pH 8,2, 5 mM MgCl2, 0,9% IGEPAL, 0,9% Tween 20, 0,02% Gelatine mit Proteinase K zu einer Endkonzentration von 0,4 mg / ml) für alle Proben auf und fügen Sie 5% extra für den Pipettenfehler hinzu. Skalieren Sie nach Bedarf.
HINWEIS: Der Lysepuffer wird am besten durch Kombination aller Inhaltsstoffe außer Proteinase K und Einfrieren in 10-50 ml Aliquots bei -20 °C hergestellt. Aliquots auftauen und vor Gebrauch bei 4 °C aufbewahren; Unmittelbar vor dem Gebrauch Proteinase K hinzufügen und gründlich mischen. Halten Sie den Lysepuffer während der Arbeit auf Eis. - Ordnen Sie die S-Platten für ein bestimmtes Streifenrohr in der richtigen Reihenfolge an, indem Sie das gedruckte Lyse-Arbeitsblatt als Leitfaden verwenden.
- Lösen Sie ein Streifenröhrchen und fügen Sie 8 μL Lysepuffer zu jeder Kappe mit einem wiederholten Pipetor hinzu. Fügen Sie den Lysepuffer jeweils zu einem Streifen von Kappen hinzu, da der Lysepuffer verdampft, wenn er bei RT belassen und freigelegt wird. Wählen Sie 3-5 Tiere von den Quellplatten (S-Platte oder N2-Stammplatte für Positivkontrollen) in die entsprechenden Kappenpositionen, die auf dem Lyse-Arbeitsblatt angegeben sind. Notieren Sie Notizen für jede S-Platte mit weniger als 5 Würmern, die zur Lyse ausgewählt wurden, im lysis_notes Abschnitt des Lyse-Arbeitsblatts.
- Nachdem Sie Nematoden in jede Position des Bandrohrs geladen haben, legen Sie die Kappenleiste wieder auf das Bandrohr. Stimmen Sie die markierte Kappe (Position 1) mit der markierten Röhre (Position 1) ab. Nach dem Verschließen zentrifugieren Sie das Streifenröhrchen kurz, bis sich die Nematoden am Boden des Röhrchens befinden.
- Legen Sie den Streifen in den -80 °C Gefrierschrank, bis er vollständig gefroren ist (mindestens 10 min). Wiederholen Sie die Schritte 7.9 bis 7.11, bis allen Streifen Nematoden zur Lyse hinzugefügt wurden. Organisieren Sie die Rohrstreifen in numerischer Reihenfolge.
- Entfernen Sie die Sätze von Streifenrohren und führen Sie das Lyseprogramm in einem Thermocycler aus: 1 h bei 60 °C, 15 min bei 95 °C, halten bei 12 °C. Wenn das Lyseprogramm abgeschlossen ist, drehen Sie die Proben bei 300 x g für 15 s bei RT herunter und lagern Sie die Lysate bei -80 ° C für bis zu 1 Woche.
- Organisieren Sie die Rohrstreifen in numerischer Reihenfolge mit 96-Well-Plattenhaltern und fügen Sie ein Etikett mit einer Proliferationsboxnummer, einem Streifenrohrnummernbereich, einem Datum und den Initialen des Forschers hinzu. Aktualisieren Sie die Genotypisierungsblattspalten 'lysis_date' und 'lysis_notes' mit Informationen aus dem Lysis-Arbeitsblatt.
8. PCR von SSU- und ITS2-Sequenzen
HINWEIS: Dieser Abschnitt enthält Anweisungen zur Durchführung von zwei separaten PCRs für jede lysierte S-Platte. Der erste Primer-Satz amplifiziert ein 500-bp-Fragment des 18S rDNA Small Subunit Gene (SSU); oECA1271 = Forward Primer TACAATGGAAGGCAGCAGGC, oECA1272 = Reverse Primer CCTCTGACTTTCGTTCTTGATTAA 12. Diese PCR wird verwendet, um die Qualität der Template-DNA zu überprüfen. Die PCR verstärkt die SSU-Region für fast alle Nematodenarten. Wenn die SSU-PCR nicht amplifiziert, deutet dieses Ergebnis darauf hin, dass die Lysequalität schlecht ist und die Lyse für diese S-Platte wiederholt werden muss. Der zweite Primer-Satz verstärkt ein 2.000-bp-Fragment des internen transkribierten Spacer-Bereichs zwischen den 5.8S- und 28S-rDNA-Genen (ITS2); oECA1687 = Vorwärtsprimer CTGCGTTACTTACCACGAATTGCARAC, oECA202 = Reverse-Primer GCGGTATTTGCTACTACCAYYAMGATCTGC3. Das ITS2 PCR-Produkt ist Sanger-sequenziert und die Sequenz wird verwendet, um Nematoden in der Gattung Caenorhabditis auf Artebene durch Sequenzähnlichkeit zu identifizieren.
- Verwenden Sie das Filterwerkzeug im Genotypisierungsblatt, um nur die S-Etiketten anzuzeigen, die für die PCR verwendet werden sollen.
- Aktualisieren Sie die pcr_plate_number und pcr_well Spalten im Genotypisierungsblatt. Um den Abbau von Lysematerial zu verhindern, werden die SSU- und ITS2-PCRs gleichzeitig ausgeführt.
- Verwenden Sie die gleiche pcr_plate_number für die ITS2- und SSU-PCRs, obwohl es sich um separate Reaktionen in separaten Platten handelt. Sie werden mit den Labels "SSU" oder "ITS2" ausgezeichnet.
- Weisen Sie acht oder weniger Streifenröhrchen eine pcr_plate_number zu (ein Streifenröhrchen pro Reihe der 96-Well-PCR-Platte, in aufsteigender Reihenfolge angeordnet, z. B. niedrigste Streifenrohrnummer oben). Weisen Sie dann jedem S-Label in den Streifenröhrchen eine pcr_plate_well zu.
HINWEIS: Die Bandrohre sind in aufsteigender Reihenfolge angeordnet, wobei die niedrigste Bandrohrnummer Zeile A und die höchste Nummer in Zeile H zugewiesen ist. Position 1 aller Bandrohre ist Spalte 1 zugeordnet. Daher wird das Streifenrohr Nummer 1, Position 1 der PCR-Platte Nummer 1, Bohrloch A01, zugewiesen. - Beschriften Sie 96-Well-PCR-Platte(n), um die Proben aufzunehmen, die für die PCR verwendet werden. Beschriften Sie jede PCR-Platte mit den folgenden Informationen: Projektname, PCR-Typ, PCR-Plattennummer und Datum der PCR (z. B. 2020FebruaryAustralia_SSU_1_20200304). Beschriften Sie die Platte auch mit den Streifenrohrnummern, die in jede Zeile geladen werden.
- Entfernen Sie das Lysematerial aus dem -80 °C Gefrierschrank und tauen Sie die Streifenröhrchen mit dem Lysematerial auf Eis auf. Während das Lysematerial auftaut, bereiten Sie ITS2- und SSU-Mastermischungen in separaten Röhrchen auf Eis vor. Die SSU- und ITS2-PCR-Rezepte finden Sie in der Supplemental Table 2.
HINWEIS: Bereiten Sie 100 Reaktionen des PCR-Master-Mixes für jede 96-Well-Platte vor, um einen Pipettierfehler zu ermöglichen. Verwenden Sie einen konischen 15 ml oder 50 ml, um den Master-Mix zu halten, wenn große Volumina verwendet werden sollen. - Wirbeln Sie den Master-Mix vorsichtig auf, bis Taq im gesamten Mix verteilt ist. Nach dem Mischen werden aliquot 38 μL des Masters zu den entsprechenden Vertiefungen der PCR-Platten auf Eis gemischt. Verwenden Sie sterile Einweg-V-Bodentröge und eine 12-Well-Mehrkanalpipette, um den Master-Mix auf die PCR-Platten zu übertragen.
- Drehen Sie die aufgetauten Lysestreifenröhrchen nach unten, um Lysematerial von den Kappen zu entfernen. Entfernen Sie vorsichtig die Deckel aller Streifenröhrchen, die in die erste PCR-Platte geladen werden. Verwenden Sie eine mehrkanalige Pipette mit geringem Volumen (entweder 12-Well oder 8-Well), um 2 μL Lysat zu der entsprechenden Vertiefung in der PCR-Platte hinzuzufügen. Pipettieren Sie das Lysat vorsichtig einmal nach oben und unten, bevor Sie die 2 μL entfernen.
HINWEIS: Überprüfen Sie die Tipps, um sicherzustellen, dass sie die Lyse vor der Übertragung enthalten. Denken Sie daran, Tipps zwischen Zeilen oder Spalten zu ändern. - Decken Sie die PCR-Platte mit PCR-Klebefolie ab und verwenden Sie eine Walze, um eine dichte Abdichtung zu erzeugen. Nachdem die Folie aufgetragen wurde, drehen Sie die PCR-Platten in einer Zentrifuge kurz nach unten. Halten Sie die Platte auf Eis, bis sie bereit ist, im Thermocycler zu laufen.
- Führen Sie die PCRs mit dem entsprechenden Thermocycler-Programm aus. Einzelheiten zu den PCR-Programmen SSU und ITS2 finden Sie in der Ergänzenden Tabelle 2.
- Wiederholen Sie die Schritte 8.4- 8.8, bis alle PCRs ausgeführt sind.
- Während die PCR-Reaktionen laufen, gießen Sie ein 100 ml 1,5% iges Agarosegel. Jedes Gel enthält Proben oder eine einzelne PCR-Platte.
- Fügen Sie 1,5 g Agarose in einen 500-ml-Kolben hinzu, fügen Sie dann 100 ml 1x TAE-Puffer hinzu (Ergänzungstabelle 3) und wirbeln Sie zum Mischen. Mikrowelle zum Auflösen und Abkühlen des Gels.
- Sobald die Lösung abgekühlt ist, fügen Sie 5 μL 10 mg / ml Ethidiumbromidlösung hinzu und mischen Sie, um zu kombinieren. Gießen Sie die Lösung in eine Gießschale mit vier 25-Well-Kämmen, so dass das Gel 96 Proben plus eine Leiter für jede Reihe im Gel aufnehmen kann.
HINWEIS: Ethidiumbromid ist ein starkes Mutagen. Verwenden Sie beim Umgang mit Ethidiumbromid einen Laborkittel, chemikalienbeständige Handschuhe und eine Chemikalienschutzbrille.
- Kurz bevor die PCR abgeschlossen ist, fügen Sie 6x Ladefarbstoff zu einem Einwegtrog hinzu und verwenden Sie eine Mehrkanalpipette, um 2 μL 6x Ladefarbstoff zu jeder Vertiefung einer neuen 96-Well-PCR-Platte hinzuzufügen. Diese Platte wird verwendet, um die Proben in das Gel zu laden. Machen Sie genug von diesen Platten, um alle Proben unterzubringen.
- Wenn die PCRs fertig sind, entfernen Sie die PCR-Platten und zentrifugieren Sie sie kurz bei 300 x g für 15 s bei RT. Lagern Sie die PCR-Platten auf Eis, bis die PCR-Produkte auf einem Gel auslaufen können.
- Um die Produkte auf einem Gel laufen zu lassen, verwenden Sie eine 12-Well-Mehrkanalpipette, um 5 μL jeder Probe in die entsprechende Vertiefung einer 96-Well-Platte mit 2 μL 6x Belastungsfarbstoff zu geben.
- Dann 6 μL dieser Mischung in jede Vertiefung eines kürzlich gegossenen Gels geben. Laden Sie 6 μL von 1 KB plus Leiter in die erste Vertiefung jeder Reihe des Gels.
HINWEIS: Um die Vertiefungen des Gels zu füllen, kann es notwendig sein, Zeile A und Reihe B von der PCR-Platte in die erste Reihe des Gels einzustreuen. Um Verwirrung zu vermeiden, notieren Sie die gel_number und gel_position im Genotypisierungsbogen für jede PCR-Probe.
- Legen Sie einen neuen Foliendeckel auf die verbleibende PCR in der/den Platte(n) und lagern Sie sie bei 4 °C. Diese Reaktionsprodukte werden in Schritt 9 für die Sequenzierung verwendet.
- Lassen Sie die PCR-Produkte 20 min lang auf dem Gel bei 120 V laufen. Stellen Sie das Gel ab und notieren Sie, welche S-Etiketten ITS2- und/oder SSU-PCR-Produkte ergeben, in den Spalten "pcr_product_its2" und "prc_product_ssu" des Genotypisierungsbogens. Kennzeichnen Sie die Anwesenheit einer Band mit einer "1"; Markieren Sie eine "0" für keine Band.
9. Identifizierung von Nematoden mit Sanger-Sequenzierung und Sequenz-BLAST
HINWEIS: Dieser Abschnitt enthält Anweisungen zum Sequenzieren der ITS2-Amplicons aus den S-Labels, zum Ausrichten dieser Sequenzen an die Datenbank des National Center for Biotechnology Information (NCBI) mithilfe des BLAST-Algorithmus und zum Parsen der BLAST-Ergebnisse, um die Nematoden auf den S-Platten zu identifizieren.
- Verwenden Sie für jede Probe, die ITS2-positiv ist, das verbleibende ITS2-PCR-Produkt für die Sanger-Sequenzierung mit dem Vorwärtsprimer oECA306 (CACTTTCAAGCAACCCGAC). Ordnen Sie an, dass die Sequenzierungsausgabedateien einfach mit einem S-Label verknüpft werden können, indem Sie die Spalten "sequencing_plate" und "sequencing_well" jedes S-Labels im Genotypisierungsblatt aufzeichnen.
- Rufen Sie die .seq-Ausgabedateien für jedes S-Label von der Sequenzierungsplattform ab. Ordnen Sie die SEQ-Dateien für ein Projekt in einem einzigen Verzeichnis mit SEQ-Dateien für jeden Sequenzierungsbatch in Unterverzeichnissen an.
- Öffnen Sie das Befehlszeilenprogramm, und navigieren Sie zum obersten Verzeichnis, das die SEQ-Dateien enthält, indem Sie den folgenden Befehl eingeben: cd . Wenn es noch nicht vorhanden ist, erstellen Sie ein zusammengeführtes FASTA für alle .seq-Dateien, indem Sie den folgenden Befehl eingeben: für dir in * /; do cd $dir; für Datei in *.seq; echo ">"$file; cat $file; done >>.. /all_seqs.fa; cd ..; fertig.
HINWEIS: Dieser Code erstellt eine zusammengeführte FASTA-Datei mit dem Namen 'all_seqs.fa' aus allen .seq-Dateien im Projektverzeichnis. Diese Datei kann im NCBI-Online-Nukleotid-BLAST-Tool verwendet werden, um die ITS2-Sequenz jedes S-Labels schnell an die Sequenzdatenbank von NCBI anzupassen. - Navigieren Sie in einem Webbrowser zur NBCI BLAST-Website20 und klicken Sie auf die Schaltfläche Datei auswählen . Wählen Sie die Datei all_seqs.fa aus, die gerade erstellt wurde, und klicken Sie dann auf den Button Ähnliche Sequenzen (BLASTn). Klicken Sie auf die BLAST-Schaltfläche unten auf der Seite, um die BLAST-Suche zu starten.
- Aktualisieren Sie das Genotypisierungsblatt mit den BLAST-Ergebnissen für jedes S-Label. Verwenden Sie das Filter-Tool, um die Aktualisierung des Google-Blatts mit Genotypisierung zu erleichtern. Klicken Sie auf Daten > Erstellen Sie einen Filter , um jeder Spaltenüberschrift eine Filterschaltfläche hinzuzufügen. Filtern Sie die sequencing_plate Spalte, um die Sequenzierungsplatten auszuwählen, die mit BLAST-Ergebnissen aktualisiert werden sollen.
- Verwenden Sie das Dropdown-Menü auf der NCBI BLAST-Ergebnisseite, um die Ergebnisse für jede ITS2-Sequenz der S-Platte zu überprüfen (Abbildung 6).
- Überprüfen Sie, ob es keine BLAST-Treffer gibt. Eine Sequenz-ID im Dropdown-Menü mit dem Präfix * hat keine Blast-Treffer. Geben Sie für diese S-Etiketten in der manual_blast_notes Spalte des Genotypisierungsblatts "Kein Treffer " ein.
- Suchen Sie nach einer möglichen neuen Caenorhabditis-Art . Klicken Sie auf den Link auf dem oberen Treffer, um die Ausrichtung zu visualisieren (Abbildung 6). Wenn der Top-Treffer (1) eine Caenorhabditis-Art ist, (2) die Ausrichtung mehr als fünf Diskrepanzen in der Mitte der Sequenz enthält und (3) die Abfrageabdeckung größer als 50% ist, deutet dieses Ergebnis darauf hin, dass das Isolat eine neue Caenorhabditis-Art sein könnte (Abbildung 7). Für diese S-Kennzeichen geben Sie die Spezies des obersten BLAST-Treffers in der Spalte "species_id" ein, geben Sie eine 1 in der Spalte "possible_new_caeno_sp" und "mögliche neue Caeno sp." in die Spalte "manual_blast_notes" zusammen mit der prozentualen Identität ein (z. B. "mögliche neue Caeno sp. 89% Identität").
- Für S-Platten-Sequenzen, die zu einer Caenorhabditis-Art BLAST-Sequenzen führen, geben Sie in der Spalte "species_id" die vollständige Gattung und den Artnamen des obersten BLAST-Treffers ein. Zum Beispiel "Caenorhabditis elegans".
- Für S-Platten-Sequenzen, die zu einer Nicht-Caenorhabditis-Art BLAST-BLAST-Sequenzen BLAST-Sequenzen werden, geben Sie in der Spalte "species_id" nur die Gattung des Top-BLAST-Treffers gefolgt von "sp." ein. Diese Notation bedeutet, dass das Isolat eine unbekannte Art innerhalb der benannten Gattung ist. Beispiel: ' Oscheius sp.'.
HINWEIS: Die ITS2-Sequenz kann nicht verwendet werden, um Isolate außerhalb der Gattung Caenorhabditis3,13 zuverlässig auf Artebene zu identifizieren. - Geben Sie 1 in die Spalte "make_strain_name" des Genotypisierungsblatts ein, wenn 'species_id' = 'Caenorhabditis elegans', 'Caenorhabditis briggsae' oder 'Caenorhabditis tropicalis' ODER 'possible_new_caeno_sp' = 1 ist.
- Benennen Sie die Stämme mit eindeutigen Namen nach den Konventionen der Caenorhabditis-Nomenklatur , d. h. eine eindeutige Laborbezeichnung, die aus 2-3 Großbuchstaben besteht, gefolgt von einer Zahl für jeden eindeutigen Stamm23. Geben Sie die Sortennamen in die Spalte "strain_name" ein.
- Nachdem Stämme benannt wurden, können sie mit etablierten Protokollen 24 kryokonserviert werden.
10. Verarbeitung der Erfassungsdaten mit dem easyFulcrum-Paket in R
HINWEIS: In diesem Schritt wird beschrieben, wie die Sammeldaten (C-Labels) und die Nematodenisolationsdaten (S-Labels) mithilfe des easyFulcrum R-Pakets miteinander verknüpft werden. Die Software enthält Funktionen, die die Fulcrum-Daten weiter mit den Genotypisierungsdaten aus dem Genotypisierungsblatt verbinden, so dass S-Label-Speziesidentitäten und Stammnamen in einem einzigen Datenrahmen organisiert sind.
- Erstellen Sie ein neues Verzeichnis mit dem Namen für das Auflistungsprojekt. Ordnen Sie die Ordnerstruktur innerhalb des Verzeichnisses so an, dass sie den im R-Paket easyFulcrum15 beschriebenen Anforderungen entspricht.
- Navigieren Sie zur Fulcrum-Website und melden Sie sich an. Exportieren Sie die Rohprojektdaten aus der Fulcrum-Datenbank mit dem Datenexport-Tool der Fulcrum-Website auf der linken Seite und aktivieren Sie die folgenden Kontrollkästchen: Projekt, Fotos einschließen, GPS-Daten einschließen, Feldabtastung und Isolierung.
HINWEIS: Die Fulcrum-Daten für das Projekt werden als fünf .csv Dateien (Comma-Separated Value, durch Kommas getrennte Werte) exportiert. Die kompletten Projektdaten werden mit dem easyFulcrum-Paket in R zu einem einzigen Datenrahmen zusammengeführt. - Verschieben Sie die fünf .csv Dateien, die aus Fulcrum exportiert wurden, in das in Schritt 10.1 erstellte Projektverzeichnis, wie in der easyFulcrum-Vignette21 beschrieben.
- Öffnen Sie eine Rstudio-Sitzung und installieren Sie das easyfulcrum-Paket in R, indem Sie die folgenden Befehle in die R-Konsole 'install.packages("devtools")' und 'devtools::install_github("AndersenLab/easyfulcrum")' eingeben.
- Öffnen Sie ein neues R-Skript und folgen Sie den Anweisungen in der easyfulcrum-Vignette, um die Erfassungsdaten zu verarbeiten21.
Ergebnisse
Dieses Protokoll wurde verwendet, um Caenorhabditis-Nematoden von mehreren Orten, einschließlich Hawaii und Kalifornien, zu sammeln. Die Isolationserfolgsrate für Caenorhabditis-Nematoden variiert je nach Entnahmeort, Klima, Probenahmeerfahrung und den untersuchten Substrattypen. Das Protokoll wurde verwendet, um die hawaiianischen Inseln umfassend zu beproben, wo neun Sammelprojekte über mehrere Jahre und Jahreszeiten durchgeführt wurden. Die Isolationserfolgsraten für selbstsüchtige Caenorhabditis-Arten sind für C. briggsae (162 von 4.506 Proben, 3,6%) und C. elegans (163 von 4.506 Proben, 3,6%) und viel niedriger für C. tropicalis (26 von 4.506 Proben, 0,58%)8. Jede der selbstsüchtigen Arten ist im Verhältnis zu den anderen Substratkategorien auf verrottenden Frucht- und Blütensubstraten angereichert. Probieren Sie verrottende Frucht- und Blütensubstrate, wenn der Forscher versucht, die Erfolgsrate zu maximieren, anstatt die Substratpräferenzen zu charakterisieren. Die Erfolgsquote variiert jedoch mit der Qualität des ausgewählten Substrats. Zum Beispiel werden unter den Frucht- und Blütensubstraten die Substrate, die zu trocken, nass oder frisch sind, wahrscheinlich keine Caenorhabditis-Nematoden ergeben.
Die Skalierbarkeit dieses Sammlungsprotokolls zeigt sich in der Anzahl der Sammlungen, die ein einzelnes Forscherpaar aus freier Wildbahn sammeln kann. Im Oktober 2018 konnten beispielsweise zwei Forscher, die dieses Entnahmeprotokoll verwendeten, in 7 Tagen insgesamt über 1.000 Proben von mehreren Standorten auf zwei hawaiianischen Inseln sammeln. Dieses Feldteam schickte die Proben über Nacht ins Labor, wo ein Team von acht Forschern über 2.000 Nematoden aus den Proben isolierte, als sie ankamen. Ein wesentlicher Vorteil dieses Protokolls besteht darin, dass es die mit der Probenahme an entfernten Standorten verbundenen Kosten minimiert, indem die Ausrüstung und das Personal vor Ort reduziert werden. Mit diesem Protokoll kann sich ein kleines Feldteam auf die Probenahme konzentrieren, während das Isolationsteam die Proben in seiner Heimatinstitution mit zerbrechlichen und schweren Geräten wie dem Sezieren von Mikroskopen und Agarplatten zur Isolierung von Nematoden verarbeiten kann. Darüber hinaus ermöglicht die Implementierung der mobilen Datenerfassungsanwendung, dass alle Felddaten, die mit den Proben verknüpft sind, direkt mit dem C-Label verknüpft werden, wodurch das Isolationsteam bei der Verarbeitung von Proben unabhängig vom Feldteam arbeiten kann.
Forscher, die dieses Sammlungsprotokoll verwenden, müssen den Aufwand berücksichtigen, der erforderlich ist, um Nematoden vor einem Sammelprojekt zu isolieren. Die Isolations- und Identifizierungsschritte sind geschwindigkeitsbegrenzend, und ein kleines Sammelteam kann Isolatoren schnell mit Proben überfordern. Darüber hinaus kann der für die Verarbeitung vieler Sammlungen erforderliche Laborraum die laufende Forschung beeinträchtigen (Abbildung 3). Darüber hinaus erfordern einige isolierte Nematoden zusätzlichen Aufwand für die Genotypisierung. Zum Beispiel können etwa 2% der Isolate nach dem ersten Lyseversuch nicht mit dem SSU-PCR-Primer-Set amplifizieren und müssen erneut lysiert werden, um sicherzustellen, dass das Lysematerial für die Amplifikation mit dem ITS2-Primer-Set geeignet ist (Abbildung 8). Darüber hinaus können etwa 3% der Isolate nach einer ersten Runde der Sanger-Sequenzierung keine Qualitätssequenzen erzeugen. Für diese Isolate ist oft eine weitere Runde Lyse, ITS2-PCR und Sanger-Sequenzierung erforderlich, was die Übergabezeit für das Isolationsteam verlängern kann. Wichtig ist, dass die Sequenzidentität allein kein ausreichender Beweis ist, um eine neue Caenorhabditis-Art zu rechtfertigen (Abbildung 7). Um die Aufzucht eines Isolats als neue Caenorhabditis-Art ordnungsgemäß zu rechtfertigen, müssen zusätzliche Anstrengungen unternommen werden, um Paarungsversuche durchzuführen und ein typisiertes Exemplar zu etablieren13. Eine formale morphologische Beschreibung des typisierten Exemplars wird ebenfalls bevorzugt, ist aber nicht erforderlich3. Zusammengenommen deuten diese Überlegungen darauf hin, dass Forscher, die dieses Sammlungsprotokoll anwenden, von Testtests der Isolations- und Identifizierungsschritte profitieren werden, um sicherzustellen, dass die Ressourcen ordnungsgemäß zugewiesen werden, bevor ein Sammelprojekt beginnt. Wichtig ist, dass auch kleine Sammelprojekte von diesem Protokoll profitieren können, da der Prozess in hohem Maße reproduzierbar ist und die Daten leicht für die Qualitätskontrolle über Laborgruppen hinweg auditiert werden können.

Abbildung 1: Substratbeispiele. (A) Eine ideale verrottende Frucht ist in der Mitte des Bildes dargestellt (1), die Frucht ist fast nicht wiederzuerkennen. Weniger verrottete Früchte werden in der Nähe gezeigt; Vermeiden Sie die Verkostung von frisch gefallenen Stöcken (2). (B) Oben (3) ist eine idealerweise zersetzte Blume dargestellt. Vermeiden Sie es, frisch gefallene Blumen zu probieren (4). (C) Die dunkle Laubstreu unter der obersten Schicht trockener Blätter ist ideal bei der Probenahme für selbstsüchtige Caenorhabditis-Nematoden (5). Vermeiden Sie die Probenahme von trockener Laubstreu (6). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Die mobile Anwendung Nematode Field Sampling. (A) Der Anfangsbildschirm nach dem Öffnen der Nematode Field Sampling-Anwendung auf einem Apple-Gerät in Fulcrum. Der rote Pfeil unten rechts zeigt auf die Schaltfläche +, mit der ein neuer Sammlungsdatensatz erstellt wurde. (B) Ein Beispiel für einen neuen Sammlungsdatensatz, der auf einem Apple-Gerät angezeigt wird. Der rote Pfeil zeigt auf das Feld "Projekt" oben auf dem Bildschirm des Sammlungsdatensatzes. Stellen Sie sicher, dass Sie das richtige Projekt auswählen, wenn Sie vor Ort eine Stichprobe durchführen. Im Projektfeld wird standardmäßig das zuletzt verwendete Projekt beim Erstellen nachfolgender Sammlungsdatensätze verwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Sammelbeutel und Entnahmeplatten, die vor dem Ausplattieren der Proben organisiert werden. Diese Abbildung zeigt die Muster in C-beschrifteten Sammelbeuteln auf der linken Seite. Jede Sammeltasche hat eine passende C-beschriftete 10 cm Platte auf der Oberseite. Auf der rechten Seite befinden sich 10 cm Sammelplatten, die Probenmaterial enthalten, nachdem es aus den Sammelbeuteln übergeben wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Eine Sammelplatte (C-Platte) mit ordnungsgemäß übertragener Probe. Eine 10 cm große C-Platte mit zersetzenden Früchten am Rand des Bakterienrasens. Das C-Label wird am Plattendeckel befestigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
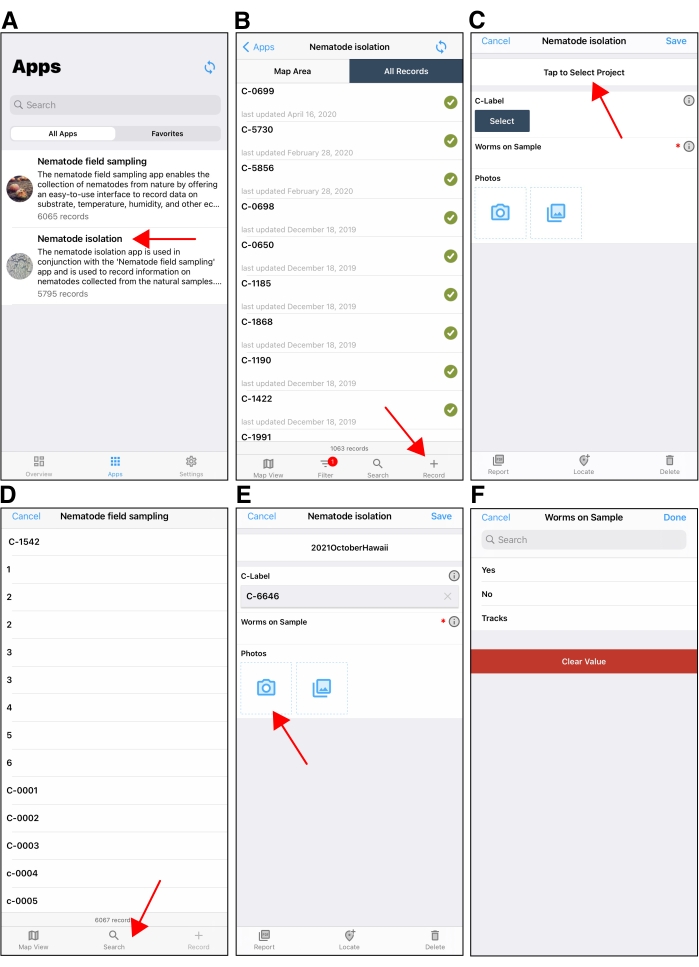
Abbildung 5: Die mobile Anwendung zur Nematodenisolation . (A) Der Anwendungsauswahlbildschirm in der mobilen Anwendung von Fulcrum. Der rote Pfeil zeigt auf die Anwendung Nematode Isolation. (B) Der erste Bildschirm nach dem Öffnen der Nematodenisolationsanwendung auf einem Apple-Gerät in Fulcrum. Der rote Pfeil unten rechts zeigt auf die Schaltfläche + , mit der ein neuer Isolationsdatensatz erstellt wurde. (C) Ein Beispiel für einen neuen Isolationsdatensatz, der auf einem Apple-Gerät angezeigt wird. Der rote Pfeil zeigt auf das Feld "Projekt" oben auf dem Bildschirm für den Isolationsdatensatz. Stellen Sie sicher, dass Sie beim Isolieren das richtige Projekt auswählen. Das Projektfeld wird standardmäßig auf das zuletzt verwendete Projekt beim Erstellen nachfolgender Isolationsdatensätze verwendet. (D) Nach dem Tippen auf das Auswahlfeld unter C-Label tippen Benutzer auf die Suchschaltfläche (roter Pfeil), um das C-Label zu finden, von dem sie Nematoden isolieren. (E) Nachdem das C-Label ausgewählt wurde, fotografiert der Benutzer das C-Kennzeichen mit der Gerätekamera. (F) Die Benutzer geben dann ein, ob sich Nematoden auf der C-Platte befinden oder nicht. S-Labels werden dem Isolationsdatensatz hinzugefügt, wenn Nematoden isoliert werden müssen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
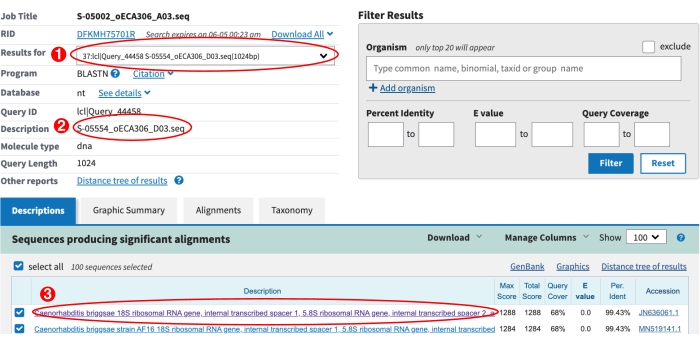
Abbildung 6: NCBI BLAST-Ergebnisseite . (1) Das Dropdown-Menü, mit dem die BLAST-Ergebnisse für alle Sequenzen angezeigt werden. (2) Die Beschreibung der aktuellen Sequenz, die aus dem Dropdown-Menü ausgewählt wurde. In diesem Fall werden die Ergebnisse für S-label S-05554 angezeigt. (3) Der Top-BLAST-Treffer für S-05554 wird angezeigt. Der violette Text zeigt an, dass auf den Link zur Visualisierung dieser Ausrichtung geklickt wurde. Bitte achten Sie darauf, die Ausrichtungen mit dem Auge zu inspizieren, um mögliche neue Caenorhabditis-Arten zu identifizieren, siehe Schritt 9.8 oben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 7: Beispiele für die Visualisierung der NCBI BLAST-Ausrichtung. (A) Ein Beispiel für die ITS2-Abfragesequenz eines Isolats, die auf eine C. kamaaina-Subjektsequenz ausgerichtet ist. (1) Die prozentuale Identität der Ausrichtung (89%), die für einen Top-BLAST-Treffer niedrig ist. (2) Eine Diskrepanz zwischen der Abfrage und der Betrefffolge (G bis A). (3) Eine Lücke von vier Basenpaaren in der Subjektsequenz, die durch den Ausrichtungsalgorithmus erzeugt wird; Lücken in der Abfrage oder im Betreff weisen auf eine schlechte Ausrichtung hin. (4) Ein verallgemeinerter Bereich in der Mitte der Ausrichtung mit vielen Diskrepanzen und Lücken. Eine Region wie diese legt nahe, dass die Abfragesequenz von einer neuen Caenorhabditis-Art stammen könnte. Gezeigt wird ein tatsächliches Ausrichtungsbeispiel einer neuen Art, C. oiwi, die 2017 entdeckt wurde. (B) Ein Beispiel für eine gute Ausrichtung zwischen der ITS2-Abfragesequenz eines Isolats und einer Subjektsequenz. (5) Die prozentuale Identität der Ausrichtung (99%), was normalerweise bedeutet, dass die Abfragesequenz von einem Isolat derselben Spezies wie das Subjekt stammt. (6) Ein zentraler Bereich der Ausrichtung mit perfekter Identität. Eine Region wie diese legt nahe, dass die Abfrageisolierung wahrscheinlich die gleiche Art wie das Subjekt ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
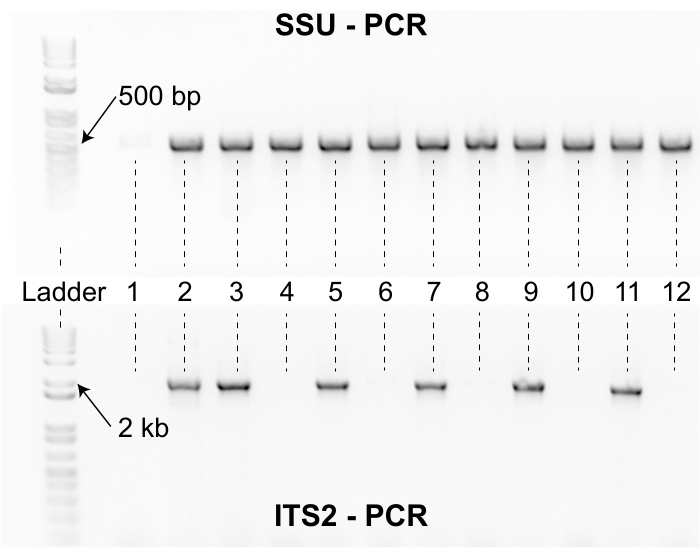
Abbildung 8: SSU- und ITS2-PCR-Produkte. Das obere Gel zeigt PCR-Produkte, die mit dem SSU-Primer-Set für 12 repräsentative Proben erzeugt wurden. Eine DNA-Leiter ist auf der linken Seite als Referenz enthalten. Die SSU PCR-Produkte für Caenorhabditis Nematoden sind ca. 500 bp lang. Die Proben 2-12 wurden mit dem SSU-Primer-Set verstärkt, aber die erste Probe nicht. Das Fehlen eines 500 bp SSU-Amplikons für Probe eins deutet darauf hin, dass das Lysematerial von schlechter Qualität war und die Probe erneut lysiert werden muss. Das untere Gel zeigt PCR-Produkte, die mit dem ITS2-Primer-Set für die gleichen 12 Proben im oberen Gel erzeugt wurden. Die Leiter und die Proben sind für beide Gele in der gleichen Ausrichtung. Sechs der 12 Proben wurden mit dem ITS2-Primer-Set nicht verstärkt. Die Proben mit SSU- und ITS2-Bändern sind Sanger-sequenziert und werden durch Sequenzähnlichkeit unter Verwendung des NCBI BLAST-Algorithmus identifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Datei 1: C-Etiketten. Eine PDF-Datei mit 2500 einzigartigen C-Labels. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: S-Etiketten. Eine PDF-Datei mit 5000 einzigartigen S-Etiketten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Tabelle 1: Feldmaterialien. Eine Verpackungsliste von Materialien, die im Feld zur Probenahme von Nematoden verwendet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 2: PCR-Rezepte und Thermocycler-Bedingungen. Eine Tabelle mit PCR-Rezepturen und Thermocycler-Bedingungen für die ITS2- und SSU-PCRs. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 3: Rezepte für Elektrophoresepuffer. Ein Rezept für 0,5 M pH 8,0 Ethylendiamintetraessigsäurelösung (EDTA) und die TRIS-Acetat-EDTA (TAE) Pufferlösung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Dieses Protokoll enthält kritische Schritte, die mit Vorsicht ausgeführt werden müssen. Zum Beispiel ist es wichtig, dass die Feld- und Isolationsteams sorgfältig das richtige Sammelprojekt in der Anwendung auswählen, bevor sie Proben aus dem Feld sammeln oder Nematoden aus den Proben im Labor isolieren. Für den Fall, dass das falsche Sammlungsprojekt ausgewählt wird, werden die fehlerhaften Datensätze am besten in der Fulcrum-Datenbank mit den Datensatzbearbeitungstools online korrigiert. Dieser Prozess kann für viele falsch platzierte Datensätze mühsam sein. Die Datenbank behält jedoch alle Änderungen an Datensätzen bei, so dass eine vollständige Überwachung von Sammlungs- und Isolationsdatensätzen möglich ist. Die anderen kritischen Schritte in diesem Protokoll umfassen die Handhabung von Proben aus dem Feld und den aus diesen Proben isolierten Nematoden. Um sicherzustellen, dass Caenorhabditis-Nematoden die Probenahme- und Versandschritte überleben, sollte die Temperatur der Proben zwischen 4 °C und 25 °C gehalten werden. Temperaturen über 25 °C können bei C. elegans14 zu Sterilität führen. Stellen Sie sicher, dass die Proben nach Möglichkeit innerhalb von fünf Tagen von Sammelbeuteln auf Entnahmeplatten übertragen werden, um den Verlust an Nematoden zu minimieren. Nachdem Nematoden isoliert wurden, ist es wichtig, dass sie genotypisiert und kryokonserviert werden, bevor sie zugrunde gehen. Es ist schwierig, lebende Nematoden auf S-Platten zu finden, die mehr als zwei bis drei Wochen alt sind, da eine pilzliche und bakterielle Kontamination die S-Platten unwirtlich machen kann.
Dieses Protokoll kann leicht modifiziert werden, um verschiedene Arten von Daten aufzunehmen, die Forscher möglicherweise im Feld sammeln möchten. Zum Beispiel ist es einfach, die Anwendung "Nematode Field Sampling" mit neuen Dateneingabefeldern über die Online-GUI von Fulcrum für die Bearbeitung von Anwendungen anzupassen. Darüber hinaus kann das Datenanalysepaket easyFulcrum diese Änderungen bei der Verarbeitung der neuen Daten berücksichtigen15. Eine weitere Änderung, die die Benutzer ansprechend finden können, ist die Verwendung einer anderen Stichprobenmethode im Feld. Anstatt diskrete Substrate zu beproben, möchten Forscher möglicherweise größere Bereiche mit mehreren Substrattypen beproben. Diese größeren Proben werden am besten im Labor mit den Baermann-Trichter- oder Tray-Extraktionsmethoden verarbeitet13. Wichtig ist, dass die Verwendung von C-Labels und S-Labels für diese Techniken weiterhin gilt und daher mit mobilen Anwendungen kompatibel ist.
Die primären Einschränkungen dieses Protokolls beziehen sich auf die Handhabungszeit von Nematoden vor der Isolierung im Labor. Erstens macht es die Verzögerungszeit zwischen der Probenentnahme und der Nematodenisolierung unmöglich, die Entwicklungsstadien von Nematoden an einer bestimmten Probe zum Zeitpunkt der Entnahme aufzuzeichnen. Zweitens sind die Häufigkeit von Männchen und die Auskreuzung in der Natur wichtige evolutionäre Fragen für sich selbst lebende Caenorhabditis-Nematoden10. Diese Methode ist nicht gut geeignet, um diese Fragen zu beantworten, da Nematoden wahrscheinlich mehrere Generationen vor der Isolation durchlaufen haben. Verzögerte Isolierung bedeutet, dass ein direkter Nachweis der männlichen Häufigkeit in der Natur unmöglich ist. Darüber hinaus bedeutet die Mehrgenerationenverzögerung während der Genotypisierungsschritte, dass der genomische Nachweis einer Auskreuzung (Heterozygotie) erodiert wird, bevor ein Nematodenstamm sequenziert werden kann. Um die Heterozygotie in der Natur zu identifizieren, werden die Nachkommen, die von einem direkt aus der Natur isolierten Nematoden produziert werden, für die Sequenzierung verwendet2. Eine weitere mögliche Einschränkung dieses Protokolls besteht darin, dass es auf die Identifizierung der selbstsüchtigen Caenorhabditis ausgerichtet ist. Dies liegt daran, dass isolierte Nematoden von sich selbst versorgenden Arten eine größere Chance haben, sich zu vermehren als obligate Auskreuzer, die sich nur vermehren, wenn ein befruchtetes Weibchen isoliert wird.
Diese Sammlungsmethode basiert auf vorhandenen Sammlungsprotokollen13,14. Der größte Fortschritt dieser Technik ist der Einsatz von mobiler Technologie und kundenspezifischer Software, um die Organisation großer Mengen ökologischer und molekularer Daten im Zusammenhang mit groß angelegten Sammelprojekten zu erleichtern. Die mit diesem Erhebungsprotokoll generierten ökologischen Daten können genutzt werden, um offene Fragen für natürliche Populationen von Caenorhabditis-Arten zu beantworten. Zum Beispiel wurden Daten, die mit dieser Methode generiert wurden, verwendet, um Nischenpräferenzen für die Art auf den Hawaii-Inseln zu entdecken. Darüber hinaus können Forscher durch die Sequenzierung der Genome von kryokonservierten Nematoden untersuchen, wie Muster genetischer Variation mit ökologischen Daten korrelieren. Forschung dieser Art kann Signaturen lokaler Anpassung in Caenorhabditis-Populationen aufdecken und wichtige Erkenntnisse über die Relevanz genetischer Variation in natürlichen Kontexten liefern8. Um ein funktionelles Verständnis vieler Gene in Caenorhabditis-Nematoden zu erlangen, sind wahrscheinlich ökologische Studien erforderlich11. Selbst für C. elegans fehlt einem großen Teil der Gene funktionelle Annotationen, obwohl es das erste mehrzellige Tier ist, das sequenziert wurde, und eines der am gründlichsten untersuchten Tiere auf der Erde. Dieses Sammlungsprotokoll wurde entwickelt, um diese Wissenslücke zu schließen, indem es die Sammlung wilder Caenorhabditis-Nematoden und die Untersuchung ihrer Ökologie und natürlichen genetischen Vielfalt erleichtert.
Offenlegungen
Die Autoren berichten von keinen Interessenkonflikten.
Danksagungen
Diese Forschung wurde durch Startkapital der Northwestern University und einen CAREER Award der National Science Foundation (IOS-1751035) unterstützt, die beide an E.C.A.
Materialien
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
Referenzen
- Frézal, L., Félix, M. -. A. C. elegans outside the Petri dish. eLife. 4, 05849 (2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339 (2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -. A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43 (2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465 (2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587 (2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -. A. Isolation of C. elegans and Related Nematodes. WormBook. , (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293 (2021).
- . Mobile data collection & workflow automation in NCAP Available from: https://www.fulcrumapp.com/ (2021)
- . Nematode Isolation Application Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021)
- . Nematode Field Sampling Application Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021)
- . JOVE wild_isolate-genotyping-template Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021)
- . Nucleotide BLAST: Search nucleotide databases using a nucleotide query Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021)
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156 (2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten