Method Article
التقاط تشكل الكروموسوم عبر مقاييس الطول
In This Article
Summary
Hi-C 3.0 هو بروتوكول Hi-C محسن يجمع بين روابط الغلوتارات الفورمالديهايد و disuccinimidyl glutarate مع مزيج من إنزيمات تقييد DpnII و DdeI لزيادة نسبة الإشارة إلى الضوضاء ودقة اكتشاف تفاعل الكروماتين.
Abstract
يستخدم التقاط شكل الكروموسوم (3C) للكشف عن تفاعلات الكروماتين ثلاثية الأبعاد. عادة ، يتم استخدام التشابك الكيميائي مع الفورمالديهايد (FA) لإصلاح تفاعلات الكروماتين. بعد ذلك ، فإن هضم الكروماتين مع إنزيم التقييد والربط اللاحق لنهايات الشظايا يحول القرب ثلاثي الأبعاد (3D) إلى منتجات ربط فريدة. أخيرا ، بعد عكس الروابط المتقاطعة وإزالة البروتين وعزل الحمض النووي ، يتم قص الحمض النووي وإعداده للتسلسل عالي الإنتاجية. تواتر ربط القرب لأزواج من المواقع هو مقياس لتكرار تمركزها المشترك في الفضاء ثلاثي الأبعاد في مجموعة الخلايا.
توفر مكتبة Hi-C المتسلسلة معلومات على مستوى الجينوم حول ترددات التفاعل بين جميع أزواج المواقع. تعتمد دقة ودقة Hi-C على التشابك الفعال الذي يحافظ على ملامسات الكروماتين والتجزئة المتكررة والموحدة للكروماتين. تصف هذه الورقة بروتوكولا محسنا في الموقع Hi-C ، Hi-C 3.0 ، يزيد من كفاءة التشابك من خلال الجمع بين اثنين من الروابط المتشابكة (الفورمالديهايد [FA] وغلوتارات disuccinimidyl [DSG]) ، يليه الهضم الدقيق باستخدام اثنين من إنزيمات التقييد (DpnII و DdeI). Hi-C 3.0 هو بروتوكول واحد للقياس الكمي الدقيق لميزات طي الجينوم على نطاقات أصغر مثل الحلقات والمجالات المرتبطة طوبولوجيا (TADs) ، بالإضافة إلى الميزات الموجودة على نطاقات أكبر على مستوى النواة مثل المقصورات.
Introduction
تم استخدام التقاط تشوه الكروموسوم منذ عام 20021. بشكل أساسي ، يعتمد كل متغير التقاط التشكل على تثبيت تفاعلات بروتين الحمض النووي والبروتين للحفاظ على تنظيم الكروماتين 3D. يتبع ذلك تجزئة الحمض النووي ، عادة عن طريق الهضم المقيد ، وأخيرا ، ربط نهايات الحمض النووي القريبة لتحويل المواضع القريبة مكانيا إلى تسلسلات الحمض النووي التساهمية الفريدة. استخدمت بروتوكولات 3C الأولية تفاعل البوليميراز المتسلسل لأخذ عينات من تفاعلات محددة "واحد لواحد". سمحت فحوصات 4C اللاحقة باكتشاف التفاعلات "واحد إلى الجميع"2 ، بينما اكتشفت 5C تفاعلات "متعدد إلى متعدد"3. جاء التقاط تكوين الكروموسوم إلى ثماره الكاملة بعد تنفيذ الجيل التالي من التسلسل عالي الإنتاجية (NGS) ، والذي سمح باكتشاف التفاعلات الجينومية "الكل إلى الكل" باستخدام Hi-C4 على مستوى الجينوم وتقنيات مماثلة مثل 3C-seq5 و TCC6 و Micro-C 7,8 (انظر أيضا مراجعة Denker و De Laat9).
في Hi-C ، تستخدم النيوكليوتيدات البيوتينيلية لتمييز 5 ′ متدلية بعد الهضم وقبل الربط (الشكل 1). وهذا يسمح باختيار الأجزاء المهضومة والمهذبة بشكل صحيح باستخدام الخرز المطلي بالستربتافيدين ، مما يميزها عن GCC10. تم تنفيذ تحديث مهم لبروتوكول Hi-C بواسطة Rao et al.11 ، الذي أجرى عملية الهضم والربط في نوى سليمة (أي في الموقع) لتقليل منتجات الربط الزائفة. علاوة على ذلك ، أدى استبدال هضم HindIII بهضم MboI (أو DpnII) إلى تقليل حجم الشظية وزيادة إمكانات دقة Hi-C. سمحت هذه الزيادة بالكشف عن الهياكل الصغيرة نسبيا وتحديد الموقع الجينومي الأكثر دقة لنقاط الاتصال ، مثل حلقات الحمض النووي بين عناصر رابطة الدول المستقلة الصغيرة ، على سبيل المثال ، الحلقات بين المواقع المرتبطة ب CTCF الناتجة عن بثق الحلقة11,12. ومع ذلك ، فإن هذه الإمكانية تأتي بتكلفة. أولا ، تتطلب الزيادة المزدوجة في الدقة زيادة أربعة أضعاف (22) في التسلسل يقرأ13. ثانيا ، تزيد أحجام الأجزاء الصغيرة من إمكانية الخلط بين الأجزاء المجاورة غير المهضومة والأجزاء المهضومة والمتدينة14. كما ذكرنا ، في Hi-C ، تختلف الأجزاء المهضومة والمهضومة عن الأجزاء غير المهضومة من خلال وجود البيوتين عند تقاطع الربط. ومع ذلك ، يلزم إزالة البيوتين المناسبة من الأطراف غير المربوطة لضمان سحب تقاطعات الربط فقطإلى أسفل 14,15.
مع انخفاض تكلفة NGS ، يصبح من الممكن دراسة طي الكروموسوم بمزيد من التفصيل. لتقليل حجم شظايا الحمض النووي ، وبالتالي زيادة الدقة ، يمكن تكييف بروتوكول Hi-C لاستخدام إنزيمات تقييد القطع بشكل متكرر16 أو لاستخدام مجموعات من إنزيمات التقييد17،18،19. بدلا من ذلك ، يمكن معايرة MNase 7,8 في Micro-C و DNase في DNase Hi-C20 لتحقيق الهضم الأمثل.
أظهر تقييم منهجي حديث لأساسيات طرق 3C أن الكشف عن ميزات طي الكروموسوم عند كل مقياس طول تحسن بشكل كبير مع التشابك المتسلسل مع 1٪ FA متبوعا ب 3 mM DSG17. علاوة على ذلك ، كان Hi-C مع هضم HindIII هو الخيار الأفضل للكشف عن ميزات الطي على نطاق واسع ، مثل المقصورات ، وأن Micro-C كان متفوقا في اكتشاف ميزات الطي على نطاق صغير مثل حلقات الحمض النووي. أدت هذه النتائج إلى تطوير استراتيجية واحدة عالية الدقة "Hi-C 3.0" ، والتي تستخدم مزيجا من الروابط المتشابكة FA و DSG متبوعة بهضم مزدوج مع DpnII و DdeI endonucleases21. يوفر Hi-C 3.0 إستراتيجية فعالة للاستخدام العام لأنه يكتشف بدقة ميزات الطي عبر جميع مقاييس الطول17. يتم تفصيل الجزء التجريبي من بروتوكول Hi-C 3.0 هنا ويتم عرض النتائج النموذجية التي يمكن توقعها بعد التسلسل.

الشكل 1: إجراء Hi-C في ست خطوات. يتم إصلاح الخلايا أولا باستخدام FA ، ثم DSG (1). ثم ، يسبق التحلل الهضم المزدوج مع DdeI و DpnII (2). يضاف البيوتين عن طريق ملء الأجزاء المتدلية ويتم ربط الأطراف الحادة القريبة (3) قبل تنقية الحمض النووي (4). تتم إزالة البيوتين من نهايات غير مرتبطة قبل صوتنة واختيار الحجم (5). أخيرا ، يسمح سحب البيوتين بربط المحول وتضخيم المكتبة بواسطة PCR (6). الاختصارات: FA = الفورمالديهايد ؛ DSG = غلوتارات ديسوسينيميديل ؛ ب = البيوتين. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Protocol
1. التثبيت عن طريق التشابك
- تثبيت الفورمالديهايد: بدءا من الخلايا في طبقة أحادية
- قم ببذر الخلايا في وسط مناسب لحصاد 5 × 106 خلايا لكل لوحة 150 مم.
ملاحظة: يمكن للمستخدمين اختيار أي حاوية مفضلة تضمن النمو الأمثل لأي خط خلايا ثديي. بالإضافة إلى ذلك ، يمكن عزل الخلايا من الأنسجة. - نضح الوسط باستخدام ماصة باستور مقترنة بمصيدة فراغ من لوحة 150 مم ، اغسل 2x مع ~ 10 مل HBSS.
- مباشرة قبل التشابك ، قم بإعداد حل تشابك FA بنسبة 1٪ في أنبوب سعة 50 مل من خلال الجمع بين 22.5 مل من HBSS و 625 ميكرولتر من 37٪ FA بتركيز نهائي بنسبة 1٪. تخلط بلطف عن طريق هزاز.
تنبيه: استخدم غطاء الدخان. الفورمالديهايد سام. - لربط الخلايا ، صب 23.125 مل من محلول FA 1٪ على كل لوحة 15 سم.
- احتضن في درجة حرارة الغرفة لمدة 10 دقائق وهز الأطباق برفق باليد كل 2 دقيقة.
- أضف 1.25 مل من 2.5 M من الجلايسين (128 mM النهائي) وقم بتدوير اللوحة برفق لإخماد تفاعل التشابك.
- احتضن في درجة حرارة الغرفة لمدة 5 دقائق واستمر في الحضانة على الجليد لمدة 15 دقيقة على الأقل لوقف التشابك.
- كشط الخلايا من لوحات مع مكشطة خلية أو شرطي المطاط.
- انقل معلق الخلية إلى أنبوب مخروطي سعة 50 مل مع ماصة. جهاز طرد مركزي عند 1000 × جم لمدة 10 دقائق في درجة حرارة الغرفة وتخلص من الطافي عن طريق الشفط.
- اغسل حبيبات الخلية مرة واحدة باستخدام 10 مل من محلول ملحي مخزن بالفوسفات (DPBS) من Dulbecco ، باستخدام ماصة للإنعاش. ثم ، جهاز طرد مركزي عند 1000 × جم لمدة 10 دقائق في درجة حرارة الغرفة. انتقل فورا إلى الربط المتشابك DSG.
ملاحظة: كن حذرا عند غسل حبيبات الخلية حيث يمكن أن تكون كريات الخلايا فضفاضة, وقد تفقد الخلايا.
- قم ببذر الخلايا في وسط مناسب لحصاد 5 × 106 خلايا لكل لوحة 150 مم.
- تثبيت الفورمالديهايد: بدءا من الخلايا المعلقة
- لديك خلايا بذر في وسط مناسب لحصاد 5 × 106 خلايا لكل وعاء.
ملاحظة: يمكن للمستخدمين اختيار أي حاوية مفضلة تضمن نمو الخلايا الأمثل لأي خط خلايا الثدييات. - مباشرة قبل الحصاد ، عد الخلايا ونقل 5 × 106 خلايا إلى أنبوب مخروطي 50 مل.
- قم بتكوير الخلايا برفق عن طريق الطرد المركزي عند 300 × جم لمدة 10 دقائق في درجة حرارة الغرفة.
- قم بإعداد محلول تشابك FA بنسبة 1٪ عن طريق إضافة 1.25 مل من 37٪ FA إلى 45 مل من HBSS واخلطه عن طريق قلب الأنبوب عدة مرات.
ملاحظة: أضف 1.25 مل من FA بالكامل دون تقسيم المبلغ.
تنبيه: الفورمالديهايد شديد السمية. - أعد تعليق حبيبات الخلية في محلول التشابك 46.25 مل من 1٪ FA الذي تم إعداده في الخطوة السابقة عن طريق السحب لأعلى ولأسفل.
- احتضن في درجة حرارة الغرفة لمدة 10 دقائق بالضبط على الدوار ، الروك ، أو عن طريق الانقلاب اليدوي اللطيف للأنبوب كل 1-2 دقيقة.
- قم بإخماد تفاعل التشابك بإضافة 2.5 مل من 2.5 M من الجلايسين (128 mM النهائي) واخلطه جيدا عن طريق قلب الأنبوب.
- احتضن لمدة 5 دقائق في درجة حرارة الغرفة ، ثم على الثلج لمدة 15 دقيقة على الأقل لإيقاف التشابك تماما.
- جهاز طرد مركزي في درجة حرارة الغرفة لتكوير الخلايا المتشابكة عند 1000 × جم لمدة 10 دقائق والتخلص من المادة الطافية عن طريق الشفط.
- اغسل الخلايا مرة واحدة ب 10 مل من DPBS ، ثم قم بالطرد المركزي عند 1000 × جم لمدة 10 دقائق في درجة حرارة الغرفة. تخلص من المادة الطافية تماما باستخدام ماصة وانتقل على الفور إلى التشابك DSG.
ملاحظة: كن حذرا عند غسل حبيبات الخلية حيث يمكن أن تكون كريات الخلايا فضفاضة, وقد تفقد الخلايا.
- لديك خلايا بذر في وسط مناسب لحصاد 5 × 106 خلايا لكل وعاء.
- التشابك مع الغلوتارات ديسوتشينيميديل
- أعد تعليق الخلايا المحببة في 9.9 مل من DPBS قبل إضافة 100 ميكرولتر من 300 mM DSG (3 mM النهائي). مزيج عن طريق الانقلاب.
ملاحظة: DSG حساس للرطوبة. من المهم تحضير مخزون جديد من 300 mM DSG في DMSO في يوم الربط المتشابك.
تنبيه: DSG في DMSO شديد السمية. - قم بربط الخلايا في درجة حرارة الغرفة لمدة 40 دقيقة على جهاز دوار.
- أضف 1.925 مل من 2.5 M من الجلايسين (400 mM النهائي) ، واقلب للخلط ، واحتضانه في درجة حرارة الغرفة لمدة 5 دقائق.
- قم بالطرد المركزي للخلايا عند 2000 × جم لمدة 15 دقيقة في درجة حرارة الغرفة.
ملاحظة: كن حذرا عند إزالة المادة الطافية من كريات الخلايا السائبة. - أعد تعليق الحبيبات في 1 مل من ألبومين مصل الأبقار 0.05٪ (BSA) -DPBS وانقلها إلى أنبوب سعة 1.7 مل.
ملاحظة: يمكن أن تساعد إضافة BSA في تقليل تكتل الخلايا. - قم بالطرد المركزي للخلايا عند 2000 × جم لمدة 15 دقيقة عند 4 درجات مئوية وقم بإزالة المادة الطافية بواسطة ماصة.
ملاحظة: لتجنب فقدان الحبيبات ، قم بإزالة المادة الطافية بسرعة وبشكل كامل. - قم بتجميد الحبيبات في النيتروجين السائل وتخزينها في -80 درجة مئوية أو انتقل فورا إلى الخطوة التالية.
- أعد تعليق الخلايا المحببة في 9.9 مل من DPBS قبل إضافة 100 ميكرولتر من 300 mM DSG (3 mM النهائي). مزيج عن طريق الانقلاب.
2. التقاط تشكيل كروموسوم
- تحلل الخلايا وهضم الكروماتين
- أعد تعليق (ماصة) حصص الخلايا المتشابكة (~ 5 × 106 خلايا) في 1 مل من محلول التحلل البارد بالثلج (الوصفة في الجدول التكميلي S1) الذي يحتوي على 10 ميكرولتر من كوكتيل مثبطات الأنزيم البروتيني ونقله إلى خالط دنس لمدة 15 دقيقة حضانة على الجليد.
ملاحظة: أضف مثبطات الأنزيم البروتيني إلى مخزن التحلل مباشرة قبل الاستخدام. - حرك ببطء المدقة A لأعلى ولأسفل 30 مرة لتجانس الخلايا على الجليد واحتضانها على الجليد لمدة 1 دقيقة للسماح للخلايا بالتبريد ، قبل 30 ضربة أخرى.
- نقل المحللة إلى أنبوب الطرد المركزي الدقيق 1.7 مل.
ملاحظة: حافظ على حركة التعليق لأنه في بعض الأحيان تلتصق الخلايا بطرف الماصة. - جهاز طرد مركزي للتعليق المتحلل عند 2500 × جم لمدة 5 دقائق في درجة حرارة الغرفة.
- تخلص من المادة الطافية ونفض الغبار أو دوامة الحبيبات الرطبة لإنعاشها. قم بإزالة أكبر قدر ممكن من المادة الطافية للحصول على مادة تشبه الزبادي مع الحد الأدنى من الكتل.
- أعد تعليق الحبيبات في 500 ميكرولتر من المخزن المؤقت المقيد 1x المثلج (من 10x ؛ انظر الوصفة في الجدول التكميلي S1) وأجهزة الطرد المركزي لمدة 5 دقائق عند 2500 × جم. كرر هذه الخطوة لغسل ثان.
ملاحظة: الحبيبات في 1x Restriction Buffer أكثر دقة من الحبيبات السابقة في محلول التحلل. - أعد تعليق الخلايا في الحجم النهائي 360 ميكرولتر من 1x Restriction Buffer عن طريق السحب بعد إضافة ~ 340 ميكرولتر إلى حجم ترحيل الحبيبات ، والذي يعتمد على حجم الخلية.
- ضع جانبا 18 ميكرولتر من كل محللة لاختبار سلامة الكروماتين (CI). قم بتخزين عينات CI في 4 درجات مئوية.
- أضف 38 ميكرولتر من كبريتات دوديسيل الصوديوم 1٪ (SDS) إلى كل أنبوب Hi-C (الحجم الإجمالي 380 ميكرولتر) واخلطه بعناية عن طريق السحب دون إدخال فقاعات.
تنبيه: SDS سامة. - احتضان العينات عند 65 درجة مئوية دون رجها لمدة 10 دقائق بالضبط لفتح الكروماتين.
- ضع الأنابيب على الثلج على الفور وقم بإعداد خليط الهضم باستخدام Triton X-100 لإخماد SDS كما هو موضح في الجدول 1.
- أضف 107 ميكرولتر من خليط الهضم إلى أنبوب Hi-C (إجمالي 487 ميكرولتر) لهضم الكروماتين طوال الليل (~ 16 ساعة) عند 37 درجة مئوية في خلاط حراري مع اهتزاز فاصل (على سبيل المثال ، 900 دورة في الدقيقة ، 30 ثانية ، 4 دقائق).
ملاحظة: تعمل إضافة Triton إلى تركيز نهائي بنسبة 1٪ على إخماد SDS.
- أعد تعليق (ماصة) حصص الخلايا المتشابكة (~ 5 × 106 خلايا) في 1 مل من محلول التحلل البارد بالثلج (الوصفة في الجدول التكميلي S1) الذي يحتوي على 10 ميكرولتر من كوكتيل مثبطات الأنزيم البروتيني ونقله إلى خالط دنس لمدة 15 دقيقة حضانة على الجليد.
- البيوتينيل من الحمض النووي ينتهي
- بعد الهضم طوال الليل ، انقل العينات إلى 65 درجة مئوية لمدة 20 دقيقة لإلغاء تنشيط نشاط النوكلياز الداخلي.
- أثناء الحضانة ، قم بإعداد مزيج رئيسي للتعبئة كما هو موضح في الجدول 2.
- بعد الحضانة ، ضع العينات على الفور على الجليد.
- ضع جانبا 10 ميكرولتر للتحكم في الهضم (DC) لكل عينة وخزنها في 4 درجات مئوية.
- قم بإزالة التكثيف من الغطاء باستخدام ماصة أو عن طريق الدوران. لكل عينة ، أضف 58 ميكرولتر من مزيج تعبئة البيوتين (إجمالي حجم العينة 535 ميكرولتر) وماصة برفق دون تشكيل فقاعات.
- احتضان العينات عند 23 درجة مئوية لمدة 4 ساعات في خلاط حراري (على سبيل المثال ، 900 دورة في الدقيقة ، 30 ثانية ، 4 دقائق).
- ربط شظايا الحمض النووي القريبة
- تحضير مزيج الربط كما هو موضح في الجدول 3 أثناء احتضان حشو البيوتين.
- أضف 665 ميكرولتر من مزيج الربط إلى كل عينة (إجمالي حجم العينة 1200 ميكرولتر). تخلط بلطف عن طريق ماصة العين.
- احتضان العينات عند 16 درجة مئوية لمدة 4 ساعات في خلاط حراري مع اهتزاز فاصل (على سبيل المثال ، 900 دورة في الدقيقة ، 30 ثانية ، 4 دقائق). قم بتخزين هذه العينات التي تحتوي على كروماتين مرتبط تساهميا عند 4 درجات مئوية لبضعة أيام.
- عكس التشابك
- ارفع أحجام عينات CI و DC إلى 50 ميكرولتر باستخدام 1x Tris Low EDTA (TLE ؛ انظر الوصفة في الجدول التكميلي S1).
- أضف 10 ميكرولتر من 10 مجم / مل من بروتيناز K إلى عينات CI و DC.
- احتضن عند 65 درجة مئوية طوال الليل مع اهتزاز الفاصل الزمني (على سبيل المثال ، 900 دورة في الدقيقة ، 30 ثانية ، 4 دقائق قبالة). بدلا من ذلك ، قم بإجراء انعكاس لمدة 30 دقيقة للربط المتشابك لعناصر التحكم هذه أثناء تنقية الحمض النووي لعينات Hi-C.
- لكل عينة Hi-C ، أضف 50 ميكرولتر من 10 مجم / مل بروتين K واحتضانها عند 65 درجة مئوية لمدة 2 ساعة على الأقل مع اهتزاز الفاصل الزمني (على سبيل المثال ، 900 دورة في الدقيقة ، 30 ثانية ، 4 دقائق).
- أضف 50 ميكرولتر أخرى من 10 مجم / مل من بروتيناز K إلى كل أنبوب Hi-C (إجمالي حجم العينة 1300 ميكرولتر) واستمر في الحضانة عند 65 درجة مئوية طوال الليل. يحفظ في درجة حرارة 4 درجات مئوية حتى تنقية الحمض النووي.
ملاحظة: يضمن تقسيم حضانة Proteinase K هضم البروتين بالكامل.
- تنقية الحمض النووي
- اترك الأنابيب تبرد من 65 درجة مئوية إلى درجة حرارة الغرفة.
- انقل كل عينة إلى أنبوب مخروطي سعة 15 مل وأضف 2.6 مل (حجم 2x) من الفينول: الكلوروفورم: كحول إيزو أميل إلى كل أنبوب.
تنبيه: الفينول: الكلوروفورم: كحول الأيزو أميل مهيج شديد السمية ويحتمل أن يكون مسرطنا. - دوامة كل أنبوب لمدة 1 دقيقة ثم نقل محتوياته إلى أنبوب قفل طور 15 مل.
- أجهزة الطرد المركزي للعينات لمدة 5 دقائق بأقصى سرعة (1500-3500 × جم) في جهاز طرد مركزي على الطاولة.
- صب الطور المائي بعناية في أنبوب طرد مركزي فائق سعة 35 مل وأضف ماء فائق النقاء إلى الحجم النهائي البالغ 1250 ميكرولتر.
ملاحظة: استخدم الأنابيب لتناسب أجهزة الطرد المركزي الفائقة المتاحة أو انقسمت إلى أنابيب طرد مركزي دقيقة متعددة. - أضف حجم 1/10عشر (~ 125 ميكرولتر) من 3 M أسيتات الصوديوم وتخلط جيدا عن طريق الانقلاب.
- أضف حجما 2.5x (~ 3.4 مل) من الإيثانول المثلج البارد بنسبة 100٪ إلى كل عينة ، وقم بموازنة الأنابيب للطرد المركزي الفائق عن طريق إضافة إيثانول 100٪ بارد ، واخلطها جيدا عن طريق الانقلاب.
- احتضان الأنابيب على الثلج الجاف لمدة ~ 15 دقيقة (تجنب التصلب).
- أجهزة الطرد المركزي للأنابيب عند 18000 × جم لمدة 30 دقيقة عند 4 درجات مئوية.
ملاحظة: بالنسبة للدوارات ذات الزاوية: حدد الأنابيب لمكان وجود الحبيبات. - باستخدام ماصة ، قم بإزالة المادة الطافية وتجاهلها تماما من الجانب غير الحبيبي.
ملاحظة: في هذه المرحلة ، يجب أن تصبح الحبيبات مرئية ويمكن تمييزها على الأنبوب ، لأنها قد لا تكون مرئية بوضوح بعد التجفيف في الخطوة التالية. - جفف العينات في الهواء لمدة 10 دقائق تقريبا أو حتى تجف بشكل واضح.
- قم بإذابة كل حبيبات في 450 ميكرولتر من 1x TLE عن طريق السحب أو الدوران ونقلها إلى وحدة تصفية الطرد المركزي 0.5 مل (CFU) مع قطع الوزن الجزيئي 3 كيلو دالتون.
- جهاز طرد مركزي CFU بأقصى سرعة لمدة 10 دقائق وتجاهل التدفق. اغسل كل أنبوب طرد مركزي فائق ب 450 ميكرولتر إضافي من 1x TLE وانقله إلى CFU الخاص به لغسله مرة أخرى.
ملاحظة: غسل CFU بهذه الطريقة يحد من فقدان الحمض النووي مع تقليل تركيز الملح. - جهاز طرد مركزي CFU بأقصى سرعة لمدة 10 دقائق وتجاهل التدفق.
- أضف 80 ميكرولتر من 1x TLE إلى العمود وقم بتحويل العمود إلى أنبوب تجميع جديد قبل الطرد المركزي لمدة 2 دقيقة بأقصى سرعة للحصول على حجم نهائي ~ 100 ميكرولتر.
- أضف 1 ميكرولتر من RnaseA (1 مجم / مل ؛ تخفيف 10 أضعاف من مخزون 10 مجم / مل) إلى كل عينة واحتضانها عند 37 درجة مئوية على كتلة حرارية أو في حمام مائي أو خلاط حراري لمدة 30 دقيقة على الأقل.
- بعد معالجة Rnase ، قم بإزالة العينات من 37 درجة مئوية وتخزينها في 4 درجات مئوية حتى خطوة مراقبة الجودة.
- التحقق من جودة الكروماتين وهضم الإنزيم وربط العينة
- قم بتبريد عينات CI و DC إلى درجة حرارة الغرفة بعد عكس الروابط المتقاطعة في الخطوة 2.4.5. بعد ذلك ، انقله إلى أنبوب قفل طور 2 مل مسبقا.
ملاحظة: تأكد من طرد محتوى قفل الطور بالطرد المركزي إلى حبيبات (السرعة القصوى لمدة 2 دقيقة). - أضف 200 ميكرولتر من الفينول: الكلوروفورم: كحول الأيزو أميل واخلط العينات عن طريق الدوامة لمدة 1 دقيقة.
- أجهزة الطرد المركزي للأنابيب لمدة 5 دقائق بأقصى سرعة.
- انقل الطور المائي من كل عينة (~ 50 ميكرولتر) إلى أنبوب ميكروفوج جديد سعة 1.7 مل.
- أضف 1 ميكرولتر من Rnase A (من 1 مجم / مل) واحتضانه عند 37 درجة مئوية لمدة 30 دقيقة على الأقل.
- قم بتحميل العينات على جل أغاروز 0.8٪ كما هو موصى به في الجدول 4.
ملاحظة: النتائج المتوقعة من مراقبة الجودة موضحة في الشكل 2. - تحديد كمية الحمض النووي عن طريق قياس الكثافة من الجل أو باستخدام Qubit أو Nanodrop.
ملاحظة: يضمن القياس الكمي الدقيق حجم الإدخال الصحيح في الجزء التالي من البروتوكول. استخدم عدة معايير بكمية معروفة لبناء منحنى قياسي.
- قم بتبريد عينات CI و DC إلى درجة حرارة الغرفة بعد عكس الروابط المتقاطعة في الخطوة 2.4.5. بعد ذلك ، انقله إلى أنبوب قفل طور 2 مل مسبقا.
الجدول 1: كواشف الهضم. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 2: كواشف تعبئة البيوتين. * لاحظ أن تغيير الإنزيمات قد يتطلب مخازن مختلفة و dNTPs البيوتينيل. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 3: كواشف مزيج الربط. اختصار: BSA = ألبومين مصل الأبقار. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 4: معلمات تحميل الجل لتقييم اختيار الجودة والحجم. الرجاء الضغط هنا لتحميل هذا الجدول.
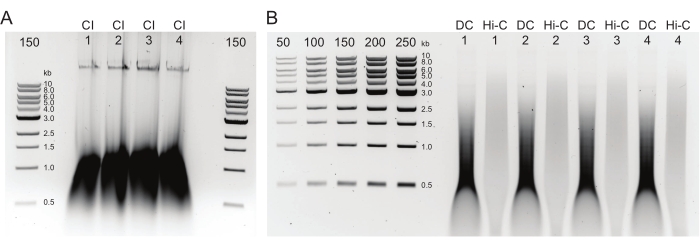
الشكل 2: جل الأغاروز يظهر نتائج مراقبة جودة تنقية ما بعد الحمض النووي النموذجية . (أ) يجب أن تشير وحدة تحكم CI إلى نطاق من الحمض النووي عالي الوزن الجزيئي. (ب) تظهر عينات DC وHi-C مجموعة من أحجام الحمض النووي (DNA). يجب أن تكون عينة Hi-C ، بعد دمجها في أجزاء أكبر ، ذات وزن جزيئي أعلى من DC. يسمح نطاق تركيز العلامات بإنشاء منحنى قياسي. لاحظ ، في هذا المثال ، تم تحميل CI على هلام منفصل ، ولكن ينصح بتحميل وتشغيل جميع العينات وعناصر التحكم معا. الاختصارات: CI = سلامة الكروماتين ؛ DC = التحكم في الهضم ؛ Hi-C = ربط القرب. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
3. إعداد مكتبة تسلسل Hi-C
- إزالة البيوتين من نهايات غير مربوطة
- تحضير تفاعلات إزالة البيوتين كما هو موضح في الجدول 5.
ملاحظة: عادة ما يكون 10 ميكروغرام من الحمض النووي كافيا ، ولكن يمكن استخدام ما يصل إلى 30 ميكروغرام. - توزيع 2 × 65 ميكرولتر من كل تفاعل 130 ميكرولتر في أنبوبي تفاعل البوليميراز المتسلسل.
- انقله إلى جهاز حراري أو جهاز PCR واحتضانه كما هو موضح في الجدول 5.
ملاحظة: يمكن تخزين العينات هنا عند 4 °C (أيام إلى أسابيع) ، -20 °C (على المدى الطويل) ، أو نقلها على الفور إلى صوتنة.
- تحضير تفاعلات إزالة البيوتين كما هو موضح في الجدول 5.
- سونيكيشن
- تجميع العينة المكررة من خطوة إزالة البيوتين (حجم العينة الإجمالي 130 ميكرولتر) في أنبوب سونيكاتور 130 ميكرولتر للصوتنة.
- قم بتقطيع العينات باستخدام المعلمات الواردة في الجدول 6 لتحقيق توزيع ضيق محكم أقل من 500 نقطة أساس.
ملاحظة: يمكن استخدام أنواع مختلفة من الصوتنة ، ولكن بالنسبة لتوزيع الأجزاء الضيق (100-500 نقطة أساس) ، قد تتطلب إعدادات الصوتنة التحسين.
- اختيار الحجم مع الخرز المغناطيسي
- ماصة الحمض النووي الصوتي من أنبوب (أنابيب) صوتي في أنبوب ربط منخفض 1.7 مل.
- أحضر كل عينة إلى حجم إجمالي قدره 500 ميكرولتر باستخدام 1x TLE. حاول أن تقرب الحجم قدر الإمكان من 500 ميكرولتر ، لأن نسبة العينة إلى خليط الخرز المغناطيسي ضرورية لاختيار الحجم.
- أضف 400 ميكرولتر من خليط الخرز المغناطيسي إلى كل أنبوب للحصول على نسبة من خليط الخرز المغناطيسي إلى حجم العينة 0.8.
ملاحظة: في ظل هذه الظروف ، تلتقط الخرز شظايا الحمض النووي >300 نقطة أساس ، والتي ستكون الجزء العلوي. سيحتوي الطافي على شظايا <300 نقطة أساس ، والتي ستكون الجزء السفلي. - امزج الأنابيب عن طريق الدوامة واحتضانها لمدة 10 دقائق في درجة حرارة الغرفة على جهاز دوار. بالنسبة لهذه النسب وغيرها من نسب اختيار الحجم ، تأكد من أن الحجم الكامل للعينة يمتزج جيدا. ابحث عن "الوضع غير المنتظم" الذي تمتلكه بعض الدوارات ، والذي يعمل بشكل جيد مع الأحجام الأصغر.
- احتضان لمدة 5 دقائق في درجة حرارة الغرفة على فاصل الجسيمات المغناطيسي (MPS).
- أثناء الحضانة ، أضف 500 ميكرولتر من خليط الخرز المغناطيسي إلى أنبوب ربط منخفض جديد سعة 1.7 ميكرولتر لكل عينة.
ملاحظة: سيتم استخدام هذه الأنابيب لاختيار الحجم التالي للجزء السفلي عن طريق توليد خليط من الخرز المغناطيسي إلى نسبة عينة 1.1: 1. - اترك الأنابيب على MPS لمدة 5 دقائق.
- قم بإزالة المادة الطافية من الخرز وأعد تعليقها باستخدام 150 ميكرولتر من خليط الخرز المغناطيسي.
ملاحظة: تتجنب هذه الخطوة تشبع الخرز بالحمض النووي عن طريق زيادة عدد الخرز دون زيادة الحجم. - انقل المادة الطافية من الخطوة 3.3.5 إلى الأنبوب المسمى المعد لتحديد الجزء السفلي (الخطوة 3.3.8).
ملاحظة: 150 ميكرولتر من خليط الخرز المغناطيسي + 400 ميكرولتر من خليط الخرز المغناطيسي 0.8x (إجمالي 550 ميكرولتر) مقسوما على 500 ميكرولتر من العينة الأولية = 1.1x خليط حبات مغناطيسية إلى نسبة العينة. - امزج أنابيب الجزء السفلي عن طريق الدوامة واحتضانها لمدة 10 دقائق في درجة حرارة الغرفة على جهاز دوار.
ملاحظة: ستربط الخرزات شظايا الحمض النووي >100 نقطة أساس ، مما ينتج عنه جزء نهائي مرتبط بالخرزة من 100-300 نقطة أساس. - ضع أنابيب الجزء السفلي على MPS لمدة 5 دقائق (درجة حرارة الغرفة).
- قم بإزالة المادة الطافية وأجهزة الطرد المركزي للأنابيب لفترة وجيزة لإزالة المادة الطافية قدر الإمكان.
- اغسل الخرز من كلا الكسرين مرتين باستخدام 200 ميكرولتر من الإيثانول بنسبة 70٪ واسترد الخرز لمدة 5 دقائق على MPS في كل مرة.
- بعد الدوران السريع في جهاز طرد مركزي ، قم بإزالة الإيثانول تماما وقم بتجفيف الخرز على MPS.
ملاحظة: يجف حتى يتبخر الكحول تماما. يجب أن تبدو الحبيبات مثل الشوكولاتة الداكنة دون تكسير (يمكن أن تستغرق ~ 10 دقائق). - أعد تعليق كلا الكسرين في 50 ميكرولتر من المخزن المؤقت 1x TLE. احتضن في درجة حرارة الغرفة لمدة 10 دقائق واضغط أو حرك الأنابيب كل دقيقتين لتحفيز الخلط والشطف.
- افصل الخرز عن الطافي على MPS لمدة 5 دقائق لكلا الكسرين.
- احتفظ بالمادة الطافية من كل عينة. ماصة الطافية في أنبوب ربط منخفض 1.7 مل.
ملاحظة: يمكن الاحتفاظ بالعينات عند 4 درجات مئوية لبضعة أيام أو عند -20 درجة مئوية على المدى الطويل. - قم بتشغيل جل الأغاروز 2٪ كما في الجدول 4 لتحديد جودة العينة وكميتها. انظر الشكل 3 للحصول على مثال لمثل هذا الهلام.
ملاحظة: إذا كان من التجربة ، صوتنة قابلة للتكرار بشكل كبير ، يمكن للمرء تخطي هذا الجل والمضي قدما في إنهاء الإصلاح على الفور. يوصى بأن يحتفظ المستخدمون بالكسور العليا حتى بعد معايرة تفاعل البوليميراز المتسلسل بالتحليل الحجمي. يمكن إنقاذ كميات الحمض النووي دون المستوى الأمثل التي تتطلب الكثير من تضخيم تفاعل البوليميراز المتسلسل من المواد الموجودة في الجزء العلوي. - حدد كمية الحمض النووي من الجل مباشرة بعد إنشاء منحنى قياسي من مدخلات سلم الحمض النووي المعروفة أو باستخدام Qubit أو Nanodrop.
- نهاية الإصلاح
- قم بإعداد مزيج الإصلاح النهائي كما في الجدول 7 (الكمية لكل تفاعل معطى).
- انقل 46 ميكرولتر المتبقية من الحمض النووي المستخلص من الجزء السفلي إلى أنابيب PCR وأضف 24 ميكرولتر من مزيج الإصلاح النهائي المحضر. احتضان في جهاز PCR ، كما هو مقترح في الجدول 7.
- بمجرد اكتمال البرنامج ، احتفظ بالعينات عند 4 درجات مئوية حتى يتم سحبها لأسفل.
- سحب منتجات ربط البيوتينيل مع الخرز المطلي بالستربتافيدين
- حدد كمية الخرز المطلي بالستربتافيدين لكل مكتبة من اختيار الحجم الكمي (الخطوة 3.3.19).
ملاحظة: يمكن لهذه الخرز المغلفة بالستربتافيدين (محلول 10 مجم / مل) ربط 20 ميكروغرام من الحمض النووي المزدوج الذي تقطعت به السبل لكل حبة مجم (= 20 ميكروغرام / 100 ميكرولتر من الخرز). استخدم 2 ميكرولتر لكل 1 ميكروغرام من الحمض النووي Hi-C ولكن لا يقل عن 10 ميكرولتر. - امزج الخرز المطلي بالستربتافيدين وماصة حجم الخرز اللازم لكل مكتبة (محسوبة في الخطوة السابقة) في أنابيب ربط منخفضة سعة 1.7 مل.
- أعد تعليق الخرز في 400 ميكرولتر من محلول غسيل توين (TWB ؛ انظر الوصفة في الجدول التكميلي S1) واحتضانها لمدة ~ 3 دقائق في درجة حرارة الغرفة على الدوار (انظر التعليمات في الخطوة 3.3.4).
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة وقم بإزالة المادة الطافية.
- اغسل الخرز عن طريق سحب 400 ميكرولتر أخرى من TWB.
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة وقم بإزالة المادة الطافية.
- أضف 400 ميكرولتر من 2x Binding Buffer (BB) (الوصفة في الجدول التكميلي S1) إلى الخرز و reuspend. بالإضافة إلى ذلك ، أضف 330 ميكرولتر من 1x TLE والحل من الإصلاح النهائي (من الخطوة 3.4.3).
- احتضان العينات لمدة 15 دقيقة في درجة حرارة الغرفة أثناء الخلط على الدوار.
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة وقم بإزالة المادة الطافية.
- أضف 400 ميكرولتر من 1x BB إلى الخرز وأعد استبداله.
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة وقم بإزالة المادة الطافية.
- أضف 100 ميكرولتر من 1x TLE لغسل الخرز.
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة وقم بإزالة المادة الطافية.
- أخيرا ، أضف 41 ميكرولتر من 1x TLE لإعادة تعليق الخرز.
- حدد كمية الخرز المطلي بالستربتافيدين لكل مكتبة من اختيار الحجم الكمي (الخطوة 3.3.19).
- أ- الذيل
- تحضير مزيج A-tailing كما في الجدول 8.
- ماصة التفاعلات في أنابيب PCR واحتضانها كما في الجدول 8.
- ضع أنابيب PCR على الثلج مباشرة بعد إزالتها من thermocycler ونقل المحتويات إلى أنابيب ربط منخفضة 1.7 مل.
- افصل الخرز عن الطافي الموجود على MPS لمدة 1 دقيقة وتخلص من المادة الطافية.
- أضف 400 ميكرولتر من مخزن الربط 1x ، المخفف من 5x T4 DNA ligase buffer بماء فائق النقاء.
- افصل الخرز عن الطافي على MPS لمدة 1 دقيقة ، ثم تخلص من المادة الطافية.
- أضف 1x مخزن ربط مؤقت إلى حجم نهائي يبلغ 40 ميكرولتر.
- محول التلدين oligos
- إعداد مخزون oligo محول من 100 ميكرومتر (الجدول 9).
ملاحظة: اطلب 250 نانومول من قلة القلة المنقى HPLC. - قم بتلدين المحولات في أنابيب PCR كما هو موضح في الجدول 9.
- استخدم جهاز PCR للدراجات الحرارية لرفع درجة الحرارة تدريجيا عند 0.5 درجة مئوية / ثانية إلى 97.5 درجة مئوية. امسك عند 97.5 درجة مئوية لمدة 2.5 دقيقة.
- استخدم جهاز PCR للدراجات الحرارية لزيادة درجة الحرارة تدريجيا عند 0.1 درجة مئوية / ثانية لمدة 775 دورة (تصل إلى 20 درجة مئوية). احفظ درجة الحرارة عند 4 درجات مئوية حتى الاستخدام مرة أخرى.
- أضف 83 ميكرولتر من 1x Rigaling Buffer (الوصفة في الجدول التكميلي S1) لتخفيف المحولات إلى 15 ميكرومتر. قم بتخزين المحولات في -20 درجة مئوية.
- إعداد مخزون oligo محول من 100 ميكرومتر (الجدول 9).
- ربط محول التسلسل
- قم بإعداد مزيج ربط المحول في أنبوب ربط منخفض سعة 1.7 مل (الجدول 10).
- Ligate لمدة 2 ساعة في درجة حرارة الغرفة.
- افصل الخرز عن الطافي الموجود على MPS لمدة 1 دقيقة وتخلص من المادة الطافية.
- أضف 400 ميكرولتر من TWB وقم بسحب الخرز لأعلى ولأسفل بعناية قبل الحضانة على الدوار لمدة 5 دقائق في درجة حرارة الغرفة. افصل الخرز عن الطافي الموجود على MPS وكرر هذه الخطوة مرة أخرى.
- افصل الخرز عن الطافي على MPS (~ 1 دقيقة) ، وتخلص من الطافي ، وأضف 200 ميكرولتر من 1x BB.
- افصل الخرز عن الطافي على MPS (~ 1 دقيقة) وتخلص من المادة الطافية.
- أضف 200 ميكرولتر من 1x قبل PCR Buffer (من 10x ؛ وصفة في الجدول التكميلي S1) وانقلها إلى أنبوب ربط منخفض جديد سعة 1.7 مل.
- افصل الخرز عن الطافي على MPS (~ 1 دقيقة) وتخلص من المادة الطافية.
- أضف 20 ميكرولتر من 1x قبل PCR Buffer واخلطه عن طريق سحب العينات.
- احتفظ بالأنابيب على الثلج أثناء الاستخدام أو خزنها في درجة حرارة 4 درجات مئوية.
- تحسين رقم دورة PCR عن طريق المعايرة بالتحليل الحجمي
- قم بإعداد 30 ميكرولتر من تفاعلات المزيج الرئيسي لكل عينة كما في الجدول 11 (الكمية لكل تفاعل معطى).
- قم بتشغيل أقل عدد من الدورات واحصل على حصة 5 ميكرولتر. قم بتشغيل 2-3 دورات إضافية على التفاعل المتبقي قبل أخذ القسمة التالية البالغة 5 ميكرولتر. كرر لجمع أربعة القسام.
- استخدم معلمات PCR من الجدول 11 لكل قسمة
- أضف 5 ميكرولتر من الماء و 2 ميكرولتر من صبغة 6x لكل عينة 5 ميكرولتر. يعمل على 2٪ هلام الأغاروز TBE (وصفة في الجدول التكميلي S1) مع 25-150 نانوغرام من سلم الوزن الجزيئي المنخفض. انظر الشكل 4 للحصول على النتائج المتوقعة.
ملاحظة: العدد الأمثل من الدورات لتضخيم PCR النهائي للمكتبة [الخطوة 3.10] هو أقل عدد من الدورات للحصول على منتج مرئي على الجل ناقص دورة واحدة.
- المكتبة النهائية لتضخيم PCR
- قم بإعداد تفاعلات 12 × 30 ميكرولتر لتضخيم كل مكتبة نهائية للتسلسل كما في الجدول 11.
- قم بتدوير تفاعلات تفاعل البوليميراز المتسلسل وفقا للجدول 11 بعد تحديد عدد الدورات بعد معايرة تفاعل البوليميراز المتسلسل (الخطوة 3.9.3).
- بمجرد اكتمال تفاعل البوليميراز المتسلسل ، قم بتجميع العينات المكررة في أنبوب microfuge منخفض الربط سعة 1.7 مل.
- ضع الأنابيب على MPS وانقل المادة الطافية إلى أنبوب microfuge منخفض الربط سعة 1.7 مل.
- أعد تعليق الخرز المتبقي المطلي بالستربتافيدين في 20 ميكرولتر من 1x قبل PCR Buffer.
ملاحظة: يمكن إعادة استخدام هذا القالب عند تخزينه في درجة حرارة 4 درجات مئوية لأيام إلى أسابيع أو على المدى الطويل عند -20 درجة مئوية.
- إزالة الاشعال مع خليط الخرز المغناطيسي
- استخدم 1x TLE buffer (وصفة في الجدول التكميلي S1) لضبط مستوى الصوت من الخطوة 3.10.4 إلى 360 ميكرولتر بالضبط.
- لكل عينة ، أضف 360 ميكرولتر من خليط الخرز المغناطيسي والماصة لأعلى ولأسفل للخلط.
- على الدوار ، امزج العينات لمدة 10 دقائق في درجة حرارة الغرفة.
- افصل الخرز عن الطافي الموجود على MPS في درجة حرارة الغرفة (3-5 دقائق).
- اغسل الخرز مرتين باستخدام 200 ميكرولتر من الإيثانول بنسبة 70٪ واسترد الخرز لمدة 5 دقائق على MPS في كل مرة.
- تدور بسرعة في جهاز طرد مركزي وماصة تماما قبالة الإيثانول. جفف الخرز في الهواء على MPS لزيادة تبخر الإيثانول.
ملاحظة: يجب أن تبدو الحبيبات مثل الشوكولاتة الداكنة دون تكسير (يمكن أن تستغرق ~ 10 دقائق). - أضف 30 ميكرولتر من الماء عالي النقاء وأعد تعليقه لتهدئة الحمض النووي لمدة 10 دقائق في درجة حرارة الغرفة. نفض الغبار الأنابيب كل 2 دقيقة للمساعدة في الخلط.
- افصل الخرز عن الطافي الموجود على MPS لمدة 5 دقائق.
- اجمع المادة الطافية من كل عينة في أنبوب جديد سعة 1.7 مل.
- قم بتشغيل 1 ميكرولتر من المكتبة على هلام 2٪ agarose TBE (وصفة في الجدول التكميلي S1) للحصول على توزيع حجم الشظية وتحديد المكتبة النهائية (الشكل 5).
ملاحظة: يمكن أن يحدث هضم ClaI فقط لتقاطعات DpnII-DpnII ويعمل كعنصر تحكم ربط إيجابي يجب أن يؤدي إلى توزيع حجم جزء أقل للمكتبة النهائية. يمكن تخزين المكتبات النهائية لبضعة أيام عند 4 درجات مئوية ، أو على المدى الطويل عند -20 درجة مئوية ، أو تخفيفها على الفور وتقديمها للتسلسل.
الجدول 5: كواشف إزالة البيوتين ودرجات الحرارة الرجاء النقر هنا لتنزيل هذا الجدول.
الجدول 6: معلمات صوتنة. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 7: كواشف الإصلاح النهائية ودرجات الحرارة. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 8: كواشف الذيل A ودرجات الحرارة. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 9: بادئات PCR و oligos ذات المهايئات المزدوجة مع الكواشف التلدين للتلدين. اختصار: 5PHOS = 5 'فوسفات. تشير العلامات النجمية إلى قواعد الحمض النووي الفوسفورية. # ادمج oligo المفهرس مع oligo العالمي للتلدين في محول مفهرس. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 10: كواشف ربط المحول. الرجاء الضغط هنا لتحميل هذا الجدول.
الجدول 11: كواشف تفاعل البوليميراز المتسلسل وبارامترات ركوب الدراجات. الرجاء الضغط هنا لتحميل هذا الجدول.

الشكل 3: جل الأغاروز يظهر نتائج اختيار ما بعد الحجم النموذجية. يتم عرض الكسور العلوية والسفلية لأربع عينات (مرقمة 1-4) من DpnII-DdeI Hi-C. يحتوي الممر الأول لكل عينة على الجزء العلوي ، المشتق من خليط خرز مغناطيسي 0.8x ، ويحتوي الحاران الثاني والثالث على تخفيف للجزء السفلي مشتق من خليط خرز مغناطيسي 1.1x. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.

الشكل 4: جل الأغاروز مع نتائج معايرة PCR. بدءا من 5 دورات من PCR ، يتم أخذ العينات بعد كل دورتين (5 و 7 و 9 و 11 دورة) لكل مكتبة من المكتبات الأربع. بناء على هذا الرقم ، تم اختيار 6 دورات كدورة مثالية لكل عينة. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
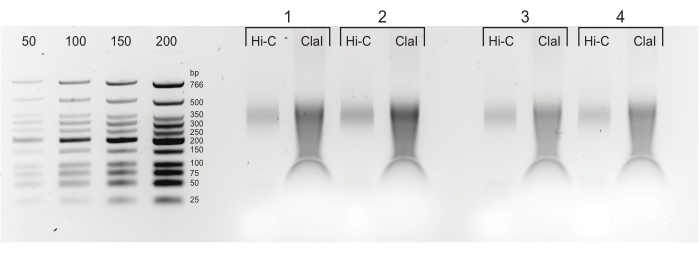
الشكل 5: منتجات PCR النهائية. بعد التنظيف واختيار الحجم ، تم تحميل منتجات PCR (Hi-C) بجوار جزء مهضوم من ClaI من نفس المكتبة (ClaI). تشير شظايا ClaI المهضومة إلى وجود روابط DpnII-DpnII المطلوبة. لاحظ أن ClaI لا يستوعب تقاطعات DpnII-DdeI ، وبالتالي ، لن تساهم جميع الروابط في تقليل الحجم من هذا القيد. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
النتائج
تم إنشاء الأشكال في هذه المخطوطة من تجربة منفصلة ومكررة للتجربة التي نشرها سابقا لافونتين وآخرون 21. بعد الحصول على بيانات تسلسل عالية الإنتاجية ، تم استخدام Open Chromatin Collective (Open2C: https://github.com/open2c) لمعالجة بيانات Hi-C. يمكن العثور على خط أنابيب مماثل على بوابة البيانات لمشروع 4D Nucleome (https://data.4dnucleome.org/resources/data-analysis/hi_c-processing-pipeline). باختصار ، تم تنفيذ جهاز تقطير خط أنابيب Nextflow (https://github.com/open2c/distiller-nf) من أجل (1) محاذاة تسلسل جزيئات Hi-C مع الجينوم المرجعي ، (2) تحليل محاذاة .sam وملفات النموذج مع أزواج Hi-C ، (3) تصفية تكرارات PCR ، و (4) تجميع الأزواج في مصفوفات bined لتفاعلات Hi-C. يمكن بعد ذلك عرض هذه المصفوفات المنسقة HDF5 ، والتي تسمى المبردات ، (1) على خادم HiGlass (https://higlass.io/) و (2) تحليلها باستخدام مجموعة كبيرة من الأدوات الحسابية مفتوحة المصدر الموجودة في مجموعة "cooltools" التي تحتفظ بها Open Chromatin Collective (https://github.com/open2c/cooltools) لاستخراج وقياس ميزات الطي مثل المقصورات و TADs والحلقات.
يمكن تقييم بعض مؤشرات الجودة لمكتبات Hi-C3.0 مباشرة بعد تعيين أزواج القراءة إلى جينوم مرجعي ، باستخدام بعض المقاييس / المؤشرات البسيطة. أولا ، عادة ~ 50٪ من أزواج القراءة المتسلسلة يمكن تعيينها بشكل فريد للخلايا البشرية. نظرا للطبيعة البوليمرية للكروموسومات ، فإن معظم هذه القراءات المعينة (~ 60٪ -90٪) تمثل تفاعلات داخل كروموسوم (رابطة الدول المستقلة) ، مع ترددات التفاعل تتحلل بسرعة مع زيادة المسافة الجينومية (الاضمحلال المعتمد على المسافة). يمكن تصور الاضمحلال المعتمد على المسافة بشكل أفضل في "مخطط التحجيم" ، والذي يوضح احتمال التلامس (لكل ذراع كروموسوم) كدالة للمسافة الجينومية. وجدنا أن استخدام روابط متشابكة وإنزيمات مختلفة يمكن أن يغير الاضمحلال المعتمد على المسافة على مسافات طويلة وقصيرة المدى17. تزيد إضافة تشابك DSG من إمكانية اكتشاف التفاعلات على مسافات قصيرة عند دمجها مع إنزيمات مثل Mnase ومجموعات من DpnII-DdeI التي تنتج شظايا أصغر (الشكل 6 أ).
يمكن أيضا ملاحظة الاضمحلال المعتمد على المسافة مباشرة من مصفوفات التفاعل 2D: تصبح التفاعلات أكثر تواترا عندما تقع بعيدا عن القطر المركزي (الشكل 6B). بالإضافة إلى ذلك ، يمكن تحديد ميزات الطي الجينومية ، مثل المقصورات ، و TADs ، والحلقات من مصفوفات Hi-C ومخططات القياس كانحرافات عن متوسط الاضمحلال العام المعتمد على المسافة على مستوى الجينوم. الأهم من ذلك ، أن التشابك مع DSG بالإضافة إلى FA يقلل من الروابط العشوائية ، والتي تكون غير مقيدة بسبب طبيعة البوليمر للكروموسومات ، وبالتالي ، من المرجح أن تحدث بين الكروموسومات (عبر) (الشكل 6C). يؤدي تقليل الربط العشوائي إلى زيادة نسب الإشارة إلى الضوضاء ، خاصة بالنسبة للتفاعلات داخل الكروموسومات والتفاعلات طويلة المدى جدا (>10-50 ميجا بايت).

الشكل 6: النتائج التمثيلية لمكتبات Hi-C المعينة والمفلترة. (أ) مخططات القياس مع احتمال الاتصال ومشتقاته لمختلف الإنزيمات ، مرتبة حسب طول الجزء (أعلى) والتشابك مع FA أو FA + DSG (أسفل). الهضم مع MNAse (microC) أو DpnII-DdeI (Hi-C 3.0) يزيد بشكل كبير من جهات الاتصال قصيرة المدى (أعلى) كما تفعل إضافة DSG إلى FA (أسفل). (B) تظهر الأعمدة خرائط حرارية Hi-C لهضم DpnII بعد تشابك FA فقط وهضم DpnII أو DdeI بعد الارتباط المتشابك FA + DSG. تظهر الأسهم البيضاء قوة متزايدة من "النقاط" بعد التشابك DSG وهضم DdeI ، مما يعني الكشف بشكل أفضل عن حلقات الحمض النووي. تظهر الصفوف أجزاء مختلفة من الكروموسوم 3 بدقة متزايدة ، محاذاة مع اللوحة C: الصف العلوي: الكروموسوم 3 بأكمله (0-198,295,559 ميجا بايت) ؛ الصف الأوسط: 186-196 ميجا بايت ؛ الصف السفلي: 191.0-191.5 ميجا بايت. (ج) الرسوم البيانية لتغطية المناطق الموضحة في A. تظهر الأسهم السوداء التغطية الأقل (٪ قراءات رابطة الدول المستقلة) للربط المتشابك FA فقط. الاختصارات: FA = الفورمالديهايد ؛ DSG = غلوتارات ديسوسينيميديل ؛ CHR = كروموسوم. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
ليست كل القراءات المعينة مفيدة. مؤشر الجودة الثاني هو عدد تكرارات PCR. من غير المرجح أن تحدث القراءات المكررة الدقيقة عن طريق الصدفة بعد الربط والصوتنة. وبالتالي ، من المحتمل أن تكون هذه القراءات ناتجة عن تضخيم تفاعل البوليميراز المتسلسل وتحتاج إلى تصفية. غالبا ما تنشأ التكرارات عندما تكون هناك حاجة إلى عدد كبير جدا من دورات PCR لتضخيم المكتبات منخفضة التعقيد. بشكل عام ، بالنسبة إلى Hi-C ، تحتاج معظم المكتبات فقط إلى 5-8 دورات من تضخيم PCR النهائي ، على النحو الذي يحدده تفاعل البوليميراز المتسلسل بالتحليل الحجمي (انظر الخطوة 3.9 ؛ الشكل 4). ومع ذلك ، يمكن الحصول على مكتبات ذات تعقيد كاف حتى بعد 14 دورة من تضخيم تفاعل البوليميراز المتسلسل.
يمكن أن تنشأ فئة أخرى من القراءات المكررة ، ما يسمى بالتكرارات الضوئية ، من عملية التضخيم على منصات تسلسل Illumina التي تستخدم خلايا تدفق منقوشة (مثل HiSeq4000). تم العثور على التكرارات الضوئية إما من التحميل الزائد لخلية التدفق ، مما يتسبب في تسمية المجموعات (الكبيرة) بمجموعتين منفصلتين ، أو من إعادة التجميع المحلي للجزيء الأصلي المزدوج بعد الجولة الأولى من تفاعل البوليميراز المتسلسل. نظرا لأن كلا النوعين من التكرارات الضوئية محليان ، يمكن تحديدهما وتمييزهما عن تكرارات PCR من خلال موقعهما على خلية التدفق. في حين أن المكتبات التي تحتوي على >15٪ من نسخ PCR المكررة ستحتاج إلى تجديد ، يمكن إعادة تحميل المكتبات ذات التكرارات الضوئية بعد تحسين عملية التحميل.
الجدول التكميلي S1: المخازن المؤقتة والحلول. الرجاء الضغط هنا لتحميل هذا الجدول.
Discussion
الخطوات الحاسمة للتعامل مع الخلايا
على الرغم من أنه من الممكن استخدام عدد أقل من خلايا الإدخال ، فقد تم تحسين هذا البروتوكول ل ~ 5 × 106 خلايا لكل حارة تسلسل (~ 400 M قراءة) لضمان التعقيد المناسب بعد التسلسل العميق. من الأفضل حساب الخلايا قبل التثبيت. لإنشاء مكتبات فائقة العمق ، نضرب بشكل عام عدد الممرات (والخلايا) حتى يتم الوصول إلى عمق القراءة المطلوب. للتثبيت الأمثل ، يجب استبدال الوسط المحتوي على المصل ب PBS قبل تثبيت FA ، ويجب إضافة المحاليل المثبتة على الفور وبدون تدرجات تركيز15,22. بالنسبة لحصاد الخلايا ، يفضل الكشط على التربسين ، لأن الانتقال من الشكل المسطح إلى الشكل الكروي بعد التربسين يمكن أن يؤثر على التشكل النووي. بعد إضافة DSG ، تضيع كريات الخلايا السائبة والمتكتلة بسهولة. كن حذرا عند التعامل مع الخلايا في هذه المرحلة وأضف ما يصل إلى 0.05٪ BSA لتقليل التكتل.
تعديلات على الطريقة
تم تطوير هذا البروتوكول باستخدام الخلايا البشرية17. ومع ذلك ، بناء على الخبرة في التقاط تكوين الكروموسوم ، يجب أن يعمل هذا البروتوكول مع معظم الخلايا حقيقية النواة. للحصول على مدخلات أقل بكثير (~ 1 × 106 خلايا) ، ننصح باستخدام نصف وحدات التخزين لإجراءات التحلل والتقاط التشكيل [الخطوات 2.1-2.4]. سيسمح هذا أيضا بإجراء عزل الحمض النووي [الخطوة 2.5] في جهاز طرد مركزي منضدية مع أنابيب سعة 1.7 مل ، مما قد يحسن التكوير لتركيزات الحمض النووي المنخفضة. سيشير القياس الكمي للحمض النووي (الخطوة 2.6.6) إلى كيفية المضي قدما. بالنسبة للكميات المنخفضة من الحمض النووي المعزول (1-5 ميكروغرام) ، نقترح تخطي اختيار الحجم (الخطوة 3.3) والمضي قدما في إزالة البيوتين بعد تقليل الحجم من 130 ميكرولتر إلى ~ 45 ميكرولتر باستخدام CFU.
تم تطوير هذا البروتوكول خصيصا لضمان بيانات عالية الجودة بعد التشابك اللاحق مع FA و DSG والهضم مع DpnII و DdeI. ومع ذلك ، فإن استراتيجيات التشابك البديلة مثل FA متبوعة ب EGS (إيثيلين جلايكول مكرر (سكسينيميديل سكسينات)) ، والتي تستخدم أيضا في ChIP-seq23 و ChIA-PET24 ، قد تعمل بشكل جيد على قدم المساواة17. وبالمثل ، يمكن استخدام تركيبات إنزيمية مختلفة ، مثل DpnII و HinfI18 أو MboI و MseI و NlaIII19 للهضم. عند تكييف مجموعات الإنزيم ، تأكد من استخدام النيوكليوتيدات البيوتينيل التي يمكنها ملء الأجزاء المتدلية المحددة بطول 5 بوصات واستخدام أفضل المخازن المؤقتة لكل كوكتيل. يأتي DpnII مع المخزن المؤقت الخاص به وتوصي الشركة المصنعة للإنزيم بمخزن مؤقت محدد لهضم DdeI. ومع ذلك ، بالنسبة للهضم المزدوج مع DpnII و DdeI في هذا البروتوكول ، يوصى باستخدام Restriction Buffer لأنه مصنف بنشاط 100٪ لكلا الإنزيمين.
استكشاف أخطاء التقاط التشكل وإصلاحها
تم تنفيذ الخطوات الرئيسية الثلاث في التقاط التشكل الكروموسومي: التشابك ، والهضم ، والربط قبل أن يتم تصور النتائج على الهلام. لتحديد جودة كل خطوة من هذه الخطوات الثلاث وتمييز المكان الذي يمكن أن تنشأ فيه المشاكل ، يتم أخذ القسمة قبل (CI) وبعد الهضم (DC) وتحميلها على الجل جنبا إلى جنب مع عينة Hi-C المربوطة (الشكل 2). يستخدم هذا الجل لتحديد جودة عينة Hi-C وما إذا كان الأمر يستحق الاستمرار في البروتوكول. بدون CI و DC ، من الصعب تحديد الخطوة (الخطوات) المحتملة دون المستوى الأمثل. تجدر الإشارة إلى أن الربط دون المستوى الأمثل قد يكون بسبب مشكلة في الربط نفسه ، أو التعبئة ، أو مشكلة في الربط المتشابك. لاستكشاف أخطاء الارتباط المتشابك وإصلاحها ، تأكد من عدم استخدام أكثر من 1 × 107 خلايا لكل مكتبة وابدأ بكواشف ربط جديدة وخلايا نظيفة (أي شطفها باستخدام PBS). للربط ، تأكد من حفظ الخلايا وخليط الربط على الجليد. أضف T4 DNA ligase قبل حضانة 4 ساعات عند 16 درجة مئوية واخلطه جيدا.
استكشاف أخطاء إعداد المكتبة وإصلاحها
إذا كانت هناك حاجة إلى أكثر من 10 دورات PCR أو لا يمكن رؤية منتج PCR على الجل بعد معايرة PCR (الشكل 4) ، فهناك بعض الخيارات لحفظ عينة Hi-C. بالعودة من معايرة PCR ، فإن الخيار الأول هو تجربة PCR مرة أخرى. إذا كان لا يزال هناك منتج غير كاف ، فمن الممكن تجربة جولة أخرى من ربط الذيل والمحول (الخطوة 3.6) بعد غسل الخرز مرتين باستخدام 1x TLE buffer. بعد هذا الربط الإضافي للذيل A والمحول، يمكن للمرء المتابعة إلى معايرة تفاعل البوليميراز المتسلسل كما كان من قبل. إذا لم يكن هناك منتج حتى الآن ، فإن الخيار الأخير هو صدى الكسر 0.8x من الخطوة 3.3 والمتابعة من هناك.
حدود ومزايا Hi-C3.0
من المهم أن ندرك أن Hi-C هي طريقة قائمة على السكان تلتقط متوسط تكرار التفاعلات بين أزواج المواقع في مجتمع الخلية. تم تصميم بعض التحليلات الحسابية لفصل مجموعات المطابقات عن السكان25 ، ولكن من حيث المبدأ ، فإن Hi-C أعمى عن الاختلافات بين الخلايا. على الرغم من أنه من الممكن إجراء Hi-C 26,27 أحادي الخلية ويمكن إجراء الاستدلالات الحسابية28 ، إلا أن Hi-C أحادي الخلية غير مناسب للحصول على معلومات 3C فائقة الدقة. هناك قيد إضافي على Hi-C وهو أنه يكتشف فقط التفاعلات الزوجية. للكشف عن تفاعلات جهات الاتصال المتعددة ، يمكن للمرء إما استخدام قواطع متكررة مقترنة بتسلسل القراءة القصيرة (Illumina)16 أو إجراء اتصالات متعددة 3C29 أو 4C30 ، باستخدام تسلسل القراءة الطويلة من منصات PacBio أو Oxford Nanopore . كما تم تطوير مشتقات Hi-C للكشف على وجه التحديد عن الاتصالات بين وعلى طول الكروماتيدات الشقيقة31,32.
على الرغم من أنه يمكن استخدام Hi-C19 و Micro-C33 لإنشاء خرائط اتصال بدقة subkilobase ، إلا أن كلاهما يتطلب قدرا كبيرا من قراءات التسلسل ويمكن أن يصبح هذا مهمة مكلفة. للوصول إلى دقة مماثلة أو حتى أعلى دون تكاليف ، يمكن تطبيق التخصيب لمناطق جينومية محددة (capture-C 34) أو تفاعلات بروتينية محددة (ChiA-PET 35 ، PLAC-seq36 ، Hi-ChIP37). تكمن قوة وسلبيات تطبيقات التخصيب هذه في أنه يتم أخذ عينات من عدد محدود فقط من التفاعلات. مع مثل هذه التخصيب ، يتم فقدان الجانب العالمي من Hi-C (وخيار التطبيع العالمي).
الأهمية والتطبيقات المحتملة ل Hi-C3.0
تم تصميم هذا البروتوكول لتمكين 3C عالي الدقة وعميق للغاية مع اكتشاف ميزات الطي واسعة النطاق في نفس الوقت مثل TADs والمقصورات17 (الشكل 6). يبدأ هذا البروتوكول ب 5 × 106 خلايا لكل أنبوب لكل مكتبة Hi-C ، والتي يجب أن تكون أكثر من مادة كافية لتسلسل ممر واحد أو ممرين على خلية تدفق للحصول على ما يصل إلى 1 مليار قراءة نهاية مزدوجة. للتسلسل فائق العمق ، يجب تحضير أنابيب متعددة من 5 × 106 خلايا ، اعتمادا على عدد القراءات المعينة ونسخ PCR. عند أعلى دقة (<1 كيلوبايت) ، توجد تفاعلات التكرار في الغالب بين مواقع CTCF ، ولكن يمكن أيضا اكتشاف تفاعلات المروج والمحسن. يمكن للقراء الرجوع إلى Akgol Oksuz et al.17 للحصول على وصف مفصل لتحليل البيانات.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
نود أن نشكر دينيس لافونتين على تطوير البروتوكول وسيرجي فينيف على المساعدة المعلوماتية الحيوية. تم دعم هذا العمل بمنحة من برنامج 4D Nucleome التابع للصندوق المشترك للمعاهد الوطنية للصحة إلى JD (U54-DK107980 ، UM1-HG011536). جي دي هو محقق في معهد هوارد هيوز الطبي.
تخضع هذه المقالة لسياسة الوصول المفتوح إلى المنشورات الخاصة ب HHMI. منح رؤساء مختبرات HHMI سابقا ترخيص CC BY 4.0 غير حصري للجمهور وترخيص من الباطن ل HHMI في مقالاتهم البحثية. وفقا لهذه التراخيص، يمكن إتاحة مخطوطة هذه المقالة المقبولة من قبل المؤلف مجانا بموجب ترخيص CC BY 4.0 فور نشرها.
Materials
| Name | Company | Catalog Number | Comments |
| 1 kb Ladder | New England Biolabs | N3232L | |
| Agarose | Invitrogen | 16500100 | |
| Agencourt AMPure XP magnetic beads , 60 mL | Beckman Coulter | A63881 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit (CFU) | EMD Millipore | UFC500396 | |
| Annealing Buffer (5x) | See recipe in supplemental materials | ||
| ATP 10 mM | ThermoFisher | R0441 | |
| Avanti J-25i High Speed Refrigerated ultra-centrifuge | Beckman Coulter | ||
| beckman ultracentrifuge tube 35 mL | Beckman Coulter | 357002 | |
| Binding Buffer (2x) | See recipe in supplemental materials | ||
| biotin-14-dATP 0.4 mM | Invitrogen | 19524-016 | |
| BSA 10 mg/mL | New England Biolabs | B9000S | dilute from 20 mg/mL |
| Cell scraper | Falcon | 353089 | |
| Cell scraper | Corning | 3008 | |
| Conical polypropylene tubes 50 mL | Denville | C1062-P | |
| Conical tube 15 mL | Denville | C1017-P | |
| Covaris micro tube AFA fiber with snap-cap 130 µL | Covaris | 520045/520077 | |
| Covaris Sonicator | Covaris | E220/E220evolution/M220 | |
| Culture flask 175 cm2 | Falcon | 353112 | |
| Culture plates 150 mm x 25 mm | Corning | 430599 | |
| dATP 1 mM | Invitrogen | 56172 | |
| dATP 10 mM | Invitrogen | 56172 | |
| dCTP 10 mM | Invitrogen | 56173 | |
| DdeI | New England Biolabs | R0175L | |
| dGTP 10 mM | Invitrogen | 56174 | |
| DMSO | Sigma | D2650-5x10ML | |
| dNTP mix 25 mM | Invitrogen | 10297117 | |
| Dounce homogenizer | DWK Life Sciences | 8853010002/8853030002 | |
| DPBS | Gibco | 14190-144 | |
| DpnII | New England Biolabs | R0543M | |
| DSG | ThermoScientific | 20593 | |
| dTTP 10 mM | Invitrogen | 56175 | |
| Ethanol 70% | Fisher | A409-4 | Diluted from 100% |
| Ethidium Bromide | Fisher | BP1302-10 | |
| Formaldehyde (37%) | Fisher | BP531-500 | |
| Gel loading dye (6x ) | New England Biolabs | B7024S | |
| Glycine in ultrapure water 2.5 M | Sigma | G8898-1KG | |
| HBSS | Gibco | 14025-092 | |
| Igepal CA-630 detergent | MP Biomedicals | 198596 | |
| Klenow DNA polymerase 5 U/µL | New England Biolabs | M0210L | |
| Klenow Fragment 3-->5’ exo-, 5 U/µL | New England Biolabs | M0212L | |
| ligation buffer (10x) | New England Biolabs | B7203S | |
| Liquid nitrogen | |||
| LoBind microcentrifuge tube 1.7 mL | Eppendorf | 22431021 | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | |
| Lysis buffer | See recipe in supplemental materials | ||
| Magnetic Particle separator | ThermoFisher | 12321D | |
| Microfuge tubes 1.7 mL | Axygen | MCT-175-C | |
| MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| NEBuffer 2.1 (10x) | New England Biolabs | B7002S | |
| NEBuffer 3.1 (10x) | New England Biolabs | B7203S | |
| PBS | Gibco | 70013-032 | |
| PCR (strip) tubes | Biorad | TBS0201/ TCS0803 | |
| PCR thermocycler | Biorad | T100 | |
| Pfu Ultra II Buffer (10x) | Agilent | Comes with Pfu Ultra | |
| PfuUltra II Fusion HS DNA Polymerase | Agilent | 600674 | |
| Phase lock tube 15 mL | Qiagen | 129065 | |
| Phase lock tubes 2 mL | Qiagen | 129056 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593-049 | |
| Protease inhibitor cocktail | ThermoFisher | 78440 | |
| Proteinase K in ultrapure water 10 mg/mL | Invitrogen | 25530-031 | |
| Refrigerated Centrifuge | Eppendorf | 5810R | |
| RNase A, DNase and protease-free 10 mg/mL | Thermo Scientific | EN0531 | |
| Rotator | Argos technologies | EW-04397-40 or rocking platform | |
| SDS 1% | Fisher | BP13111 | |
| Sodium acetate pH = 5.2, 3 M | Sigma | ||
| Sub-Cell GT Horizontal Electrophoresis System | Biorad | 1704401 | |
| T4 DNA ligase 1 U/µL | Invitrogen | 100004817 | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase 3 U/µL | New England Biolabs | M0203L | |
| T4 ligation buffer (5x) | Invitrogen | Y90001 | |
| T4 polynucleotide kinase 10 U/µL | New England Biolabs | M0201L | |
| Tabletop centrifuge | Eppendorf | 5425 | |
| TBE buffer | See recipe in supplemental materials | ||
| Tris Low EDTA Buffer (TLE) | See recipe in supplemental materials | ||
| Triton X-100 (10%) | Sigma | 93443 | |
| Truseq adapter oligos | Integrated DNA Technologies (IDT)) | https://www.idtdna.com/site/order/oligoentry | 250 nmole and HPLC purified |
| Tween 20 detergent | Fisher | 9005-64-5 | |
| Tween Wash Buffer | See recipe in supplemental materials | ||
| Vortex | Scientific Industries | (G560)SI-0236 |
References
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- Kalhor, R., Tjong, H., Jayathilaka, N., Alber, F., Chen, L. Genome architectures revealed by tethered chromosome conformation capture and population-based modeling. Nature Biotechnology. 30 (1), 90-98 (2012).
- Hsieh, T. H., et al. Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162 (1), 108-119 (2015).
- Hsieh, T. -H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Rodley, C. D., Bertels, F., Jones, B., O’Sullivan, J. M. Global identification of yeast chromosome interactions using Genome conformation capture. Fungal Genetics and Biology. 46 (11), 879-886 (2009).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Alipour, E., Marko, J. F. Self-organization of domain structures by DNA-loop-extruding enzymes. Nucleic Acids Research. 40 (22), 11202-11212 (2012).
- Lajoie, B. R., Dekker, J., Kaplan, N. The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods. 72, 65-75 (2015).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- Golloshi, R., Sanders, J. T., McCord, R. P. Iteratively improving Hi-C experiments one step at a time. Methods. 142, 47-58 (2018).
- Darrow, E. M., et al. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture. Proceedings of the National Academy of Sciences of the United States of America. 113 (31), 4504-4512 (2016).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Ghuryeid, J., et al. Integrating Hi-C links with assembly graphs for chromosome-scale assembly. PLoS Computational Biology. 15 (8), 1007273(2019).
- Gu, H., et al. Fine-mapping of nuclear compartments using ultra-deep Hi-C shows that active promoter and enhancer elements localize in the active A compartment even when adjacent sequences do not. bioRxiv. , (2021).
- Ramani, V., et al. Mapping 3D genome architecture through in situ DNase Hi-C. Nature Protocols. 11 (11), 2104-2121 (2016).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved Protocol for Genome-Wide Chromosome Conformation Capture. Current Protocols. 1 (7), 198(2021).
- Belton, J. -M. M., et al. Hi-C: A comprehensive technique to capture the conformation of genomes. Methods. 58 (3), 268-276 (2012).
- Truch, J., Telenius, J., Higgs, D. R., Gibbons, R. J. How to tackle challenging ChIP-Seq, with long-range cross-linking, Using ATRX as an example. Methods in Molecular Biology. 1832, Clifton, N.J. 105-130 (2018).
- Wang, P., et al. In situ chromatin interaction analysis using paired-end tag sequencing. Current Protocols. 1 (8), 174(2021).
- Tjong, H., et al. Population-based 3D genome structure analysis reveals driving forces in spatial genome organization. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1663-1672 (2016).
- Nagano, T., et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547 (7661), 61-67 (2017).
- Ramani, V., et al. Massively multiplex single-cell Hi-C. Nature Methods. 14 (3), 263-266 (2017).
- Meng, L., Wang, C., Shi, Y., Luo, Q. Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data. Nature Communications. 12 (1), 4369(2021).
- Tavares-Cadete, F., Norouzi, D., Dekker, B., Liu, Y., Dekker, J. Multi-contact 3C reveals that the human genome during interphase is largely not entangled. Nature Structural & Molecular Biology. 27 (12), 1105-1114 (2020).
- Vermeulen, C., et al. Multi-contact 4C: long-molecule sequencing of complex proximity ligation products to uncover local cooperative and competitive chromatin topologies. Nature Protocols. 15 (2), 364-397 (2020).
- Oomen, M. E., Hedger, A. K., Watts, J. K., Dekker, J. Detecting chromatin interactions between and along sister chromatids with SisterC. Nature Methods. 17 (10), 1002-1009 (2020).
- Mitter, M., et al. Conformation of sister chromatids in the replicated human genome. Nature. 586 (7827), 139-144 (2020).
- Krietenstein, N., et al. Ultrastructural Details of Mammalian Chromosome Architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462 (7269), 58-64 (2009).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved