Method Article
길이 척도에 걸친 염색체 형태 포착
요약
Hi-C 3.0은 포름알데히드 및 디숙신이미딜글루타레이트 가교결합제를 DpnII 및 DdeI 제한 효소의 칵테일과 결합하여 신호 대 잡음비와 염색질 상호 작용 감지의 분해능을 높이는 개선된 Hi-C 프로토콜입니다.
초록
염색체 형태 캡처(3C)는 3차원 염색질 상호 작용을 감지하는 데 사용됩니다. 일반적으로 포름알데히드(FA)와의 화학적 가교는 염색질 상호 작용을 수정하는 데 사용됩니다. 그런 다음 제한 효소를 사용한 염색질 분해와 단편 말단의 후속 재생은 3차원(3D) 근접성을 고유한 결찰 산물로 변환합니다. 마지막으로, 가교결합의 역전, 단백질 제거 및 DNA 분리 후, DNA를 전단하고 고처리량 시퀀싱을 위해 준비합니다. 유전자좌 쌍의 근접 결찰 빈도는 세포 집단의 3 차원 공간에서의 공동 국소화 빈도의 척도입니다.
시퀀싱된 Hi-C 라이브러리는 모든 유전자좌 쌍 간의 상호 작용 빈도에 대한 게놈 전체 정보를 제공합니다. Hi-C의 분해능과 정밀도는 염색질 접촉과 염색질의 빈번하고 균일한 단편화를 유지하는 효율적인 가교에 의존합니다. 이 논문에서는 두 개의 가교제(포름알데히드[FA] 및 디숙신이미딜 글루타레이트[DSG])를 결합한 후 두 가지 제한 효소(DpnII 및 DdeI)를 사용하여 더 미세하게 분해하여 가교 효율성을 높이는 개선된 현장 Hi-C 프로토콜인 Hi-C 3.0에 대해 설명합니다. Hi-C 3.0은 루프 및 토폴로지 연관 도메인(TAD)과 같은 더 작은 규모에서 게놈 접힘 특징과 구획과 같은 더 큰 핵 전체 규모의 특징을 정확하게 정량화하기 위한 단일 프로토콜입니다.
서문
염색체 형태 포착은 2002 년부터 사용되었습니다1. 기본적으로 모든 형태 포착 변형은 3D 염색질 조직을 보존하기 위해 DNA-단백질 및 단백질-단백질 상호 작용의 고정에 의존합니다. 그 다음에는 일반적으로 제한 소화에 의한 DNA 단편화가 뒤따르고, 마지막으로 주변 DNA의 종교화가 말단에 이르러 공간적으로 근위 유전자좌를 고유한 공유 DNA 서열로 변환합니다. 초기 3C 프로토콜은 PCR을 사용하여 특정 "일대일" 상호 작용을 샘플링했습니다. 후속 4C 분석은 "일대일" 상호작용2의 검출을 허용한 반면, 5C는 "다대다" 상호작용3을 검출하였다. 염색체 형태 캡처는 차세대 고처리량 시퀀싱(NGS)을 구현한 후 완전한 결실을 맺었으며, 이를 통해 게놈 전체의 Hi-C4 및 3C-seq5, TCC6 및 Micro-C 7,8과 같은 유사한 기술을 사용하여 "올-투-올" 게놈 상호 작용을 검출할 수 있었습니다(Denker 및 De Laat9의 검토 참조).
Hi-C에서 비오틴화 뉴클레오티드는 소화 후 및 결찰 전에 5' 돌출부를 표시하는 데 사용됩니다(그림 1). 이를 통해 스트렙타비딘 코팅 비드를 사용하여 적절하게 소화되고 재생산된 단편을 선택할 수 있어 GCC10과 차별화됩니다. Hi-C 프로토콜에 대한 중요한 업데이트는 가짜 결찰 생성물을 줄이기 위해 손상되지 않은 핵(즉, 제자리)에서 소화 및 알코올화를 수행한 Rao et al.11에 의해 구현되었습니다. 또한 HindIII 분해를 MboI (또는 DpnII) 분해로 대체하면 단편 크기가 감소하고 Hi-C의 분해능 잠재력이 증가했습니다. 이러한 증가는 상대적으로 작은 스케일의 구조의 검출 및 작은 시스-요소 사이의 DNA 루프, 예를 들어, 루프 압출(11,12)에 의해 생성된 CTCF-결합 부위 사이의 루프와 같은 접촉점의 보다 정확한 게놈 국소화를 가능하게 하였다. 그러나 이러한 잠재력에는 대가가 따릅니다. 첫째, 해상도가 2배 증가하려면 시퀀싱 읽기13이 4배(22) 증가해야 합니다. 둘째, 작은 단편 크기는 소화되지 않은 이웃 단편을 소화되고 종교화된 단편(14)으로 착각할 가능성을 증가시킨다. 언급 한 바와 같이, Hi-C에서, 소화 된 단편과 종교화 된 단편은 결찰 접합부에 비오틴이 존재함으로써 소화되지 않은 단편과 다르다. 그러나, 결찰 접합부만 아래로 당겨지도록 하기 위해 의무가 없는 말단에서 적절한 비오틴 제거가 필요하다 14,15.
NGS의 비용이 감소함에 따라 염색체 접힘을 더 자세히 연구하는 것이 가능해졌습니다. DNA 단편의 크기를 감소시키고 그에 따라 분해능을 증가시키기 위해, Hi-C 프로토콜은 더 빈번하게 절단 제한 효소 16을 사용하거나 제한 효소17, 18,19의 조합을 사용하도록 적응될 수 있다. 대안적으로, 마이크로-C 중의 MNase 7,8 및 DNase Hi-C20 중의 DNase는 최적의 소화를 달성하기 위해 적정될 수 있다.
3C 방법의 기초에 대한 최근의 체계적인 평가는 모든 길이 척도에서 염색체 접힘 특징의 검출이 1 % FA와 3mM DSG17을 사용한 순차적 가교로 크게 향상되었음을 보여주었습니다. 또한 HindIII 분해가 가능한 Hi-C는 구획과 같은 대규모 접힘 특징을 감지하는 데 가장 적합한 옵션이었으며 Micro-C는 DNA 루프와 같은 소규모 접힘 특징을 감지하는 데 탁월했습니다. 이러한 결과는 FA 및 DSG 가교결합제의 조합과 DpnII 및 DdeI 엔도뉴클레아제21을 사용한 이중 분해를 사용하는 단일 고해상도 "Hi-C 3.0" 전략의 개발로 이어졌습니다. Hi-C 3.0은 모든 길이 스케일에서 접힘 형상을 정확하게 감지하기 때문에 일반적인 사용에 효과적인 전략을 제공합니다17. Hi-C 3.0 프로토콜의 실험 부분은 여기에 자세히 설명되어 있으며 시퀀싱 후 예상 할 수있는 일반적인 결과가 표시됩니다.

그림 1: 6단계의 Hi-C 절차. 셀은 먼저 FA로 고정 된 다음 DSG (1)로 고정됩니다. 그런 다음 용해는 DdeI 및 DpnII로 이중 소화에 선행합니다 (2). 비오틴은 오버행 필인에 의해 첨가되고 근위 무딘 끝은 DNA 정제 (4) 전에 결찰됩니다 (3). 비오틴은 초음파 처리 및 크기 선택 전에 의무화되지 않은 끝에서 제거됩니다 (5). 마지막으로, 비오틴의 풀다운은 PCR에 의한 어댑터 결찰 및 라이브러리 증폭을 허용합니다(6). 약어 : FA = 포름 알데히드; DSG = 디숙신이미딜글루타레이트; B = 비오틴. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 가교에 의한 고정
- 포름 알데히드 고정 : 단층의 세포에서 시작
- 세포를 적절한 배지에 시딩하여 150mm 플레이트당 5 × 106 개의 세포를 수확하게 합니다.
참고: 사용자는 모든 포유류 세포주의 최적 성장을 보장하는 선호하는 용기를 선택할 수 있습니다. 또한 세포는 조직으로부터 분리 될 수 있습니다. - 150mm 플레이트의 진공 트랩에 연결된 파스퇴르 피펫으로 배지를 흡입하고 ~10mL HBSS로 2회 세척합니다.
- 가교 결합 직전에 22.5mL의 HBSS와 625μL의 37% FA를 최종 1% 농도로 결합하여 50mL 튜브에서 1% FA 가교 용액을 준비합니다. 흔들어서 부드럽게 섞는다.
주의 : 흄 후드를 사용하십시오. 포름 알데히드는 독성이 있습니다. - 세포를 가교결합시키기 위해, 23.125 mL의 1% FA 용액을 각각의 15 cm 플레이트에 붓는다.
- 실온에서 10분 동안 배양하고 2분마다 손으로 접시를 부드럽게 흔듭니다.
- 1.25mL의 2.5M 글리신(최종 128mM)을 추가하고 플레이트를 부드럽게 소용돌이쳐 가교 반응을 켄칭합니다.
- 실온에서 5분 동안 배양하고 가교결합을 중지하기 위해 최소 15분 동안 얼음 위에서 계속 배양합니다.
- 세포 스크레이퍼 또는 고무 경찰관으로 플레이트에서 세포를 긁어냅니다.
- 세포 현탁액을 피펫을 사용하여 50mL 원뿔형 튜브로 옮깁니다. 실온에서 10분 동안 1,000× g 으로 원심분리하고 흡인하여 상청액을 버립니다.
- 세포 펠릿을 Dulbecco의 인산염 완충 식염수(DPBS) 10mL로 한 번 세척하고 피펫을 사용하여 재현탁합니다. 이어서, 실온에서 10 분 동안 1,000×g으로 원심분리한다. 즉시 DSG 가교로 진행합니다.
알림: 셀 펠릿이 느슨해지고 셀이 손실될 수 있으므로 셀 펠릿을 세척할 때 주의하십시오.
- 세포를 적절한 배지에 시딩하여 150mm 플레이트당 5 × 106 개의 세포를 수확하게 합니다.
- 포름 알데히드 고정 : 현탁액의 세포에서 시작
- 세포를 적절한 배지에 파종하여 용기 당 5 × 106 세포를 수확합니다.
참고: 사용자는 모든 포유류 세포주의 최적 세포 성장을 보장하는 선호하는 용기를 선택할 수 있습니다. - 수확 직전에 세포를 세고 5 × 106 세포를 50mL 원추형 튜브로 옮깁니다.
- 실온에서 10 분 동안 300×g에서 원심분리하여 세포를 부드럽게 펠릿화합니다.
- HBSS 45mL에 37% FA 1.25mL를 첨가하여 1% FA 가교 용액을 준비하고 튜브를 여러 번 뒤집어 혼합합니다.
알림: 양을 나누지 않고 전체 1.25mL의 FA를 추가합니다.
주의 : 포름 알데히드는 독성이 강합니다. - 이전 단계에서 준비한 1% FA 가교 용액 46.25mL에 세포 펠릿을 위아래로 피펫팅하여 재현탁합니다.
- 실온에서 회전기, 로커에서 정확히 10분 동안 배양하거나 1-2분마다 튜브를 부드럽게 수동으로 뒤집어 배양합니다.
- 2.5 M 글리신(최종 128 mM 최종) 2.5 mL를 첨가하여 가교 반응을 켄칭하고 튜브를 뒤집어 잘 혼합합니다.
- 실온에서 5분 동안 배양한 다음 얼음 위에서 최소 15분 동안 가교를 완전히 중지합니다.
- 실온에서 원심분리하여 가교결합된 세포를 1,000 × g 에서 10분 동안 펠릿화하고 흡인하여 상청액을 버린다.
- 10mL의 DPBS로 세포를 한 번 씻은 다음 실온에서 1,000 × g 에서 10 분 동안 원심 분리합니다. 피펫을 사용하여 상청액을 완전히 버리고 즉시 DSG 가교결합을 진행한다.
알림: 셀 펠릿이 느슨해지고 셀이 손실될 수 있으므로 셀 펠릿을 세척할 때 주의하십시오.
- 세포를 적절한 배지에 파종하여 용기 당 5 × 106 세포를 수확합니다.
- 디숙신이미딜글루타레이트와의 가교 결합
- 펠렛화된 세포를 9.9mL의 DPBS에 재현탁시킨 후 100μL의 300mM DSG(3mM 최종)를 추가합니다. 반전으로 혼합하십시오.
참고: DSG는 습기에 민감합니다. 가교 당일에 DMSO에서 300mM DSG의 새로운 재고를 준비하는 것이 중요합니다.
주의 : DMSO의 DSG는 독성이 강합니다. - 세포를 실온에서 회전기에서 40분 동안 가교결합시킨다.
- 1.925mL의 2.5M 글리신(최종 400mM)을 첨가하고, 뒤집어 혼합하고, 실온에서 5분 동안 배양한다.
- 실온에서 15 분 동안 2,000×g에서 세포를 원심분리합니다.
알림: 느슨한 세포 펠릿에서 상청액을 제거할 때 주의하십시오. - 펠릿을 0.05% 소 혈청 알부민(BSA)-DPBS 1mL에 재현탁하고 1.7mL 튜브로 옮깁니다.
참고: BSA를 추가하면 세포 응집을 줄이는 데 도움이 될 수 있습니다. - 세포를 2,000 × g 에서 4°C에서 15분 동안 원심분리하고, 피펫으로 상청액을 제거한다.
알림: 펠릿 손실을 방지하려면 상청액을 빠르고 완벽하게 제거하십시오. - 펠릿을 액체 질소에 스냅 동결하고 -80 ° C에서 보관하거나 즉시 다음 단계로 진행하십시오.
- 펠렛화된 세포를 9.9mL의 DPBS에 재현탁시킨 후 100μL의 300mM DSG(3mM 최종)를 추가합니다. 반전으로 혼합하십시오.
2. 염색체 형태 포착
- 세포 용해 및 염색질 소화
- 10μL의 프로테아제 억제제 칵테일을 포함하는 1mL의 얼음-냉장 용해 완충액(보충 표 S1의 레시피)에 가교결합된 세포 분취량(~5 ×10 6개 세포)을 재현탁(피펫)하고 dounce 균질화기로 옮겨 얼음 위에서 15분 배양합니다.
알림: 사용 직전에 용해 완충액에 프로테아제 억제제를 추가하십시오. - 유봉 A를 천천히 위아래로 30 번 움직여 얼음 위에서 세포를 균질화하고 얼음 위에서 1 분 동안 배양하여 세포가 식을 수 있도록하고 30 회 더 스트로크합니다.
- 용해물을 1.7mL 미세 원심분리 튜브로 옮깁니다.
참고: 때때로 세포가 피펫 팁에 달라붙기 때문에 현탁액을 계속 움직이십시오. - 용해된 현탁액을 실온에서 5 분 동안 2,500×g으로 원심분리합니다.
- 상청액을 버리고 젖은 펠릿을 튕기거나 와동시켜 다시 부유시킵니다. 가능한 한 많은 상청액을 제거하여 최소한의 덩어리로 요구르트와 같은 물질을 얻으십시오.
- 펠릿을 500μL의 얼음처럼 차가운 1x 제한 버퍼(10x부터, 보충 표 S1의 레시피 참조)에 재현탁하고 2,500×g에서 5분 동안 원심분리 합니다. 두 번째 세척을 위해이 단계를 반복하십시오.
알림: 1x 제한 버퍼의 펠릿은 용해 완충액의 이전 펠릿보다 더 세분화되어 있습니다. - 세포 크기에 따라 달라지는 펠릿의 캐리오버 부피에 ~340μL를 추가한 후 피펫팅을 통해 1x 제한 버퍼의 최종 부피 360μL에 세포를 재현탁합니다.
- 염색질 무결성(CI)을 테스트하기 위해 각 용해물 18μL를 따로 보관합니다. CI 샘플을 4°C에서 보관한다.
- 각 Hi-C 튜브(총 부피 380μL)에 1% 도데실 황산나트륨(SDS) 38μL를 넣고 기포가 생기지 않고 피펫팅하여 조심스럽게 혼합합니다.
주의 : SDS는 독성이 있습니다. - 염색질을 열기 위해 정확히 10분 동안 흔들지 않고 65°C에서 샘플을 배양합니다.
- 즉시 튜브를 얼음 위에 놓고 Triton X-100으로 분해 혼합물을 준비하여 표 1에 설명된 대로 SDS를 담금질합니다.
- 분해 혼합물 107μL를 Hi-C-튜브(총 487μL)에 추가하여 간격 진탕(예: 900rpm, 30초 켜기, 4분 꺼짐)이 있는 열혼합기에서 37°C에서 밤새(~16시간) 염색질을 분해합니다.
참고: 트리톤을 최종 농도 1%로 첨가하면 SDS를 소멸시키는 역할을 합니다.
- 10μL의 프로테아제 억제제 칵테일을 포함하는 1mL의 얼음-냉장 용해 완충액(보충 표 S1의 레시피)에 가교결합된 세포 분취량(~5 ×10 6개 세포)을 재현탁(피펫)하고 dounce 균질화기로 옮겨 얼음 위에서 15분 배양합니다.
- DNA의 비오티닐화 종료
- 밤새 분해한 후 샘플을 65°C로 20분 동안 옮겨 나머지 엔도뉴클레아제 활성을 비활성화합니다.
- 인큐베이션 동안, 표 2에 나타낸 바와 같이 필인 마스터 믹스를 제조한다.
- 배양 후 샘플을 즉시 얼음 위에 놓습니다.
- 각 샘플에 대해 10 μL 분해 제어(DC)를 따로 보관하고 4°C에서 보관하십시오.
- 피펫으로 또는 회전하여 뚜껑에서 응결을 제거하십시오. 각 샘플에 58μL의 비오틴 필인 믹스(총 샘플 부피 535μL)를 추가하고 기포를 형성하지 않고 부드럽게 피펫을 추가합니다.
- 샘플을 23°C에서 4시간 동안 열혼합기에서 인큐베이션합니다(예를 들어, 900rpm, 30초 켜짐, 4분 꺼짐).
- 근위 DNA 단편의 결찰
- 표 3에 나타낸 바와 같이 비오틴 필-인이 인큐베이션되는 동안 라이게이션 믹스를 준비한다.
- 각 샘플에 665μL의 결찰 혼합물을 추가합니다(총 샘플 부피 1,200μL). 피펫팅으로 부드럽게 섞습니다.
- 샘플을 16°C에서 4시간 동안 열혼합기에서 인터벌 쉐이크(예를 들어, 900rpm, 30초 켜짐, 4분 꺼짐)로 인큐베이션한다. 공유 결합 염색질이 포함된 이 샘플을 4°C에서 며칠 동안 보관하십시오.
- 가교의 반전
- 1x Tris Low EDTA(TLE; 보충 표 S1의 레시피 참조)를 사용하여 CI 및 DC 샘플의 부피를 50μL로 가져옵니다.
- 10 μL의 10 mg/mL 프로테이나제 K를 CI 및 DC 샘플에 추가합니다.
- 65°C에서 밤새 간격 진탕(예: 900rpm, 30초 켜짐, 4분 꺼짐)으로 배양합니다. 대안적으로, Hi-C 샘플의 DNA 정제 동안 이들 대조군에 대해 30분 가교결합의 역전을 수행한다.
- 각 Hi-C 샘플에 10mg/mL 프로테이나제 K 50μL를 추가하고 간격 진탕(예: 900rpm, 30초 켜기, 4분 꺼짐)으로 최소 2시간 동안 65°C에서 배양합니다.
- 각 Hi-C 튜브(총 시료량 1,300μL)에 10mg/mL 프로테이나제 K 50μL를 더 추가하고 65°C에서 밤새 배양을 계속합니다. DNA 정제가 이루어질 때까지 4°C에서 보관한다.
참고: 프로테이나제 K 배양을 분할하면 전체 단백질 소화가 보장됩니다.
- DNA 정제
- 튜브를 65 ° C에서 실온으로 식히십시오.
- 각 샘플을 15mL 원뿔형 튜브로 옮기고 각 튜브에 2.6mL(부피 2배)의 페놀:클로로포름:이소아밀 알코올을 추가합니다.
주의 : 페놀 : 클로로포름 : 이소 아밀 알코올은 매우 독성이 강한 자극제이며 잠재적으로 발암 성입니다. - 각 튜브를 1분 동안 볼텍스한 다음 내용물을 15mL 위상 잠금 튜브로 옮깁니다.
- 벤치탑 원심분리기에서 최대 속도(1,500-3,500 × g)로 샘플을 5분 동안 원심분리합니다.
- 수성 상을 35mL 초원심분리 튜브에 조심스럽게 붓고 최종 부피 1,250μL가 되도록 초순수를 추가합니다.
알림: 튜브를 사용하여 사용 가능한 초원심분리기에 맞추거나 여러 개의 미세 원심분리기 튜브로 분할합니다. - 3M 아세트산 나트륨의 1/10부피 (~ 125 μL)를 추가하고 반전으로 잘 섞습니다.
- 각 샘플에 2.5x 부피 (~ 3.4mL)의 얼음처럼 차가운 100 % 에탄올을 첨가하고 얼음처럼 차가운 100 % 에탄올을 첨가하여 초 원심 분리를 위해 튜브의 균형을 맞추고 반전으로 잘 혼합합니다.
- 튜브를 드라이 아이스에서 ~ 15 분 동안 배양하십시오 (응고를 피하십시오).
- 튜브를 18,000 ×g에서 4 ° C에서 30 분 동안 원 심 분리합니다.
알림: 각진 로터의 경우: 펠릿이 있을 위치에 튜브를 표시하십시오. - 피펫을 사용하여 펠릿이 아닌 쪽에서 상청액을 완전히 제거하고 폐기합니다.
알림: 이 시점에서 펠릿이 보여야 하며 다음 단계에서 건조 후 명확하게 보이지 않을 수 있으므로 튜브에 표시할 수 있습니다. - 샘플을 약 10분 동안 또는 눈에 띄게 건조될 때까지 자연 건조합니다.
- 피펫팅 또는 소용돌이를 통해 각 펠릿을 450μL의 1x TLE에 용해시키고 3kDa 분자량 컷오프를 사용하여 0.5mL 원심 필터 장치(CFU)로 옮깁니다.
- CFU를 최대 속도로 10분 동안 원심분리하고 플로우스루를 폐기합니다. 각 초원심분리 튜브를 450μL의 1x TLE로 추가로 세척하고 CFU로 옮겨 다시 세척합니다.
알림: 이 방법으로 CFU를 세척하면 염분 농도를 줄이면서 DNA 손실을 제한합니다. - CFU를 최대 속도로 10분 동안 원심분리하고 플로우스루를 폐기합니다.
- 컬럼에 80μL의 1x TLE를 추가하고 컬럼을 새 수집 튜브로 돌린 후 최대 속도로 2분 동안 원심분리하여 ~100μL의 최종 부피를 얻습니다.
- 각 시료에 1μL의 RnaseA(1mg/mL, 10mg/mL 스톡의 10배 희석)를 추가하고 37°C의 열 블록, 수조 또는 써모믹서에서 최소 30분 동안 배양합니다.
- Rnase 처리 후, 샘플을 37°C에서 제거하고 품질 관리 단계까지 4°C에서 보관한다.
- 염색질 품질, 효소 소화 및 시료 결찰 확인
- CI 및 DC 샘플을 2.4.5단계에서 가교결합을 반전시킨 후 실온으로 냉각시킨다. 그런 다음 사전 회전된 2mL 위상 잠금 튜브로 옮깁니다.
알림: 위상 잠금 내용물이 펠릿으로 원심 분리되었는지 확인하십시오(최대 속도 2분). - 페놀:클로로포름:이소아밀 알코올 200μL를 넣고 1분 동안 볼텍싱하여 샘플을 혼합합니다.
- 최대 속도로 5분 동안 튜브를 원심분리합니다.
- 각 시료(~50μL)의 수성상을 새로운 1.7mL 미세원심분리 튜브로 옮깁니다.
- 1 μL의 Rnase A (1 mg / mL에서)를 추가하고 37 ° C에서 최소 30 분 동안 배양합니다.
- 샘플을 표 4에서 권장하는 대로 0.8% 아가로스 겔 상에 로딩한다.
참고: 품질 관리에서 예상되는 결과는 그림 2에 나와 있습니다. - 젤의 농도계 또는 큐비트 또는 나노드롭을 사용하여 DNA를 정량화합니다.
알림: 정확한 정량화는 프로토콜의 다음 부분에서 올바른 입력 볼륨을 보장합니다. 알려진 수량의 여러 표준을 사용하여 표준 곡선을 구성합니다.
- CI 및 DC 샘플을 2.4.5단계에서 가교결합을 반전시킨 후 실온으로 냉각시킨다. 그런 다음 사전 회전된 2mL 위상 잠금 튜브로 옮깁니다.
표 1: 소화 시약. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 비오틴 필인 시약. *효소를 변경하려면 다른 완충액과 비오티닐화 dNTP가 필요할 수 있습니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 3: 결찰 혼합 시약. 약어 : BSA = 소 혈청 알부민. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 4: 품질 및 크기 선택 평가를 위한 겔 로딩 매개변수. 이 표를 다운로드하려면 여기를 클릭하십시오.
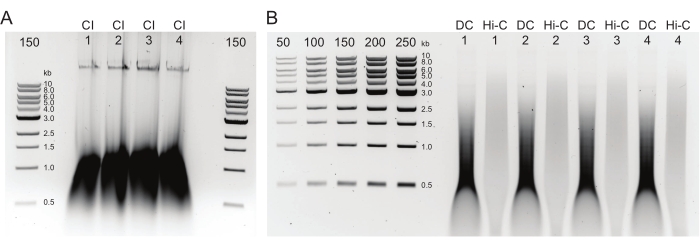
그림 2: 일반적인 DNA 정제 후 품질 관리 결과를 보여주는 아가로스 겔 . (A) CI 대조군은 고 분자량 DNA의 밴드를 나타내야합니다. (B) DC 및 Hi-C 샘플은 다양한 DNA 크기를 보여줍니다. 더 큰 조각으로 결합 된 Hi-C 샘플은 DC보다 분자량이 높아야합니다. 마커의 농도 범위는 표준 곡선을 생성 할 수있게합니다. 이 예에서는 CI가 별도의 젤에로드되었지만 모든 샘플과 대조군을 함께로드하고 실행하는 것이 좋습니다. 약어 : CI = 염색질 무결성; DC = 소화 제어; Hi-C = 근접 결찰. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. Hi-C 시퀀싱 라이브러리 준비
- 의무가없는 끝에서 비오틴 제거
- 표 5와 같이 비오틴 제거 반응을 준비한다.
참고: 일반적으로 10μg의 DNA로 충분하지만 최대 30μg을 사용할 수 있습니다. - 각 130 μL 반응에서 2 x 65 μL 분취량을 2개의 PCR 튜브에 분배합니다.
- 열순환기 또는 PCR 기계로 옮기고 표 5에 설명된 대로 배양합니다.
참고: 샘플은 4 °C (일에서 주), -20 °C (장기간)에서 여기에 저장하거나 즉시 초음파 처리로 옮길 수 있습니다.
- 표 5와 같이 비오틴 제거 반응을 준비한다.
- 쥡니다
- 비오틴 제거 단계 (130 μL 총 샘플 볼륨)에서 샘플 복제본을 초음파 처리를 위해 130 μL 초음파 처리기 튜브에 풀링합니다.
- 표 6에 주어진 매개 변수를 사용하여 샘플을 초음파 처리하여 500bp 미만의 좁은 분포를 달성하십시오.
노트: 다른 초음파 처리기 유형을 사용할 수 있지만 좁은 조각 분포 (100-500 bp)의 경우 초음파 처리기 설정에 최적화가 필요할 수 있습니다.
- 마그네틱 비드로 사이즈 선택
- 초음파 처리기 튜브에서 초음파 처리 된 DNA를 1.7 mL 낮은 결합 튜브로 피펫합니다.
- 1x TLE를 사용하여 각 샘플을 총 부피 500μL로 가져옵니다. 크기 선택에 시료와 마그네틱 비드 혼합물의 비율이 필수적이므로 부피를 가능한 한 500μL에 가깝게 만드십시오.
- 각 튜브에 400μL의 마그네틱 비드 혼합물을 추가하여 0.8의 샘플 부피에 대한 마그네틱 비드 혼합물의 비율을 얻습니다.
참고: 이러한 조건에서 비드는 >300bp의 DNA 단편을 포착하며, 이는 상부 분획이 됩니다. 상청액은 <300 bp의 단편을 포함 할 것이며, 이는 하부 분획이 될 것이다. - 튜브를 볼텍싱하여 혼합하고 회전 장치에서 실온에서 10분 동안 배양합니다. 이 비율과 다른 크기 선택 비율의 경우 샘플의 전체 부피가 잘 혼합되는지 확인하십시오. 일부 로터의 "불규칙한 모드"를 찾으십시오.이 모드는 더 작은 볼륨에 적합합니다.
- 자성 입자 분리기(MPS) 상에서 실온에서 5분 동안 배양한다.
- 배양하는 동안 500μL의 자성 비드 혼합물을 각 샘플에 대해 새로운 1.7μL의 저결합 튜브에 추가합니다.
참고: 이 튜브는 1.1:1의 마그네틱 비드 혼합물 대 샘플 비율을 생성하여 하부 분획의 다음 크기 선택에 사용됩니다. - 튜브를 MPS에 5분 동안 그대로 두십시오.
- 비드로부터 상청액을 제거하고 150 μL의 자성 비드 혼합물로 재현탁시킨다.
참고: 이 단계는 부피를 늘리지 않고 비드 수를 늘려 DNA로 비드가 포화되는 것을 방지합니다. - 단계 3.3.5의 상청액을 준비된 표지된 튜브로 옮겨 하부 분획을 선택한다(단계 3.3.8).
참고: 150μL의 마그네틱 비드 혼합물 + 400μL의 0.8x 마그네틱 비드 혼합물(총 550μL)을 500μL의 초기 샘플로 나눈 값 = 1.1x 마그네틱 비드 혼합물 대 샘플 비율. - 하부 분획 튜브를 볼텍싱하여 혼합하고 회전 장치에서 실온에서 10분 동안 배양합니다.
참고: 비드는 DNA 단편에 >100bp를 결합하여 100-300bp의 최종 비드 결합 분획을 생성합니다. - 하부 분획 튜브를 MPS에 5분(실온) 동안 놓습니다.
- 상청액을 제거하고 튜브를 짧게 원심분리하여 상청액을 최대한 더 제거합니다.
- 200 μL의 70% 에탄올을 사용하여 두 분획으로부터 비드를 두 번 세척하고, 매번 MPS에서 5분 동안 비드를 회수한다.
- 원심 분리기에서 빠르게 회전 한 후 에탄올을 완전히 제거하고 MPS에서 비드를 추가로 건조시킵니다.
알림: 알코올이 완전히 증발 할 때까지 건조하십시오. 펠릿은 깨지지 않고 다크 초콜릿처럼 보일 것입니다(~10분 소요될 수 있음). - 두 분획을 50μL의 1x TLE 완충액에 재현탁시킨다. 실온에서 10분 동안 배양하고 격분으로 튜브를 두드리거나 튕겨 혼합 및 용리를 자극합니다.
- 비드를 MPS 상의 상청액으로부터 분리하여 두 분획 모두에 대해 5분 동안 사용한다.
- 각 샘플에서 상청액을 보관하십시오. 상청액을 1.7mL의 저결합 튜브에 피펫팅합니다.
알림: 샘플은 며칠 동안 4 ° C에서 또는 장기간 -20 ° C에서 보관할 수 있습니다. - 표 4와 같이 2% 아가로스 겔을 실행하여 시료의 질과 양을 결정하였다. 이러한 겔의 예는 그림 3을 참조하십시오.
알림: 경험에서 초음파 처리가 재현성이 높으면이 젤을 건너 뛰고 즉시 수리를 종료 할 수 있습니다. 사용자는 적정 PCR 후까지 상부 분획을 유지하는 것이 좋습니다. 많은 PCR 증폭이 필요한 차선의 DNA 양은 상부 분획의 물질로부터 구출 될 수 있습니다. - 알려진 DNA 래더 입력에서 표준 곡선을 생성한 직후 또는 큐비트 또는 나노드롭을 사용하여 겔에서 DNA의 양을 정량화합니다.
- 수리 종료
- 최종 수리 믹스를 표 7 과 같이 준비한다(주어진 반응당 양).
- 나머지 46μL의 용출된 DNA를 하부 분획에서 PCR 튜브로 옮기고 준비된 최종 복구 믹스 24μL를 추가합니다. 표 7에 제안된 바와 같이 PCR 기계에서 배양한다.
- 프로그램이 완료되면 풀다운될 때까지 샘플을 4°C에 보관합니다.
- 스트렙타비딘 코팅 비드를 사용한 비오티닐화 결찰 제품의 풀다운
- 정량화된 크기 선택으로부터 각 라이브러리에 대한 스트렙타비딘 코팅 비드의 양을 결정한다(단계 3.3.19).
참고: 이 스트렙타비딘 코팅 비드(10mg/mL 용액)는 mg 비드당 20μg의 이중 가닥 DNA에 결합할 수 있습니다(= 20μg/100μL 비드). Hi-C DNA 1μg당 2μL를 사용하되 10μL 이상은 사용하지 마십시오. - 스트렙타비딘 코팅 비드를 혼합하고 각 라이브러리에 필요한 비드의 부피(이전 단계에서 계산)를 개별 1.7mL 저결합 튜브에 피펫합니다.
- 400μL의 트윈 세척 완충액(TWB, 보충 표 S1의 레시피 참조)에 비드를 재현탁하고 회전 장치에서 실온에서 ~3분 동안 배양합니다(3.3.4단계의 지침 참조).
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 제거한다.
- 400μL의 TWB를 추가로 피펫팅하여 비드를 세척합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 제거한다.
- 400μL의 2x 결합 완충액(BB)( 보충 표 S1의 레시피)을 비드에 추가하고 재현탁합니다. 또한 330μL의 1x TLE와 최종 복구의 용액을 추가합니다(3.4.3단계에서).
- 샘플을 회전제에서 혼합하면서 실온에서 15분 동안 배양합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 제거한다.
- 400μL의 1x BB를 비드에 추가하고 재현탁합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 제거한다.
- 1x TLE 100μL를 추가하여 비드를 세척합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 제거한다.
- 마지막으로 41μL의 1x TLE를 추가하여 비드를 재현탁합니다.
- 정량화된 크기 선택으로부터 각 라이브러리에 대한 스트렙타비딘 코팅 비드의 양을 결정한다(단계 3.3.19).
- A-테일링
- 표 8과 같이 A-테일링 믹스를 준비한다.
- 반응물을 PCR 튜브에 넣고 표 8과 같이 배양한다.
- 써모사이클러에서 꺼낸 직후 PCR 튜브를 얼음 위에 놓고 내용물을 1.7mL 저결합 튜브로 옮깁니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 버린다.
- 초순수로 5x T4 DNA 리가아제 완충액에서 희석한 400μL의 1x 결찰 완충액을 추가합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리한 다음, 상청액을 버린다.
- 1x 라이게이션 버퍼를 최종 부피 40μL에 추가합니다.
- 어닐링 어댑터 올리고
- 100 μM의 어댑터 올리고 스톡을 준비합니다(표 9).
참고: HPLC 정제 올리고 250nmole을 주문하십시오. - 어댑터를 표 9에 설명된 대로 PCR 튜브에서 어닐링합니다.
- PCR 열순환기를 사용하여 0.5°C/s에서 97.5°C까지 온도를 점진적으로 높입니다. 97.5 ° C에서 2.5 분 동안 유지하십시오.
- PCR 열순환기를 사용하여 775 사이클 (20 ° C에 도달) 동안 0.1 ° C / s에서 점차적으로 온도를 높입니다. 더 사용할 때까지 온도를 4 ° C로 유지하십시오.
- 83μL의 1x 어닐링 버퍼( 보충 표 S1의 레시피)를 추가하여 어댑터를 15μM로 희석합니다. 어댑터를 -20°C에 보관하십시오.
- 100 μM의 어댑터 올리고 스톡을 준비합니다(표 9).
- 시퀀싱 어댑터 결찰
- 어댑터 결찰 혼합물을 1.7mL 저결합 튜브에 준비합니다(표 10).
- 실온에서 2시간 동안 결찰합니다.
- 1분 동안 MPS 상의 상청액으로부터 비드를 분리하고 상청액을 버린다.
- 400μL의 TWB를 추가하고 실온에서 5분 동안 회전기에서 배양하기 전에 비드를 조심스럽게 위아래로 피펫합니다. MPS의 상청액에서 비드를 분리하고 이 단계를 한 번 더 반복합니다.
- MPS의 상청액에서 비드를 분리하고(~1분), 상청액을 버리고 200μL의 1x BB를 추가합니다.
- MPS의 상청액에서 비드를 분리하고(~1분) 상청액을 버립니다.
- 200μL의 1x pre-PCR 버퍼(10x, 보충 표 S1의 레시피)를 추가하고 새로운 1.7mL 저결합 튜브로 옮깁니다.
- MPS의 상청액에서 비드를 분리하고(~1분) 상청액을 버립니다.
- 20μL의 1x pre-PCR 버퍼를 추가하고 피펫팅으로 혼합합니다.
- 사용 중에는 튜브를 얼음 위에 보관하거나 4 ° C에서 보관하십시오.
- 적정에 의한 PCR 주기 수 최적화
- 표 11과 같이 샘플당 30μL의 마스터 혼합 반응을 설정합니다(주어진 반응당 양).
- 가장 적은 수의 사이클을 실행하고 5μL 분취량을 취합니다. 다음 5 μL 분취량을 취하기 전에 나머지 반응에 대해 추가 2-3 사이클을 실행하십시오. 4 개의 부분 표본을 수집하려면 반복하십시오.
- 각 부분 표본에 대해 표 11 의 PCR 매개 변수를 사용하십시오.
- 각 5μL 샘플에 5μL의 물과 2μL의 6x 염료를 추가합니다. 2-1 ng의 저 분자량 사다리가있는 2 % 아가 로스 TBE 젤 ( 보충 표 S150의 레시피)에서 실행하십시오. 예상 결과는 그림 4 를 참조하십시오.
참고: 최종 라이브러리 PCR 증폭을 위한 최적의 사이클 수[단계 3.10]는 겔에서 가시적인 생성물을 얻기 위한 가장 낮은 사이클 수에서 1사이클을 뺀 값입니다.
- 최종 라이브러리 PCR 증폭
- 표 11과 같이 시퀀싱을 위해 각 최종 라이브러리를 증폭하기 위해 12 x 30 μL 반응을 설정합니다.
- PCR 적정 후 사이클 횟수를 결정한 후 표 11 에 따라 PCR 반응을 순환한다(단계 3.9.3).
- PCR이 완료되면 복제 샘플을 1.7mL 저결합 미세원심분리 튜브에 풀링합니다.
- 튜브를 MPS에 놓고 상청액을 새로운 1.7mL 저결합 미세원심분리 튜브로 옮깁니다.
- 남은 스트렙타비딘 코팅 비드를 20μL의 1x pre-PCR 버퍼에 재현탁합니다.
참고: 이 템플릿은 4°C에서 며칠에서 몇 주 동안 보관하거나 -20°C에서 장기간 보관할 때 재사용할 수 있습니다.
- 마그네틱 비드 혼합물로 프라이머 제거
- 1x TLE 버퍼( 보충 표 S1의 레시피)를 사용하여 3.10.4단계의 부피를 정확히 360μL로 조정합니다.
- 각 샘플에 360μL의 마그네틱 비드 혼합물과 피펫을 위아래로 추가하여 혼합합니다.
- 회전 장치에서 샘플을 실온에서 10분 동안 혼합합니다.
- 실온(3-5분)에서 MPS 상의 상청액으로부터 비드를 분리한다.
- 200 μL의 70% 에탄올을 사용하여 비드를 두 번 세척하고 매번 MPS에서 5분 동안 비드를 회수합니다.
- 원심 분리기에서 빠르게 회전하고 에탄올을 완전히 피펫팅합니다. 에탄올을 더 증발시키기 위해 MPS의 공기 중에서 비드를 건조시킵니다.
알림: 펠릿은 깨지지 않고 다크 초콜릿처럼 보일 것입니다(~10분 소요될 수 있음). - 30μL의 초순수를 넣고 실온에서 10분 동안 DNA를 용리하기 위해 재현탁합니다. 혼합을 돕기 위해 2분마다 튜브를 튕깁니다.
- 5분 동안 MPS의 상청액에서 비드를 분리합니다.
- 새로운 1.7mL 튜브에서 각 샘플의 상청액을 수집합니다.
- 2% 아가로스 TBE( 보충 표 S1의 레시피) 겔에서 1μL의 라이브러리를 실행하여 단편 크기 분포를 얻고 최종 라이브러리를 정량화했습니다(그림 5).
참고: ClaI 분해는 DpnII-DpnII 접합에 대해서만 발생할 수 있으며 최종 라이브러리에 대한 단편 크기 분포를 낮추는 양성 결찰 제어 역할을 합니다. 최종 라이브러리는 4°C에서 며칠 동안, -20°C에서 장기간 보관하거나, 즉시 희석하여 시퀀싱을 위해 제출할 수 있습니다.
표 5: 비오틴 제거 시약 및 온도 이 표를 다운로드하려면 여기를 클릭하십시오.
표 6 : 초음파 처리를위한 매개 변수. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 7: 수리 시약 및 온도 종료. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 8: A-테일링 시약 및 온도. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 9: 어닐링을 위한 시약 어닐링이 있는 PCR 프라이머 및 쌍을 이루는 말단 어댑터 올리고. 약어 : 5PHOS = 5 '인산염. 별표는 인산화 된 DNA 염기를 나타냅니다. # 인덱싱된 올리고를 범용 올리고와 결합하여 인덱싱된 어댑터로 어닐링합니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 10: 어댑터 결찰 시약. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 11: PCR 시약 및 사이클링 파라미터. 이 표를 다운로드하려면 여기를 클릭하십시오.

그림 3: 일반적인 사후 크기 선택 결과를 보여주는 아가로스 겔. DpnII-DdeI Hi-C의 4 개 샘플 (번호 1-4)에 대한 상부 및 하부 분획이 표시됩니다. 각 샘플에 대한 첫 번째 레인은 0.8x 자성 비드 혼합물에서 파생된 상부 분획을 포함하고, 두 번째 및 세 번째 레인은 1.1x 자성 비드 혼합물에서 파생된 하부 분획의 희석액을 포함합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: PCR 적정 결과가 있는 아가로스 겔. PCR의 5 사이클부터 시작하여 4 개의 라이브러리 각각에 대해 2 사이클 (5, 7, 9 및 11 사이클)마다 샘플을 채취합니다. 이 그림에 기초하여, 6 사이클이 각 샘플에 대한 최적 사이클로서 선택되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
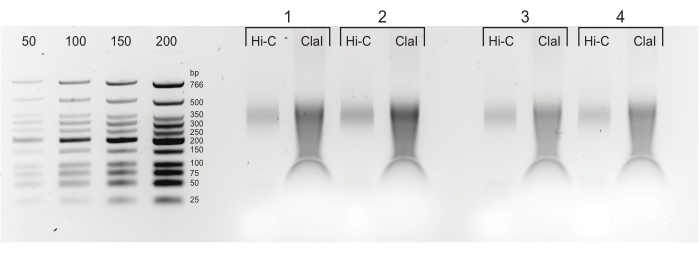
그림 5: 최종 PCR 산물. 세척 및 크기 선택 후, PCR 산물 (Hi-C)을 동일한 라이브러리 (ClaI)의 ClaI-분해된 분획 옆에 로딩하였다. ClaI- 소화 된 단편은 인기있는 DpnII-DpnII 결찰의 존재를 나타낸다. ClaI는 DpnII-DdeI 접합을 소화하지 않으므로 모든 결찰이이 제한에서 크기 감소에 기여하는 것은 아닙니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
이 원고의 수치는 이전에 Lafontaine et al.21에 의해 출판된 것과 별도의 복제 실험에서 생성되었습니다. 고처리량 시퀀싱 데이터를 얻은 후 오픈 크로마틴 집단 (Open2C: https://github.com/open2c)을 사용하여 Hi-C 데이터를 처리했습니다. 유사한 파이프라인은 4D Nucleome 프로젝트(https://data.4dnucleome.org/resources/data-analysis/hi_c-processing-pipeline)의 데이터 포털에서 찾을 수 있습니다. 간단히 말해서, Nextflow 파이프라인 증류기는 (https://github.com/open2c/distiller-nf) (1) Hi-C 분자의 서열을 참조 게놈에 정렬하고, (2) .sam 정렬을 구문 분석하고 Hi-C 쌍으로 파일을 형성하고, (3) PCR 중복을 필터링하고, (4) Hi-C 상호 작용의 빈 매트릭스로 쌍을 집계합니다. 쿨러라고 하는 이러한 HDF5 형식의 매트릭스는 (1) HiGlass 서버(https://higlass.io/)에서 볼 수 있으며, (2) 구획, TAD 및 루프와 같은 접는 특징을 추출하고 정량화하기 위해 Open Chromatin Collective(https://github.com/open2c/cooltools)에서 유지 관리하는 "cooltools" 컬렉션에 있는 대규모 오픈 소스 계산 도구 세트를 사용하여 분석할 수 있습니다.
Hi-C3.0 라이브러리의 일부 품질 지표는 몇 가지 간단한 메트릭/지표를 사용하여 판독 쌍을 참조 게놈에 매핑한 직후에 평가할 수 있습니다. 첫째, 전형적으로 ~50%의 서열화된 판독 쌍이 인간 세포에 대해 고유하게 맵핑될 수 있다. 염색체의 고분자 특성으로 인해 이러한 매핑된 판독의 대부분(~60%-90%)은 염색체(cis) 내의 상호 작용을 나타내며 상호 작용 빈도는 게놈 거리가 증가함에 따라 빠르게 감소합니다(거리 의존적 붕괴). 거리 의존적 붕괴는 게놈 거리의 함수로 접촉 확률(염색체 팔당)을 보여주는 "스케일링 플롯"에서 가장 잘 시각화할 수 있습니다. 우리는 다른 가교제와 효소의 사용이 장거리 및 단거리 거리에서 거리 의존적 붕괴를 변경할 수 있음을 발견했습니다17. DSG 가교결합을 추가하면 Mnase와 같은 효소 및 더 작은 단편을 생성하는 DpnII-DdeI의 조합과 결합할 때 단거리에서 상호 작용의 검출 가능성이 증가합니다(그림 6A).
거리 의존적 감쇠는 2D 상호 작용 매트릭스에서 직접 관찰 할 수도 있습니다 : 상호 작용은 중앙 대각선에서 멀리 떨어져있을 때 더 드물어집니다 (그림 6B). 또한 구획, TAD 및 루프와 같은 게놈 폴딩 특징은 Hi-C 매트릭스 및 스케일링 플롯에서 일반적인 게놈 전체 평균 거리 의존적 붕괴로부터의 편차로 식별 할 수 있습니다. 중요하게도, FA 외에 DSG와의 가교는 염색체의 중합체 특성으로 인해 제한되지 않는 무작위 결찰을 감소시키고, 따라서 염색체 사이(트랜스에서)에서 발생할 가능성이 더 높다는 것입니다(그림 6C). 무작위 결찰을 줄이면 특히 염색체 간 및 매우 긴 범위(>10-50Mb) 염색체 내 상호 작용의 경우 신호 대 잡음비가 증가합니다.

그림 6: 매핑 및 필터링된 Hi-C 라이브러리의 대표적인 결과 . (A) 다양한 효소에 대한 접촉 확률 및 그 유도체가 포함된 스케일링 플롯으로, 단편 길이(상단) 및 FA 또는 FA + DSG와의 가교결합(하단)으로 정렬됩니다. MNAse(마이크로C) 또는 DpnII-DdeI(Hi-C 3.0)를 사용한 분해는 FA(하단)에 DSG를 추가하는 것과 마찬가지로 단거리 접촉(상단)을 크게 증가시킵니다. (B) 열은 FA 가교 후 DpnII 분해 및 FA + DSG 가교 후 DpnII 또는 DdeI 분해의 Hi-C 히트 맵을 보여줍니다. 흰색 화살표는 DSG 가교 및 DdeI 소화 후 "점"의 강도가 증가하는 것을 보여 주며 이는 DNA 루프의 더 나은 검출을 의미합니다. 행은 패널 C와 정렬하여 증가하는 해상도로 염색체 3의 다른 부분을 보여줍니다: 맨 위 줄: 전체 염색체 3(0-198,295,559Mb); 중간 줄 : 186-196 Mb; 맨 아래 줄: 191.0-191.5Mb. (C) A에 표시된 지역에 대한 커버리지 그래프. 검은색 화살표는 FA 전용 가교결합에 대한 낮은 커버리지(%cis 읽기)를 나타냅니다. 약어 : FA = 포름 알데히드; DSG = 디숙신이미딜글루타레이트; CHR = 염색체. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
매핑된 모든 읽기가 유용한 것은 아닙니다. 두 번째 품질 지표는 PCR 중복 수입니다. 정확한 중복 읽기는 결찰 및 초음파 처리 후 우연히 발생할 가능성이 거의 없습니다. 따라서 이러한 판독은 PCR 증폭으로 인한 것일 수 있으며 필터링해야 합니다. 복잡성이 낮은 라이브러리를 증폭하기 위해 너무 많은 PCR 주기가 필요할 때 중복이 자주 발생합니다. 일반적으로 Hi-C의 경우 대부분의 라이브러리는 적정 PCR에 의해 결정된 최종 PCR 증폭의 5-8주기만 필요합니다(3.9단계 참조; 그림 4). 그러나, 충분한 복잡성을 갖는 라이브러리는 PCR 증폭의 14 사이클 후에도 수득 될 수있다.
소위 광학 복제라고 하는 중복 읽기의 또 다른 범주는 패턴화된 플로우 셀(예: HiSeq4000)을 사용하는 Illumina 시퀀싱 플랫폼의 증폭 프로세스에서 발생할 수 있습니다. 광학 중복은 유동 세포에 과부하가 걸려 (큰) 클러스터를 두 개의 개별 클러스터라고 부르거나 첫 번째 PCR 후 원래 쌍을 이루는 말단 분자의 국소 재 클러스터링에서 발견됩니다. 두 가지 유형의 광학 복제는 모두 국소적이기 때문에 유동 세포에서의 위치에 따라 PCR 복제와 식별하고 구별할 수 있습니다. PCR 중복이 >15%인 라이브러리는 재생성이 필요한 반면, 광학 중복이 있는 라이브러리는 로딩 프로세스를 최적화한 후 다시 로드할 수 있습니다.
보충 표 S1: 버퍼 및 용액. 이 표를 다운로드하려면 여기를 클릭하십시오.
토론
세포 취급을 위한 중요한 단계
더 적은 수의 입력 셀을 사용할 수 있지만 이 프로토콜은 딥 시퀀싱 후 적절한 복잡성을 보장하기 위해 시퀀싱 레인당 ~5 × 106개의 셀(~400M 읽기)에 최적화되었습니다. 세포는 고정 전에 가장 잘 계산됩니다. 울트라딥 라이브러리 생성을 위해 일반적으로 원하는 판독 깊이에 도달할 때까지 레인(및 셀)의 수를 곱합니다. 최적의 고정을 위해, FA 고정 전에 혈청 함유 배지를 PBS로 교체해야하며, 고정 용액은 농도 구배15,22없이 즉시 첨가되어야한다. 세포 수확의 경우, 트립신 화 후 더 평평한 모양에서 구형으로의 전환이 핵 형태에 영향을 미칠 수 있기 때문에 트립신 화보다 스크래핑이 선호됩니다. DSG를 첨가 한 후에는 느슨하고 덩어리진 세포 펠릿이 쉽게 손실됩니다. 이 단계에서 셀을 취급할 때 주의하고 응집을 줄이기 위해 최대 0.05% BSA를 추가하십시오.
방법 수정
이 프로토콜은 인간 세포17을 사용하여 개발되었습니다. 그러나 염색체 형태 포획 경험에 비추어 볼 때 이 프로토콜은 대부분의 진핵 세포에서 작동해야 합니다. 상당히 낮은 입력(~1 × 106 셀)의 경우 용해 및 형태 캡처 절차[단계 2.1-2.4]에 볼륨의 절반을 사용하는 것이 좋습니다. 이것은 또한 1.7mL 튜브가 있는 탁상용 원심분리기에서 DNA 분리[단계 2.5]를 수행할 수 있게 하여 낮은 DNA 농도에 대한 펠릿화를 개선할 수 있습니다. DNA의 정량화 (2.6.6 단계)는 진행 방법을 나타냅니다. 소량의 분리 된 DNA (1-5 μg)의 경우 크기 선택 (3.3 단계)을 건너 뛰고 CFU로 부피를 130 μL에서 ~ 45 μL로 줄인 후 비오틴 제거를 진행하는 것이 좋습니다.
이 프로토콜은 FA 및 DSG와의 후속 가교 및 DpnII 및 DdeI와의 분해 후 고품질 데이터를 보장하기 위해 특별히 개발되었습니다. 그러나 FA에 이어 EGS (에틸렌 글리콜 비스 (숙신이 미딜 숙시 네이트))와 같은 대안적인 가교 전략은 ChIP-seq23 및 ChIA-PET24에서도 사용되며17도 똑같이 잘 작동 할 수 있습니다. 유사하게, 상이한 효소 조합, 예컨대 DpnII 및 HinfI18 또는 MboI, MseI, 및 NlaIII19가 소화에 사용될 수 있다. 효소 조합을 조정할 때 특정 5' 오버행을 채울 수 있는 비오티닐화 뉴클레오티드를 사용하고 각 칵테일에 가장 최적의 완충액을 사용해야 합니다. DpnII는 자체 완충액과 함께 제공되며 효소 제조업체는 DdeI 분해를 위한 특정 완충액을 권장합니다. 그러나 이 프로토콜에서 DpnII 및 DdeI를 사용한 이중 분해의 경우 제한 버퍼는 두 효소 모두에 대해 100% 활성으로 평가되기 때문에 권장됩니다.
형태 캡처 문제 해결
염색체 형태 포착의 세 가지 주요 단계인 가교, 소화 및 종교화는 결과를 젤에 시각화하기 전에 모두 수행되었습니다. 이 세 단계 각각의 품질을 결정하고 문제가 발생할 수 있는 부분을 식별하기 위해 분해 전후(CI) 분취량을 가져와 결찰된 Hi-C 샘플과 함께 젤에 로드합니다(그림 2). 이 젤은 Hi-C 샘플의 품질과 프로토콜을 계속할 가치가 있는지 여부를 결정하는 데 사용됩니다. CI 및 DC가 없으면 잠재적인 차선의 단계를 정확히 찾아내기가 어렵습니다. 차선의 결찰은 결찰 자체의 문제, 채우기 또는 가교 문제로 인한 것일 수 있습니다. 가교 문제를 해결하려면 라이브러리당 1개 × 10개 7개 이상의 세포를 사용하지 말고 새로운 가교 시약과 깨끗한 세포(즉, PBS로 헹궈짐)로 시작하십시오. 결찰을 위해 세포와 결찰 혼합물이 얼음 위에 보관되어 있는지 확인하십시오. 16°C에서 4시간 인큐베이션 직전에 T4 DNA 리가아제를 첨가하고 잘 혼합한다.
라이브러리 준비 문제 해결
10회 이상의 PCR 주기가 필요하거나 PCR 적정 후 겔에서 PCR 산물을 볼 수 없는 경우(그림 4), Hi-C 샘플을 저장할 수 있는 몇 가지 옵션이 있습니다. PCR 적정에서 다시 작업하면 첫 번째 옵션은 PCR을 다시 시도하는 것입니다. 여전히 생성물이 충분하지 않은 경우, 비드를 1x TLE 완충액으로 2회 세척한 후 A-테일링 및 어댑터 라이게이션(단계 3.6)의 또 다른 라운드를 시도할 수 있다. 이 추가 A-테일링 및 어댑터 결찰 후 이전과 같이 PCR 적정을 진행할 수 있습니다. 여전히 제품이 없으면 마지막 옵션은 3.3 단계에서 0.8x 분수를 다시 음압하고 거기에서 진행하는 것입니다.
Hi-C3.0의 한계와 장점
Hi-C는 세포 집단에서 유전자좌 쌍 간의 상호 작용의 평균 빈도를 캡처하는 집단 기반 방법이라는 것을 인식하는 것이 중요합니다. 일부 전산 분석은 집단25에서 형태의 조합을 풀도록 설계되었지만 원칙적으로 Hi-C는 세포 간의 차이를 인식하지 못합니다. 단일 셀 Hi-C(26,27)를 수행할 수 있고 계산 추론(28)을 수행할 수 있지만, 단일 셀 Hi-C는 초고해상도 3C 정보를 얻기에 적합하지 않습니다. Hi-C의 추가 제한 사항은 쌍별 상호 작용만 감지한다는 것입니다. 다중 접촉 상호 작용을 감지하기 위해 짧은 읽기 시퀀싱(Illumina)16과 결합된 빈번한 커터를 사용하거나 PacBio 또는 Oxford Nanopore 플랫폼의 긴 읽기 시퀀싱을 사용하여 다중 접촉 3C29 또는 4C30을 수행할 수 있습니다. 자매 염색분체 사이의 접촉을 특이적으로 감지하기 위한 Hi-C 유도체도 개발되었습니다.31,32.
Hi-C19 및 Micro-C33을 사용하여 서브킬로베이스 해상도에서 접촉 맵을 생성할 수 있지만 둘 다 많은 양의 시퀀싱 읽기가 필요하며 이는 비용이 많이 드는 작업이 될 수 있습니다. 비용 없이 유사하거나 더 높은 분해능을 얻기 위해 특정 게놈 영역(캡처-C 34) 또는 특정 단백질 상호 작용(ChiA-PET 35, PLAC-seq36, Hi-ChIP37)에 대한 농축을 적용할 수 있습니다. 이러한 보강 응용 프로그램의 강점과 단점은 제한된 수의 상호 작용만 샘플링된다는 것입니다. 이러한 보강으로 Hi-C의 글로벌 측면 (및 글로벌 정규화 옵션)이 손실됩니다.
Hi-C3.0의 중요성과 잠재적 응용 분야
이 프로토콜은 고분해능, 초심층 3C를 가능하게 하는 동시에 TAD 및 구획(17 )과 같은 대규모 폴딩 기능을 감지하도록 설계되었습니다(그림 6). 이 프로토콜은 각 Hi-C 라이브러리에 대해 튜브당 5개 × 10개6 개의 셀로 시작하며, 이는 최대 10억 개의 쌍 최종 판독을 얻기 위해 플로우 셀에서 하나 또는 두 개의 레인을 시퀀싱하기에 충분한 재료여야 합니다. 초심층 시퀀싱의 경우 매핑된 판독 및 PCR 중복 수에 따라 5 × 106 세포의 다중 튜브를 준비해야 합니다. 최고 해상도(<1kb)에서 루핑 상호작용은 대부분 CTCF 부위 사이에서 발견되지만 프로모터-인핸서 상호작용도 검출할 수 있습니다. 독자는 데이터 분석에 대한 자세한 설명을 위해 Akgol Oksuz et al.17 을 참조할 수 있습니다.
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
프로토콜 개발을 위해 Denis Lafontaine과 생물 정보학 지원을 위해 Sergey Venev에게 감사드립니다. 이 연구는 국립 보건원 공통 기금 4D 뉴클레옴 프로그램에서 JD(U54-DK107980, UM1-HG011536)로의 보조금으로 지원되었습니다. JD는 Howard Hughes Medical Institute의 연구원입니다.
이 문서는 HHMI의 출판물에 대한 오픈 액세스 정책의 적용을 받습니다. HHMI 연구소장은 이전에 대중에게 비독점적 CC BY 4.0 라이선스를 부여하고 연구 논문에서 HHMI에 대한 재라이선스 라이선스를 부여했습니다. 이러한 라이선스에 따라 저자가 수락한 이 논문의 원고는 출판 즉시 CC BY 4.0 라이선스에 따라 무료로 사용할 수 있습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 kb Ladder | New England Biolabs | N3232L | |
| Agarose | Invitrogen | 16500100 | |
| Agencourt AMPure XP magnetic beads , 60 mL | Beckman Coulter | A63881 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit (CFU) | EMD Millipore | UFC500396 | |
| Annealing Buffer (5x) | See recipe in supplemental materials | ||
| ATP 10 mM | ThermoFisher | R0441 | |
| Avanti J-25i High Speed Refrigerated ultra-centrifuge | Beckman Coulter | ||
| beckman ultracentrifuge tube 35 mL | Beckman Coulter | 357002 | |
| Binding Buffer (2x) | See recipe in supplemental materials | ||
| biotin-14-dATP 0.4 mM | Invitrogen | 19524-016 | |
| BSA 10 mg/mL | New England Biolabs | B9000S | dilute from 20 mg/mL |
| Cell scraper | Falcon | 353089 | |
| Cell scraper | Corning | 3008 | |
| Conical polypropylene tubes 50 mL | Denville | C1062-P | |
| Conical tube 15 mL | Denville | C1017-P | |
| Covaris micro tube AFA fiber with snap-cap 130 µL | Covaris | 520045/520077 | |
| Covaris Sonicator | Covaris | E220/E220evolution/M220 | |
| Culture flask 175 cm2 | Falcon | 353112 | |
| Culture plates 150 mm x 25 mm | Corning | 430599 | |
| dATP 1 mM | Invitrogen | 56172 | |
| dATP 10 mM | Invitrogen | 56172 | |
| dCTP 10 mM | Invitrogen | 56173 | |
| DdeI | New England Biolabs | R0175L | |
| dGTP 10 mM | Invitrogen | 56174 | |
| DMSO | Sigma | D2650-5x10ML | |
| dNTP mix 25 mM | Invitrogen | 10297117 | |
| Dounce homogenizer | DWK Life Sciences | 8853010002/8853030002 | |
| DPBS | Gibco | 14190-144 | |
| DpnII | New England Biolabs | R0543M | |
| DSG | ThermoScientific | 20593 | |
| dTTP 10 mM | Invitrogen | 56175 | |
| Ethanol 70% | Fisher | A409-4 | Diluted from 100% |
| Ethidium Bromide | Fisher | BP1302-10 | |
| Formaldehyde (37%) | Fisher | BP531-500 | |
| Gel loading dye (6x ) | New England Biolabs | B7024S | |
| Glycine in ultrapure water 2.5 M | Sigma | G8898-1KG | |
| HBSS | Gibco | 14025-092 | |
| Igepal CA-630 detergent | MP Biomedicals | 198596 | |
| Klenow DNA polymerase 5 U/µL | New England Biolabs | M0210L | |
| Klenow Fragment 3-->5’ exo-, 5 U/µL | New England Biolabs | M0212L | |
| ligation buffer (10x) | New England Biolabs | B7203S | |
| Liquid nitrogen | |||
| LoBind microcentrifuge tube 1.7 mL | Eppendorf | 22431021 | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | |
| Lysis buffer | See recipe in supplemental materials | ||
| Magnetic Particle separator | ThermoFisher | 12321D | |
| Microfuge tubes 1.7 mL | Axygen | MCT-175-C | |
| MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| NEBuffer 2.1 (10x) | New England Biolabs | B7002S | |
| NEBuffer 3.1 (10x) | New England Biolabs | B7203S | |
| PBS | Gibco | 70013-032 | |
| PCR (strip) tubes | Biorad | TBS0201/ TCS0803 | |
| PCR thermocycler | Biorad | T100 | |
| Pfu Ultra II Buffer (10x) | Agilent | Comes with Pfu Ultra | |
| PfuUltra II Fusion HS DNA Polymerase | Agilent | 600674 | |
| Phase lock tube 15 mL | Qiagen | 129065 | |
| Phase lock tubes 2 mL | Qiagen | 129056 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593-049 | |
| Protease inhibitor cocktail | ThermoFisher | 78440 | |
| Proteinase K in ultrapure water 10 mg/mL | Invitrogen | 25530-031 | |
| Refrigerated Centrifuge | Eppendorf | 5810R | |
| RNase A, DNase and protease-free 10 mg/mL | Thermo Scientific | EN0531 | |
| Rotator | Argos technologies | EW-04397-40 or rocking platform | |
| SDS 1% | Fisher | BP13111 | |
| Sodium acetate pH = 5.2, 3 M | Sigma | ||
| Sub-Cell GT Horizontal Electrophoresis System | Biorad | 1704401 | |
| T4 DNA ligase 1 U/µL | Invitrogen | 100004817 | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase 3 U/µL | New England Biolabs | M0203L | |
| T4 ligation buffer (5x) | Invitrogen | Y90001 | |
| T4 polynucleotide kinase 10 U/µL | New England Biolabs | M0201L | |
| Tabletop centrifuge | Eppendorf | 5425 | |
| TBE buffer | See recipe in supplemental materials | ||
| Tris Low EDTA Buffer (TLE) | See recipe in supplemental materials | ||
| Triton X-100 (10%) | Sigma | 93443 | |
| Truseq adapter oligos | Integrated DNA Technologies (IDT)) | https://www.idtdna.com/site/order/oligoentry | 250 nmole and HPLC purified |
| Tween 20 detergent | Fisher | 9005-64-5 | |
| Tween Wash Buffer | See recipe in supplemental materials | ||
| Vortex | Scientific Industries | (G560)SI-0236 |
참고문헌
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- Kalhor, R., Tjong, H., Jayathilaka, N., Alber, F., Chen, L. Genome architectures revealed by tethered chromosome conformation capture and population-based modeling. Nature Biotechnology. 30 (1), 90-98 (2012).
- Hsieh, T. H., et al. Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162 (1), 108-119 (2015).
- Hsieh, T. -. H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Rodley, C. D., Bertels, F., Jones, B., O’Sullivan, J. M. Global identification of yeast chromosome interactions using Genome conformation capture. Fungal Genetics and Biology. 46 (11), 879-886 (2009).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Alipour, E., Marko, J. F. Self-organization of domain structures by DNA-loop-extruding enzymes. Nucleic Acids Research. 40 (22), 11202-11212 (2012).
- Lajoie, B. R., Dekker, J., Kaplan, N. The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods. 72, 65-75 (2015).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- Golloshi, R., Sanders, J. T., McCord, R. P. Iteratively improving Hi-C experiments one step at a time. Methods. 142, 47-58 (2018).
- Darrow, E. M., et al. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture. Proceedings of the National Academy of Sciences of the United States of America. 113 (31), 4504-4512 (2016).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Ghuryeid, J., et al. Integrating Hi-C links with assembly graphs for chromosome-scale assembly. PLoS Computational Biology. 15 (8), 1007273 (2019).
- Gu, H., et al. Fine-mapping of nuclear compartments using ultra-deep Hi-C shows that active promoter and enhancer elements localize in the active A compartment even when adjacent sequences do not. bioRxiv. , (2021).
- Ramani, V., et al. Mapping 3D genome architecture through in situ DNase Hi-C. Nature Protocols. 11 (11), 2104-2121 (2016).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved Protocol for Genome-Wide Chromosome Conformation Capture. Current Protocols. 1 (7), 198 (2021).
- Belton, J. -. M. M., et al. Hi-C: A comprehensive technique to capture the conformation of genomes. Methods. 58 (3), 268-276 (2012).
- Truch, J., Telenius, J., Higgs, D. R., Gibbons, R. J. How to tackle challenging ChIP-Seq, with long-range cross-linking, Using ATRX as an example. Methods in Molecular Biology. 1832, 105-130 (2018).
- Wang, P., et al. In situ chromatin interaction analysis using paired-end tag sequencing. Current Protocols. 1 (8), 174 (2021).
- Tjong, H., et al. Population-based 3D genome structure analysis reveals driving forces in spatial genome organization. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1663-1672 (2016).
- Nagano, T., et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547 (7661), 61-67 (2017).
- Ramani, V., et al. Massively multiplex single-cell Hi-C. Nature Methods. 14 (3), 263-266 (2017).
- Meng, L., Wang, C., Shi, Y., Luo, Q. Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data. Nature Communications. 12 (1), 4369 (2021).
- Tavares-Cadete, F., Norouzi, D., Dekker, B., Liu, Y., Dekker, J. Multi-contact 3C reveals that the human genome during interphase is largely not entangled. Nature Structural & Molecular Biology. 27 (12), 1105-1114 (2020).
- Vermeulen, C., et al. Multi-contact 4C: long-molecule sequencing of complex proximity ligation products to uncover local cooperative and competitive chromatin topologies. Nature Protocols. 15 (2), 364-397 (2020).
- Oomen, M. E., Hedger, A. K., Watts, J. K., Dekker, J. Detecting chromatin interactions between and along sister chromatids with SisterC. Nature Methods. 17 (10), 1002-1009 (2020).
- Mitter, M., et al. Conformation of sister chromatids in the replicated human genome. Nature. 586 (7827), 139-144 (2020).
- Krietenstein, N., et al. Ultrastructural Details of Mammalian Chromosome Architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462 (7269), 58-64 (2009).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유