Method Article
跨长度尺度捕获染色体构象
摘要
Hi-C 3.0 是一种改进的 Hi-C 方案,它将甲醛和二琥珀酰亚胺戊二酸酯交联剂与 DpnII 和 DdeI 限制性内切酶的混合物相结合,以提高信噪比和染色质相互作用检测的分辨率。
摘要
染色体构象捕获(3C)用于检测三维染色质相互作用。通常,与甲醛(FA)的化学交联用于固定染色质相互作用。然后,使用限制性内切酶消化染色质并随后连接片段末端,将三维(3D)接近转化为独特的连接产物。最后,在逆转交联、蛋白质去除和DNA分离后,剪切DNA并准备用于高通量测序。成对位点的邻近连接频率是它们在细胞群中三维空间中共定位频率的量度。
测序的Hi-C文库提供了所有位点对之间相互作用频率的全基因组信息。Hi-C的分辨率和精度依赖于维持染色质接触和染色质频繁均匀碎裂的高效交联。本文描述了一种改进的 原位 Hi-C方案Hi-C 3.0,该方案通过结合两种交联剂(甲醛[FA]和二琥珀酰亚胺戊二酸[DSG])来提高交联效率,然后使用两种限制性内切酶(DpnII和DdeI)进行更精细的消化。Hi-C 3.0 是一种单一协议,用于准确定量较小尺度(如环和拓扑关联域 (TAD))的基因组折叠特征,以及较大细胞核范围尺度(如区室)的特征。
引言
染色体构象捕获自 2002 年以来一直使用1.从根本上说,每个构象捕获变体都依赖于DNA-蛋白质和蛋白质-蛋白质相互作用的固定来保持3D染色质组织。接下来是DNA片段化,通常是限制性酶切,最后是连接附近的DNA末端,将空间近端位点转化为独特的共价DNA序列。最初的3C方案使用PCR对特定的"一对一"相互作用进行采样。随后的4C测定允许检测"一对多"相互作用2,而5C检测"多对多"相互作用3。染色体构象捕获在实施下一代高通量测序(NGS)后完全实现,这允许使用全基因组Hi-C 4和类似技术(如3C-seq5,TCC6和Micro-C 7,8)检测"全对所有人"基因组相互作用(另见Denker和De Laat9的评论)。
在Hi-C中,生物素化的核苷酸用于在消化后和连接之前标记5′突出部分(图1)。这允许使用链霉亲和素包被的磁珠选择正确消化和连接的片段,使其与GCC10区分开来。Rao等人11对Hi-C方案进行了重要更新,他们在完整细胞核(即 原位)中进行消化和连接以减少虚假连接产物。此外,用MboI(或DpnII)消化代替HindIII酶切减少了片段大小并提高了Hi-C的分离潜力。这种增加允许检测相对较小的结构和接触点的更精确的基因组定位,例如小顺式元件之间的DNA环,例如,由环挤出产生的CTCF结合位点之间的环11,12。然而,这种潜力是有代价的。首先,分辨率提高两倍需要测序读数增加四倍(22)13。其次,小片段尺寸增加了将未消化的相邻片段误认为消化和连接的片段的可能性14。如前所述,在Hi-C中,消化和连接的片段与未消化的片段的不同之处在于连接连接处存在生物素。然而,需要从未连接的末端适当去除生物素,以确保仅将连接连接向下拉14,15。
随着NGS成本的降低,更详细地研究染色体折叠变得可行。为了减小DNA片段的大小,从而提高分辨率,Hi-C方案可以适应使用更频繁的切割限制性内切酶16或使用限制性内切酶17,18,19的组合。或者,可以滴定Micro-C中的MNase7,8和DNaseHi-C 20中的DNase以达到最佳消化。
最近对 3C 方法基础的系统评估表明,通过 1% FA 的顺序交联和 3 mM DSG17,在每个长度尺度上对染色体折叠特征的检测都得到了显着改善。此外,具有HindIII消化的Hi-C是检测大规模折叠特征(如隔室)的最佳选择,而Micro-C在检测DNA环等小规模折叠特征方面具有优势。这些结果导致了单一高分辨率"Hi-C 3.0"策略的开发,该策略使用FA和DSG交联剂的组合,然后与DpnII和DdeI核酸内切酶21进行双重酶解。Hi-C 3.0 为一般用途提供了一种有效的策略,因为它可以准确检测所有长度尺度上的折叠特征17。此处详细介绍了Hi-C 3.0协议的实验部分,并显示了测序后可以预期的典型结果。

图 1:Hi-C 程序分为六个步骤。 首先用FA固定细胞,然后用DSG(1)固定细胞。然后,裂解先于DdeI和DpnII的双重酶切(2)。通过突出填充添加生物素,并在DNA纯化之前连接近端钝端(3)(4)。在超声处理和尺寸选择之前,将生物素从未连接的末端去除(5)。最后,生物素的下拉允许通过PCR进行接头连接和文库扩增(6)。缩写:FA = 甲醛;DSG = 戊二酸二琥珀酰亚胺酯;B = 生物素。 请点击此处查看此图的大图。
研究方案
1. 交联固定
- 甲醛固定:从单层细胞开始
- 将细胞接种在适当的培养基中,以每150mm板收获5×106 个细胞。
注意:用户可以选择任何首选容器,以确保任何哺乳动物细胞系的最佳生长。此外,可以从组织中分离细胞。 - 用巴斯德移液器从 150 mm 板中与真空阱耦合,吸出培养基,用 ~10 mL HBSS 洗涤 2 次。
- 在交联之前,将 22.5 mL HBSS 和 625 μL 37% FA 混合至最终浓度为 1%,在 50 mL 管中制备 1% FA 交联溶液。摇晃轻轻混合。
注意:使用通风橱;甲醛有毒。 - 为了交联细胞,将 23.125 mL 的 1% FA 溶液倒入每个 15 cm 平板上。
- 在室温下孵育10分钟,每2分钟用手轻轻摇动板。
- 加入 1.25 mL 的 2.5 M 甘氨酸(最终 128 mM),轻轻旋转板以淬灭交联反应。
- 在室温下孵育5分钟,并继续在冰上孵育至少15分钟以停止交联。
- 用细胞刮刀或橡胶警察从板上刮下细胞。
- 用移液管将细胞悬液转移到 50 mL 锥形管中。在室温下以1,000× g 离心10分钟,并通过抽吸弃掉上清液。
- 用 10 mL Dulbecco 的磷酸盐缓冲盐水 (DPBS) 洗涤细胞沉淀一次,使用移液管重悬。然后,在室温下以1,000× g 离心10分钟。立即进行 DSG 交联。
注意:清洗细胞沉淀时要小心,因为细胞沉淀可能会松散,细胞可能会丢失。
- 将细胞接种在适当的培养基中,以每150mm板收获5×106 个细胞。
- 甲醛固定:从悬浮细胞开始
- 将细胞接种在适当的培养基中,以每容器收获5×106 个细胞。
注意:用户可以选择任何首选容器,以确保任何哺乳动物细胞系的最佳细胞生长。 - 在收获前,立即计数细胞并将 5 × 10个 6 个细胞转移到 50 mL 锥形管中。
- 通过在室温下以300× g 离心10分钟来轻轻沉淀细胞。
- 通过将 1.25 mL 的 37% FA 加入 45 mL 的 HBSS 中来制备 1% FA 交联溶液,并通过倒置管数次混合。
注意:加入整个 1.25 mL FA,不要拆分量。
注意:甲醛具有剧毒。 - 通过上下移液将细胞沉淀重悬于上一步制备的 46.25 mL 1% FA 交联溶液中。
- 在室温下在旋转器,摇臂上孵育10分钟,或每1-2分钟轻轻手动倒置试管。
- 通过加入 2.5 mL 的 2.5 M 甘氨酸(最终 128 mM)淬灭交联反应,并通过倒置管充分混合。
- 在室温下孵育5分钟,然后在冰上孵育至少15分钟以完全停止交联。
- 在室温下离心以1,000× g 沉淀交联细胞10分钟,并通过抽吸弃去上清液。
- 用10mL DPBS洗涤细胞一次,然后在室温下以1,000 × g 离心10分钟。使用移液管完全弃去上清液,并立即进行DSG交联。
注意:清洗细胞沉淀时要小心,因为细胞沉淀可能会松散,细胞可能会丢失。
- 将细胞接种在适当的培养基中,以每容器收获5×106 个细胞。
- 与戊二酸二琥珀酰亚胺交联
- 将沉淀的细胞重悬于 9.9 mL DPBS 中,然后加入 100 μL 300 mM DSG(3 mM 最终)。倒置混合。
注意:DSG 对湿气敏感。重要的是在交联当天在DMSO中制备300 mM DSG的新储备液。
注意:DMSO中的DSG具有剧毒。 - 在室温下在旋转器上交联细胞40分钟。
- 加入 1.925 mL 的 2.5 M 甘氨酸(400 mM 最终),倒置混合,并在室温下孵育 5 分钟。
- 在室温下以2,000× g 离心细胞15分钟。
注意:从松散细胞沉淀中去除上清液时要小心。 - 将沉淀重悬于 1 mL 0.05% 牛血清白蛋白 (BSA)-DPBS 中,并转移到 1.7 mL 管中。
注意:添加BSA有助于减少细胞聚集。 - 在4°C下以2,000× g 离心细胞15分钟,并通过移液除去上清液。
注意:为避免丢失沉淀,请快速完全除去上清液。 - 将沉淀在液氮中快速冷冻并储存在-80°C或立即进行下一步。
- 将沉淀的细胞重悬于 9.9 mL DPBS 中,然后加入 100 μL 300 mM DSG(3 mM 最终)。倒置混合。
2. 染色体构象捕获
- 细胞裂解和染色质消化
- 将交联细胞等分试样(~5×106 个细胞)重悬(移液)在含有10μL蛋白酶抑制剂混合物的1mL冰冷裂解缓冲液( 补充表S1中的配方)中,并转移到dounce匀浆器中在冰上孵育15分钟。
注意:使用前立即将蛋白酶抑制剂添加到裂解缓冲液中。 - 缓慢地上下移动杵A30次以使冰上的细胞均质化,并在冰上孵育1分钟以使细胞冷却,然后再进行30次冲程。
- 将裂解物转移到 1.7 mL 微量离心管中。
注意:保持悬浮液移动,因为有时细胞会粘在移液器吸头中。 - 在室温下以2,500× g 离心裂解的悬浮液5分钟。
- 弃去上清液并轻弹或涡旋湿沉淀以重悬。尽可能多地去除上清液,以获得团块最少的酸奶状物质。
- 将沉淀重悬于500μL冰冷的1x限制性缓冲液(从10x开始;参见 补充表S1中的配方)中,并以2,500 × g离心5分钟。重复此步骤进行第二次洗涤。
注意:1x限制性缓冲液中的沉淀比裂解缓冲液中的先前沉淀更颗粒。 - 将细胞重悬于终体积为 360 μL 的 1x 限制性缓冲液中,方法是在向沉淀的残留体积中加入 ~340 μL 后移液,具体取决于细胞大小。
- 留出 18 μL 的每种裂解物用于测试染色质完整性 (CI)。将CI样品储存在4°C。
- 向每个 Hi-C 管(总体积为 380 μL)中加入 38 μL 1% 十二烷基硫酸钠 (SDS),并通过移液小心混合,不要引入气泡。
注意:SDS有毒。 - 将样品在65°C下孵育而不摇动正好10分钟以打开染色质。
- 立即将试管放在冰上,并用Triton X-100制备消化混合物以淬灭SDS,如 表1中所述。
- 将 107 μL 消化混合物加入 Hi-C 管(总共 487 μL)中,以在间隔振荡(例如,900 rpm、30 秒开、4 分钟关)的热混合器中在 37 °C 下消化染色质过夜(~16 小时)。
注意:将Triton添加到1%的最终浓度中有助于淬灭SDS。
- 将交联细胞等分试样(~5×106 个细胞)重悬(移液)在含有10μL蛋白酶抑制剂混合物的1mL冰冷裂解缓冲液( 补充表S1中的配方)中,并转移到dounce匀浆器中在冰上孵育15分钟。
- DNA末端的生物素化
- 消化过夜后,将样品转移到65°C20分钟以失活剩余的核酸内切酶活性。
- 在孵育期间,制备填充预混液,如 表2所示。
- 孵育后,立即将样品放在冰上。
- 为每个样品留出10μL消化对照(DC),并储存在4°C。
- 用移液管或旋转从盖子上去除冷凝物。向每个样品中加入 58 μL 生物素填充混合物(总样品体积 535 μL),轻轻移液,不形成气泡。
- 将样品在23°C下在热混合器中孵育4小时(例如,900rpm,30秒开,4分钟关闭)。
- 近端DNA片段的连接
- 在生物素填充物孵育时如 表3 所示制备连接混合物。
- 向每个样品中加入 665 μL 的连接混合物(总样品体积 1,200 μL)。通过移液轻轻混合。
- 将样品在16°C下在间隔振荡的热混合器中孵育4小时(例如,900rpm,30秒开,4分钟关闭)。将这些含有共价连接的染色质的样品在4°C下储存几天。
- 逆转交联
- 使用 1x Tris 低 EDTA 将 CI 和 DC 样品的体积降至 50 μL(TLE;参见 补充表 S1 中的配方)。
- 向 CI 和 DC 样品中加入 10 μL 10 mg/mL 蛋白酶 K。
- 在65°C孵育过夜,间隔振荡(例如,900rpm,30秒开,4分钟关)。或者,在Hi-C样品的DNA纯化过程中对这些对照进行30分钟的交联逆转。
- 向每个 Hi-C 样品中加入 50 μL 10 mg/mL 蛋白酶 K,并在 65 °C 下孵育至少 2 小时,间隔振荡(例如,900 rpm,30 秒开,4 分钟关闭)。
- 向每个 Hi-C 管(总样品体积 1,300 μL)中再加入 50 μL 的 10 mg/mL 蛋白酶 K,并继续在 65 °C 下孵育过夜。储存在4°C直至DNA纯化。
注意:拆分蛋白酶K孵育可确保总蛋白质消化。
- 脱氧核糖核酸纯化
- 让试管从65°C冷却至室温。
- 将每个样品转移到 15 mL 锥形管中,并向每个管中加入 2.6 mL(2x 体积)苯酚:氯仿:异戊醇。
注意:苯酚:氯仿:异戊醇是一种剧毒刺激物,可能致癌。 - 涡旋每个管1分钟,然后将其内容物转移到15 mL锁相管中。
- 在台式离心机中以最大速度(1,500-3,500× g)离心样品5分钟。
- 小心地将水相倒入 35 mL 超速离心管中,并加入超纯水至最终体积为 1,250 μL。
注意:使用管子安装可用的超速离心机或分成多个微量离心管。 - 加入 1/10体积 (~125 μL) 的 3 M 乙酸钠,并通过倒置充分混合。
- 向每个样品中加入 2.5 倍体积 (~3.4 mL) 的冰冷 100% 乙醇,通过加入冰冷的 100% 乙醇平衡管进行超速离心,并通过倒置充分混合。
- 将试管在干冰上孵育~15分钟(避免凝固)。
- 将试管在4°C下以18,000 ×g 离心30分钟。
注意:对于倾斜的转子:标记颗粒所在位置的管子。 - 使用移液器,从非沉淀侧完全取出并丢弃上清液。
注意:此时,颗粒应该变得可见并且可以在管子上标记,因为在下一步干燥后可能不清晰可见。 - 将样品风干约10分钟或直到它们明显干燥。
- 通过移液或旋流将每个沉淀溶解在 450 μL 的 1x TLE 中,并转移到 0.5 mL 离心过滤单元 (CFU) 中,截止分子量为 3 kDa。
- 以最大速度离心CFU10分钟,然后丢弃流出物。用额外的 450 μL 1x TLE 洗涤每个超速离心管,并转移到其 CFU 进行另一次洗涤。
注意:以这种方式清洗CFU可以限制DNA损失,同时降低盐浓度。 - 以最大速度离心CFU10分钟,然后丢弃流出物。
- 向色谱柱中加入 80 μL 1x TLE,然后将色谱柱旋转到新的收集管中,然后以最大速度离心 2 分钟以获得 ~100 μL 的最终体积。
- 向每个样品中加入 1 μL RnaseA(1 mg/mL;10 mg/mL 储备液的 10 倍稀释),并在 37 °C 下在加热块、水浴或热混合器中孵育至少 30 分钟。
- Rnase处理后,从37°C取出样品并储存在4°C直至质量控制步骤。
- 检查染色质质量、酶消化和样品连接
- 在步骤2.4.5中反转交联后,将CI和DC样品冷却至室温。然后,转移到预纺的 2 mL 锁相管中。
注意:确保将锁相内容物离心到沉淀中(最大速度2分钟)。 - 加入 200 μL 苯酚:氯仿:异戊醇,涡旋 1 分钟混合样品。
- 以最大速度离心试管5分钟。
- 将每个样品(~50 μL)的水相转移到新的1.7 mL微量离心管中。
- 加入 1 μL Rnase A(来自 1 mg/mL),并在 37 °C 下孵育至少 30 分钟。
- 按照 表4中的建议将样品加载到0.8%琼脂糖凝胶上。
注意:质量控制的预期结果如图 2所示。 - 通过凝胶中的密度测定或使用Qubit或Nanodrop来定量DNA。
注意:准确的定量可确保在协议的下一部分获得正确的输入音量。使用具有已知数量的多个标准品来构建标准曲线。
- 在步骤2.4.5中反转交联后,将CI和DC样品冷却至室温。然后,转移到预纺的 2 mL 锁相管中。
表1:消化试剂。请按此下载此表格。
表 2:生物素填充试剂。 *请注意,更换酶可能需要不同的缓冲液和生物素化的dNTP。 请按此下载此表格。
表3:连接混合试剂。 缩写:BSA = 牛血清白蛋白。 请按此下载此表格。
表4:用于质量和尺寸选择评估的凝胶上样参数。请按此下载此表格。
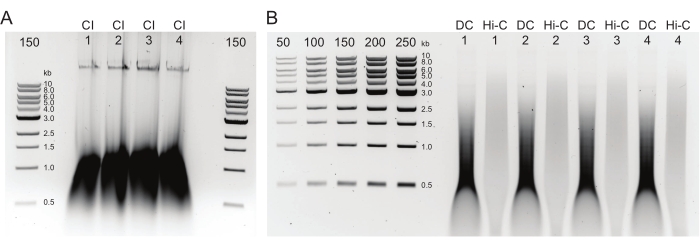
图2:琼脂糖凝胶显示典型的DNA后纯化质量控制结果 。 (A)CI对照应指示高分子量DNA的条带。(B)DC和Hi-C样品显示一系列DNA大小。Hi-C样品已合并成更大的片段,其分子量应高于DC。标记物的浓度范围允许生成标准曲线。请注意,在本例中,CI上样于单独的凝胶上,但建议将所有样品和对照一起上样并运行。缩写:CI = 染色质完整性;DC = 消解控制;Hi-C = 邻近连接。 请点击此处查看此图的大图。
3. Hi-C测序文库制备
- 从未结扎的末端去除生物素
- 制备生物素去除反应,如 表5所示。
注意:通常,10μg的DNA就足够了,但最多可以使用30μg。 - 将每个 130 μL 反应中的 2 x 65 μL 等分试样分配到两个 PCR 管中。
- 转移到热循环仪或PCR仪中,如 表5所述孵育。
注意:样品可以储存在4°C(几天到几周),-20°C(长期),或立即进行超声处理。
- 制备生物素去除反应,如 表5所示。
- 超声处理
- 将生物素去除步骤(总样品体积为 130 μL )中的样品重复物汇集到 130 μL 超声仪管中进行超声处理。
- 使用 表6 中给出的参数对样品进行超声处理,以实现低于500 bp的紧密窄分布。
注意:可以使用不同的超声仪类型,但对于窄片段分布(100-500 bp),超声仪设置可能需要优化。
- 使用磁珠选择尺寸
- 将超声处理后的 DNA 从超声仪管中移液到 1.7 mL 低结合管中。
- 用 1x TLE 使每个样品的总体积为 500 μL。尽量使体积尽可能接近 500 μL,因为样品与磁珠混合物的比例对于尺寸选择至关重要。
- 向每个试管中加入 400 μL 磁珠混合物,以获得磁珠混合物与样品体积 0.8 的比率。
注意:在这些条件下,磁珠捕获DNA片段>300 bp,这将是上层。上清液将包含片段<300 bp,这将是下级分。 - 通过涡旋混合管,并在室温下在旋转器上孵育10分钟。对于此尺寸和其他尺寸选择比例,请确保样品的全体积混合良好。寻找某些转子具有的"不稳定模式",该模式适用于较小的体积。
- 在室温下在磁粉分离器(MPS)上孵育5分钟。
- 孵育期间,将 500 μL 磁珠混合物加入新鲜的 1.7 μL 低结合管中,用于每个样品。
注意:这些试管将通过生成1.1:1的磁珠混合物与样品的比例来用于下馏分的下一个尺寸选择。 - 将试管留在MPS上5分钟。
- 从磁珠中取出上清液,并用 150 μL 磁珠混合物重悬。
注意:此步骤通过增加磁珠数量而不增加体积来避免磁珠与DNA饱和。 - 将上清液从步骤3.3.5转移到准备选择下级分的标记管中(步骤3.3.8)。
注意:150 μL 磁珠混合物 + 400 μL 0.8x 磁珠混合物(总共 550 μL)除以 500 μL 初始样品 = 1.1x 磁珠混合物与样品的比例。 - 通过涡旋混合下馏分管,并在室温下在旋转器上孵育10分钟。
注意:磁珠将结合 DNA 片段>100 bp,导致最终磁珠结合部分为 100-300 bp。 - 将下馏分管放在MPS上5分钟(室温)。
- 取出上清液并短暂离心管以尽可能多地进一步除去上清液。
- 使用 200 μL 70% 乙醇从两个级分中洗涤磁珠两次,每次在 MPS 上回收磁珠 5 分钟。
- 在离心机中快速旋转后,完全除去乙醇并进一步干燥MPS上的珠子。
注意:干燥直到酒精完全蒸发。颗粒应该看起来像黑巧克力而不会破裂(可能需要~10分钟)。 - 将两个级分重悬于 50 μL 1x TLE 缓冲液中。在室温下孵育10分钟,每隔一分钟敲击或轻弹试管以刺激混合和洗脱。
- 将珠子与MPS上的上清液分离5分钟。
- 保留每个样品的上清液。将上清液移液到 1.7 mL 低结合管中。
注意:样品可以在4°C下保存几天或在-20°C下长期保存。 - 如 表4 所示运行2%琼脂糖凝胶以确定样品质量和数量。有关这种凝胶的示例,请参见 图3 。
注意:如果根据经验,超声处理具有高度可重复性,则可以跳过此凝胶并立即结束修复。建议用户在滴定PCR之前保留上限。需要大量PCR扩增的次优DNA量可以从上级分的材料中拯救出来。 - 从已知的DNA分子量标准输入生成标准曲线后,或使用Qubit或Nanodrop直接量化凝胶中的DNA量。
- 端修复
- 如 表7 (每次反应给出的量)制备末端修复混合物。
- 将剩余的 46 μL 洗脱 DNA 从下级分转移到 PCR 管中,并加入 24 μL 制备的末端修复混合物。在PCR仪中孵育,如 表7所示。
- 程序完成后,将样品保持在4°C直至下拉。
- 使用链霉亲和素包被珠下拉生物素化连接产物
- 从定量的尺寸选择中确定每个文库的链霉亲和素包被磁珠的量(步骤3.3.19)。
注意:这些链霉亲和素包被的磁珠(10 mg / mL溶液)每mg磁珠(= 20 μg / 100 μL磁珠)可结合20 μg双链DNA。每 1 μg Hi-C DNA 使用 2 μL,但不少于 10 μL。 - 混合链霉亲和素包被的磁珠,并将每个文库所需的磁珠体积(在上一步中计算)移液到单独的 1.7 mL 低结合管中。
- 将珠子重悬于 400 μL 吐温洗涤缓冲液 (TWB;参见 补充表 S1 中的配方)中,并在室温下在旋转器上孵育 ~3 分钟(参见步骤 3.3.4 中的说明)。
- 将珠子与MPS上的上清液分离1分钟,并除去上清液。
- 通过移液另外 400 μL TWB 清洗磁珠。
- 将珠子与MPS上的上清液分离1分钟,并除去上清液。
- 向磁珠中加入 400 μL 2x 结合缓冲液 (BB)( 补充表 S1 中的配方)并重悬。此外,加入 330 μL 的 1x TLE 和来自末端修复的溶液(来自步骤 3.4.3)。
- 将样品在室温下孵育15分钟,同时在旋转器上混合。
- 将珠子与MPS上的上清液分离1分钟,并除去上清液。
- 向磁珠中加入 400 μL 的 1x BB 并重悬。
- 将珠子与MPS上的上清液分离1分钟,并除去上清液。
- 加入 100 μL 的 1x TLE 以洗涤珠子。
- 将珠子与MPS上的上清液分离1分钟,并除去上清液。
- 最后,加入 41 μL 的 1x TLE 以重悬磁珠。
- 从定量的尺寸选择中确定每个文库的链霉亲和素包被磁珠的量(步骤3.3.19)。
- A尾
- 如 表8所示制备A尾混合物。
- 将反应物移液到PCR管中并孵育,如 表8所示。
- 从热循环仪中取出后,立即将PCR管放在冰上,并将内容物转移到1.7 mL低结合管中。
- 将珠子与MPS上的上清液分离1分钟,弃去上清液。
- 加入 400 μL 1x 连接缓冲液,用超纯水从 5x T4 DNA 连接酶缓冲液中稀释。
- 将珠子与MPS上的上清液分离1分钟,然后弃去上清液。
- 将 1x 连接缓冲液加入 40 μL 的最终体积。
- 退火适配器寡核苷酸
- 制备100μM的适配器寡核苷酸储备液(表9)。
注意:订购250 nmole的HPLC纯化寡核苷酸。 - 如 表9所述在PCR管中退火适配器。
- 使用PCR热循环仪以0.5°C/s的速度逐渐升温至97.5°C。 在97.5°C保持2.5分钟。
- 使用PCR热循环仪以0.1°C/s的速度逐渐升温775次(达到20°C)。将温度保持在4°C,直到进一步使用。
- 加入 83 μL 1x 退火缓冲液( 补充表 S1 中的配方)将适配器稀释至 15 μM。 将适配器储存在-20°C。
- 制备100μM的适配器寡核苷酸储备液(表9)。
- 测序衔接连接
- 在 1.7 mL 低结合管中制备适配器连接混合物(表 10)。
- 在室温下连接2小时。
- 将珠子与MPS上的上清液分离1分钟,弃去上清液。
- 加入 400 μL TWB,小心地上下移液珠子,然后在室温下在旋转器上孵育 5 分钟。将珠子与MPS上的上清液分离,然后再次重复此步骤。
- 将珠子与MPS上的上清液分离(~1分钟),弃去上清液,并加入200μL的1x BB。
- 将珠子与MPS上的上清液分离(~1分钟)并弃去上清液。
- 加入 200 μL 1x PCR 前缓冲液(来自 10x; 补充表 S1 中的配方),并转移到新的 1.7 mL 低结合管中。
- 将珠子与MPS上的上清液分离(~1分钟)并弃去上清液。
- 加入 20 μL 1x PCR 前缓冲液,并通过移液混合。
- 使用时将试管放在冰上或储存在4°C。
- 通过滴定优化PCR循环次数
- 如 表11 所示,每个样品设置30 μL预混反应(每次反应的量)。
- 运行最少的循环次数,取 5 μL 等分试样。在取下一个 5 μL 等分试样之前,在剩余的反应上再运行 2-3 个循环。重复以收集四个等分试样。
- 对每个等分试样使用 表11 中的PCR参数。
- 向每个 5 μL 样品中加入 5 μL 水和 2 μL 6x 染料。在2%琼脂糖TBE凝胶( 配方在补充表S1中)和25-150 ng低分子量分子量标准上运行。有关预期结果,请参见 图 4 。
注意:最终文库PCR扩增[步骤3.10]的最佳循环数是在凝胶上获得可见产物的最小循环数减去一个循环。
- 最终文库PCR扩增
- 设置12 x 30 μL反应以扩增每个最终文库以进行测序,如 表11所示。
- 在确定PCR滴定后的循环次数(步骤3.9.3)后,根据 表11 循环PCR反应。
- PCR 完成后,将复制样品汇集到 1.7 mL 低结合微量离心管中。
- 将试管放在MPS上,并将上清液转移到新的1.7 mL低结合微量离心管中。
- 将剩余的链霉亲和素包被的磁珠重悬于 20 μL 1x PCR 前缓冲液中。
注意:该模板在4°C下储存数天至数周或在-20°C下长期储存时可以重复使用。
- 用磁珠混合物去除引物
- 使用 1x TLE 缓冲液( 补充表 S1 中的配方)将体积从步骤 3.10.4 精确调整到 360 μL。
- 向每个样品中加入 360 μL 磁珠混合物,上下移液混合。
- 在旋转器上,在室温下混合样品10分钟。
- 在室温(3-5分钟)下将珠子与MPS上的上清液分离。
- 使用 200 μL 70% 乙醇洗涤珠子两次,每次在 MPS 上回收珠子 5 分钟。
- 在离心机中快速旋转并完全移走乙醇。在MPS上的空气中干燥珠子以进一步蒸发乙醇。
注意:颗粒应该看起来像黑巧克力而不会破裂(可能需要~10分钟)。 - 加入 30 μL 超纯水并重悬以在室温下洗脱 DNA 10 分钟。每 2 分钟轻弹一次试管以帮助混合。
- 将珠子与MPS上的上清液分离5分钟。
- 将每个样品的上清液收集在新鲜的 1.7 mL 管中。
- 在 2% 琼脂糖 TBE( 补充表 S1 中的配方)凝胶上运行 1 μL 文库,以获得片段大小分布并定量最终文库(图 5)。
注意:ClaI消化只能发生在DpnII-DpnII连接处,并作为阳性连接对照,应导致最终文库的片段大小分布较低。最终文库可以在4°C下储存几天,在-20°C下长期保存,或立即稀释并提交测序。
表5:生物素去除试剂和温度 请点击此处下载此表。
表 6:超声处理参数。请按此下载此表格。
表 7:末端修复试剂和温度。请按此下载此表格。
表 8:A尾试剂和温度。请按此下载此表格。
表 9:PCR 引物和配对末端适配器寡核苷酸,试剂退火退火。 缩写:5PHOS = 5' 磷酸盐。星号表示硫代磷酸化的DNA碱基。 #将索引寡核苷酸与通用寡核苷酸合并以退火到索引适配器中。 请按此下载此表格。
表 10:接头连接试剂。请按此下载此表格。
表11:PCR试剂和循环参数。请按此下载此表格。

图 3:琼脂糖凝胶显示了典型的尺寸后选择结果。 显示了四个样品(编号为1-4)的DpnII-DdeI Hi-C的上限和下部。每个样品的第一个泳道包含来自 0.8x 磁珠混合物的上级分,第二和第三泳道包含来自 1.1x 磁珠混合物的下级分的稀释液。 请点击此处查看此图的大图。

图 4:具有 PCR 滴定结果的琼脂糖凝胶。 从 5 个 PCR 循环开始,每 2 个循环(5、7、9 和 11 个循环)对四个文库中的每一个采集样品。根据该图,选择6个循环作为每个样品的最佳循环。 请点击此处查看此图的大图。
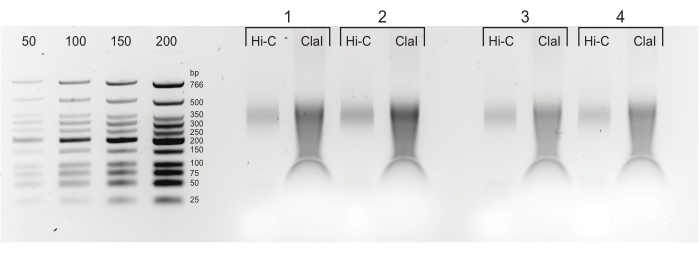
图 5:最终 PCR 产物。 清洁和尺寸选择后,将PCR产物(Hi-C)加载到同一文库(ClaI)的ClaI酶切部分旁边。ClaI消化片段表明存在广受欢迎的DpnII-DpnII连接。请注意,ClaI不能消化DpnII-DdeI连接,因此,并非所有连接都有助于减小此限制的尺寸。 请点击此处查看此图的大图。
结果
这份手稿中的数字是由Lafontaine等人之前发表的一个单独的重复实验产生的,21。获得高通量测序数据后,使用 Open Chromatin Collective (Open2C:https://github.com/open2c)处理Hi-C数据。类似的管道可以在4D Nucleome项目(https://data.4dnucleome.org/resources/data-analysis/hi_c-processing-pipeline)的数据门户上找到。简而言之,Nextflow管道蒸馏器(https://github.com/open2c/distiller-nf)的实施是为了(1)将Hi-C分子的序列与参考基因组对齐,(2)使用Hi-C对解析.sam比对和形成文件,(3)过滤PCR重复项,以及(4)将配对聚合成Hi-C相互作用的分箱矩阵。然后,可以(1)在HiGlass服务器(https://higlass.io/)上查看这些HDF5格式的矩阵,以及(2)使用开放染色质集体(https://github.com/open2c/cooltools)维护的"cooltools"集合中存在的大量开源计算工具进行分析,以提取和量化折叠特征,例如隔室,TAD和环。
Hi-C3.0文库的一些质量指标可以在将读对映射到参考基因组后立即进行评估,使用一些简单的指标/指标。首先,通常~50%的测序读对可以唯一地定位于人类细胞。由于染色体的聚合性质,大多数这些映射的读段(~60%-90%)代表染色体内的相互作用(顺式),相互作用频率随着基因组距离的增加而迅速衰减(距离依赖性衰变)。距离依赖性衰变可以在"缩放图"中最好地可视化,该图将接触概率(每个染色体臂)显示为基因组距离的函数。我们发现,使用不同的交联剂和酶可以改变长距离和短距离的距离依赖性衰变17。当与Mnase等酶和产生较小片段的DpnII-DdeI组合结合使用时,DSG交联的添加提高了短距离相互作用的可检测性(图6A)。
距离依赖性衰减也可以直接从 2D 交互矩阵中观察到:当远离中心对角线时,相互作用变得更加不频繁(图 6B)。此外,可以从Hi-C矩阵和缩放图中识别基因组折叠特征,例如区室,TAD和环,作为与一般全基因组平均距离依赖性衰减的偏差。重要的是,除了FA之外,与DSG交联可减少随机连接,由于染色体的聚合物性质,随机连接不受限制,因此更有可能发生在染色体之间(反式)(图6C)。减少随机连接会导致信噪比增加,特别是对于染色体间和超长距离(>10-50 Mb)染色体内相互作用。

图 6:映射和过滤的 Hi-C 库的代表性结果。 (A)具有各种酶的接触概率及其衍生物的缩放图,按片段长度(顶部)排序,并与FA或FA + DSG(底部)交联。使用MNAse(microC)或DpnII-DdeI(Hi-C 3.0)进行消化可显着增加短距离接触(顶部),将DSG添加到FA(底部)也是如此。(B)列显示了仅FA交联后DpnII消化和FA + DSG交联后DpnII或DdeI消化的Hi-C热图。白色箭头显示DSG交联和DdeI消化后"点"的强度增加,这意味着可以更好地检测DNA环。行以递增的分辨率显示 3 号染色体的不同部分,与图 C 对齐:第一行:整个 3 号染色体 (0-198,295,559 Mb);中间排:186-196 Mb;底行:191.0-191.5 Mb。 (C) A 中描述区域的覆盖率图。黑色箭头显示仅 FA 交联的较低覆盖率(%顺式读数)。缩写:FA = 甲醛;DSG = 戊二酸二琥珀酰亚胺酯;CHR = 染色体。请点击此处查看此图的大图。
并非所有映射读取都很有用。第二个质量指标是PCR重复的数量。在连接和超声处理后,不太可能偶然发生精确的重复读数。因此,这种读取可能是由PCR扩增引起的,需要过滤掉。当需要太多PCR循环来扩增低复杂度文库时,通常会出现重复。通常,对于Hi-C,大多数文库只需要5-8个循环的最终PCR扩增,通过滴定PCR确定(见步骤3.9; 图4)。然而,即使在14个PCR扩增循环后,也可以获得具有足够复杂性的文库。
另一类重复读取,即所谓的光学重复,可能来自使用图案化流通池(如HiSeq4000)的Illumina测序平台上的扩增过程。光学重复来自流通池过载,导致(大)簇被称为两个单独的簇,或者在第一轮PCR后原始配对末端分子的局部重新聚集。由于这两种类型的光学重复都是局部的,因此可以通过它们在流通池上的位置来识别它们并将其与PCR重复进行区分。PCR重复>15%的文库需要再生,而具有光学重复的文库可以在优化加载过程后重新加载。
补充表S1:缓冲液和溶液。请按此下载此表格。
讨论
细胞处理的关键步骤
虽然可以使用较少数量的输入细胞,但该协议已针对每个测序通道(~400 M读数)~5×106 个细胞进行了优化,以确保深度测序后适当的复杂性。细胞最好在固定前计数。对于超深库的生成,我们通常会乘以通道(和单元)的数量,直到达到所需的读取深度。为了获得最佳固定效果,在FA固定之前,应用PBS替换含血清的培养基,并应立即添加固定剂溶液,且没有浓度梯度15,22。对于细胞收获,刮擦优于胰蛋白酶消化,因为胰蛋白酶消化后从扁平到球形的转变可能会影响核构象。加入DSG后,松散和团块状细胞沉淀容易丢失。在此阶段处理细胞时要小心,并添加0.05%的BSA以减少结块。
对方法的修改
该协议是使用人类细胞17开发的。然而,根据染色体构象捕获的经验,该方案应该适用于大多数真核细胞。对于显着较低的输入(~1×106 个细胞),我们建议使用一半的体积进行裂解和构象捕获程序[步骤2.1-2.4]。这也将允许在带有1.7 mL管的台式离心机中进行DNA分离[步骤2.5],这可以改善低DNA浓度的沉淀。DNA的定量(步骤2.6.6)将指示如何进行。对于少量分离的DNA(1-5μg),我们建议跳过大小选择(步骤3.3),并在使用CFU将体积从130μL减少到~45μL后继续去除生物素。
该协议是专门为确保随后与FA和DSG交联以及与DpnII和DdeI消化后的高质量数据而开发的。然而,替代交联策略,如FA和EGS(乙二醇双(琥珀酸琥珀酰亚胺酯)),也用于ChIP-seq23 和ChIA-PET24,可能同样有效17。同样,不同的酶组合,如DpnII和HinfI18 或MboI,MseI和NlaIII19 可用于消化。在调整酶组合时,请务必使用可以填充特定 5' 突出部的生物素化核苷酸,并为每种鸡尾酒使用最佳缓冲液。DpnII 自带缓冲液,酶制造商推荐使用 DdeI 消解的特定缓冲液。然而,对于该方案中使用DpnII和DdeI的双重酶切,建议使用限制性缓冲液,因为它对两种酶的额定活性为100%。
构象捕获故障排除
染色体构象捕获的三个关键步骤:交联、消化和连接,在凝胶上可视化结果之前已经完成。为了确定这三个步骤中每个步骤的质量并识别可能出现问题的位置,在消化(CI)和消化(DC)之前和之后取等分试样,并与连接的Hi-C样品一起加载到凝胶上(图2)。该凝胶用于确定Hi-C样品的质量以及是否值得继续使用该方案。如果没有CI和DC,就很难确定潜在的次优步骤。值得注意的是,次优连接可能是由于连接本身、填充或交联问题造成的。要排除交联故障,请确保每个文库使用不超过1×107 个细胞,并从新鲜交联试剂和清洁细胞(即用PBS冲洗)开始。对于连接,请确保将细胞和连接混合物保存在冰上。在16°C孵育4小时之前加入T4 DNA连接酶并充分混合。
库准备疑难解答
如果需要超过10个PCR循环或在PCR滴定后凝胶上看不到PCR产物(图4),则有几种选择可以保存Hi-C样品。从PCR滴定回来,第一种选择是再次尝试PCR。如果仍然没有足够的产品,可以在用1x TLE缓冲液洗涤磁珠两次后尝试另一轮A尾和适配器连接(步骤3.6)。在这种额外的A尾和适配器连接之后,可以像以前一样进行PCR滴定。如果仍然没有产品,最后一个选择是重新分配步骤 3.3 中的 0.8x 分数并从那里继续。
Hi-C3.0的局限性和优势
重要的是要认识到Hi-C是一种基于群体的方法,可以捕获细胞群中位点对之间相互作用的平均频率。一些计算分析旨在从群体25中解开构象组合,但原则上,Hi-C对细胞之间的差异视而不见。虽然可以执行单细胞Hi-C 26,27并且可以进行计算推理28,但单细胞Hi-C不适合获得超高分辨率的3C信息。Hi-C 的另一个限制是它只检测成对交互。为了检测多触点相互作用,可以使用频繁的切割机结合短读长测序(Illumina)16,或者使用来自PacBio或Oxford Nanopore平台的长读长测序进行多接触3C29或4C30。还开发了专门检测姐妹染色单体之间和沿姊妹染色单体接触的Hi-C衍生物31,32。
尽管Hi-C19和Micro-C33可用于生成亚千碱基分辨率的接触图,但两者都需要大量的测序读取,这可能成为一项昂贵的工作。为了在不花费成本的情况下获得相似甚至更高的分辨率,可以应用特定基因组区域(捕获-C 34)或特定蛋白质相互作用(ChiA-PET 35,PLAC-seq36,Hi-ChIP37)的富集。这些富集应用程序的优缺点是仅对有限数量的交互进行采样。有了这样的丰富,Hi-C的全局方面(以及全局规范化的选择)就失去了。
Hi-C3.0的重要性和潜在应用
该协议旨在实现高分辨率、超深 3C,同时检测大规模折叠特征,如 TAD 和隔室17 (图 6)。该协议从每个Hi-C文库的每个管5个×10个6个细胞 开始,这应该有足够的材料对流通池上的一个或两个泳道进行测序,以获得多达10亿个配对端读取。对于超深测序,应制备多管5×106 细胞,具体取决于映射读段的数量和PCR重复数。在最高分辨率(<1 kb)下,CTCF位点之间主要发现环状相互作用,但也可以检测到启动子-增强子相互作用。读者可以参考Akgol Oksuz等人17 ,了解数据分析的详细说明。
披露声明
作者没有利益冲突需要披露。
致谢
我们要感谢Denis Lafontaine的协议开发和Sergey Venev的生物信息学帮助。这项工作得到了美国国立卫生研究院共同基金4D核组计划对JD的资助(U54-DK107980,UM1-HG011536)。J.D.是霍华德休斯医学研究所的研究员。
本文受 HHMI 的出版物开放获取政策的约束。HHMI实验室负责人此前已向公众授予非排他性CC BY 4.0许可证,并在其研究文章中向HHMI授予可再许可许可证。根据这些许可证,本文作者接受的手稿可以在出版后立即在CC BY 4.0许可证下免费提供。
材料
| Name | Company | Catalog Number | Comments |
| 1 kb Ladder | New England Biolabs | N3232L | |
| Agarose | Invitrogen | 16500100 | |
| Agencourt AMPure XP magnetic beads , 60 mL | Beckman Coulter | A63881 | |
| Amicon Ultra-0.5 Centrifugal Filter Unit (CFU) | EMD Millipore | UFC500396 | |
| Annealing Buffer (5x) | See recipe in supplemental materials | ||
| ATP 10 mM | ThermoFisher | R0441 | |
| Avanti J-25i High Speed Refrigerated ultra-centrifuge | Beckman Coulter | ||
| beckman ultracentrifuge tube 35 mL | Beckman Coulter | 357002 | |
| Binding Buffer (2x) | See recipe in supplemental materials | ||
| biotin-14-dATP 0.4 mM | Invitrogen | 19524-016 | |
| BSA 10 mg/mL | New England Biolabs | B9000S | dilute from 20 mg/mL |
| Cell scraper | Falcon | 353089 | |
| Cell scraper | Corning | 3008 | |
| Conical polypropylene tubes 50 mL | Denville | C1062-P | |
| Conical tube 15 mL | Denville | C1017-P | |
| Covaris micro tube AFA fiber with snap-cap 130 µL | Covaris | 520045/520077 | |
| Covaris Sonicator | Covaris | E220/E220evolution/M220 | |
| Culture flask 175 cm2 | Falcon | 353112 | |
| Culture plates 150 mm x 25 mm | Corning | 430599 | |
| dATP 1 mM | Invitrogen | 56172 | |
| dATP 10 mM | Invitrogen | 56172 | |
| dCTP 10 mM | Invitrogen | 56173 | |
| DdeI | New England Biolabs | R0175L | |
| dGTP 10 mM | Invitrogen | 56174 | |
| DMSO | Sigma | D2650-5x10ML | |
| dNTP mix 25 mM | Invitrogen | 10297117 | |
| Dounce homogenizer | DWK Life Sciences | 8853010002/8853030002 | |
| DPBS | Gibco | 14190-144 | |
| DpnII | New England Biolabs | R0543M | |
| DSG | ThermoScientific | 20593 | |
| dTTP 10 mM | Invitrogen | 56175 | |
| Ethanol 70% | Fisher | A409-4 | Diluted from 100% |
| Ethidium Bromide | Fisher | BP1302-10 | |
| Formaldehyde (37%) | Fisher | BP531-500 | |
| Gel loading dye (6x ) | New England Biolabs | B7024S | |
| Glycine in ultrapure water 2.5 M | Sigma | G8898-1KG | |
| HBSS | Gibco | 14025-092 | |
| Igepal CA-630 detergent | MP Biomedicals | 198596 | |
| Klenow DNA polymerase 5 U/µL | New England Biolabs | M0210L | |
| Klenow Fragment 3-->5’ exo-, 5 U/µL | New England Biolabs | M0212L | |
| ligation buffer (10x) | New England Biolabs | B7203S | |
| Liquid nitrogen | |||
| LoBind microcentrifuge tube 1.7 mL | Eppendorf | 22431021 | |
| Low Molecular Weight DNA Ladder | New England Biolabs | N3233L | |
| Lysis buffer | See recipe in supplemental materials | ||
| Magnetic Particle separator | ThermoFisher | 12321D | |
| Microfuge tubes 1.7 mL | Axygen | MCT-175-C | |
| MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| NEBuffer 2.1 (10x) | New England Biolabs | B7002S | |
| NEBuffer 3.1 (10x) | New England Biolabs | B7203S | |
| PBS | Gibco | 70013-032 | |
| PCR (strip) tubes | Biorad | TBS0201/ TCS0803 | |
| PCR thermocycler | Biorad | T100 | |
| Pfu Ultra II Buffer (10x) | Agilent | Comes with Pfu Ultra | |
| PfuUltra II Fusion HS DNA Polymerase | Agilent | 600674 | |
| Phase lock tube 15 mL | Qiagen | 129065 | |
| Phase lock tubes 2 mL | Qiagen | 129056 | |
| Phenol:chloroform:isoamyl alcohol | Invitrogen | 15593-049 | |
| Protease inhibitor cocktail | ThermoFisher | 78440 | |
| Proteinase K in ultrapure water 10 mg/mL | Invitrogen | 25530-031 | |
| Refrigerated Centrifuge | Eppendorf | 5810R | |
| RNase A, DNase and protease-free 10 mg/mL | Thermo Scientific | EN0531 | |
| Rotator | Argos technologies | EW-04397-40 or rocking platform | |
| SDS 1% | Fisher | BP13111 | |
| Sodium acetate pH = 5.2, 3 M | Sigma | ||
| Sub-Cell GT Horizontal Electrophoresis System | Biorad | 1704401 | |
| T4 DNA ligase 1 U/µL | Invitrogen | 100004817 | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase | New England Biolabs | M0203L | |
| T4 DNA polymerase 3 U/µL | New England Biolabs | M0203L | |
| T4 ligation buffer (5x) | Invitrogen | Y90001 | |
| T4 polynucleotide kinase 10 U/µL | New England Biolabs | M0201L | |
| Tabletop centrifuge | Eppendorf | 5425 | |
| TBE buffer | See recipe in supplemental materials | ||
| Tris Low EDTA Buffer (TLE) | See recipe in supplemental materials | ||
| Triton X-100 (10%) | Sigma | 93443 | |
| Truseq adapter oligos | Integrated DNA Technologies (IDT)) | https://www.idtdna.com/site/order/oligoentry | 250 nmole and HPLC purified |
| Tween 20 detergent | Fisher | 9005-64-5 | |
| Tween Wash Buffer | See recipe in supplemental materials | ||
| Vortex | Scientific Industries | (G560)SI-0236 |
参考文献
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research. 16 (10), 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- Kalhor, R., Tjong, H., Jayathilaka, N., Alber, F., Chen, L. Genome architectures revealed by tethered chromosome conformation capture and population-based modeling. Nature Biotechnology. 30 (1), 90-98 (2012).
- Hsieh, T. H., et al. Mapping nucleosome resolution chromosome folding in yeast by micro-C. Cell. 162 (1), 108-119 (2015).
- Hsieh, T. -H. S., Fudenberg, G., Goloborodko, A., Rando, O. J. Micro-C XL: assaying chromosome conformation from the nucleosome to the entire genome. Nature Methods. 13 (12), 1009-1011 (2016).
- Denker, A., de Laat, W. The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development. 30 (12), 1357-1382 (2016).
- Rodley, C. D., Bertels, F., Jones, B., O’Sullivan, J. M. Global identification of yeast chromosome interactions using Genome conformation capture. Fungal Genetics and Biology. 46 (11), 879-886 (2009).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Alipour, E., Marko, J. F. Self-organization of domain structures by DNA-loop-extruding enzymes. Nucleic Acids Research. 40 (22), 11202-11212 (2012).
- Lajoie, B. R., Dekker, J., Kaplan, N. The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods. 72, 65-75 (2015).
- Belaghzal, H., Dekker, J., Gibcus, J. H. Hi-C 2.0: An optimized Hi-C procedure for high-resolution genome-wide mapping of chromosome conformation. Methods. 123, 56-65 (2017).
- Golloshi, R., Sanders, J. T., McCord, R. P. Iteratively improving Hi-C experiments one step at a time. Methods. 142, 47-58 (2018).
- Darrow, E. M., et al. Deletion of DXZ4 on the human inactive X chromosome alters higher-order genome architecture. Proceedings of the National Academy of Sciences of the United States of America. 113 (31), 4504-4512 (2016).
- Akgol Oksuz, B., et al. Systematic evaluation of chromosome conformation capture assays. Nature Methods. 18 (9), 1046-1055 (2021).
- Ghuryeid, J., et al. Integrating Hi-C links with assembly graphs for chromosome-scale assembly. PLoS Computational Biology. 15 (8), 1007273(2019).
- Gu, H., et al. Fine-mapping of nuclear compartments using ultra-deep Hi-C shows that active promoter and enhancer elements localize in the active A compartment even when adjacent sequences do not. bioRxiv. , (2021).
- Ramani, V., et al. Mapping 3D genome architecture through in situ DNase Hi-C. Nature Protocols. 11 (11), 2104-2121 (2016).
- Lafontaine, D. L., Yang, L., Dekker, J., Gibcus, J. H. Hi-C 3.0: Improved Protocol for Genome-Wide Chromosome Conformation Capture. Current Protocols. 1 (7), 198(2021).
- Belton, J. -M. M., et al. Hi-C: A comprehensive technique to capture the conformation of genomes. Methods. 58 (3), 268-276 (2012).
- Truch, J., Telenius, J., Higgs, D. R., Gibbons, R. J. How to tackle challenging ChIP-Seq, with long-range cross-linking, Using ATRX as an example. Methods in Molecular Biology. 1832, Clifton, N.J. 105-130 (2018).
- Wang, P., et al. In situ chromatin interaction analysis using paired-end tag sequencing. Current Protocols. 1 (8), 174(2021).
- Tjong, H., et al. Population-based 3D genome structure analysis reveals driving forces in spatial genome organization. Proceedings of the National Academy of Sciences of the United States of America. 113 (12), 1663-1672 (2016).
- Nagano, T., et al. Cell-cycle dynamics of chromosomal organization at single-cell resolution. Nature. 547 (7661), 61-67 (2017).
- Ramani, V., et al. Massively multiplex single-cell Hi-C. Nature Methods. 14 (3), 263-266 (2017).
- Meng, L., Wang, C., Shi, Y., Luo, Q. Si-C is a method for inferring super-resolution intact genome structure from single-cell Hi-C data. Nature Communications. 12 (1), 4369(2021).
- Tavares-Cadete, F., Norouzi, D., Dekker, B., Liu, Y., Dekker, J. Multi-contact 3C reveals that the human genome during interphase is largely not entangled. Nature Structural & Molecular Biology. 27 (12), 1105-1114 (2020).
- Vermeulen, C., et al. Multi-contact 4C: long-molecule sequencing of complex proximity ligation products to uncover local cooperative and competitive chromatin topologies. Nature Protocols. 15 (2), 364-397 (2020).
- Oomen, M. E., Hedger, A. K., Watts, J. K., Dekker, J. Detecting chromatin interactions between and along sister chromatids with SisterC. Nature Methods. 17 (10), 1002-1009 (2020).
- Mitter, M., et al. Conformation of sister chromatids in the replicated human genome. Nature. 586 (7827), 139-144 (2020).
- Krietenstein, N., et al. Ultrastructural Details of Mammalian Chromosome Architecture. Molecular Cell. 78 (3), 554-565 (2020).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46 (2), 205-212 (2014).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462 (7269), 58-64 (2009).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。