Method Article
عوالم مصغرة للسماد كبيئات متنوعة ميكروبيا تشبه الطبيعة لأبحاث الميكروبيوم في Caenorhabditis elegans
In This Article
Summary
تجلب العوالم المصغرة للسماد التنوع الميكروبي الموجود في الطبيعة إلى المختبر لتسهيل أبحاث الميكروبيوم في Caenorhabditis elegans. فيما يلي بروتوكولات لإعداد تجارب العالم المصغر ، حيث تظهر التجارب القدرة على تعديل التنوع الميكروبي البيئي لاستكشاف العلاقات بين التنوع الميكروبي البيئي وتكوين ميكروبيوم الأمعاء الدودية.
Abstract
تظهر الديدان الخيطية Caenorhabditis elegans كنموذج مفيد لدراسة الآليات الجزيئية الكامنة وراء التفاعلات بين المضيفين وميكروبيوم الأمعاء. في حين أن التجارب مع البكتيريا ذات الخصائص الجيدة أو المجتمعات البكتيرية المحددة يمكن أن تسهل تحليل الآليات الجزيئية ، فإن دراسة الديدان الخيطية في سياقها الميكروبي الطبيعي أمر ضروري لاستكشاف تنوع هذه الآليات. في الوقت نفسه ، فإن عزل الديدان عن البرية ليس ممكنا دائما ، وحتى عندما يكون ذلك ممكنا ، فإن أخذ العينات من البرية يقيد استخدام مجموعة الأدوات الجينية المتاحة بخلاف ذلك لأبحاث C. elegans. يصف البروتوكول التالي طريقة لدراسات الميكروبيوم باستخدام عوالم مصغرة للسماد للنمو داخل المختبر في بيئات متنوعة ميكروبيا وشبيهة بالطبيعة.
يمكن إثراء التربة من مصادر محلية بالمنتجات لتنويع المجتمعات الميكروبية التي تربى فيها الديدان والتي يتم حصادها وغسلها وتعقيمها سطحيا لإجراء تحليلات لاحقة. تظهر التجارب التمثيلية القدرة على تعديل المجتمع الميكروبي في تربة مشتركة عن طريق إثرائها بمنتجات مختلفة وتثبت كذلك أن الديدان التي تربى في هذه البيئات المتميزة تجمع ميكروبيوم الأمعاء المماثل المتميز عن بيئاتها الخاصة ، مما يدعم فكرة ميكروبيوم الأمعاء الأساسي الخاص بالأنواع. بشكل عام ، توفر العوالم المصغرة للسماد بيئات معملية شبيهة بالطبيعة لأبحاث الميكروبيوم كبديل للمجتمعات الميكروبية الاصطناعية أو لعزل الديدان الخيطية البرية.
Introduction
تظهر الديدان الخيطية Caenorhabditis elegans كنموذج مفيد لدراسة التفاعلات بين المضيفين وميكروبيوم الأمعاء 1,2. كنموذج ، فإنه يوفر العديد من المزايا. أولا ، من السهل الحصول على الحيوانات الخالية من الجراثيم أو gnotobiotic وصيانتها ؛ يمكن استخدام المبيض لقتل الديدان الجاذبة والميكروبات المرتبطة بها ، تاركا بيضها المقاوم للتبييض دون أن يصاب بأذى لينمو كمجموعات متزامنة مع العمر يمكن أن تستعمرها البكتيريا ذات الأهمية 3,4. بالإضافة إلى ذلك ، عندما تزرع في وجود البكتيريا ، فإن C. elegans ، وهي بكتيريا ، تبتلع البكتيريا التي تمت مواجهتها ، مع هضم الأنواع الحساسة أو إفرازها ، في حين أن الأنواع المقاومة والمستمرة تستعمر أمعاء الدودة بثبات. علاوة على ذلك ، فإن C. elegans هي في الغالب خنثى ، وتنتج مجموعات من ذرية متطابقة وراثيا ، مما يقلل من التباين الجيني المربك. إلى جانب توافر سلالات الديدان الطافرة والمحورة وراثيا ، فإن العمل مع C. elegans يقدم للباحثين نموذجا جنوبيوتيا وقابلا للتتبع وراثيا للتحقيق في الأسس الجزيئية للتفاعلات بين المضيف والميكروب5،6،7،8.
في حين أن التجارب على البكتيريا ذات الخصائص الجيدة يمكن أن تسهل تحليل الآليات الجزيئية ، فإن تحديد ودراسة البكتيريا التي تتفاعل معها الديدان في الطبيعة أمر ضروري لاستكشاف تنوع هذه الآليات ، وكشف السياق الطبيعي لوظيفتها ، وفهم القوى الانتقائية التي شكلت تطورها. خارج المختبر ، تم العثور على C. elegans عالميا في المناخات المعتدلة الرطبة ، حيث يعتقد أن السكان يخضعون لدورة حياة "الازدهار والكساد" ، والتي تتميز بالنمو السكاني السريع عندما تكون الموارد وفيرة ، يليها تحول تنموي إلى dauers رائدة وتتحمل الإجهاد عندما يتم استنفاد الموارد9. على الرغم من اعتبارها نيماتودا في التربة ، إلا أن مجموعات C. elegans المتكاثرة في البرية هي الأكثر شيوعا تتغذى على المواد العضوية المتحللة مثل الزهور أو الفواكه المتعفنة ، حيث تكون مجموعات البكتيريا وفيرة ومتنوعة.
حددت الدراسات التي أجريت على ميكروبيوم الأمعاء في الديدان الخيطية المعزولة من البرية مجتمعات بكتيرية متنوعة ، لكنها مميزة ، 10,11 ، تم دعم تكوينها بشكل أكبر من خلال الدراسات التي أجريت مع الديدان التي نشأت في بيئات مصغرة شبيهة بالطبيعة 12,13. مكنت هذه الدراسات معا من تحديد ميكروبيوم الأمعاء الدوديالأساسي 2. في حين أن أخذ عينات من مجموعات C. elegans في البرية يمثل الفحص الأكثر مباشرة للتفاعلات الطبيعية بين الدودة والميكروب ، إلا أنه غير ممكن في كل مكان وزمان ، لأنه يقتصر على المناطق والمواسم ذات الأمطار الوفيرة10,11. بدلا من ذلك ، بدلا من عزل الديدان عن بيئتها الطبيعية ، تجلب التجارب التي تستخدم العوالم المصغرة الموائل الطبيعية إلى المختبر6،8،12،13،14،15. يتم تحضير بيئات العالم المصغر من التربة المصنفة مع العديد من الفواكه أو الخضروات ، مما يتيح مزيدا من التنويع لمجتمع التربة الأولي. وهي توفر طرقا تجريبية قابلة للتتبع تجمع بين التنوع الميكروبي وبيئة التربة البرية ثلاثية الأبعاد مع المزايا التجريبية لمرفق مختبر خاضع للرقابة وسلالات دودة محددة وراثيا. يوضح البروتوكول أدناه بالتفصيل الخطوات التي ينطوي عليها العمل مع العوالم المصغرة للسماد ، مما يدل على استخدامها في فهم تجميع ميكروبيوم الأمعاء الدودي المميز من بيئات متنوعة.
Protocol
1. تحضير السماد
- احصل على السماد أو تربة الحديقة من أي مصدر مناسب وقم بتخزينه داخل المختبر في حاوية بلاستيكية قياسية للمطبخ مع فتحات مقطوعة في الغطاء للسماح بدخول الهواء. سد الثقوب بالصوف القطني لإبعاد ذباب الفاكهة واللافقاريات الأخرى (الشكل 1 أ).
ملاحظة: خمسمائة جرام من التربة (التركيب في حاوية سعة 1.5 جالون ، الأبعاد: 30 سم × 20 سم × 10 سم) ستوفر مادة كافية ل 12 عالما صغيرا. - قم بإثراء السماد أو التربة بالمنتجات المفرومة أو خليط من المنتجات المختلفة بنسبة كتلة 1: 2 من المنتجات إلى التربة.
- احتضن لمدة 7-14 يوما عند 20-25 درجة مئوية ، واخلطها مرة واحدة يوميا وأضف وسط M9 حسب الضرورة للحفاظ على الرطوبة دون جعلها موحلة.
ملاحظة: التربة غير المخصبة بالمنتجات عادة لا تدعم نمو C. elegans ، ولكن أي منتج محدد يجب استخدامه متروك للباحث ، مع العديد من الأنواع والخلطات القادرة على دعم نمو الديدان. سيعزز التخصيب بمنتجات مختلفة تنويع المجتمع البكتيري بطرق مختلفة ، مما يتيح دراسة تجميع ميكروبيوم الأمعاء من نقاط انطلاق مختلفة (انظر قسم المناقشة).
2. إعداد السماد microalsms
- لكل صورة مصغرة ، أضف 10 جم من السماد المخصب إلى دورق زجاجي سعة 30 مل مغطى بورق القصدير والأوتوكلاف (الشكل 1 ب).
- لتحضير المستخلص الميكروبي لتجديد السماد المعقم ، ابدأ بإضافة 30 جم من نفس السماد المستخدم في الخطوة 2.1 إلى كل أنبوب من ثلاثة أنابيب سعة 50 مل واملأها ب M9. دوامة لمدة دقيقة واحدة (الشكل 1C).
ملاحظة: يجب أن يوفر هذا ما يكفي من البكتيريا لتسعة عوالم مصغرة ، كل منها يدعم تطوير مئات الديدان الخيطية. - أجهزة الطرد المركزي للأنابيب عند 560 × جم لمدة 5 دقائق في درجة حرارة الغرفة (RT).
- مع الانتباه إلى عدم إزعاج الحبيبات ، قم بإزالة المواد الطافية باستخدام ماصة مصلية وادمجها في أنبوب جديد سعة 50 مل.
- ركز المستخلص البكتيري عن طريق الطرد المركزي بأقصى سرعة (2000 × جم) لمدة 15 دقيقة في RT. أعد تعليق الكريات في M9 بما يكفي للحصول على 200 ميكرولتر لكل صورة مصغرة و 200 ميكرولتر أكثر لإضافتها إلى لوحة ستكون بمثابة وكيل مرئي لتطور الديدان داخل العوالم الدقيقة.
ملاحظة: على سبيل المثال ، بالنسبة لتسعة عوالم مصغرة ، أعد تعليق الحبيبات الميكروبية في 2 مل من M9. - أضف 200 ميكرولتر من المستخلص الميكروبي المركز إلى كل دورق من السماد المعقم ، وكذلك إلى لوحة NGM التي ستكون بمثابة لوحة الوكيل المرئية.
- احتضان العوالم المصغرة ولوحة الوكيل لمدة 24 ساعة عند 20-25 درجة مئوية قبل إضافة الديدان.
3. تربية الديدان في عوالم مصغرة السماد
- أضف 500-1000 بيضة أو يرقات L13 إلى كل صورة مصغرة وإلى لوحة الوكيل (الخطوة 2.7) لبدء التجربة (الشكل 1D).
ملاحظة: تستخدم التجارب الموصوفة هنا الديدان البرية من النوع N2. ومع ذلك ، يمكن استخدام سلالات C . elegans الأخرى (وربما الديدان الخيطية الأخرى). - ارفع الديدان إلى مرحلة البلوغ عند 20 درجة مئوية (عادة 3 أيام).
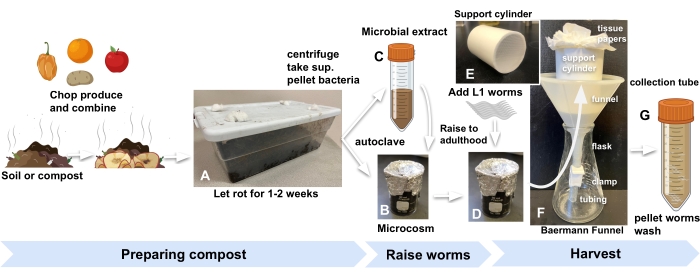
الشكل 1: تحضير العوالم المصغرة للسماد، وتربية الديدان، والحصاد . (أ) إثراء التربة المحلية أو السماد بالمنتجات واحتضانها لمدة 2 أسابيع. اجمع بين (B) التربة المخصبة المعقمة مع (C) المستخلص الميكروبي و (D) الاحتضان لمدة 24 ساعة على الأقل قبل إضافة ديدان L1s المتزامنة إلى العوالم المصغرة لبدء التجربة. (ه، واو) عندما تكون جاهزا للحصاد ، أضف السماد من العالم المصغر إلى أسطوانة دعم قمع بيرمان وقم بتغطيتها ب M9. (ز) بعد 15 دقيقة، حرر الراشح في أنبوب سعة 50 مل. اختصار: sup. = طاف طاف. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
4. تحضير قمع بيرمان لحصاد الديدان
- قم بتجميع قمع بيرمان عن طريق ربط 5-8 سم من الأنابيب المطاطية الصلبة بنهاية قمع بلاستيكي.
- حرك مشبكا على الأنبوب وأغلقه.
- ضع في القمع أنبوبا أسطوانيا PVC بطول 7 سم وقطر 5 سم مع شبكة نايلون 1 مم ملتصقة في قاعها (الشكل 1E). قم بتبطين الأسطوانة بورقتين من المناديل الورقية.
- ضع قمع بيرمان في دورق (الشكل 1F).
5. حصاد الديدان من العوالم المصغرة وجمع عينات التربة المعنية
- أضف 20 مل من M9 إلى العالم المصغر الذي نشأت فيه الديدان ، وقم بتحريك الخليط ، ثم صب الخليط من الدورق في الأسطوانة المبطنة بورق المناديل في إعداد قمع بيرمان. أضف المزيد من M9 لغمر السماد بالكامل في القمع.
ملاحظة: تصل مجموعات C. elegans (N2) إلى مرحلة البلوغ في عوالم مصغرة للسماد بمعدل مماثل كما هو الحال في ألواح الآجار القياسية المصنفة بالإشريكية القولونية. لمزيد من الدقة في حصاد الديدان في مرحلة نمو معينة ، راجع لوحة الوكيل من الخطوة 3.3. - بعد 30 دقيقة ، قم بفك المشبك لتحرير الراشح الذي يحتوي على الديدان المحصودة في أنبوب سعة 50 مل (الشكل 1G).
- أضف المزيد من M9 إلى الأسطوانة وكرر الجولة الثانية لحصاد المزيد من الديدان ، ومرة أخرى إذا كانت هناك حاجة إلى ديدان إضافية.
ملاحظة: عند تكرار جولات الحصاد ، احرص على عدم المساس بسلامة المناديل الورقية ، مما قد يسمح بمرور جزيئات التربة. يجب أن تعزل أربع جولات حصاد كحد أقصى ما لا يقل عن 50٪ من الديدان المضافة أصلا إلى العالم المصغر دون المساس بسلامة المناديل الورقية. - تركيز الديدان عن طريق الطرد المركزي في 560 × غرام لمدة 2 دقيقة (RT). إزالة 35 مل من الطافي مع ماصة المصلية.
- انقل ال 15 مل المتبقية إلى أنبوب سعة 15 مل وأجهزة طرد مركزي مرة أخرى عند 560 × جم لمدة دقيقة واحدة لزيادة تركيز الديدان. إزالة 14 مل من الطافي مع ماصة المصلية.
- في موازاة ذلك ، اجمع 1 غرام من التربة المصغرة المتبقية في أنبوب سعة 1.5 مل. معالجة عينات التربة التي تحتوي على المجتمع البكتيري البيئي على الفور أو تخزينها في -20 درجة مئوية لاستخراج الأحماض النووية لاحقا ، كما هو موضح أدناه لعينات الديدان.
6. غسل وتعقيم سطح الديدان المحصودة
- انقل 1 مل من الديدان المركزة المحصودة من الخطوة 5.5 إلى أنبوب سعة 1.5 مل باستخدام ماصة زجاجية. احتضان لمدة 2 دقيقة للسماح للديدان بالاستقرار في قاع الأنبوب. قم بإزالة المادة الطافية ، تاركا قاع 100 ميكرولتر دون إزعاج.
- اغسل 6x ب 1.5 مل من M9 + T (0.025٪ Triton-X في M9) ، مما يسمح للديدان بالاستقرار في القاع في كل مرة.
- انقل الديدان المغسولة بحجم 100 ميكرولتر إلى أنبوب جديد سعة 1.5 مل باستخدام ماصة زجاجية.
ملاحظة: في هذه المرحلة من البروتوكول ، يجب أن يكون قد مر ما يقرب من 30 دقيقة منذ الغسيل الأول (الخطوة 6.2). يوصى بالسماح للديدان بالبقاء بدون طعام لمدة 1 ساعة على الأقل قبل إضافة الليفاميزول للسماح بإفراز البكتيريا العابرة والهضم الكامل للبكتيريا الغذائية. - أضف 100 ميكرولتر من 25 مللي مول ليفاميزول هيدروكلوريد لشل الديدان. احتضان لمدة 5 دقائق في RT.
- أضف 200 ميكرولتر من محلول التبييض بنسبة 4٪. احتضان لمدة 2 دقيقة.
- قم بإزالة المادة الطافية ، واترك الجزء السفلي 150 ميكرولتر دون إزعاج ، واغسل 3x باستخدام M9 + T ، على النحو الوارد أعلاه.
ملاحظة: لتقليل تلوث العينات ، يوصى باستخدام M9 + T المصفى (من خلال مرشح 0.2 ميكرومتر) وارتداء القفازات من هذه النقطة فصاعدا. - بعد الغسيل ، خذ 50 ميكرولتر من 150 ميكرولتر المتبقية من آخر غسلة ولوحة على لوحة رطل ، مع الحضانة عند 25 درجة مئوية لمدة 48 ساعة لتأكيد الإزالة الفعالة للبكتيريا الخارجية.
ملاحظة: يسمح بمراقبة ما يصل إلى 30 مستعمرة في الغسيل الأخير (تمثل 60 خلية بكتيرية خارجية إجمالي ما تبقى في العينة) ومن المتوقع أن يكون لها مساهمة هامشية فقط في تكوين الميكروبيوم الذي تم تحليله ، بالنظر إلى آلاف البكتيريا التي تستعمر عادة كل دودة بالغة15. عند ملاحظة المزيد ، قد تتعرض سلامة العينة للخطر. - استخدم الديدان المعقمة سطحيا على الفور (على سبيل المثال ، لاستخراج البكتيريا الحية للزراعة [عدد CFU]) أو تخزينها عند -20 درجة مئوية لاستخراج الأحماض النووية لاحقا.
7. استخراج الحمض النووي
ملاحظة: تصف الخطوات التالية استخراج الحمض النووي للديدان المحصودة باستخدام مجموعة تجارية مصممة لاستخراج الحمض النووي الميكروبي من التربة (انظر جدول المواد) ، مع التعديلات الموضحة أدناه لتسهيل استخراج الحمض النووي الميكروبي من الديدان.
- نقل العينة التي تحتوي على ديدان معقمة سطحيا (تذويب في RT إذا لزم الأمر) إلى الأنابيب التي توفرها المجموعة ، واستبدال الخرز الزجاجي المقدم بخرز زركونيا قطره 30-50 1 مم تقريبا (انظر جدول المواد).
ملاحظة: تكون غلة الحمض النووي المرصودة أعلى مع حبات الزركونيا مقارنة بالخرز الزجاجي الذي توفره المجموعة. على الرغم من أن السبب وراء هذه الملاحظة لا يزال غير محدد ، إلا أن حبات الزركونيا قد تفتح بشرة الديدان الخيطية بشكل أكثر كفاءة ، مما يؤدي إلى إطلاق المزيد من البكتيريا. - بالنسبة لعينات السماد ، أضف ما يقرب من 250 مجم من التربة المجمعة إلى أنابيب المجموعة دون استبدال الخرز الزجاجي.
- بعد إضافة محلول المخزن المؤقت الذي توفره المجموعة إلى جميع العينات ، قم بالتجانس باستخدام خالط كهربائي في RT (انظر جدول المواد) لجولتين من 2000 دورة في الدقيقة لمدة 30 ثانية لكل منهما ، مع التوقف مؤقتا لمدة 30 ثانية بينهما.
- أكمل خطوات تنقية الحمض النووي المتبقية وفقا لبروتوكول المجموعة. قم بتخفيف عينات الدودة في ما لا يزيد عن 50 ميكرولتر من محلول الشطف لضمان تركيزات الحمض النووي العالية بما يكفي للتسلسل.
النتائج
لاستكشاف القدرة على تنويع مجتمع العوالم المصغرة للتربة ، قارنا المجتمعات الميكروبية في عوالم مصغرة للسماد تم تحضيرها عن طريق إثراء نفس التربة الأولية ، وهو سماد صناعي متاح من مدينة بيركلي ، كاليفورنيا ، مع منتجات مختلفة: التفاح أو الفلفل الحلو أو البرتقال أو البطاطس (كل مجموعة في ثلاث نسخ). قمنا كذلك بمقارنة المجتمعات الميكروبية لكل بيئة سماد مع ميكروبيوم الأمعاء من النوع البري C. elegans الذي نشأ في العالم المصغر المعني. تم إجراء التحليل باستخدام عينات الحمض النووي المستخرجة من حوالي 500 بالغ معقم سطحيا لكل صورة مصغرة ومن عينات سماد 250 مجم من العوالم المصغرة المعنية.
اعتمد توصيف التربة البيئية وميكروبيوم الأمعاء الدودية على تسلسل الجيل التالي لمنطقة V4 من جين 16S rRNA البكتيري. تم إعداد مكتبة التسلسل باستخدام المجموعات القياسية وتم تنفيذها وفقا لتعليمات الشركات المصنعة ، مع إجراء التسلسل على جهاز تسلسل تجاري (انظر جدول المواد). تمت معالجة التسلسلات غير المتعددة باستخدام DADA2 ، والتصنيف المخصص بناء على قاعدة البيانات المرجعية SILVA v132 ، وتحليلها باستخدام phyloseq16،17،18 (انظر الملف التكميلي 1 ، الشكل التكميلي S1 ، الشكل التكميلي S2 ، الشكل التكميلي S3 ، الجدول التكميلي S1 ، والجدول التكميلي S2 للحصول على وصف مفصل للتسلسل والتحليل ؛ يتوفر خط الأنابيب الحسابي الكامل في GitHub [https://github.com/kennytrang/CompostMicrocosms]). تتوفر البيانات الأولية في أرشيف قراءة تسلسل NCBI (معرف المشروع الحيوي PRJNA856419).
في المتوسط ، تم الحصول على 73220 تسلسلا لكل عينة. تمثل هذه التسلسلات 15027 متغيرا من تسلسل الأمبليكون (ASVs) ، تغطي 27 شعبة و 216 عائلة ، بما في ذلك العائلات التي تعتبر جزءا من ميكروبيوم الأمعاء C. elegans الأساسي 13 ، مثل Rhizobiaceae و Burkholderiaceae و Bacillaceae. كانت البكتيريا المعوية ، و Pseudomonadaceae ، التي وجد سابقا أنها أعضاء مهيمنة ، أقلية هذه المرة ، لكنها لا تزال مخصبة (2-10 أضعاف) مقارنة ببيئات التربة الخاصة بها. أظهرت المقارنات القائمة على مسافات UniFrac19,20 غير المرجحة والمرجحة إمكانية استنساخ جيدة بين ثلاثية العالم المصغر المخصب بنفس المنتج ، كما يتضح من التجميع الوثيق. في المقابل ، فإن ميكروبيوم التربة البيئي المخصب بمنتجات مختلفة متجمعة بعيدا عن بعضها البعض ، مما يدل على القدرة على تنويع مجتمع ميكروبي أولي من خلال إضافة منتجات مختلفة (الشكل 2).
في مقارنات ميكروبيوم الأمعاء الدودية والمجتمعات البيئية ، أظهر تحليل الإحداثيات الرئيسي (PCoA) مع مسافات UniFrac غير المرجحة أو المرجحة تجمعا متميزا لميكروبات الأمعاء الدودية بعيدا عن بيئاتها الخاصة لكل نوع من أنواع العالم المصغر (الشكل 2). في حين أن PCoA القائم على مسافات UniFrac غير المرجحة لم يميز بين ميكروبيوم التربة والديدان (الشكل 2 أ) ، فإن التجميع القائم على المسافات المرجحة كشف عن فصل واضح بين أمعاء الدودة وميكروبيوم السماد (الشكل 2 ب). تدعم هذه النتائج عملية تعمل فيها ترشيح المضيف على التوافر البيئي لتشكيل ميكروبيوم الأمعاء الذي لا يختلف تماما عن مصدره البيئي فيما يتعلق بوجود الأصناف ولكنه يعدل وفرتها عن طريق إثراء مجموعة فرعية من الأصناف المتاحة ، مما يؤدي في النهاية إلى ميكروبيوم أمعاء الدودة الأساسية المشتركة بين الديدان التي تربى في بيئات مختلفة.

الشكل 2: ميكروبيوم الأمعاء الدودية يتجمع بعيدا عن البيئات الميكروبية المتنوعة المنتجات الخاصة بكل منها. تم تحديد تكوين الميكروبيوم باستخدام تسلسل 16S ، وتم تجميع المجتمعات من العوالم المصغرة المخصبة بالمنتجات المحددة أو من الديدان التي أثيرت فيها باستخدام PCoA بناء على (A) مسافات UniFrac غير مرجحة أو (B). المحاور الموضحة هي تلك التي تفسر أكبر تباين في تكوين المجتمع بين العينات (N = 3 لكل نوع من أنواع العالم المصغر). الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
الملف التكميلي 1: الجيل التالي من التسلسل وتحليل البيانات. فيما يلي خطوات إعداد المكتبة والتسلسل داخل المختبر وتحليل البيانات. الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي S1: مثال على الرسم البياني لمراقبة الجودة للقراءات العكسية من عينة واحدة. يظهر المحور X (دورة) موضع النوكليوتيدات على طول قراءة التسلسل. يظهر المحور Y الأيسر نقاط الجودة. تمثل خريطة الحرارة ذات التدرج الرمادي تردد درجة الجودة في كل موضع نيوكليوتيدات. يصور الخط الأخضر متوسط درجة الجودة في كل موضع نيوكليوتيدات. يصور الخط البرتقالي العلوي أرباع توزيع نقاط الجودة ؛ يصور الخط الأحمر السفلي النسبة المئوية لقراءات التسلسل التي مددت موضع النوكليوتيدات (المحور Y الأيمن ، هنا 100٪). الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي S2: معدلات الخطأ للعينات المختلفة. يجب أن ينخفض تكرار الخطأ في العينات المختلفة (النقاط السوداء) مع زيادة نقاط الجودة لكل استبدال محتمل لزوج القاعدة الموضح ، مما يعكس الاتجاه المتوقع. الرجاء الضغط هنا لتنزيل هذا الملف.
الشكل التكميلي S3: مثال على PCoA بناء على مسافات UniFrac المرجحة. تمثل أسماء المجموعات الموضحة في وسيلة الإيضاح المنتجات المستخدمة لإثراء السماد المستخدم في العوالم المصغرة المختلفة. الرجاء الضغط هنا لتنزيل هذا الملف.
الجدول التكميلي S1: تصفية التسلسل المتسلسل. أ عدد قراءات التسلسل قبل التصفية. b-d يمثل كل عمود عدد قراءات التسلسل المتبقية بعد خطوة الترشيح: تصفية القراءات منخفضة الجودة (الخطوة 2.5) ، وخوارزمية تقليل الضوضاء التي يقوم بها dada () (الخطوة 2.8) ، ودمج القراءات الأمامية والعكسية (الخطوة 2.9) ، وإزالة الوهم (الخطوة 2.11). الرجاء الضغط هنا لتنزيل هذا الملف.
الجدول التكميلي S2: جدول البيانات الوصفية. الرجاء الضغط هنا لتنزيل هذا الملف.
Discussion
يصف البروتوكول المقدم هنا طريقة لدراسة ميكروبيوم الأمعاء للديدان الخيطية التي تربى في بيئات شبيهة بالطبيعة ، ويقدم نهجا بديلا لعزل الديدان عن الطبيعة أو تربيتها على المجتمعات الاصطناعية.
تعكس الآلاف من الأنواع البكتيرية المحتملة التي تم التقاطها في تجربة العالم المصغر التمثيلي التنوع الميكروبي الذي تطورت معه الديدان وتثبت قدرة خط أنابيب العالم المصغر على الجمع بين مزايا العمل مع كائن حي مضيف نموذجي ومزايا العمل مع المجتمعات الميكروبية الطبيعية المتنوعة.
تظهر النتائج التمثيلية أن إثراء التربة المشتركة بأنواع مختلفة من المنتجات يعدل التنوع الميكروبي البيئي ، مما يسلط الضوء على نطاق التنوع الميكروبي المتاح للاستكشاف باستخدام خط الأنابيب هذا. اختيار الإنتاج ليس مهما بشكل خاص. استخدمت الأعمال السابقة الموز والتفاح والبرتقال والفراولة وأوراق الشاي الأخضر والبطاطس لإثراء التربة ، مما أدى إلى كفاءة مماثلة في رفع الديدان. كما تم استخدام المنتجات المختلطة بشكل فعال. السمة الرئيسية هي أن المنتجات المختلفة ستنوع تربة معينة بطرق متميزة.
على الرغم من التباين الواسع في التنوع الميكروبي البيئي ، فإن ميكروبيوم الأمعاء الدودية تختلف اختلافا أقل بكثير ، مما يلخص أهمية مكانة الأمعاء الدودية وترشيح المضيف لتجميع ميكروبيوم الأمعاء الأساسي الذي يختلف عن بيئة التربة13. يتم ملاحظة هذا النمط بشكل أفضل مع تحليل UniFrac PCoA المرجح ، مما يدل على أن الاختلافات بين التربة البيئية وميكروبيوم الأمعاء الدودية تنبع في الغالب من الاختلافات في الوفرة النسبية للأصناف الرئيسية.
على الرغم من أن البروتوكول الموصوف هنا يركز على حصاد الديدان لتسلسل 16S ، يمكن استخدام العوالم المصغرة لاستكشاف أسئلة إضافية ذات أهمية. على سبيل المثال ، يمكن فحص الديدان التي يتم حصادها من العوالم المصغرة لاحقا لمعرفة تأثير ميكروبيوم الأمعاء المتنوع على مقاومة المضيف لمختلف الظروف المعاكسة ، بما في ذلك مسببات الأمراض أو السموم. وبدلا من ذلك، يمكن عزل الأنواع والسلالات البكتيرية الجديدة واستزراعها من الديدان المحصودة على الأرض، مما يوسع التنوع التصنيفي والوظيفي للبكتيريا المتاحة لإجراء التجارب عليها.
بينما تسعى طرق البحث في البيئات المختبرية إلى الاتساق والتكاثر ، فإن العمل مع العوالم المصغرة يستفيد من الاختلاف الطبيعي لاستكشاف تفاعلات الميكروب المضيف في سياق يشبه الطبيعة. ومع ذلك ، فإن هذا الاختلاف يطرح أيضا بعض التحديات. قد تتطلب بعض أنواع التربة التي تحتوي على مستويات عالية من اللافقاريات الداخلية خطوات إضافية للطرد المركزي والترشيح والفحص للقضاء بشكل فعال على الكائنات الحية غير المرغوب فيها من إعداد العالم المصغر. بالإضافة إلى ذلك ، قد تؤدي الوفرة الميكروبية المنخفضة في التربة إلى تكوين دواير غير مرغوب فيه في مجموعات الديدان ، مما يتطلب زيادة في المستخلص الميكروبي أو إثراء التربة بكمية أكبر من المنتجات. مع كل تجربة مصغرة يتم إجراؤها ، يواصل الباحثون استكشاف التنوع التصنيفي والوظيفي الكامل الذي توفره الطبيعة ، مما يتيح اكتشاف أصناف ميكروبية جديدة وقدرات وظيفية تتراوح من مقاومة العدوى إلى الحماية من الكائنات الحية الغريبة البيئية.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للإعلان عن أنه ذو صلة بمحتوى هذه المقالة.
Acknowledgements
تم دعم العمل الموصوف في هذه المخطوطة من خلال منح المعاهد الوطنية للصحة R01OD024780 و R01AG061302. تم دعم KT أيضا من خلال زمالة بحثية صيفية للطلاب الجامعيين من جامعة كاليفورنيا ، بيركلي ، بتمويل من مؤسسة روز هيلز. تم الحصول على تصاميم الرسوم المتحركة في الشكل 1 من BioRender.com.
Materials
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
References
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved