Method Article
Microcosmos de composto como ambientes microbianamente diversos e naturais para pesquisa de microbioma em Caenorhabditis elegans
Neste Artigo
Resumo
Os microcosmos de composto trazem a diversidade microbiana encontrada na natureza para o laboratório para facilitar a pesquisa do microbioma em Caenorhabditis elegans. Aqui são fornecidos protocolos para a criação de experimentos de microcosmos, com os experimentos demonstrando a capacidade de modular a diversidade microbiana ambiental para explorar as relações entre a diversidade microbiana ambiental e a composição do microbioma intestinal do verme.
Resumo
O nematoide Caenorhabditis elegans está emergindo como um modelo útil para estudar os mecanismos moleculares subjacentes às interações entre hospedeiros e seus microbiomas intestinais. Embora experimentos com bactérias bem caracterizadas ou comunidades bacterianas definidas possam facilitar a análise de mecanismos moleculares, estudar nematoides em seu contexto microbiano natural é essencial para explorar a diversidade de tais mecanismos. Ao mesmo tempo, o isolamento de vermes da natureza nem sempre é viável e, mesmo quando possível, a amostragem da natureza restringe o uso do kit de ferramentas genéticas de outra forma disponível para a pesquisa de C. elegans. O protocolo a seguir descreve um método para estudos de microbioma utilizando microcosmos de composto para o crescimento em laboratório em ambientes microbianamente diversos e naturais.
O solo de origem local pode ser enriquecido com produtos para diversificar as comunidades microbianas nas quais os vermes são criados e dos quais são colhidos, lavados e esterilizados superficialmente para análises subsequentes. Experimentos representativos demonstram a capacidade de modular a comunidade microbiana em um solo comum, enriquecendo-o com diferentes produtos e demonstram ainda que os vermes criados nesses ambientes distintos montam microbiomas intestinais semelhantes distintos de seus respectivos ambientes, apoiando a noção de um microbioma intestinal central específico da espécie. No geral, os microcosmos de composto fornecem ambientes naturais em laboratório para a pesquisa do microbioma como uma alternativa às comunidades microbianas sintéticas ou ao isolamento de nematoides selvagens.
Introdução
O nematoide Caenorhabditis elegans está emergindo como um modelo útil para o estudo das interações entre hospedeiros e seus microbiomas intestinais 1,2. Como modelo, oferece várias vantagens. Em primeiro lugar, os animais livres de germes ou gnotobióticos são fáceis de obter e manter; A água sanitária pode ser usada para matar vermes gravídicos e micróbios associados, deixando seus ovos resistentes à água sanitária ilesos para crescer como populações sincronizadas com a idade que podem ser colonizadas por bactérias de interesse 3,4. Além disso, quando cultivada na presença de bactérias, C. elegans, um bacterívoro, ingere as bactérias encontradas, com espécies suscetíveis digeridas ou excretadas, enquanto espécies resistentes e persistentes colonizam de forma estável o intestino do verme. Além disso, C. elegans é principalmente hermafrodita, produzindo populações de progênie geneticamente idêntica, o que reduz a variação genética confusa. Juntamente com a disponibilidade de cepas de vermes mutantes e transgênicos, o trabalho com C. elegans oferece aos pesquisadores um modelo gnotobiótico e geneticamente tratável para investigar os fundamentos moleculares das interações hospedeiro-micróbio 5,6,7,8.
Embora experimentos com bactérias bem caracterizadas possam facilitar a análise de mecanismos moleculares, identificar e estudar as bactérias com as quais os vermes interagem na natureza são essenciais para explorar a diversidade de tais mecanismos, desvendar o contexto natural para sua função e entender as forças seletivas que moldaram sua evolução. Fora do laboratório, C. elegans é encontrada globalmente em climas temperados úmidos, onde se acredita que as populações passem por um ciclo de vida de "boom-and-bust", caracterizado pelo rápido crescimento populacional quando os recursos são abundantes, seguido por uma mudança de desenvolvimento para dauers pioneiros e tolerantes ao estresse quando os recursos estão esgotados9. Embora considerado um nematoide do solo, as populações de C. elegans em proliferação selvagem são mais comumente encontradas alimentando-se de material orgânico em decomposição, como flores ou frutos em decomposição, onde as populações bacterianas são abundantes e diversificadas.
Estudos do microbioma intestinal em nematoides isolados da natureza identificaram comunidades bacterianas diversas, porémcaracterísticas10,11, cuja composição foi ainda mais apoiada por estudos realizados com vermes criados em ambientes de microcosmos semelhantes aos naturais12,13. Juntos, tais estudos permitiram o delineamento de um microbioma intestinal de vermes centrais2. Considerando que a amostragem de populações de C. elegans na natureza representa o exame mais direto das interações verme-micróbio natural, não é viável em todos os lugares e a qualquer momento, pois é limitada a regiões e estações com ampla precipitação10,11. Alternativamente, em vez de isolar vermes de seu habitat natural, experimentos usando microcosmos trazem o habitat natural para o laboratório 6,8,12,13,14,15. Os ambientes de microcosmos são preparados a partir do solo compostado com várias frutas ou legumes, o que permite uma maior diversificação da comunidade de solo inicial. Eles oferecem métodos experimentais tratáveis que combinam a diversidade microbiana e o ambiente tridimensional do solo selvagem com as vantagens experimentais de uma instalação de laboratório controlada e cepas de vermes geneticamente definidas. O protocolo abaixo detalha as etapas envolvidas no trabalho com microcosmos de composto, demonstrando seu uso na compreensão da montagem de um microbioma intestinal característico de diversos ambientes.
Protocolo
1. Preparação do composto
- Obtenha composto ou solo de jardim de qualquer fonte conveniente e armazene dentro do laboratório em um recipiente de plástico de cozinha padrão com furos cortados na tampa para deixar o ar entrar. Tapar os orifícios com algodão para manter as moscas da fruta e outros invertebrados fora (Figura 1A).
NOTA: Quinhentos gramas de solo (encaixe em um recipiente de 1,5 galão, dimensões: 30 cm x 20 cm x 10 cm) fornecerão material suficiente para 12 microcosmos. - Enriquecer o composto ou o solo com produtos picados ou uma mistura de produtos diferentes numa proporção de massa de 1:2 de produtos para solo.
- Incubar durante 7-14 dias a 20-25 °C, misturando uma vez por dia e adicionando M9 meio conforme necessário para manter a humidade sem a tornar lamacenta.
NOTA: Solos não enriquecidos com produtos geralmente não suportam o crescimento de C. elegans , mas qual produto específico a ser usado cabe ao pesquisador, com muitos tipos e misturas capazes de suportar o crescimento de vermes. O enriquecimento com diferentes produtos promoverá a diversificação da comunidade bacteriana de diferentes maneiras, permitindo o estudo da montagem do microbioma intestinal a partir de diferentes pontos de partida (veja a seção de discussão).
2. Preparação de microcosmos de composto
- Para cada microcosmo, adicionar 10 g de composto enriquecido a um copo de vidro de 30 mL coberto com folha de estanho e autoclave (Figura 1B).
- Para preparar o extrato microbiano para reabastecer o composto autoclavado, comece adicionando 30 g do mesmo composto usado na etapa 2.1 a cada um dos três tubos de 50 mL e encha com M9. Vórtice por 1 min (Figura 1C).
NOTA: Isso deve fornecer bactérias suficientes para nove microcosmos, cada um apoiando o desenvolvimento de centenas de nematoides. - Centrifugar os tubos a 560 × g durante 5 min à temperatura ambiente (RT).
- Prestando atenção para não perturbar o pellet, retire os sobrenadantes com uma pipeta sorológica e misture em um novo tubo de 50 mL.
- Concentrar o extrato bacteriano centrifugando na velocidade máxima (2.000 × g) por 15 min no RT. Resuspenda os pellets em M9 suficiente para ter 200 μL para cada microcosmo e mais 200 μL para adicionar a uma placa que servirá como um proxy visível do desenvolvimento de vermes dentro dos microcosmos.
NOTA: Por exemplo, para nove microcosmos, ressuspeite o pellet microbiano em 2 mL de M9. - Adicione 200 μL do extrato microbiano concentrado a cada copo de composto autoclavado, bem como a uma placa NGM que servirá como placa proxy visível.
- Incubar microcosmos e placa proxy durante 24 h a 20-25 °C antes da adição de vermes.
3. Criação de vermes em microcosmos de composto
- Adicionar 500-1000 ovos ou larvas L13 a cada microcosmo e à placa proxy (etapa 2.7) para iniciar o experimento (Figura 1D).
NOTA: Os experimentos descritos aqui utilizam worms do tipo selvagem N2. No entanto, outras cepas de C. elegans (e potencialmente outros nematoides) podem ser usadas. - Elevar os vermes até à idade adulta a 20 °C (normalmente 3 dias).
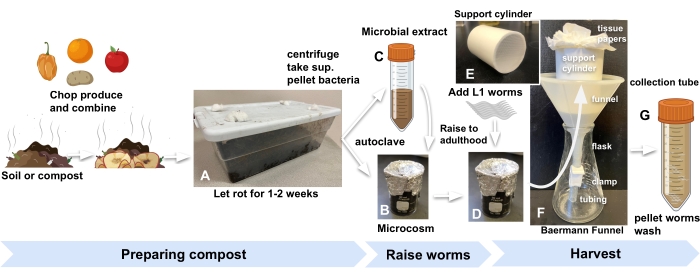
Figura 1: Preparação de microcosmos de compostagem, criação de vermes e colheita . (A) Enriqueça o solo local ou composto com produtos e incube por 2 semanas. Combinar (B) solo enriquecido autoclavado com (C) extrato microbiano e (D) incubar por pelo menos 24 h antes de adicionar vermes L1s sincronizados a microcosmos para iniciar o experimento. (E, F) Quando estiver pronto para colher, adicione o composto do microcosmo em um cilindro de suporte de funil Baermann e cubra com M9. (G) Após 15 min, libertar o filtrado num tubo de 50 ml. Abreviação: sup. = sobrenadante. Por favor, clique aqui para ver uma versão maior desta figura.
4. Preparando um funil Baermann para a colheita de minhocas
- Monte um funil Baermann anexando 5-8 cm de tubo de borracha rígida ao final de um funil de plástico.
- Deslize um grampo sobre a tubulação e feche o grampo.
- Colocar no funil um tubo cilíndrico de PVC cilíndrico de 7 cm de comprimento e 5 cm de diâmetro, com uma malha de nylon de 1 mm colada no fundo (Figura 1E). Forre o cilindro com duas folhas de papel de seda.
- Colocar a ampola de Baermann num balão (figura 1F).
5. Colheita de vermes de microcosmos e coleta das respectivas amostras de solo
- Adicione 20 mL de M9 ao microcosmo em que os vermes foram levantados, agite a mistura e, em seguida, despeje a mistura do copo no cilindro revestido de papel de tecido na configuração do funil Baermann. Adicione mais M9 para submergir completamente o composto no funil.
NOTA: As populações de C. elegans (N2) atingem a idade adulta em microcosmos de composto a uma taxa semelhante à das placas de ágar padrão semeadas com Escherichia coli. Para maior precisão na colheita de vermes em um estágio específico de desenvolvimento, consulte a placa proxy da etapa 3.3. - Após 30 min, desaperte o grampo para liberar o filtrado contendo os vermes colhidos em um tubo de 50 mL (Figura 1G).
- Adicione mais M9 ao cilindro e repita para uma segunda rodada para colher mais vermes e, mais uma vez, se forem necessários vermes adicionais.
NOTA: Ao repetir as rodadas de colheita, tenha cuidado para não comprometer a integridade do papel de seda, o que poderia permitir a passagem de partículas do solo. Um máximo de quatro rodadas de colheita deve isolar pelo menos 50% dos vermes originalmente adicionados ao microcosmo sem comprometer a integridade do papel tissue. - Concentrar os vermes centrifugando a 560 × g durante 2 min (RT). Remover 35 ml do sobrenadante com uma pipeta serológica.
- Transfira os 15 mL restantes para um tubo de 15 mL e centrifugar novamente a 560 × g por 1 min para concentrar ainda mais os vermes. Remover 14 mL do sobrenadante com uma pipeta sorológica.
- Em paralelo, coletar 1 g do solo restante do microcosmo em um tubo de 1,5 mL. Processar as amostras de solo contendo a comunidade bacteriana ambiental imediatamente ou armazená-las a -20 °C para posterior extração de ácidos nucleicos, conforme descrito abaixo para amostras de vermes.
6. Lavagem e esterilização superficial de vermes colhidos
- Transfira 1 mL dos vermes concentrados e colhidos da etapa 5,5 para um tubo de 1,5 mL usando uma pipeta de vidro. Incube por 2 minutos para permitir que os vermes se depositem no fundo do tubo. Remova o sobrenadante, deixando os 100 μL inferiores intactos.
- Lave 6x com 1,5 mL de M9+T (0,025% Triton-X em M9), permitindo que os vermes se depositem no fundo a cada vez.
- Transfira os vermes lavados em um volume de 100 μL para um novo tubo de 1,5 mL usando uma pipeta de vidro.
NOTA: Neste ponto do protocolo, cerca de 30 minutos devem ter passado desde a primeira lavagem (etapa 6.2). Permitir que os vermes permaneçam sem alimentos por pelo menos 1 h antes da adição de levamisol é recomendado para permitir a excreção de bactérias transitórias e a digestão completa de bactérias alimentares. - Adicionar 100 μL de cloridrato de levamisol a 25 mM para paralisar os vermes. Incubar por 5 min no RT.
- Adicionar 200 μL de solução de lixívia a 4%. Incubar por 2 min.
- Remova o sobrenadante, deixando o fundo mais 150 μL sem ser perturbado, e lave 3x com M9 + T, como acima.
NOTA: Para minimizar a contaminação das amostras, recomenda-se o uso de M9+T filtrado (através de um filtro de 0,2 μm) e o uso de luvas a partir deste ponto. - Após as lavagens, tomar 50 μL dos restantes 150 μL da última lavagem e placa sobre uma placa LB, incubando a 25 °C durante 48 h para confirmar a remoção eficaz de bactérias externas.
NOTA: A observação de até 30 colônias na última lavagem (representando o total de 60 células bacterianas externas remanescentes na amostra) é permitida e espera-se que tenha apenas uma contribuição marginal para a composição do microbioma analisado, dadas as milhares de bactérias que tipicamente colonizam cada verme adulto15. Quando mais são observados, a integridade da amostra pode ser comprometida. - Use os vermes esterilizados na superfície imediatamente (por exemplo, para extrair bactérias vivas para cultivo [contagens de UFC]) ou armazene a -20 °C para a extração subsequente de ácidos nucleicos.
7. Extração de DNA
NOTA: As etapas a seguir descrevem a extração de DNA dos vermes colhidos usando um kit comercial projetado para a extração de DNA microbiano do solo (consulte Tabela de Materiais), com modificações descritas abaixo para facilitar a extração de DNA microbiano de vermes.
- Transfira a amostra contendo minhocas esterilizadas superficialmente (descongele em RT, se necessário) para os tubos fornecidos pelo kit, substituindo as contas de vidro fornecidas por contas de zircônia de aproximadamente 30-50 1 mm de diâmetro (ver Tabela de Materiais).
NOTA: Os rendimentos de DNA observados são maiores com contas de zircônia do que com as contas de vidro fornecidas pelo kit. Embora a razão por trás dessa observação permaneça indeterminada, as esferas de zircônia podem abrir a cutícula do nematoide de forma mais eficiente, liberando mais bactérias. - Para amostras de compostagem, adicione cerca de 250 mg do solo coletado aos tubos do kit sem substituir as contas de vidro.
- Após a adição da solução tampão fornecida pelo kit a todas as amostras, homogeneizar com um homogeneizador de potência no RT (ver Tabela de Materiais) por duas rodadas de 2.000 rpm por 30 s cada, pausando por 30 s no meio.
- Complete as etapas restantes de purificação do DNA de acordo com o protocolo do kit. Eluir as amostras de vermes em não mais de 50 μL de tampão de eluição para garantir concentrações de DNA que sejam altas o suficiente para o sequenciamento.
Resultados
Para explorar a capacidade de diversificar a comunidade de microcosmos do solo, comparamos as comunidades microbianas em microcosmos de composto preparados enriquecendo o mesmo solo inicial, um composto de grau industrial disponível na cidade de Berkeley, Califórnia, com diferentes produtos: maçãs, pimentões, laranjas ou batatas (cada um definido em triplicado). Além disso, comparamos as comunidades microbianas de cada ambiente de composto com o microbioma intestinal de C. elegans do tipo selvagem criados no respectivo microcosmo. A análise foi realizada com amostras de DNA extraídas de aproximadamente 500 adultos esterilizados superficialmente por microcosmo e de amostras de composto de 250 mg dos respectivos microcosmos.
A caracterização dos microbiomas ambientais do solo e do intestino de vermes baseou-se no sequenciamento de próxima geração da região V4 do gene 16S rRNA bacteriano. A preparação da biblioteca de sequenciamento foi realizada utilizando os kits padrão e realizada de acordo com as instruções dos fabricantes, com o sequenciamento realizado em um sequenciador comercial (ver Tabela de Materiais). As sequências desmultiplexadas foram processadas usando DADA2, atribuída taxonomia com base no banco de dados de referência SILVA v132, e analisadas com filoseq16,17,18 (ver Arquivo Suplementar 1, Figura Suplementar S1, Figura Suplementar S2, Figura Suplementar S3, Tabela Suplementar S1 e Tabela Suplementar S2 para uma descrição pormenorizada da sequenciação e análise; o pipeline computacional completo está disponível no GitHub [https://github.com/kennytrang/CompostMicrocosms]). Os dados brutos estão disponíveis no NCBI Sequence Read Archive (Bioproject ID PRJNA856419).
Em média, foram obtidas 73.220 sequências por amostra. Essas sequências representam 15.027 variantes de sequência amplicon (ASVs), abrangendo 27 filos e 216 famílias, incluindo famílias consideradas parte do núcleo do microbioma intestinal de C. elegans 13, como Rhizobiaceae, Burkholderiaceae e Bacillaceae. Enterobacteriaceae e Pseudomonadaceae, que anteriormente se encontravam como membros dominantes, eram uma minoria desta vez, mas ainda estavam enriquecidas (2-10 vezes) em comparação com seus respectivos ambientes de solo. Comparações baseadas em distâncias UniFrac19,20 não ponderadas e ponderadas demonstraram boa reprodutibilidade entre triplicados de microcosmos enriquecidos com o mesmo produto, conforme indicado por agrupamento próximo. Em contraste, os microbiomas ambientais do solo enriquecidos com diferentes produtos agrupados entre si, demonstrando a capacidade de diversificar uma comunidade microbiana inicial através da adição de diferentes produtos (Figura 2).
Em comparações de microbiomas intestinais de vermes e comunidades ambientais, a análise de coordenadas principais (PCoA) com distâncias UniFrac não ponderadas ou ponderadas mostrou agrupamento distinto de microbiomas intestinais de vermes longe do de seus respectivos ambientes para cada tipo de microcosmo (Figura 2). Enquanto a PCoA baseada em distâncias UniFrac não ponderadas não distinguiu entre os microbiomas do solo e do verme (Figura 2A), o agrupamento baseado em distâncias ponderadas revelou uma clara separação dos microbiomas do intestino do verme e do composto (Figura 2B). Esses resultados suportam um processo no qual a filtragem do hospedeiro opera na disponibilidade ambiental para moldar um microbioma intestinal que não é completamente distinto de sua fonte ambiental no que diz respeito à presença de táxons, mas modula sua abundância enriquecendo para um subconjunto dos táxons disponíveis, resultando em um microbioma intestinal de verme central compartilhado entre vermes criados em diferentes ambientes.

Figura 2: Microbiomas intestinais de vermes agrupando-se longe de seus respectivos ambientes microbianos diversificados de produção. A composição do microbioma foi determinada com sequenciamento de 16S, e as comunidades de microcosmos enriquecidos com o produto designado ou de vermes criados neles foram agrupadas usando PCoA com base em (A) distâncias UniFrac não ponderadas ou (B) ponderadas. Os eixos mostrados são os que explicam a maior variação na composição da comunidade entre as amostras (N = 3 para cada tipo de microcosmo). Por favor, clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar 1: Sequenciamento de próxima geração e análise de dados. Aqui são apresentadas as etapas para a preparação da biblioteca, o sequenciamento em laboratório e a análise de dados. Clique aqui para baixar este arquivo.
Figura Suplementar S1: Um exemplo de um gráfico de controle de qualidade para as leituras inversas de uma amostra. O eixo X (ciclo) mostra a posição do nucleotídeo ao longo da sequência lida. O eixo Y esquerdo mostra o índice de qualidade. O mapa de calor em escala de cinzas representa a frequência do escore de qualidade em cada posição de nucleotídeos; a linha verde representa a mediana do escore de qualidade em cada posição de nucleotídeos; a linha laranja superior representa os quartis da distribuição do índice de qualidade; a linha vermelha inferior representa a porcentagem de leituras de sequência que estenderam essa posição de nucleotídeos (eixo Y direito, aqui 100%). Clique aqui para baixar este arquivo.
Figura S2 suplementar: Taxas de erro para diferentes amostras. A frequência de erro nas diferentes amostras (pontos pretos) deve diminuir com o aumento do escore de qualidade para cada possível substituição de par de bases representada, refletindo a tendência esperada. Clique aqui para baixar este arquivo.
Figura Suplementar S3: Um exemplo de PCoA baseado em distâncias UniFrac ponderadas. Os nomes dos grupos mostrados na legenda representam o produto usado para enriquecer o composto usado nos diferentes microcosmos. Clique aqui para baixar este arquivo.
Tabela Suplementar S1: Filtragem sequencial de sequências. um Número de leituras de sequência antes da filtragem. b-d Cada coluna representa o número de leituras de sequência restantes após uma etapa de filtração: filtrando leituras de baixa qualidade (etapa 2.5), algoritmo de denoising realizado por dada() (etapa 2.8), mesclando leituras para frente e para trás (etapa 2.9) e removendo quimeras (etapa 2.11). Clique aqui para baixar este arquivo.
Tabela Suplementar S2: Tabela de metadados. Clique aqui para baixar este arquivo.
Discussão
O protocolo aqui apresentado descreve um método para estudar o microbioma intestinal de nematoides criados em ambientes naturais, oferecendo uma abordagem alternativa para o isolamento de vermes da natureza ou para criá-los em comunidades sintéticas.
As milhares de espécies bacterianas potenciais capturadas no experimento representativo do microcosmo refletem a diversidade microbiana com a qual os vermes evoluíram e demonstram a capacidade do pipeline de microcosmos de combinar as vantagens de trabalhar com um organismo hospedeiro modelo e as de trabalhar com comunidades microbianas naturais e diversas.
Os resultados representativos demonstram que o enriquecimento de um solo comum com diferentes tipos de produtos modula a diversidade microbiana ambiental, destacando a gama de diversidade microbiana disponível para exploração utilizando esse gasoduto. A escolha do produto não é particularmente importante. Trabalhos anteriores usaram bananas, maçãs, laranjas, morangos, folhas de chá verde e batatas para enriquecer o solo, resultando em vermes semelhantes aumentando a eficiência. Produtos mistos também têm sido utilizados de forma eficaz. A principal característica é que diferentes produtos diversificarão um determinado solo de maneiras distintas.
Apesar da ampla variação na diversidade microbiana ambiental, os microbiomas intestinais dos vermes variam consideravelmente menos, recapitulando a importância do nicho intestinal do verme e da filtragem do hospedeiro para a montagem de um microbioma intestinal central distinto do ambiente do solo13. Esse padrão é melhor observado com a análise ponderada UniFrac PCoA, demonstrando que as diferenças entre o solo ambiental e os microbiomas intestinais de vermes derivam principalmente de diferenças na abundância relativa de táxons-chave.
Embora o protocolo descrito aqui se concentre na colheita de vermes para sequenciamento 16S, os microcosmos podem ser usados para explorar questões adicionais de interesse. Por exemplo, vermes colhidos de microcosmos podem ser posteriormente examinados quanto ao efeito de um microbioma intestinal diversificado na resistência do hospedeiro a várias condições adversas, incluindo patógenos ou toxinas. Alternativamente, novas espécies e cepas bacterianas podem ser isoladas e cultivadas a partir de vermes colhidos no solo, expandindo a diversidade taxonômica e funcional de bactérias disponíveis para realizar experimentos.
Enquanto os métodos de pesquisa em ambientes de laboratório buscam consistência e reprodutibilidade, o trabalho com microcosmos aproveita a variação natural para explorar as interações hospedeiro-micróbio em um contexto natural. No entanto, essa variação também apresenta alguns desafios. Alguns solos que incluem altos níveis de invertebrados endógenos podem exigir etapas adicionais de centrifugação, filtração e exame para eliminar efetivamente organismos indesejados da preparação do microcosmo. Além disso, a baixa abundância microbiana no solo pode induzir a formação indesejada de dauer em populações de vermes, exigindo um aumento no extrato microbiano ou enriquecendo o solo com uma maior quantidade de produtos. Com cada experimento de microcosmo realizado, os pesquisadores continuam a explorar toda a diversidade taxonômica e funcional fornecida pela natureza, permitindo a descoberta de novos táxons microbianos e habilidades funcionais que vão desde a resistência à infecção até a proteção contra xenobióticos ambientais.
Divulgações
Os autores não têm conflitos de interesse para declarar que são relevantes para o conteúdo deste artigo.
Agradecimentos
O trabalho descrito neste manuscrito foi apoiado pelos subsídios do NIH R01OD024780 e R01AG061302. K.T. foi ainda apoiado por uma bolsa de pesquisa de graduação de verão da Universidade da Califórnia, Berkeley, financiada pela Fundação Rose Hills. Os desenhos animados da Figura 1 foram obtidos a partir de BioRender.com.
Materiais
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
Referências
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285 (2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604 (2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849 (2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622 (2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343 (2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217 (2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados