Method Article
堆肥微观世界作为秀丽隐杆线虫微生物组研究的微生物多样化、类似自然的环境
摘要
堆肥微观世界将自然界中发现的微生物多样性带入实验室,以促进 秀丽隐杆线虫的微生物组研究。这里提供了用于建立微观实验的协议,实验展示了调节环境微生物多样性的能力,以探索环境微生物多样性与蠕虫肠道微生物组组成之间的关系。
摘要
线虫秀 丽隐杆线虫 正在成为研究宿主与其肠道微生物组之间相互作用的分子机制的有用模型。虽然使用表征良好的细菌或确定的细菌群落进行实验可以促进分子机制的分析,但在自然微生物环境中研究线虫对于探索这些机制的多样性至关重要。同时,从野外分离蠕虫并不总是可行的,即使可能,从野外取样也限制了遗传工具包的使用,否则可用于 秀丽隐杆线虫 研究。以下协议描述了一种利用堆肥微观世界在微生物多样化和类似自然的环境中进行实验室内生长的微生物组研究方法。
当地采购的土壤可以用农产品来丰富,以使饲养蠕虫的微生物群落多样化,并从中收获、清洗和表面消毒以进行后续分析。代表性实验证明了通过用不同的农产品丰富来调节普通土壤中的微生物群落的能力,并进一步证明在这些不同环境中饲养的蠕虫组装了与各自环境不同的相似肠道微生物组,支持物种特异性核心肠道微生物组的概念。总体而言,堆肥微观世界为微生物组研究提供了类似自然的实验室环境,作为合成微生物群落或野生线虫分离的替代方案。
引言
线虫秀丽隐杆线虫正在成为研究宿主与其肠道微生物组之间相互作用的有用模型1,2。作为一种模型,它提供了几个优点。首先,无菌或侏儒动物易于获得和饲养;漂白剂可用于杀死妊娠蠕虫和相关微生物,使其抗漂白剂卵不受伤害地生长为年龄同步的种群,可以被感兴趣的细菌定植3,4。此外,当在细菌存在下生长时,秀丽隐杆线虫(一种细菌食者)摄取遇到的细菌,易感物种被消化或排泄,而耐药性和持久性物种则稳定地定植在蠕虫肠道中。此外,秀丽隐杆线虫大多是雌雄同体的,产生基因相同的后代种群,这减少了混杂的遗传变异。再加上突变和转基因蠕虫菌株的可用性,与秀丽隐杆线虫合作为研究人员提供了一个GNTOBIO和遗传可处理的模型,以研究宿主 - 微生物相互作用的分子基础5,6,7,8。
虽然对表征良好的细菌的实验可以促进分子机制的分析,但识别和研究蠕虫在自然界中与之相互作用的细菌对于探索这些机制的多样性、揭示其功能的自然背景以及了解塑造其进化的选择性力量至关重要。在实验室之外, 秀丽隐杆线虫 在全球范围内被发现在潮湿的温带气候中,那里的种群被认为经历了"繁荣与萧条"的生命周期,其特征是资源丰富时人口快速增长,然后在资源枯竭时向开拓性、抗压的 dauers 发展转变9.虽然被认为是土壤线虫,但野生增殖的 秀丽隐杆线虫 种群最常见于腐烂的有机物质,如腐烂的花朵或水果,其中细菌种群丰富多样。
对从野外分离的线虫中的肠道微生物组的研究已经确定了多种但具有特征的细菌群落10,11,其组成得到了在自然微观环境中饲养的蠕虫进行的研究的进一步支持12,13。总之,这些研究能够描绘出核心蠕虫肠道微生物组2。虽然对野生秀丽隐杆线虫种群的采样代表了对天然蠕虫-微生物相互作用的最直接检查,但它并非随时随地可行,因为它仅限于降水充足的地区和季节10,11。或者,不是将蠕虫与它们的自然栖息地隔离开来,而是使用微观世界的实验将自然栖息地带入实验室6,8,12,13,14,15。微观环境由含有各种水果或蔬菜的土壤堆肥制备而成,这使得起始土壤群落进一步多样化。它们提供了易于处理的实验方法,将微生物多样性和三维野生土壤环境与受控实验室设施和遗传定义的蠕虫菌株的实验优势相结合。下面的协议详细介绍了使用堆肥微观世界所涉及的步骤,展示了它们在理解来自不同环境的特征蠕虫肠道微生物组组装中的用途。
研究方案
1. 堆肥的制备
- 从任何方便的来源获取堆肥或花园土壤,并将实验室内存放在标准厨房塑料容器中,盖子上切孔以让空气进入。用棉絮堵住孔,以防止果蝇和其他无脊椎动物进入(图1A)。
注意:500克土壤(装在1.5加仑的容器中,尺寸:30厘米x 20厘米x 10厘米)将为12个微观世界提供足够的材料。 - 用切碎的农产品或不同农产品的混合物以 1:2 的质量比丰富堆肥或土壤。
- 在20-25°C下孵育7-14天,每天混合一次,并根据需要加入M9培养基以保持水分而不会使其变得浑浊。
注意:没有富含农产品的土壤通常不支持 秀丽隐杆线 虫的生长,但使用哪种特定农产品取决于研究人员,有许多类型和混合物能够支持蠕虫的生长。不同产品的富集将以不同的方式促进细菌群落多样化,从而能够从不同的起点研究肠道微生物组组装(见讨论部分)。
2. 堆肥微观世界的制备
- 对于每个微观世界,将 10 g 富集堆肥添加到覆盖有锡箔和高压釜的 30 mL 玻璃烧杯中(图 1B)。
- 为了制备微生物提取物以补充高压灭菌的堆肥,首先将30 g在步骤2.1中使用的相同堆肥添加到三个50 mL管中的每一个中,并填充M9。涡旋1分钟(图1C)。
注意:这应该为九个微观世界提供足够的细菌,每个微观世界支持数百种线虫的发育。 - 在室温(RT)下以560× g 离心管5分钟。
- 注意不要干扰沉淀,用血清移液管去除上清液,并合并到新的50 mL管中。
- 通过在室温下以最大速度(2,000 × g)离心 15 分钟来浓缩细菌提取物。 将沉淀重悬于足够的 M9 中,每个微观世界有 200 μL,再增加 200 μL 添加到板中,该板将作为微观世界中蠕虫发展的可见代理。
注意:例如,对于九个微观世界,将微生物沉淀重悬于 2 mL 的 M9 中。 - 将 200 μL 浓缩微生物提取物添加到高压灭菌堆肥的每个烧杯中,以及将用作可见代理板的 NGM 板中。
- 在加入蠕虫之前,在20-25°C下孵育微观和代理板24小时。
3. 堆肥微观世界中的蠕虫
- 向每个显微镜和代理板(步骤2.7)中加入500-1000个卵或L1幼虫3 以开始实验(图1D)。
注意:此处描述的实验利用N2野生型蠕虫。然而,可以使用其他 秀丽隐杆 线虫菌株(以及潜在的其他线虫)。 - 在20°C(通常为3天)下将蠕虫饲养至成年。
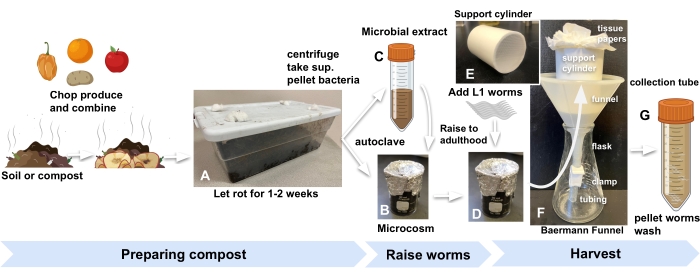
图1:准备堆肥微观世界,饲养蠕虫和收获 。 (A)用农产品丰富当地土壤或堆肥,并孵育2周。将(B)高压灭菌的富集土壤与(C)微生物提取物和(D)孵育至少24小时,然后将同步的L1s蠕虫添加到微观世界中以开始实验。(东、 女)准备收获时,将显微镜中的堆肥添加到贝尔曼漏斗支撑筒中,并用 M9 覆盖。(G)15分钟后,将滤液释放到50mL管中。缩写:sup. = 上清液。 请点击此处查看此图的大图。
4. 准备用于收割蠕虫的贝尔曼漏斗
- 通过将 5-8 厘米的刚性橡胶管连接到塑料漏斗的末端来组装贝尔曼漏斗。
- 将夹子滑到管子上并将其夹紧。
- 将7厘米长,直径5厘米的圆柱形PVC管放在漏斗中,底部粘有1毫米尼龙网(图1E)。在圆筒上铺上两张薄纸。
- 将贝尔曼漏斗放入烧瓶中(图1F)。
5. 从微观世界中收获蠕虫并收集相应的土壤样本
- 将 20 mL M9 加入到提升蠕虫的微观世界中,搅拌混合物,然后将烧杯中的混合物倒入贝尔曼漏斗设置中的薄纸衬里圆筒中。添加更多 M9 以将堆肥完全淹没在漏斗中。
注意: 秀丽隐杆线虫 (N2)种群在堆肥微观世界中以与接种大 肠杆菌的标准琼脂平板相似的速度达到成年期。有关在特定发育阶段收获蠕虫的进一步准确性,请参阅步骤 3.3 中的代理板。 - 30分钟后,松开夹子,将含有收获的蠕虫的滤液释放到50mL管中(图1G)。
- 向圆柱体中添加更多M9并重复第二轮以收获更多蠕虫,如果需要额外的蠕虫,则再次添加一次。
注意:重复收获轮次时,请注意不要损害薄纸的完整性,这可能会让土壤颗粒通过。最多四轮收获应分离至少50%的最初添加到微观世界中的蠕虫,而不会影响薄纸的完整性。 - 通过以560× g 离心2分钟(RT)来浓缩蠕虫。用血清移液管除去 35 mL 上清液。
- 将剩余的 15 mL 转移到 15 mL 管中,再次以 560 × g 离心 1 分钟以进一步浓缩蠕虫。用血清移液管除去14mL上清液。
- 同时,将 1 g 剩余的微观土壤收集到 1.5 mL 管中。立即处理含有环境细菌群落的土壤样品或将其储存在-20°C,以便以后提取核酸,如下所述蠕虫样品。
6. 收获的蠕虫的清洗和表面灭菌
- 使用玻璃移液管将步骤5.5中的1 mL浓缩收获的蠕虫转移到1.5 mL管中。孵育2分钟,让蠕虫沉淀在管底部。除去上清液,使底部 100 μL 不受干扰。
- 用 1.5 mL M9+T(M9 中的 0.025% Triton-X )洗涤 6 次,让蠕虫每次都沉降在底部。
- 使用玻璃移液管将体积为 100 μL 的洗涤蠕虫转移到新的 1.5 mL 管中。
注意:在协议中的这一点上,自第一次洗涤(步骤6.2)以来应该已经过去了大约30分钟。建议在添加左旋咪唑之前允许蠕虫在没有食物的情况下保持至少1小时,以允许短暂细菌的排泄和食物细菌的完全消化。 - 加入 100 μL 25 mM 左旋咪唑盐酸盐以麻痹蠕虫。在室温下孵育5分钟。
- 加入 200 μL 4% 漂白剂溶液。孵育2分钟。
- 除去上清液,保持最底部的 150 μL 不受干扰,并如上所述用 M9+T 洗涤 3 次。
注意:为了尽量减少样品的污染,建议使用过滤的M9 + T(通过0.2μm过滤器)并从此时戴上手套。 - 洗涤后,从最后一次洗涤中取出剩余的150μL中的50μL,并在LB板上进行板,在25°C下孵育48小时以确认有效去除外部细菌。
注意:允许在最后一次洗涤中观察多达30个菌落(代表样品中剩余的60个外部细菌细胞),并且预计对分析的微生物组组成只有很小的贡献,因为通常定植每个成虫的数千个细菌15。当观察到更多时,样品完整性可能会受到影响。 - 立即使用表面灭菌的蠕虫(例如,提取活细菌进行培养[CFU计数])或储存在-20°C以随后提取核酸。
7. 脱氧核糖核酸提取
注意:以下步骤描述了使用设计用于从土壤中提取微生物DNA的商业试剂盒(见 材料表)提取收获的蠕虫的DNA,并进行如下所述的修改以促进从蠕虫中提取微生物DNA。
- 将含有表面灭菌蠕虫的样品(如有必要,在室温下解冻)转移到试剂盒提供的管中,用大约 30-50 个直径为 1 mm 的氧化锆珠替换提供的玻璃珠(参见 材料表)。
注意:氧化锆珠观察到的DNA产量高于试剂盒提供的玻璃珠。虽然这一观察背后的原因仍未确定,但氧化锆珠可能会更有效地打开线虫角质层,释放更多的细菌。 - 对于堆肥样品,在不更换玻璃珠的情况下,将大约 250 毫克收集的土壤添加到试剂盒的管中。
- 将试剂盒提供的缓冲溶液添加到所有样品中后,在室温下用功率均质器(参见 材料表)均质两轮,每轮 2,000 rpm,每次 30 秒,中间暂停 30 秒。
- 根据试剂盒方案完成剩余的DNA纯化步骤。在不超过 50 μL 的洗脱缓冲液中洗脱蠕虫样品,以确保 DNA 浓度足够高以进行测序。
结果
为了探索使土壤微观世界群落多样化的能力,我们比较了堆肥微观世界中的微生物群落,这些微生物群落是通过富集相同的初始土壤(加利福尼亚州伯克利市提供的工业级堆肥)制备的,具有不同的产品:苹果、甜椒、橙子或土豆(每套一式三份)。我们进一步比较了每个堆肥环境的微生物群落与各自微观世界中饲养的野生型 秀丽隐杆线虫 的肠道微生物组。使用从每个微观世界大约500名表面灭菌的成年人中提取的DNA样品和从各自微观世界的250mg堆肥样品中提取的分析。
环境土壤和蠕虫肠道微生物组的表征依赖于细菌16S rRNA基因V4区域的下一代测序。测序文库制备使用标准试剂盒实现,并根据制造商的说明进行,并在商业测序仪上进行测序(参见材料表)。使用DADA2处理解复用序列,根据SILVA v132参考数据库分配分类法,并使用phyloseq16,17,18进行分析(参见补充文件1,补充图S1,补充图S2,补充图S3,补充表S1和补充表S2 用于测序和分析的详细说明;完整的计算管道可在 GitHub [https://github.com/kennytrang/CompostMicrocosms]中找到)。原始数据可在NCBI序列读取档案(生物项目ID PRJNA856419)中找到。
平均每个样品获得73,220个序列。这些序列代表 15,027 个扩增子序列变体 (ASV),跨越 27 个门和 216 个科,包括被认为是秀丽隐杆线虫肠道微生物组核心 13 一部分的家族,例如根茎科、伯克霍尔德科和杆菌科。以前发现的肠杆菌科和假单胞菌科是优势成员,这次是少数,但与各自的土壤环境相比仍然富集(2-10倍)。基于未加权和加权UniFrac19,20距离的比较表明,富含相同产品的微观一式三份之间具有良好的重现性,如紧密聚类所示。相比之下,富含不同农产品的环境土壤微生物组彼此聚集在一起,证明了通过添加不同农产品使初始微生物群落多样化的能力(图2)。
在蠕虫肠道微生物组和环境群落的比较中,具有未加权或加权UniFrac距离的主坐标分析(PCoA)显示,对于每种微观世界类型,蠕虫肠道微生物组与各自环境的聚类明显不同(图2)。虽然基于未加权UniFrac距离的PCoA没有区分土壤和蠕虫微生物组(图2A),但基于加权距离的聚类揭示了蠕虫肠道和堆肥微生物组的明显分离(图2B)。这些结果支持宿主过滤对环境可用性起作用的过程,以形成肠道微生物组,该微生物组在分类群的存在方面与其环境来源并不完全不同,但通过富集可用分类群的子集来调节它们的丰度,最终导致在不同环境中饲养的蠕虫之间共享的核心蠕虫肠道微生物组。

图2:蠕虫肠道微生物组聚集在各自产生多样化的微生物环境中。 通过16S测序确定微生物组组成,并使用基于(A)未加权或(B)加权UniFrac距离的PCoA对富含指定产品的微观世界或在其中饲养的蠕虫的群落进行聚类。所示轴是解释样本之间群落组成最大差异的轴(每种微观世界类型N = 3)。 请点击此处查看此图的大图。
补充文件1:二代测序和数据分析。 这里介绍文库制备、实验室测序和数据分析的步骤。 请点击此处下载此文件。
补充图S1:一个样品反向读数的质量控制图示例。 X轴(循环)显示沿序列读取的核苷酸位置。左侧 Y 轴显示质量得分。灰度热图表示每个核苷酸位置的质量得分频率;绿线表示每个核苷酸位置的中位数质量得分;顶部的橙色线表示质量得分分布的四分位数;底部红线描述了扩展核苷酸位置的序列读数的百分比(右Y轴,此处为100%)。 请点击此处下载此文件。
补充图S2:不同样本的错误率。 对于所描述的每个可能的碱基对替换,不同样本(黑点)中的错误频率应随着质量得分的增加而降低,从而反映预期趋势。 请点击此处下载此文件。
补充图S3:基于加权UniFrac距离的PCoA示例。 图例中显示的组名代表用于丰富不同微观世界中使用的堆肥的农产品。 请点击此处下载此文件。
补充表S1:顺序序列过滤。 一个过滤前的序列读取数。b-d 每列表示过滤步骤后剩余的序列读取数:过滤掉低质量读取(步骤 2.5)、由 dada() 执行的去噪算法(步骤 2.8)、合并正向和反向读取(步骤 2.9)和删除嵌合体(步骤 2.11)。请点击此处下载此文件。
补充表 S2:元数据表。请点击此处下载此文件。
讨论
这里介绍的协议描述了一种研究在自然环境中饲养的线虫肠道微生物组的方法,提供了一种将蠕虫与自然界隔离或在合成群落上饲养蠕虫的替代方法。
在代表性的微观实验中捕获的数千种潜在细菌物种反映了蠕虫进化的微生物多样性,并证明了微观管道将使用模型宿主生物的优势与与自然,多样化的微生物群落合作的优势相结合的能力。
代表性结果表明,用不同的农产品类型丰富公共土壤可以调节环境微生物多样性,突出了使用该管道可用于勘探的微生物多样性范围。产品选择并不是特别重要。以前的工作使用香蕉,苹果,橙子,草莓,绿茶叶和土豆来丰富土壤,从而产生类似的蠕虫饲养效率。混合产品也得到了有效利用。关键特征是不同的产品将以不同的方式使给定的土壤多样化。
尽管环境微生物多样性差异很大,但蠕虫肠道微生物组的变化要小得多,这概括了蠕虫肠道生态位和宿主过滤对于组装与其土壤环境不同的核心肠道微生物组的重要性13。这种模式在加权UniFrac PCoA分析中观察到最好,表明环境土壤和蠕虫肠道微生物组之间的差异主要源于关键分类群相对丰度的差异。
尽管此处描述的协议侧重于收集蠕虫以进行16S测序,但微观世界可用于探索其他感兴趣的问题。例如,随后可以检查从微观世界中收获的蠕虫,以确定多种肠道微生物组对宿主对各种不利条件(包括病原体或毒素)的抵抗力的影响。或者,可以从地面收获的蠕虫中分离和培养新的细菌种类和菌株,从而扩大可用于进行实验的细菌的分类和功能多样性。
虽然实验室环境中的研究方法寻求一致性和可重复性,但微观世界的工作利用自然变异在类似自然的背景下探索宿主 - 微生物相互作用。然而,这种变化也带来了一些挑战。一些含有高水平内源性无脊椎动物的土壤可能需要额外的离心、过滤和检查步骤,以有效地从微观制备中消除不需要的生物。此外,土壤中微生物丰度低可能会在蠕虫种群中诱导不希望的dauer形成,需要增加微生物提取物或用更多的农产品丰富土壤。随着每个微观实验的进行,研究人员继续探索自然界提供的全部分类学和功能多样性,从而能够发现新的微生物分类群和功能能力,从抗感染到对环境异生的保护。
披露声明
作者没有与本文内容相关的利益冲突声明。
致谢
本手稿中描述的工作得到了NIH拨款R01OD024780和R01AG061302的支持。K.T.得到了加州大学伯克利分校的暑期本科生研究奖学金的进一步支持,该奖学金由玫瑰山基金会资助。 图1 中的卡通设计是从 BioRender.com 获得的。
材料
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
参考文献
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。