Method Article
Microcosmi di compost come ambienti microbicamente diversi e simili a quelli naturali per la ricerca sul microbioma in Caenorhabditis elegans
In questo articolo
Riepilogo
I microcosmi di compost portano la diversità microbica presente in natura in laboratorio per facilitare la ricerca sul microbioma in Caenorhabditis elegans. Qui sono forniti protocolli per la creazione di esperimenti di microcosmo, con gli esperimenti che dimostrano la capacità di modulare la diversità microbica ambientale per esplorare le relazioni tra diversità microbica ambientale e composizione del microbioma intestinale del verme.
Abstract
Il nematode Caenorhabditis elegans sta emergendo come un modello utile per studiare i meccanismi molecolari alla base delle interazioni tra gli ospiti e i loro microbiomi intestinali. Mentre gli esperimenti con batteri ben caratterizzati o comunità batteriche definite possono facilitare l'analisi dei meccanismi molecolari, lo studio dei nematodi nel loro contesto microbico naturale è essenziale per esplorare la diversità di tali meccanismi. Allo stesso tempo, l'isolamento dei vermi dalla natura non è sempre fattibile e, anche quando possibile, il campionamento dalla natura limita l'uso del kit di strumenti genetici altrimenti disponibile per la ricerca su C. elegans. Il seguente protocollo descrive un metodo per gli studi sul microbioma che utilizza microcosmi di compost per la crescita in laboratorio in ambienti microbicamente diversi e naturali.
Il suolo di provenienza locale può essere arricchito con prodotti per diversificare le comunità microbiche in cui i vermi vengono allevati e da cui vengono raccolti, lavati e sterilizzati in superficie per le successive analisi. Esperimenti rappresentativi dimostrano la capacità di modulare la comunità microbica in un suolo comune arricchendolo con prodotti diversi e dimostrano ulteriormente che i vermi allevati in questi ambienti distinti assemblano microbiomi intestinali simili distinti dai rispettivi ambienti, supportando la nozione di un microbioma intestinale centrale specifico per specie. Nel complesso, i microcosmi di compost forniscono ambienti naturali in laboratorio per la ricerca sul microbioma come alternativa alle comunità microbiche sintetiche o all'isolamento dei nematodi selvatici.
Introduzione
Il nematode Caenorhabditis elegans sta emergendo come un modello utile per studiare le interazioni tra gli ospiti e i loro microbiomi intestinali 1,2. Come modello, offre diversi vantaggi. In primo luogo, gli animali privi di germi o gnotobiotici sono facili da ottenere e mantenere; La candeggina può essere usata per uccidere i vermi gravidi e i microbi associati, lasciando illese le loro uova resistenti alla candeggina per crescere come popolazioni sincronizzate per età che possono essere colonizzate da batteri di interesse 3,4. Inoltre, quando coltivato in presenza di batteri, C. elegans, un batterivoro, ingerisce i batteri incontrati, con specie sensibili digerite o escrete, mentre le specie resistenti e persistenti colonizzano stabilmente l'intestino del verme. Inoltre, i C. elegans sono per lo più ermafroditi, producendo popolazioni di progenie geneticamente identica, che riduce la variazione genetica confondente. Insieme alla disponibilità di ceppi di vermi mutanti e transgenici, lavorare con C. elegans offre ai ricercatori un modello gnotobiotico e geneticamente trattabile per studiare le basi molecolari delle interazioni ospite-microbo5,6,7,8.
Mentre gli esperimenti con batteri ben caratterizzati possono facilitare l'analisi dei meccanismi molecolari, identificare e studiare i batteri con cui i vermi interagiscono in natura sono essenziali per esplorare la diversità di tali meccanismi, svelare il contesto naturale per la loro funzione e comprendere le forze selettive che hanno modellato la loro evoluzione. Al di fuori del laboratorio, C. elegans si trova a livello globale in climi temperati umidi, dove si pensa che le popolazioni subiscano un ciclo di vita "boom-and-bust", caratterizzato da una rapida crescita della popolazione quando le risorse sono abbondanti, seguita da uno spostamento evolutivo verso pionieristici e tolleranti allo stress quando le risorse sono esaurite9. Sebbene sia considerato un nematode del suolo, le popolazioni selvatiche proliferanti di C. elegans si trovano più comunemente a nutrirsi di materiale organico in decomposizione come fiori o frutti in decomposizione, dove le popolazioni batteriche sono abbondanti e diversificate.
Gli studi sul microbioma intestinale nei nematodi isolati in natura hanno identificato comunità batteriche diverse, ma caratteristiche, 10,11, la cui composizione è stata ulteriormente supportata da studi condotti con vermi allevati in ambienti microcosmo simili a quelli naturali 12,13. Insieme, tali studi hanno permesso la delineazione di un microbioma intestinale del verme principale2. Mentre il campionamento delle popolazioni di C. elegans in natura rappresenta l'esame più diretto delle interazioni naturali verme-microbo, non è fattibile ovunque e in qualsiasi momento, poiché è limitato a regioni e stagioni con abbondanti precipitazioni10,11. In alternativa, invece di isolare i vermi dal loro habitat naturale, gli esperimenti che utilizzano microcosmi portano l'habitat naturale in laboratorio 6,8,12,13,14,15. Gli ambienti del microcosmo sono preparati da terreno compostato con vari tipi di frutta o verdura, il che consente un'ulteriore diversificazione della comunità del suolo di partenza. Offrono metodi sperimentali trattabili che combinano la diversità microbica e l'ambiente tridimensionale del suolo selvaggio con i vantaggi sperimentali di una struttura di laboratorio controllata e ceppi di vermi geneticamente definiti. Il protocollo seguente descrive in dettaglio i passaggi coinvolti nel lavoro con i microcosmi del compost, dimostrando il loro uso nella comprensione dell'assemblaggio di un caratteristico microbioma intestinale di vermi da ambienti diversi.
Protocollo
1. Preparazione del compost
- Ottenere compost o terriccio da giardino da qualsiasi fonte conveniente e conservare all'interno del laboratorio in un contenitore di plastica da cucina standard con fori tagliati nel coperchio per far entrare l'aria. Tappare i fori con cotone idrofilo per tenere lontani i moscerini della frutta e altri invertebrati (Figura 1A).
NOTA: Cinquecento grammi di terreno (adatto in un contenitore da 1,5 galloni, dimensioni: 30 cm x 20 cm x 10 cm) forniranno materiale sufficiente per 12 microcosmi. - Arricchire il compost o il terreno con prodotti tritati o una miscela di prodotti diversi in un rapporto di massa 1: 2 tra prodotti e terreno.
- Incubare per 7-14 giorni a 20-25 °C, mescolando una volta al giorno e aggiungendo M9 mezzo se necessario per mantenere l'umidità senza renderla fangosa.
NOTA: I terreni non arricchiti con prodotti di solito non supportano la crescita di C. elegans , ma quale prodotto specifico utilizzare spetta al ricercatore, con molti tipi e miscele in grado di supportare la crescita del verme. L'arricchimento con prodotti diversi promuoverà la diversificazione della comunità batterica in modi diversi, consentendo lo studio dell'assemblaggio del microbioma intestinale da diversi punti di partenza (vedere la sezione di discussione).
2. Preparazione di microcosmi di compost
- Per ciascun microcosmo, aggiungere 10 g di compost arricchito in un becher di vetro da 30 ml ricoperto di carta stagnola e autoclave (figura 1B).
- Per preparare l'estratto microbico per reintegrare il compost autoclavato, iniziare aggiungendo 30 g dello stesso compost utilizzato nella fase 2.1 a ciascuno dei tre tubi da 50 ml e riempire con M9. Vortice per 1 minuto (Figura 1C).
NOTA: Questo dovrebbe fornire abbastanza batteri per nove microcosmi, ognuno dei quali supporta lo sviluppo di centinaia di nematodi. - Centrifugare i tubi a 560 × g per 5 minuti a temperatura ambiente (RT).
- Facendo attenzione a non disturbare il pellet, rimuovere i surnatanti con una pipetta sierologica e combinare in un nuovo tubo da 50 ml.
- Concentrare l'estratto batterico centrifugando alla massima velocità (2.000 × g) per 15 minuti a RT. Risospendere i pellet in sufficiente M9 per avere 200 μL per ogni microcosmo e 200 μL in più da aggiungere a una piastra che servirà come proxy visibile dello sviluppo dei vermi all'interno dei microcosmi.
NOTA: Ad esempio, per nove microcosmi, risospendere il pellet microbico in 2 ml di M9. - Aggiungere 200 μL dell'estratto microbico concentrato a ciascun becher di compost autoclavato, nonché a una piastra NGM che fungerà da piastra proxy visibile.
- Incubare microcosmi e piastra proxy per 24 h a 20-25 °C prima dell'aggiunta di vermi.
3. Allevare vermi nei microcosmi del compost
- Aggiungere 500-1000 uova o larve L13 a ciascun microcosmo e alla piastra proxy (passo 2.7) per iniziare l'esperimento (Figura 1D).
NOTA: Gli esperimenti qui descritti utilizzano vermi wild-type N2. Tuttavia, altri ceppi di C. elegans (e potenzialmente altri nematodi) possono essere utilizzati. - Portare i vermi all'età adulta a 20 °C (in genere 3 giorni).
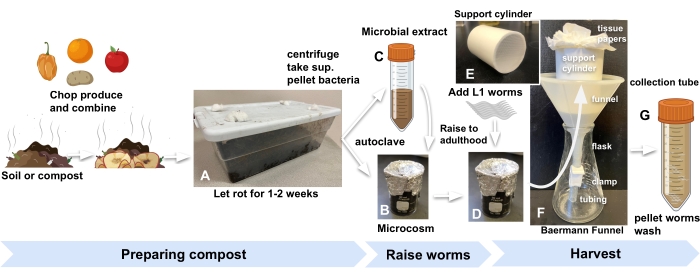
Figura 1: Preparazione dei microcosmi del compost, allevamento di vermi e raccolta . (A) Arricchire il suolo locale o il compost con prodotti e incubare per 2 settimane. Combinare (B) terreno arricchito in autoclave con (C) estratto microbico e (D) incubare per almeno 24 ore prima di aggiungere vermi L1s sincronizzati ai microcosmi per iniziare l'esperimento. (E, F) Quando è pronto per la raccolta, aggiungere il compost dal microcosmo in un cilindro di supporto a imbuto Baermann e coprire con M9. (G) Dopo 15 minuti, rilasciare il filtrato in una provetta da 50 ml. Abbreviazione: sup. = supernatante. Fare clic qui per visualizzare una versione ingrandita di questa figura.
4. Preparazione di un imbuto Baermann per la raccolta dei vermi
- Assemblare un imbuto Baermann attaccando 5-8 cm di tubo di gomma rigida all'estremità di un imbuto di plastica.
- Far scorrere un morsetto sul tubo e bloccarlo.
- Posizionare nell'imbuto un tubo cilindrico in PVC lungo 7 cm e diametro 5 cm con una rete di nylon da 1 mm incollata sul fondo (Figura 1E). Foderare il cilindro con due fogli di carta velina.
- Inserire l'imbuto Baermann in un matraccio (Figura 1F).
5. Raccolta di vermi da microcosmi e raccolta dei rispettivi campioni di terreno
- Aggiungere 20 ml di M9 al microcosmo in cui sono stati sollevati i vermi, agitare la miscela e quindi versare la miscela dal becher nel cilindro rivestito di carta velina nella configurazione dell'imbuto Baermann. Aggiungi più M9 per immergere completamente il compost nell'imbuto.
NOTA: Le popolazioni di C. elegans (N2) raggiungono l'età adulta in microcosmi di compost ad un ritmo simile a quello delle piastre di agar standard seminate con Escherichia coli. Per ulteriori accuratezze nella raccolta dei vermi in una specifica fase di sviluppo, fare riferimento alla piastra proxy del passaggio 3.3. - Dopo 30 minuti, sganciare il morsetto per rilasciare il filtrato contenente i vermi raccolti in una provetta da 50 ml (Figura 1G).
- Aggiungi più M9 al cilindro e ripeti per un secondo round per raccogliere più vermi, e ancora una volta se sono necessari altri vermi.
NOTA: quando si ripetono i cicli di raccolta, fare attenzione a non compromettere l'integrità della carta velina, che potrebbe consentire il passaggio delle particelle di terreno. Un massimo di quattro cicli di raccolta dovrebbero isolare almeno il 50% dei vermi originariamente aggiunti al microcosmo senza compromettere l'integrità della carta velina. - Concentrare i vermi centrifugando a 560 × g per 2 minuti (RT). Rimuovere 35 mL di surnatante con una pipetta sierologica.
- Trasferire i restanti 15 ml in una provetta da 15 ml e centrifugare nuovamente a 560 × g per 1 minuto per concentrare ulteriormente i vermi. Rimuovere 14 ml del surnatante con una pipetta sierologica.
- In parallelo, raccogliere 1 g del terreno del microcosmo rimanente in un tubo da 1,5 ml. Trattare immediatamente i campioni di terreno contenenti la comunità batterica ambientale o conservarli a -20 °C per la successiva estrazione degli acidi nucleici, come descritto di seguito per i campioni di vermi.
6. Lavaggio e sterilizzazione superficiale dei vermi raccolti
- Trasferire 1 mL dei vermi concentrati e raccolti dal punto 5.5 a un tubo da 1,5 mL usando una pipetta di vetro. Incubare per 2 minuti per consentire ai vermi di depositarsi sul fondo del tubo. Rimuovere il surnatante, lasciando indisturbati i 100 μL inferiori.
- Lavare 6 volte con 1,5 ml di M9+T (0,025% Triton-X in M9), lasciando che i vermi si depositino sul fondo ogni volta.
- Trasferire i vermi lavati in un volume di 100 μL in una nuova provetta da 1,5 mL utilizzando una pipetta di vetro.
NOTA: A questo punto del protocollo, dovrebbero essere trascorsi circa 30 minuti dal primo lavaggio (passaggio 6.2). Si raccomanda di lasciare che i vermi rimangano senza cibo per almeno 1 ora prima dell'aggiunta di levamisolo per consentire l'escrezione di batteri transitori e la completa digestione dei batteri alimentari. - Aggiungere 100 μL di 25 mM di levamisolo cloridrato per paralizzare i vermi. Incubare per 5 minuti a RT.
- Aggiungere 200 μL di soluzione di candeggina al 4%. Incubare per 2 min.
- Rimuovere il surnatante, lasciando indisturbato il fondo da 150 μL, e lavare 3x con M9+T, come sopra.
NOTA: Per ridurre al minimo la contaminazione dei campioni, si consiglia di utilizzare M9+T filtrato (attraverso un filtro da 0,2 μm) e di indossare guanti da questo punto in poi. - Dopo i lavaggi, prelevare 50 μL dei restanti 150 μL dall'ultimo lavaggio e placcare su una piastra LB, incubando a 25 °C per 48 ore per confermare l'effettiva rimozione dei batteri esterni.
NOTA: L'osservazione di un massimo di 30 colonie nell'ultimo lavaggio (che rappresentano 60 cellule batteriche esterne totali rimanenti nel campione) è consentita e ci si aspetta che abbia solo un contributo marginale alla composizione del microbioma analizzata, date le migliaia di batteri che tipicamente colonizzano ogni verme adulto15. Quando se ne osservano di più, l'integrità del campione potrebbe essere compromessa. - Utilizzare immediatamente i vermi sterilizzati in superficie (ad esempio, per estrarre batteri vivi per la coltura [conteggi CFU]) o conservare a -20 °C per la successiva estrazione di acidi nucleici.
7. Estrazione del DNA
NOTA: I seguenti passaggi descrivono l'estrazione del DNA dei vermi raccolti utilizzando un kit commerciale progettato per l'estrazione di DNA microbico dal suolo (vedi Tabella dei materiali), con le modifiche descritte di seguito per facilitare l'estrazione del DNA microbico dai vermi.
- Trasferire il campione contenente vermi sterilizzati in superficie (sbrinare a RT se necessario) nelle provette fornite dal kit, sostituendo le perle di vetro fornite con circa 30-50 perle di zirconia del diametro di 1 mm (vedi Tabella dei materiali).
NOTA: Le rese di DNA osservate sono più elevate con le perle di zirconia rispetto alle perle di vetro fornite dal kit. Sebbene la ragione alla base di questa osservazione rimanga indeterminata, le perle di zirconia possono aprire la cuticola del nematode in modo più efficiente, rilasciando più batteri. - Per i campioni di compost, aggiungere circa 250 mg del terreno raccolto ai tubi del kit senza sostituire le perle di vetro.
- Dopo l'aggiunta della soluzione tampone fornita dal kit a tutti i campioni, omogeneizzare con un omogeneizzatore di potenza a RT (vedi Tabella dei materiali) per due giri di 2.000 giri/min per 30 s ciascuno, fermandosi per 30 s in mezzo.
- Completare le restanti fasi di purificazione del DNA secondo il protocollo del kit. Eluire i campioni di vermi in non più di 50 μL di tampone di eluizione per garantire concentrazioni di DNA sufficientemente elevate per il sequenziamento.
Risultati
Per esplorare la capacità di diversificare la comunità dei microcosmi del suolo, abbiamo confrontato le comunità microbiche nei microcosmi di compost preparati arricchendo lo stesso terreno iniziale, un compost di livello industriale disponibile dalla città di Berkeley, in California, con prodotti diversi: mele, peperoni, arance o patate (ciascuno impostato in triplice copia). Abbiamo inoltre confrontato le comunità microbiche di ciascun ambiente di compostaggio con il microbioma intestinale di C. elegans selvatici allevati nel rispettivo microcosmo. L'analisi è stata eseguita con campioni di DNA estratti da circa 500 adulti sterilizzati in superficie per microcosmo e da campioni di compost da 250 mg dei rispettivi microcosmi.
La caratterizzazione dei microbiomi ambientali del suolo e dell'intestino dei vermi si è basata sul sequenziamento di nuova generazione della regione V4 del gene rRNA batterico 16S. La preparazione della libreria di sequenziamento è stata ottenuta utilizzando i kit standard ed eseguita secondo le istruzioni dei produttori, con il sequenziamento eseguito su un sequenziatore commerciale (vedi Tabella dei materiali). Le sequenze demultiplexate sono state elaborate utilizzando DADA2, tassonomia assegnata basata sul database di riferimento SILVA v132 e analizzate con phyloseq16,17,18 (vedi file supplementare 1, figura supplementare S1, figura supplementare S2, figura supplementare S3, tabella supplementare S1 e tabella supplementare S2 per una descrizione dettagliata del sequenziamento e dell'analisi; la pipeline computazionale completa è disponibile in GitHub [https://github.com/kennytrang/CompostMicrocosms]). I dati grezzi sono disponibili presso l'NCBI Sequence Read Archive (Bioproject ID PRJNA856419).
In media, sono state ottenute 73.220 sequenze per campione. Queste sequenze rappresentano 15.027 varianti di sequenza amplicon (ASV), che coprono 27 phyla e 216 famiglie, comprese le famiglie considerate parte del microbioma intestinale13 di C. elegans, come Rhizobiaceae, Burkholderiaceae e Bacillaceae. Enterobacteriaceae e Pseudomonadaceae, che in precedenza erano risultati essere membri dominanti, erano una minoranza questa volta, ma erano ancora arricchiti (2-10 volte) rispetto ai rispettivi ambienti del suolo. I confronti basati su distanze UniFrac19,20 non ponderate e ponderate hanno dimostrato una buona riproducibilità tra microcosmo triplicati arricchiti con lo stesso prodotto, come indicato dal clustering ravvicinato. Al contrario, i microbiomi ambientali del suolo arricchiti con prodotti diversi raggruppati l'uno dall'altro, dimostrando la capacità di diversificare una comunità microbica iniziale attraverso l'aggiunta di prodotti diversi (Figura 2).
Nel confronto tra microbiomi intestinali di vermi e comunità ambientali, l'analisi delle coordinate principali (PCoA) con distanze UniFrac non ponderate o ponderate ha mostrato un raggruppamento distinto di microbiomi intestinali di vermi lontano da quello dei rispettivi ambienti per ciascun tipo di microcosmo (Figura 2). Mentre il PCoA basato su distanze UniFrac non ponderate non distingueva tra microbiomi del suolo e dei vermi (Figura 2A), il raggruppamento basato su distanze ponderate ha rivelato una chiara separazione dei microbiomi dell'intestino del verme e del compost (Figura 2B). Questi risultati supportano un processo in cui il filtraggio dell'ospite opera sulla disponibilità ambientale per modellare un microbioma intestinale che non è completamente distinto dalla sua fonte ambientale per quanto riguarda la presenza di taxa, ma modula la loro abbondanza arricchendo per un sottoinsieme dei taxa disponibili, risultando in definitiva in un microbioma intestinale di vermi centrale condiviso tra vermi allevati in ambienti diversi.

Figura 2: I microbiomi intestinali dei vermi si raggruppano lontano dai rispettivi ambienti microbici diversificati per i prodotti. La composizione del microbioma è stata determinata con il sequenziamento 16S e le comunità di microcosmi arricchiti con i prodotti designati o da vermi allevati in essi sono state raggruppate utilizzando PCoA basato su (A) distanze UniFrac non ponderate o (B) ponderate. Gli assi mostrati sono quelli che spiegano la maggiore variazione nella composizione della comunità tra i campioni (N = 3 per ogni tipo di microcosmo). Fare clic qui per visualizzare una versione ingrandita di questa figura.
File supplementare 1: sequenziamento di nuova generazione e analisi dei dati. Di seguito sono presentati i passaggi per la preparazione della libreria, il sequenziamento in laboratorio e l'analisi dei dati. Clicca qui per scaricare questo file.
Figura supplementare S1: Un esempio di grafico di controllo qualità per le letture inverse da un campione. L'asse X (ciclo) mostra la posizione del nucleotide lungo la sequenza letta. L'asse Y sinistro mostra il punteggio di qualità. La mappa di calore in scala di grigi rappresenta la frequenza del punteggio di qualità in ciascuna posizione nucleotidica; La linea verde rappresenta il punteggio di qualità mediano in ciascuna posizione nucleotidica; la linea arancione superiore rappresenta i quartili della distribuzione del punteggio di qualità; la linea rossa inferiore rappresenta la percentuale di letture di sequenza che hanno esteso quella posizione nucleotidica (asse Y destro, qui 100%). Clicca qui per scaricare questo file.
Figura supplementare S2: Tassi di errore per diversi campioni. La frequenza di errore nei diversi campioni (punti neri) dovrebbe diminuire con l'aumentare del punteggio di qualità per ogni possibile sostituzione della coppia di basi rappresentata, riflettendo la tendenza prevista. Clicca qui per scaricare questo file.
Figura supplementare S3: Un esempio di PCoA basato su distanze UniFrac ponderate. I nomi dei gruppi riportati nella legenda rappresentano i prodotti utilizzati per arricchire il compost utilizzato nei diversi microcosmi. Clicca qui per scaricare questo file.
Tabella supplementare S1: Filtraggio sequenziale delle sequenze. un Numero di letture della sequenza prima del filtraggio. b-d Ogni colonna rappresenta il numero di letture di sequenza rimanenti dopo una fase di filtraggio: filtraggio delle letture di bassa qualità (passaggio 2.5), algoritmo di denoising eseguito da dada() (passaggio 2.8), unione di letture in avanti e indietro (passaggio 2.9) e rimozione delle chimere (passaggio 2.11). Clicca qui per scaricare questo file.
Tabella supplementare S2: tabella dei metadati. Clicca qui per scaricare questo file.
Discussione
Il protocollo qui presentato descrive un metodo per studiare il microbioma intestinale dei nematodi allevati in ambienti naturali, offrendo un approccio alternativo all'isolamento dei vermi dalla natura o all'allevamento su comunità sintetiche.
Le migliaia di potenziali specie batteriche catturate nell'esperimento rappresentativo del microcosmo riflettono la diversità microbica con cui i vermi si sono evoluti e dimostrano la capacità della pipeline del microcosmo di combinare i vantaggi di lavorare con un organismo ospite modello e quelli di lavorare con comunità microbiche naturali e diverse.
I risultati rappresentativi dimostrano che arricchire un suolo comune con diversi tipi di prodotti modula la diversità microbica ambientale, evidenziando la gamma di diversità microbica disponibile per l'esplorazione utilizzando questa pipeline. La scelta dei prodotti non è particolarmente importante. Il lavoro precedente ha utilizzato banane, mele, arance, fragole, foglie di tè verde e patate per arricchire il terreno, con conseguente efficienza di aumento del verme simile. Anche i prodotti misti sono stati utilizzati in modo efficace. La caratteristica chiave è che prodotti diversi diversificheranno un determinato terreno in modi distinti.
Nonostante l'ampia variazione della diversità microbica ambientale, i microbiomi intestinali dei vermi variano considerevolmente meno, ricapitolando l'importanza della nicchia intestinale del verme e del filtraggio dell'ospite per l'assemblaggio di un microbioma intestinale centrale distinto da quello del suo ambiente suolo13. Questo modello è osservato meglio con l'analisi ponderata UniFrac PCoA, dimostrando che le differenze tra il suolo ambientale e i microbiomi intestinali dei vermi derivano principalmente da differenze nell'abbondanza relativa di taxa chiave.
Sebbene il protocollo qui descritto si concentri sulla raccolta di vermi per il sequenziamento 16S, i microcosmi possono essere utilizzati per esplorare ulteriori domande di interesse. Ad esempio, i vermi raccolti dai microcosmi possono essere successivamente esaminati per l'effetto di un microbioma intestinale diversificato sulla resistenza dell'ospite a varie condizioni avverse, inclusi agenti patogeni o tossine. In alternativa, nuove specie e ceppi batterici possono essere isolati e coltivati da vermi macinati, espandendo la diversità tassonomica e funzionale dei batteri disponibili per eseguire esperimenti.
Mentre i metodi di ricerca in ambienti di laboratorio cercano coerenza e riproducibilità, il lavoro con i microcosmi sfrutta la variazione naturale per esplorare le interazioni ospite-microbo in un contesto simile a quello naturale. Tuttavia, questa variazione pone anche alcune sfide. Alcuni terreni che includono alti livelli di invertebrati endogeni possono richiedere ulteriori fasi di centrifugazione, filtrazione ed esame per eliminare efficacemente gli organismi indesiderati dalla preparazione del microcosmo. Inoltre, la bassa abbondanza microbica nel suolo può indurre la formazione indesiderata di dauer nelle popolazioni di vermi, richiedendo un aumento dell'estratto microbico o arricchendo il suolo con una maggiore quantità di prodotti. Con ogni esperimento di microcosmo eseguito, i ricercatori continuano a esplorare l'intera diversità tassonomica e funzionale fornita dalla natura, consentendo la scoperta di nuovi taxa microbici e abilità funzionali che vanno dalla resistenza alle infezioni alla protezione contro gli xenobiotici ambientali.
Divulgazioni
Gli autori non hanno conflitti di interesse per dichiarare che sono rilevanti per il contenuto di questo articolo.
Riconoscimenti
Il lavoro descritto in questo manoscritto è stato supportato dalle sovvenzioni NIH R01OD024780 e R01AG061302. K.T. è stato inoltre supportato da una Summer Undergraduate Research Fellowship dell'Università della California, Berkeley, finanziata dalla Rose Hills Foundation. I disegni dei cartoni animati nella Figura 1 sono stati ottenuti da BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
Riferimenti
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon