Method Article
Microcosmos de compost como entornos microbianamente diversos y naturales para la investigación del microbioma en Caenorhabditis elegans
En este artículo
Resumen
Los microcosmos de compost llevan la diversidad microbiana que se encuentra en la naturaleza al laboratorio para facilitar la investigación del microbioma en Caenorhabditis elegans. Aquí se proporcionan protocolos para establecer experimentos de microcosmos, y los experimentos demuestran la capacidad de modular la diversidad microbiana ambiental para explorar las relaciones entre la diversidad microbiana ambiental y la composición del microbioma intestinal del gusano.
Resumen
El nematodo Caenorhabditis elegans está emergiendo como un modelo útil para estudiar los mecanismos moleculares subyacentes a las interacciones entre los huéspedes y sus microbiomas intestinales. Si bien los experimentos con bacterias bien caracterizadas o comunidades bacterianas definidas pueden facilitar el análisis de los mecanismos moleculares, el estudio de los nematodos en su contexto microbiano natural es esencial para explorar la diversidad de tales mecanismos. Al mismo tiempo, el aislamiento de gusanos de la naturaleza no siempre es factible e, incluso cuando es posible, el muestreo de la naturaleza restringe el uso del conjunto de herramientas genéticas disponibles para la investigación de C. elegans. El siguiente protocolo describe un método para estudios de microbiomas que utilizan microcosmos de compost para el crecimiento en el laboratorio en entornos microbianamente diversos y naturales.
El suelo de origen local puede enriquecerse con productos para diversificar las comunidades microbianas en las que se crían los gusanos y de las cuales se cosechan, lavan y esterilizan en la superficie para análisis posteriores. Experimentos representativos demuestran la capacidad de modular la comunidad microbiana en un suelo común enriqueciéndolo con diferentes productos y demuestran además que los gusanos criados en estos distintos ambientes ensamblan microbiomas intestinales similares distintos de sus respectivos entornos, apoyando la noción de un microbioma intestinal central específico de la especie. En general, los microcosmos de compost proporcionan entornos naturales en el laboratorio para la investigación del microbioma como una alternativa a las comunidades microbianas sintéticas o al aislamiento de nematodos silvestres.
Introducción
El nematodo Caenorhabditis elegans está emergiendo como un modelo útil para estudiar las interacciones entre los huéspedes y sus microbiomasintestinales 1,2. Como modelo, ofrece varias ventajas. En primer lugar, los animales libres de gérmenes o gnotobióticos son fáciles de obtener y mantener; La lejía puede ser utilizada para matar gusanos grávidos y microbios asociados, dejando sus huevos resistentes a la lejía ilesos para crecer como poblaciones sincronizadas por edad que pueden ser colonizadas por bacterias de interés 3,4. Además, cuando se cultiva en presencia de bacterias, C. elegans, un bacterívoro, ingiere las bacterias encontradas, con especies susceptibles digeridas o excretadas, mientras que las especies resistentes y persistentes colonizan de manera estable el intestino del gusano. Además, C. elegans son en su mayoría hermafroditas, produciendo poblaciones de progenie genéticamente idéntica, lo que reduce la variación genética de confusión. Junto con la disponibilidad de cepas de gusanos mutantes y transgénicos, trabajar con C. elegans ofrece a los investigadores un modelo gnotobiótico y genéticamente tratable para investigar los fundamentos moleculares de las interacciones huésped-microbio 5,6,7,8.
Si bien los experimentos con bacterias bien caracterizadas pueden facilitar el análisis de los mecanismos moleculares, identificar y estudiar las bacterias con las que interactúan los gusanos en la naturaleza es esencial para explorar la diversidad de tales mecanismos, desentrañar el contexto natural de su función y comprender las fuerzas selectivas que han dado forma a su evolución. Fuera del laboratorio, C. elegans se encuentra globalmente en climas templados húmedos, donde se cree que las poblaciones experimentan un ciclo de vida de "auge y caída", caracterizado por un rápido crecimiento de la población cuando los recursos son abundantes, seguido de un cambio de desarrollo hacia pioneros y tolerantes al estrés cuando los recursos se agotan9. Aunque se considera un nematodo del suelo, las poblaciones de C. elegans que proliferan en la naturaleza se encuentran más comúnmente alimentándose de material orgánico en descomposición, como flores o frutas podridas, donde las poblaciones bacterianas son abundantes y diversas.
Los estudios del microbioma intestinal en nematodos aislados de la naturaleza han identificado comunidades bacterianas diversas, pero características, 10,11, cuya composición fue respaldada por estudios realizados con gusanos criados en ambientes de microcosmos naturales 12,13. Juntos, tales estudios permitieron la delineación de un microbioma intestinal del gusano central2. Mientras que el muestreo de las poblaciones de C. elegans en la naturaleza representa el examen más directo de las interacciones naturales gusano-microbio, no es factible en todas partes y en cualquier momento, ya que se limita a regiones y estaciones con abundante precipitación10,11. Alternativamente, en lugar de aislar a los gusanos de su hábitat natural, los experimentos con microcosmos llevan el hábitat natural al laboratorio 6,8,12,13,14,15. Los ambientes de microcosmos se preparan a partir de suelo compostado con diversas frutas o verduras, lo que permite una mayor diversificación de la comunidad de suelo inicial. Ofrecen métodos experimentales manejables que combinan la diversidad microbiana y el entorno tridimensional del suelo silvestre con las ventajas experimentales de una instalación de laboratorio controlada y cepas de gusanos genéticamente definidas. El siguiente protocolo detalla los pasos involucrados en el trabajo con microcosmos de compost, demostrando su uso para comprender el ensamblaje de un microbioma intestinal de gusano característico de diversos entornos.
Protocolo
1. Preparación de compost
- Obtenga compost o tierra de jardín de cualquier fuente conveniente y guárdela dentro del laboratorio en un recipiente de plástico de cocina estándar con agujeros cortados en la tapa para dejar entrar el aire. Tapa los agujeros con algodón para mantener alejadas a las moscas de la fruta y otros invertebrados (Figura 1A).
NOTA: Quinientos gramos de tierra (caben en un recipiente de 1.5 galones, dimensiones: 30 cm x 20 cm x 10 cm) proporcionarán suficiente material para 12 microcosmos. - Enriquezca el compost o el suelo con productos picados o una mezcla de diferentes productos en una proporción de masa de 1: 2 de productos a tierra.
- Incubar durante 7-14 días a 20-25 °C, mezclar una vez al día y añadir medio M9 según sea necesario para mantener la humedad sin enturbiarla.
NOTA: Los suelos no enriquecidos con productos generalmente no admiten el crecimiento de C. elegans , pero el producto específico a utilizar depende del investigador, con muchos tipos y mezclas capaces de soportar el crecimiento de gusanos. El enriquecimiento con diferentes productos promoverá la diversificación de la comunidad bacteriana de diferentes maneras, permitiendo el estudio del ensamblaje del microbioma intestinal desde diferentes puntos de partida (consulte la sección de discusión).
2. Preparación de microcosmos de compost
- Para cada microcosmos, agregue 10 g de compost enriquecido a un vaso de precipitados de vidrio de 30 ml cubierto con papel de aluminio y autoclave (Figura 1B).
- Para preparar el extracto microbiano para reponer el compost esterilizado en autoclave, comience agregando 30 g del mismo compost utilizado en el paso 2.1 a cada uno de los tres tubos de 50 ml y llénelo con M9. Vórtice durante 1 min (Figura 1C).
NOTA: Esto debería proporcionar suficientes bacterias para nueve microcosmos, cada uno apoyando el desarrollo de cientos de nematodos. - Centrifugar los tubos a 560 × g durante 5 min a temperatura ambiente (RT).
- Prestando atención a no perturbar el pellet, retire los sobrenadantes con una pipeta serológica y combine en un nuevo tubo de 50 ml.
- Concentrar el extracto bacteriano centrifugando a velocidad máxima (2.000 × g) durante 15 min a RT. Resuspender los gránulos en suficiente M9 para tener 200 μL para cada microcosmos y 200 μL más para agregar a una placa que servirá como un proxy visible del desarrollo del gusano dentro del microcosmos.
NOTA: Por ejemplo, para nueve microcosmos, resuspenda el pellet microbiano en 2 ml de M9. - Agregue 200 μL del extracto microbiano concentrado a cada vaso de precipitados de compost esterilizado en autoclave, así como a una placa NGM que servirá como placa proxy visible.
- Incubar el microcosmos y la placa sustitutiva durante 24 h a 20-25 °C antes de añadir los gusanos.
3. Cría de lombrices en microcosmos de compost
- Agregue 500-1000 huevos o larvas L13 a cada microcosmos y a la placa proxy (paso 2.7) para comenzar el experimento (Figura 1D).
NOTA: Los experimentos descritos aquí utilizan gusanos de tipo salvaje N2. Sin embargo, se pueden usar otras cepas de C. elegans (y potencialmente otros nematodos). - Elevar los gusanos a la edad adulta a 20 °C (típicamente 3 días).
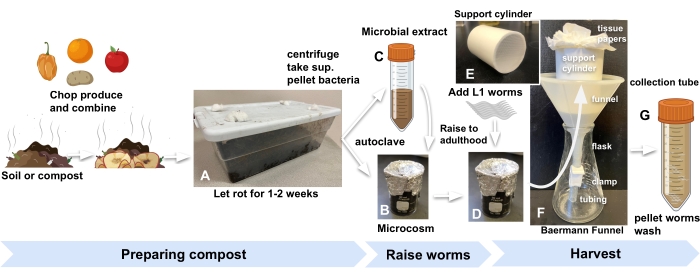
Figura 1: Preparación de microcosmos de compost, cría de gusanos y cosecha . (A) Enriquecer el suelo local o el compost con productos e incubar durante 2 semanas. Combine (B) suelo enriquecido esterilizado en autoclave con (C) extracto microbiano y (D) incube durante al menos 24 h antes de agregar gusanos L1s sincronizados al microcosmos para comenzar el experimento. (E, F) Cuando esté listo para cosechar, agregue compost del microcosmos en un cilindro de soporte de embudo Baermann y cubra con M9. (G) Después de 15 min, liberar el filtrado en un tubo de 50 ml. Abreviatura: sup. = sobrenadante. Haga clic aquí para ver una versión más grande de esta figura.
4. Preparación de un embudo Baermann para cosechar gusanos
- Ensamble un embudo Baermann uniendo 5-8 cm de tubo de goma rígido al extremo de un embudo de plástico.
- Deslice una abrazadera sobre el tubo y cierre la abraza.
- Coloque en el embudo un tubo cilíndrico de PVC de 7 cm de largo y 5 cm de diámetro con una malla de nylon de 1 mm pegada en su parte inferior (Figura 1E). Forre el cilindro con dos hojas de papel de seda.
- Colocar el embudo de Baermann en un matraz (figura 1F).
5. Cosechar gusanos del microcosmos y recolectar las muestras de suelo respectivas
- Agregue 20 ml de M9 al microcosmos en el que se criaron los gusanos, agite la mezcla y luego vierta la mezcla del vaso de precipitados en el cilindro forrado de papel de seda en la configuración del embudo Baermann. Agregue más M9 para sumergir completamente el compost en el embudo.
NOTA: Las poblaciones de C. elegans (N2) alcanzan la edad adulta en microcosmos de compost a un ritmo similar al de las placas de agar estándar sembradas con Escherichia coli. Para una mayor precisión en la recolección de gusanos en una etapa específica del desarrollo, consulte la placa proxy del paso 3.3. - Después de 30 minutos, desabroche la pinza para liberar el filtrado que contiene los gusanos cosechados en un tubo de 50 ml (Figura 1G).
- Agregue más M9 al cilindro y repita durante una segunda ronda para cosechar más gusanos, y una vez más si se requieren gusanos adicionales.
NOTA: Cuando repita las rondas de cosecha, tenga cuidado de no comprometer la integridad del papel de seda, lo que podría permitir el paso de partículas de tierra. Un máximo de cuatro rondas de cosecha deben aislar al menos el 50% de los gusanos originalmente agregados al microcosmos sin comprometer la integridad del papel tisú. - Concentrar los gusanos centrifugando a 560 × g durante 2 min (RT). Extraer 35 ml del sobrenadante con una pipeta serológica.
- Transfiera los 15 ml restantes a un tubo de 15 ml y centrifugar nuevamente a 560 × g durante 1 minuto para concentrar aún más los gusanos. Extraer 14 ml del sobrenadante con una pipeta serológica.
- Paralelamente, recoja 1 g del suelo del microcosmos restante en un tubo de 1,5 ml. Procesar inmediatamente las muestras de suelo que contienen la comunidad bacteriana ambiental o almacenarlas a -20 °C para su posterior extracción de ácidos nucleicos, como se describe a continuación para muestras de lombrices.
6. Lavado y esterilización superficial de gusanos cosechados
- Transfiera 1 ml de los gusanos concentrados cosechados del paso 5.5 a un tubo de 1,5 ml utilizando una pipeta de vidrio. Incubar durante 2 minutos para permitir que los gusanos se asienten en el fondo del tubo. Retire el sobrenadante, dejando los 100 μL inferiores sin perturbaciones.
- Lave 6x con 1.5 ml de M9 + T (0.025% Triton-X en M9), permitiendo que los gusanos se asienten en el fondo cada vez.
- Transfiera los gusanos lavados en un volumen de 100 μL a un nuevo tubo de 1,5 ml con una pipeta de vidrio.
NOTA: En este punto del protocolo, deberían haber pasado aproximadamente 30 minutos desde el primer lavado (paso 6.2). Se recomienda permitir que los gusanos permanezcan sin alimentos durante al menos 1 h antes de la adición de levamisol para permitir la excreción de bacterias transitorias y la digestión completa de las bacterias de los alimentos. - Añadir 100 μL de clorhidrato de levamisol a 25 mM para paralizar los gusanos. Incubar durante 5 min en RT.
- Añadir 200 μL de solución de lejía al 4%. Incubar durante 2 min.
- Retire el sobrenadante, dejando los 150 μL más bajos sin alterar, y lave 3 veces con M9 + T, como se indica anteriormente.
NOTA: Para minimizar la contaminación de las muestras, se recomienda utilizar M9+T filtrado (a través de un filtro de 0,2 μm) y usar guantes a partir de este momento. - Después de los lavados, tomar 50 μL de los 150 μL restantes del último lavado y planchar en una placa LB, incubando a 25 °C durante 48 h para confirmar la eliminación efectiva de las bacterias externas.
NOTA: Se permite observar hasta 30 colonias en el último lavado (que representan 60 células bacterianas externas que quedan en total en la muestra) y se espera que tenga solo una contribución marginal a la composición del microbioma analizado, dadas las miles de bacterias que típicamente colonizan cada gusano adulto15. Cuando se observan más, la integridad de la muestra puede verse comprometida. - Use los gusanos esterilizados en la superficie inmediatamente (por ejemplo, para extraer bacterias vivas para el cultivo [recuentos de UFC]) o guárdelos a -20 °C para la posterior extracción de ácidos nucleicos.
7. Extracción de ADN
NOTA: Los siguientes pasos describen la extracción de ADN de los gusanos cosechados utilizando un kit comercial diseñado para la extracción de ADN microbiano del suelo (ver Tabla de materiales), con modificaciones descritas a continuación para facilitar la extracción de ADN microbiano de gusanos.
- Transfiera la muestra que contiene gusanos esterilizados en superficie (descongele en RT si es necesario) a los tubos proporcionados por el kit, reemplazando las perlas de vidrio proporcionadas con aproximadamente 30-50 perlas de zirconia de 1 mm de diámetro (consulte la Tabla de materiales).
NOTA: Los rendimientos de ADN observados son mayores con las perlas de zirconia que con las cuentas de vidrio proporcionadas por el kit. Aunque la razón detrás de esta observación sigue siendo indeterminada, las perlas de zirconia pueden romper la cutícula del nematodo de manera más eficiente, liberando más bacterias. - Para muestras de compost, agregue aproximadamente 250 mg de la tierra recolectada a los tubos del kit sin reemplazar las perlas de vidrio.
- Después de la adición de la solución tampón proporcionada por el kit a todas las muestras, homogeneice con un homogeneizador de potencia en RT (consulte la Tabla de materiales) durante dos rondas de 2.000 rpm durante 30 s cada una, haciendo una pausa durante 30 s en el medio.
- Complete los pasos restantes de purificación de ADN de acuerdo con el protocolo del kit. Eluya las muestras de gusanos en no más de 50 μL de tampón de elución para garantizar concentraciones de ADN lo suficientemente altas para la secuenciación.
Resultados
Para explorar la capacidad de diversificar la comunidad de microcosmos del suelo, comparamos las comunidades microbianas en microcosmos de compost preparados enriqueciendo el mismo suelo inicial, un compost de grado industrial disponible en la ciudad de Berkeley, California, con diferentes productos: manzanas, pimientos, naranjas o papas (cada uno por triplicado). Además, comparamos las comunidades microbianas de cada entorno de compost con el microbioma intestinal de C. elegans de tipo silvestre criados en el microcosmos respectivo. El análisis se realizó con muestras de ADN extraídas de aproximadamente 500 adultos esterilizados en la superficie por microcosmos y de muestras de compost de 250 mg de los respectivos microcosmos.
La caracterización de los microbiomas ambientales del suelo y del intestino de los gusanos se basó en la secuenciación de próxima generación de la región V4 del gen bacteriano 16S rRNA. La preparación de la biblioteca de secuenciación se logró utilizando los kits estándar y se realizó de acuerdo con las instrucciones del fabricante, con la secuenciación realizada en un secuenciador comercial (ver Tabla de materiales). Las secuencias demultiplexadas se procesaron utilizando DADA2, se les asignó una taxonomía basada en la base de datos de referencia SILVA v132 y se analizaron con phyloseq16,17,18 (ver Archivo Suplementario 1, Figura Suplementaria S1, Figura Suplementaria S2, Figura Suplementaria S3, Tabla Suplementaria S1 y Tabla Suplementaria S2 para una descripción detallada de la secuenciación y el análisis; la canalización computacional completa está disponible en GitHub [https://github.com/kennytrang/CompostMicrocosms]). Los datos sin procesar están disponibles en el NCBI Sequence Read Archive (ID de bioproyecto PRJNA856419).
En promedio, se obtuvieron 73.220 secuencias por muestra. Estas secuencias representan 15.027 variantes de secuencia de amplicón (ASV), que abarcan 27 filos y 216 familias, incluidas familias consideradas parte del núcleo del microbioma intestinal13 de C. elegans, como Rhizobiaceae, Burkholderiaceae y Bacillaceae. Enterobacteriaceae, y Pseudomonadaceae, que previamente se encontró que eran miembros dominantes, eran una minoría esta vez, pero todavía estaban enriquecidos (2-10 veces) en comparación con sus respectivos ambientes de suelo. Las comparaciones basadas en distancias UniFrac19,20 no ponderadas y ponderadas demostraron una buena reproducibilidad entre triplicados de microcosmos enriquecidos con el mismo producto, como lo indica la agrupación cercana. En contraste, los microbiomas ambientales del suelo enriquecidos con diferentes productos se agruparon entre sí, lo que demuestra la capacidad de diversificar una comunidad microbiana inicial mediante la adición de diferentes productos (Figura 2).
En las comparaciones de microbiomas intestinales de gusanos y comunidades ambientales, el análisis de coordenadas principales (PCoA) con distancias UniFrac no ponderadas o ponderadas mostró una agrupación distinta de microbiomas intestinales de lombrices lejos de la de sus respectivos entornos para cada tipo de microcosmos (Figura 2). Mientras que el PCoA basado en distancias UniFrac no ponderadas no distinguió entre los microbiomas del suelo y del gusano (Figura 2A), la agrupación basada en distancias ponderadas reveló una clara separación del intestino del gusano y los microbiomas de compost (Figura 2B). Estos resultados apoyan un proceso en el que el filtrado del huésped opera según la disponibilidad ambiental para dar forma a un microbioma intestinal que no es completamente distinto de su fuente ambiental con respecto a la presencia de taxones, pero modula su abundancia enriqueciendo un subconjunto de los taxones disponibles, lo que finalmente resulta en un microbioma intestinal del gusano central compartido entre gusanos criados en diferentes entornos.

Figura 2: Microbiomas intestinales de gusanos que se agrupan lejos de sus respectivos entornos microbianos diversificados para productos. La composición del microbioma se determinó con secuenciación 16S, y las comunidades de microcosmos enriquecidos con el producto designado o de gusanos criados en ellos se agruparon utilizando PCoA basado en (A) distancias UniFrac no ponderadas o (B) ponderadas. Los ejes mostrados son los que explican la mayor variación en la composición de la comunidad entre muestras (N = 3 para cada tipo de microcosmos). Haga clic aquí para ver una versión más grande de esta figura.
Archivo complementario 1: Secuenciación de próxima generación y análisis de datos. Aquí se presentan los pasos para la preparación de la biblioteca, la secuenciación en el laboratorio y el análisis de datos. Haga clic aquí para descargar este archivo.
Figura suplementaria S1: Un ejemplo de un gráfico de control de calidad para las lecturas inversas de una muestra. El eje X (ciclo) muestra la posición del nucleótido a lo largo de la secuencia leída. El eje Y izquierdo muestra la puntuación de calidad. El mapa de calor en escala de grises representa la frecuencia de la puntuación de calidad en cada posición de nucleótido; La línea verde representa la puntuación de calidad mediana en cada posición de nucleótido; La línea naranja superior representa los cuartiles de la distribución de la puntuación de calidad; la línea roja inferior representa el porcentaje de lecturas de secuencia que extendieron esa posición de nucleótido (eje Y derecho, aquí 100%). Haga clic aquí para descargar este archivo.
Figura suplementaria S2: Tasas de error para diferentes muestras. La frecuencia de error en las diferentes muestras (puntos negros) debería disminuir con el aumento de la puntuación de calidad para cada posible sustitución de pares de bases representada, lo que refleja la tendencia esperada. Haga clic aquí para descargar este archivo.
Figura suplementaria S3: Un ejemplo de PCoA basado en distancias UniFrac ponderadas. Los nombres de los grupos que se muestran en la leyenda representan el producto utilizado para enriquecer el compost utilizado en los diferentes microcosmos. Haga clic aquí para descargar este archivo.
Tabla suplementaria S1: Filtrado secuencial de secuencias. un Número de lecturas de secuencia antes del filtrado. b-d Cada columna representa el número de lecturas de secuencia restantes después de un paso de filtración: filtrado de lecturas de baja calidad (paso 2.5), eliminación de ruido del algoritmo realizado por dada() (paso 2.8), fusión de lecturas hacia adelante y hacia atrás (paso 2.9) y eliminación de quimeras (paso 2.11). Haga clic aquí para descargar este archivo.
Tabla suplementaria S2: Tabla de metadatos. Haga clic aquí para descargar este archivo.
Discusión
El protocolo presentado aquí describe un método para estudiar el microbioma intestinal de nematodos criados en ambientes naturales, ofreciendo un enfoque alternativo para el aislamiento de gusanos de la naturaleza o para criarlos en comunidades sintéticas.
Las miles de especies bacterianas potenciales capturadas en el experimento representativo del microcosmos reflejan la diversidad microbiana con la que los gusanos han evolucionado y demuestran la capacidad de la tubería de microcosmos para combinar las ventajas de trabajar con un organismo huésped modelo y las de trabajar con comunidades microbianas naturales y diversas.
Los resultados representativos demuestran que enriquecer un suelo común con diferentes tipos de productos modula la diversidad microbiana ambiental, destacando la gama de diversidad microbiana disponible para la exploración utilizando esta tubería. La elección de productos no es particularmente importante. Trabajos anteriores han utilizado plátanos, manzanas, naranjas, fresas, hojas de té verde y papas para enriquecer el suelo, lo que resulta en una eficiencia similar de crianza de gusanos. Los productos mixtos también se han utilizado de manera efectiva. La característica clave es que diferentes productos diversificarán un suelo dado de distintas maneras.
A pesar de la amplia variación en la diversidad microbiana ambiental, los microbiomas intestinales de los gusanos varían considerablemente menos, recapitulando la importancia del nicho del intestino del gusano y el filtrado del huésped para el ensamblaje de un microbioma intestinal central que es distinto del de su entorno del suelo13. Este patrón se observa mejor con el análisis ponderado de UniFrac PCoA, lo que demuestra que las diferencias entre el suelo ambiental y los microbiomas intestinales de gusanos provienen principalmente de las diferencias en la abundancia relativa de taxones clave.
Aunque el protocolo descrito aquí se centra en la cosecha de gusanos para la secuenciación 16S, los microcosmos se pueden utilizar para explorar cuestiones adicionales de interés. Por ejemplo, los gusanos cosechados de microcosmos pueden examinarse posteriormente para determinar el efecto de un microbioma intestinal diverso en la resistencia del huésped a diversas condiciones adversas, incluidos patógenos o toxinas. Alternativamente, nuevas especies y cepas bacterianas pueden aislarse y cultivarse a partir de gusanos cosechados en el suelo, ampliando la diversidad taxonómica y funcional de las bacterias disponibles para realizar experimentos.
Mientras que los métodos de investigación en entornos de laboratorio buscan consistencia y reproducibilidad, el trabajo con microcosmos aprovecha la variación natural para explorar las interacciones huésped-microbio en un contexto natural. Sin embargo, esta variación también plantea algunos desafíos. Algunos suelos que incluyen altos niveles de invertebrados endógenos pueden requerir pasos adicionales de centrifugación, filtración y examen para eliminar eficazmente los organismos no deseados de la preparación del microcosmos. Además, la baja abundancia microbiana en el suelo puede inducir la formación de dauer no deseados en las poblaciones de gusanos, lo que requiere un aumento en el extracto microbiano o enriquecer el suelo con una mayor cantidad de productos. Con cada experimento de microcosmos realizado, los investigadores continúan explorando toda la diversidad taxonómica y funcional proporcionada por la naturaleza, lo que permite el descubrimiento de nuevos taxones microbianos y capacidades funcionales que van desde la resistencia a las infecciones hasta la protección contra los xenobióticos ambientales.
Divulgaciones
Los autores no tienen conflictos de intereses para declarar que son relevantes para el contenido de este artículo.
Agradecimientos
El trabajo descrito en este manuscrito fue apoyado por las subvenciones de los NIH R01OD024780 y R01AG061302. K.T. fue apoyado además por una beca de investigación de pregrado de verano de la Universidad de California, Berkeley, financiada por la Fundación Rose Hills. Los diseños de dibujos animados en la Figura 1 se obtuvieron de BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
Referencias
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados