Method Article
Caenorhabditis elegans의 마이크로바이옴 연구를 위한 미생물적으로 다양하고 자연과 유사한 환경으로서의 소우주를 퇴비화
요약
퇴비 소우주는 자연에서 발견되는 미생물 다양성을 실험실로 가져와 Caenorhabditis elegans의 미생물 군집 연구를 용이하게합니다. 여기에는 소우주 실험을 설정하기위한 프로토콜이 제공되며, 실험은 환경 미생물 다양성과 벌레 장내 미생물 구성 간의 관계를 탐구하기 위해 환경 미생물 다양성을 조절하는 능력을 보여줍니다.
초록
선충류 Caenorhabditis elegans 는 숙주와 장내 미생물 군집 사이의 상호 작용의 기초가되는 분자 메커니즘을 연구하는 데 유용한 모델로 부상하고 있습니다. 잘 특성화 된 박테리아 또는 정의 된 박테리아 군집을 사용한 실험은 분자 메커니즘의 분석을 용이하게 할 수 있지만, 천연 미생물 맥락에서 선충류를 연구하는 것은 그러한 메커니즘의 다양성을 탐구하는 데 필수적입니다. 동시에 야생에서 벌레를 분리하는 것이 항상 가능한 것은 아니며 가능한 경우에도 야생에서 샘플링하면 C. elegans 연구에 사용할 수있는 유전 툴킷의 사용이 제한됩니다. 다음 프로토콜은 미생물적으로 다양하고 자연과 유사한 환경에서 실험실 내 성장을 위해 퇴비 소우주를 활용하는 마이크로바이옴 연구 방법을 설명합니다.
현지에서 조달 된 토양은 농산물을 풍부하게하여 벌레가 사육되고 후속 분석을 위해 수확, 세척 및 표면 멸균되는 미생물 군집을 다양화할 수 있습니다. 대표적인 실험은 다양한 농산물로 미생물 군집을 풍부하게함으로써 공통 토양에서 미생물 군집을 조절하는 능력을 입증하고, 이러한 별개의 환경에서 자란 벌레가 각각의 환경과 구별되는 유사한 장내 미생물 군집을 조립하여 종 별 핵심 장내 미생물 군집의 개념을 뒷받침한다는 것을 보여줍니다. 전반적으로 퇴비 소우주는 합성 미생물 군집 또는 야생 선충류의 분리에 대한 대안으로 미생물 군집 연구를위한 자연과 같은 실험실 내 환경을 제공합니다.
서문
선충류 Caenorhabditis elegans는 숙주와 장내 미생물 군집 사이의 상호 작용을 연구하는 데 유용한 모델로 부상하고 있습니다 1,2. 모델로서 몇 가지 이점을 제공합니다. 첫째, 무균 또는 gnotobiotic 동물은 쉽게 얻고 유지할 수 있습니다. 표백제는 중력충 및 관련 미생물을 죽이는 데 사용할 수 있으며, 표백제 내성 계란을 무사히 남겨두고 관심 박테리아에 의해 식민지화될 수 있는 연령 동기화 개체군으로 성장할 수 있습니다3,4. 또한 박테리아가있는 상태에서 자라면 박테리아 인 C. elegans는 민감한 종을 소화하거나 배설하는 박테리아를 섭취하고 내성이 있고 지속적인 종은 벌레 내장을 안정적으로 식민지화합니다. 또한 C. elegans는 대부분 자웅동체이며 유전적으로 동일한 자손 개체군을 생산하여 혼란스러운 유전적 변이를 줄입니다. 돌연변이 및 형질전환 웜 균주의 가용성과 함께 C. elegans와의 협력은 연구자들에게 숙주-미생물 상호 작용의 분자적 토대를 조사하기 위한 gnotobiotic 및 유전적으로 다루기 쉬운 모델을 제공합니다 5,6,7,8.
잘 특성화 된 박테리아를 사용한 실험은 분자 메커니즘의 분석을 용이하게 할 수 있지만, 벌레가 자연에서 상호 작용하는 박테리아를 식별하고 연구하는 것은 그러한 메커니즘의 다양성을 탐구하고, 기능에 대한 자연적 맥락을 풀고, 진화를 형성 한 선택적 힘을 이해하는 데 필수적입니다. 실험실 밖에서 C. elegans 는 습한 온대 기후에서 전 세계적으로 발견되며, 인구는 자원이 풍부할 때 급속한 인구 증가를 특징으로 하는 "붐 앤 버스트" 수명 주기를 겪고 자원이 고갈되면 선구적이고 스트레스에 강한 다우어로의 발달 전환이 뒤따르는 것으로 생각됩니다9. 토양 선충으로 간주되지만 야생 증식하는 C. elegans 개체군은 박테리아 개체군이 풍부하고 다양한 썩은 꽃이나 과일과 같은 분해되는 유기 물질을 먹는 것으로 가장 일반적으로 발견됩니다.
야생에서 분리 된 선충류의 장내 미생물 군집에 대한 연구는 다양하면서도 특징적인 박테리아 군집10,11을 확인했으며, 그 구성은 자연과 유사한 소우주 환경12,13에서 자란 벌레로 수행 된 연구에 의해 더욱 뒷받침되었습니다. 함께, 그러한 연구는 핵심 벌레 장내 미생물 군집2의 묘사를 가능하게했습니다. 야생에서 C. elegans 개체군의 샘플링은 자연 벌레-미생물 상호 작용에 대한 가장 직접적인 검사를 나타내는 반면, 강수량이 충분한 지역과 계절로 제한되기 때문에 언제 어디서나 가능합니다10,11. 또는 자연 서식지에서 벌레를 분리하는 대신 소우주를 사용한 실험은 자연 서식지를 실험실 6,8,12,13,14,15로 가져옵니다. 소우주 환경은 다양한 과일이나 채소로 퇴비화 된 토양으로 준비되므로 시작 토양 공동체를 더욱 다양화할 수 있습니다. 그들은 미생물 다양성과 3차원 야생 토양 환경을 통제된 실험실 시설 및 유전적으로 정의된 벌레 균주의 실험적 이점과 결합한 다루기 쉬운 실험 방법을 제공합니다. 아래 프로토콜은 퇴비 소우주 작업과 관련된 단계를 자세히 설명하여 다양한 환경에서 특징적인 벌레 장내 미생물군집의 조립을 이해하는 데 사용하는 방법을 보여줍니다.
프로토콜
1. 퇴비의 준비
- 편리한 출처에서 퇴비 또는 정원 토양을 얻고 공기가 들어올 수 있도록 뚜껑에 구멍이 뚫린 표준 주방 플라스틱 용기에 실험실 내부에 보관하십시오. 초파리와 다른 무척추 동물을 막기 위해 탈지면으로 구멍을 막습니다 (그림 1A).
알림: 500g의 토양(1.5갤런 용기에 장착, 치수: 30cm x 20cm x 10cm)은 12개의 소우주에 충분한 재료를 제공합니다. - 다진 농산물 또는 다른 농산물의 혼합물로 퇴비 또는 토양을 농산물 대 토양의 1:2 질량비로 풍부하게 합니다.
- 20-25 ° C에서 7-14 일 동안 배양하고 하루에 한 번 혼합하고 필요에 따라 M9 배지를 추가하여 진흙 투성이가되지 않고 수분을 유지합니다.
참고 : 농산물이 풍부하지 않은 토양은 일반적으로 C. elegans 성장을 지원하지 않지만 사용할 특정 농산물은 연구자에게 달려 있으며 웜 성장을 지원할 수있는 많은 유형과 혼합물이 있습니다. 다양한 농산물을 농축하면 다양한 방식으로 박테리아 군집 다양 화를 촉진하여 다양한 출발점에서 장내 미생물 군집 조립을 연구 할 수 있습니다 (토론 섹션 참조).
2. 퇴비 소우주의 준비
- 각 소우주에 대해 주석 호일과 오토클레이브로 덮인 30mL 유리 비커에 농축 퇴비 10g을 추가합니다(그림 1B).
- 오토클레이브 퇴비를 보충하기 위해 미생물 추출물을 준비하려면 먼저 2.1단계에서 사용한 것과 동일한 퇴비 30g을 3개의 50mL 튜브 각각에 추가하고 M9로 채웁니다. 1 분 동안 와류 (그림 1C).
참고: 이것은 각각 수백 개의 선충류의 발달을 지원하는 9개의 소우주에 충분한 박테리아를 제공해야 합니다. - 실온(RT)에서 5분 동안 560× g 의 튜브를 원심분리합니다.
- 펠릿을 방해하지 않도록주의하면서 혈청 학적 피펫으로 상청액을 제거하고 새로운 50mL 튜브에 결합하십시오.
- RT에서 15분 동안 최대 속도(2,000× g)로 원심분리하여 박테리아 추출물을 농축합니다. 각 소우주에 대해 200μL가 되도록 충분한 M9에 펠릿을 재현탁하고 소우주 내부의 벌레 발달의 가시적인 대리인 역할을 할 플레이트에 추가하기 위해 200μL를 더 추가합니다.
참고: 예를 들어, 9개의 소우주의 경우 미생물 펠릿을 2mL의 M9에 재현탁합니다. - 농축 미생물 추출물 200μL를 오토클레이브 퇴비의 각 비커와 가시적 프록시 플레이트 역할을 할 NGM 플레이트에 추가합니다.
- 소우주와 프록시 플레이트를 20-25 ° C에서 24 시간 동안 배양 한 후 벌레를 첨가하십시오.
3. 퇴비 소우주에서 벌레 키우기
- 500-1000 개의 알 또는 L1 유충3 을 각 소우주와 프록시 플레이트 (2.7 단계)에 추가하여 실험을 시작합니다 (그림 1D).
참고: 여기에 설명된 실험은 N2 야생형 웜을 사용합니다. 그러나 다른 C. elegans 균주 (및 잠재적으로 다른 선충류)를 사용할 수 있습니다. - 벌레를 20 ° C (일반적으로 3 일)에서 성충으로 올립니다.
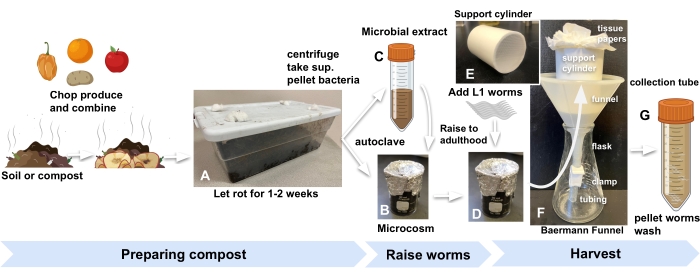
그림 1: 퇴비 소우주 준비, 벌레 키우기, 수확 . (A) 농산물로 지역 토양이나 퇴비를 풍부하게하고 2 주 동안 배양하십시오. (B) 오토클레이브 농축 토양을 (C) 미생물 추출물과 결합하고 (D) 동기화된 L1s 웜을 소우주에 추가하기 전에 최소 24시간 동안 배양하여 실험을 시작합니다. (E, F) 수확 할 준비가되면 소우주의 퇴비를 Baermann 깔때기지지 실린더에 넣고 M9로 덮습니다. (G) 15분 후, 여과액을 50mL 튜브에 넣는다. 약어 : sup. = 상청액. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 벌레 수확을위한 베어 만 깔때기 준비
- 플라스틱 깔때기 끝에 5-8cm의 단단한 고무 튜브를 부착하여 Baermann 깔때기를 조립합니다.
- cl을 밀어 넣습니다.amp 튜브에 밀어 넣고 cl을 닫습니다.
- 깔때기에 길이 7cm, 지름 5cm의 원통형 PVC 파이프를 놓고 바닥에 1mm 나일론 메쉬를 붙입니다 (그림 1E). 실린더에 두 장의 티슈 페이퍼를 깔아줍니다.
- Baermann 깔때기를 플라스크에 넣습니다(그림 1F).
5. 소우주에서 벌레를 수확하고 각각의 토양 샘플을 수집합니다.
- 벌레가 자란 소우주에 M9 20mL를 넣고 혼합물을 교반한 다음 비커의 혼합물을 Baermann 깔때기 설정의 티슈 페이퍼가 늘어선 실린더에 붓습니다. 깔때기에 퇴비를 완전히 담그려면 M9를 더 추가하십시오.
참고: C. elegans (N2) 개체군은 대장균이 파종 된 표준 한천 플레이트와 유사한 속도로 퇴비 소우주에서 성인기에 도달합니다. 특정 발달 단계에서 벌레를 수확하는 데 더 많은 정확성을 얻으려면 3.3 단계의 프록시 플레이트를 참조하십시오. - 30분 후 클램프를 풀어 수확한 벌레가 들어 있는 여과액을 50mL 튜브에 넣습니다(그림 1G).
- 실린더에 M9를 더 추가하고 두 번째 라운드를 반복하여 더 많은 웜을 수확하고 추가 웜이 필요한 경우 한 번 더 수확합니다.
알림: 수확 라운드를 반복 할 때 토양 입자가 통과 할 수있는 티슈 페이퍼의 무결성을 손상시키지 않도록주의하십시오. 최대 4 번의 수확 라운드는 티슈 페이퍼 무결성을 손상시키지 않으면 서 소우주에 원래 추가 된 벌레의 최소 50 %를 분리해야합니다. - 560 × g 에서 2 분 (RT) 동안 원심 분리하여 웜을 농축하십시오. 혈청학적 피펫으로 상청액 35mL를 제거합니다.
- 나머지 15mL를 15mL 튜브로 옮기고 560× g 에서 1분 동안 다시 원심분리하여 웜을 더 농축합니다. 혈청 학적 피펫으로 상청액 14mL를 제거합니다.
- 동시에 나머지 소우주 토양 1g을 1.5mL 튜브에 수집합니다. 환경 박테리아 군집을 포함하는 토양 샘플을 즉시 처리하거나 웜 샘플에 대해 아래에 설명된 대로 나중에 핵산을 추출하기 위해 -20°C에서 보관하십시오.
6. 수확 된 벌레의 세척 및 표면 살균
- 5.5단계에서 수확한 농축된 벌레 1mL를 유리 피펫을 사용하여 1.5mL 튜브로 옮깁니다. 벌레가 튜브 바닥에 정착 할 수 있도록 2 분 동안 배양하십시오. 상청액을 제거하고 바닥 100 μL를 그대로 둡니다.
- 1.5mL의 M9+T(M9의 0.025% 트리톤-X)로 6x 세척하여 매번 벌레가 바닥에 가라앉도록 합니다.
- 세척된 웜을 100μL 부피로 유리 피펫을 사용하여 새 1.5mL 튜브로 옮깁니다.
알림: 프로토콜의 이 시점에서 첫 번째 세척 이후 약 30분이 경과해야 합니다(6.2단계). 일시적인 박테리아의 배설과 음식 박테리아의 완전한 소화를 허용하기 위해 levamisole을 첨가하기 전에 벌레가 적어도 1 시간 동안 음식없이 남아 있도록 허용하는 것이 좋습니다. - 100μL의 25mM 레바미솔 염산염을 추가하여 벌레를 마비시킵니다. RT에서 5 분 동안 배양하십시오.
- 200 % 표백제 용액 4 μL를 첨가하십시오. 2 분 동안 배양하십시오.
- 상청액을 제거하고 맨 아래 150μL를 그대로 두고 위와 같이 M9+T로 3x 세척합니다.
알림: 샘플의 오염을 최소화하려면 여과된 M9+T(0.2μm 필터 통과)를 사용하고 이 시점부터 장갑을 착용하는 것이 좋습니다. - 세척 후 마지막 세척에서 남은 150μL 중 50μL를 취하여 LB 플레이트에 플레이트하고 25°C에서 48시간 동안 배양하여 외부 박테리아의 효과적인 제거를 확인합니다.
참고: 마지막 세척에서 최대 30개의 콜로니(샘플에 남아 있는 총 60개의 외부 박테리아 세포를 나타냄)를 관찰하는 것이 허용되며 일반적으로 각 성충(15)에 서식하는 수천 개의 박테리아를 고려할 때 분석된 마이크로바이옴 구성에 미미한 기여만 있을 것으로 예상됩니다. 더 많은 것이 관찰되면 샘플 무결성이 손상될 수 있습니다. - 표면 멸균된 웜을 즉시(예: 배양을 위한 살아있는 박테리아 추출[CFU 카운트]) 사용하거나 후속 핵산 추출을 위해 -20°C에서 보관하십시오.
7. DNA 추출
알림: 다음 단계는 토양에서 미생물 DNA를 추출하도록 설계된 상용 키트( 재료 표 참조)를 사용하여 수확된 벌레의 DNA 추출을 설명하고 벌레에서 미생물 DNA를 쉽게 추출할 수 있도록 아래에 설명된 수정 사항을 설명합니다.
- 표면 멸균된 웜(필요한 경우 RT에서 제상)이 포함된 샘플을 키트에서 제공하는 튜브로 옮기고 제공된 유리 비드를 약 30-50 직경 1mm 지르코니아 비드로 교체합니다( 재료 표 참조).
참고: 관찰된 DNA 수율은 키트에서 제공하는 유리 비드보다 지르코니아 비드에서 더 높습니다. 이 관찰의 이유는 아직 밝혀지지 않았지만 지르코니아 비드는 선충 큐티클을 더 효율적으로 열어 더 많은 박테리아를 방출할 수 있습니다. - 퇴비 샘플의 경우 유리 구슬을 교체하지 않고 수집 된 토양 약 250mg을 키트의 튜브에 추가하십시오.
- 키트에 의해 제공된 완충 용액을 모든 샘플에 첨가한 후, RT에서 파워 균질화기( 재료 표 참조)로 각각 30초 동안 2,000rpm의 두 라운드 동안 균질화하고 그 사이에 30초 동안 일시 중지합니다.
- 키트 프로토콜에 따라 나머지 DNA 정제 단계를 완료하십시오. 50μL 이하의 용리 완충액에서 웜 샘플을 용리하여 시퀀싱에 충분히 높은 DNA 농도를 보장합니다.
결과
토양 소우주 군집을 다양화하는 능력을 탐구하기 위해 우리는 캘리포니아 버클리시에서 구할 수있는 산업 등급 퇴비 인 동일한 초기 토양을 풍부하게하여 준비된 퇴비 소우주의 미생물 군집을 사과, 피망, 오렌지 또는 감자 (각각 3 배 세트). 우리는 또한 각 퇴비 환경의 미생물 군집을 각각의 소우주에서 자란 야생형 C. elegans 의 장내 미생물 군집과 비교했습니다. 분석은 소우주 당 약 500 명의 표면 멸균 된 성인과 각 소우주의 250mg 퇴비 샘플에서 추출한 DNA 샘플로 수행되었습니다.
환경 토양 및 벌레 장내 미생물군집의 특성 분석은 박테리아 16S rRNA 유전자의 V4 영역의 차세대 시퀀싱에 의존했습니다. 시퀀싱 라이브러리 준비는 표준 키트를 사용하여 달성되었으며 제조업체의 지침에 따라 수행되었으며 시퀀싱은 상용 시퀀서에서 수행되었습니다(재료 표 참조). 역다중화된 서열은 DADA2를 사용하여 처리하고, SILVA v132 참조 데이터베이스를 기반으로 분류법을 할당하고, 계통16,17,18로 분석하였다(보충 파일 1, 보충 그림 S1, 보충 그림 S2, 보충 그림 S3, 보충 표 S1 및 보충 표 S2 참조) 시퀀싱 및 분석에 대한 자세한 설명; 전체 계산 파이프라인은 GitHub [https://github.com/kennytrang/CompostMicrocosms]에서 사용할 수 있습니다. 원시 데이터는 NCBI 서열 판독 아카이브(바이오프로젝트 ID PRJNA856419)에서 확인할 수 있습니다.
평균적으로 샘플 당 73,220 개의 서열이 얻어졌습니다. 이러한 서열은 Rhizobiaceae, Burkholderiaceae 및 Bacillaceae와 같은 핵심 C. elegans 장내 미생물군집13의 일부로 간주되는 패밀리를 포함하여 27개의 문과 216개 패밀리에 걸쳐 15,027개의 앰플리콘 서열 변이체(ASV)를 나타냅니다. 이전에 지배적 인 구성원으로 밝혀진 장내 세균과 슈도모나다과가 이번에는 소수 였지만 각각의 토양 환경에 비해 여전히 풍부했습니다 (2-10 배). 가중치가 적용되지 않은 UniFrac19,20 거리를 기반으로 한 비교는 가까운 클러스터링으로 표시된 바와 같이 동일한 농산물이 풍부한 소우주 삼중 중 사이에서 우수한 재현성을 보여주었습니다. 대조적으로, 서로 다른 농산물이 풍부한 환경 토양 미생물 군집은 서로 멀리 떨어져 모여 서로 다른 농산물을 추가하여 초기 미생물 군집을 다양 화할 수있는 능력을 보여줍니다 (그림 2).
웜 장내 마이크로바이옴과 환경 커뮤니티를 비교한 결과, 가중치가 적용되지 않거나 가중된 UniFrac 거리를 사용한 주요 좌표 분석(PCoA)은 각 소우주 유형에 대한 각 환경의 클러스터링에서 웜 장내 마이크로바이옴의 뚜렷한 클러스터링을 보여주었습니다(그림 2). 가중치가 적용되지 않은 UniFrac 거리를 기반으로 한 PCoA는 토양과 벌레 미생물군집을 구별하지 못했지만(그림 2A), 가중 거리를 기반으로 한 클러스터링은 벌레 장과 퇴비 미생물군집의 명확한 분리를 보여주었습니다(그림 2B). 이러한 결과는 숙주 필터링이 환경 가용성에 따라 작동하여 분류군의 존재와 관련하여 환경 소스와 완전히 구별되지는 않지만 사용 가능한 분류군의 하위 집합을 풍부하게하여 풍부함을 조절하여 궁극적으로 다른 환경에서 자란 웜간에 공유되는 핵심 웜 장내 미생물 군집을 형성하는 프로세스를 지원합니다.

그림 2: 각각의 농산물이 다양한 미생물 환경에서 멀어지는 벌레 장내 미생물군집. 마이크로바이옴 구성은 16S 시퀀싱으로 결정되었으며, 지정된 농산물이 풍부한 소우주 또는 그 안에서 자란 벌레의 군집은 (A) 비가중 또는 (B) 가중 UniFrac 거리를 기반으로 PCoA를 사용하여 클러스터링되었습니다. 표시된 축은 샘플 간의 군집 구성의 가장 큰 변화를 설명하는 축입니다(각 소우주 유형에 대해 N = 3). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1: 차세대 시퀀싱 및 데이터 분석. 다음은 라이브러리 준비, 실험실 내 시퀀싱 및 데이터 분석을 위한 단계입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S1: 한 샘플에서 역판독에 대한 품질 관리 그래프의 예. X축(cycle)은 판독된 서열을 따른 뉴클레오티드 위치를 나타낸다. 왼쪽 Y축은 품질평가점수를 보여줍니다. 그레이스케일 히트맵은 각 뉴클레오티드 위치에서 품질 점수의 빈도를 나타냅니다. 녹색 선은 각 뉴클레오티드 위치에서의 중간 품질 점수를 나타냅니다. 맨 위 주황색 선은 품질 평가 점수 분포의 사분위수를 나타냅니다. 하단 빨간색 선은 해당 뉴클레오티드 위치를 확장한 서열 판독의 백분율을 나타냅니다(오른쪽 Y축, 여기서는 100%). 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S2: 다양한 샘플에 대한 오류율. 다른 샘플(검은색 점)의 오류 빈도는 예상되는 추세를 반영하여 묘사된 각 가능한 염기쌍 대체에 대한 품질 점수가 증가함에 따라 감소해야 합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 S3: 가중 UniFrac 거리를 기반으로 한 PCoA의 예. 범례에 표시된 그룹 이름은 다른 소우주에서 사용되는 퇴비를 풍부하게하는 데 사용되는 농산물을 나타냅니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 S1: 순차 시퀀스 필터링. ᅡ 필터링하기 전에 읽은 시퀀스 수입니다. b-d 각 열은 낮은 품질의 읽기 필터링(2.5단계), dada()에 의해 수행된 노이즈 제거 알고리즘(2.8단계), 정방향 및 역방향 읽기 병합(2.9단계) 및 키메라 제거(2.11단계)와 같은 필터링 단계 후에 남은 시퀀스 읽기 수를 나타냅니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 S2: 메타데이터 표. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
여기에 제시된 프로토콜은 자연과 유사한 환경에서 자란 선충류의 장내 미생물 군집을 연구하는 방법을 설명하여 자연에서 벌레를 분리하거나 합성 공동체에서 키우는 대안적인 접근 방식을 제공합니다.
대표적인 소우주 실험에서 포착된 수천 종의 잠재적 박테리아는 웜이 진화한 미생물 다양성을 반영하고 모델 숙주 유기체로 작업하는 이점과 자연적이고 다양한 미생물 군집으로 작업하는 이점을 결합하는 소우주 파이프라인의 능력을 보여줍니다.
대표적인 결과는 다양한 농산물 유형으로 공통 토양을 풍부하게하는 것이 환경 미생물 다양성을 조절하고이 파이프 라인을 사용하여 탐사에 사용할 수있는 미생물 다양성의 범위를 강조한다는 것을 보여줍니다. 농산물 선택은 특별히 중요하지 않습니다. 이전 연구에서는 바나나, 사과, 오렌지, 딸기, 녹차 잎 및 감자를 사용하여 토양을 풍부하게하여 유사한 벌레 사육 효율성을 얻었습니다. 혼합 농산물도 효과적으로 사용되었습니다. 주요 특징은 다양한 농산물이 주어진 토양을 뚜렷한 방식으로 다양화한다는 것입니다.
환경 미생물 다양성의 광범위한 변화에도 불구하고, 웜 장내 미생물군집은 상당히 덜 다양하며, 토양 환경과 구별되는 핵심 장내 미생물군집의 조립을 위한 벌레 장내 틈새 및 숙주 필터링의 중요성을 되풀이합니다13. 이 패턴은 가중 UniFrac PCoA 분석에서 가장 잘 관찰되며, 환경 토양과 벌레 장내 미생물군집 간의 차이는 주로 주요 분류군의 상대적 풍부도의 차이에서 비롯된다는 것을 보여줍니다.
여기에 설명된 프로토콜은 16S 시퀀싱을 위해 웜을 수확하는 데 중점을 두고 있지만 소우주를 사용하여 관심 있는 추가 질문을 탐색할 수 있습니다. 예를 들어, 소우주에서 수확 한 벌레는 병원균이나 독소를 포함한 다양한 불리한 조건에 대한 숙주 저항성에 대한 다양한 장내 미생물 군집의 효과에 대해 조사 할 수 있습니다. 또는 새로운 박테리아 종 및 균주를 지상 수확 된 벌레에서 분리하고 배양하여 실험을 수행 할 수있는 박테리아의 분류 학적 및 기능적 다양성을 확장 할 수 있습니다.
실험실 환경에서의 연구 방법은 일관성과 재현성을 추구하지만, 소우주와의 작업은 자연적 변화를 활용하여 자연과 유사한 맥락에서 숙주-미생물 상호 작용을 탐구합니다. 그럼에도 불구하고 이러한 변형은 또한 몇 가지 문제를 제기합니다. 높은 수준의 내인성 무척추 동물을 포함하는 일부 토양은 소우주 준비에서 원하지 않는 유기체를 효과적으로 제거하기 위해 추가 원심 분리, 여과 및 검사 단계가 필요할 수 있습니다. 또한 토양의 미생물 풍부도가 낮 으면 벌레 개체군에서 바람직하지 않은 다우어 형성을 유도하여 미생물 추출물을 증가시키거나 더 많은 양의 농산물로 토양을 풍부하게 할 수 있습니다. 각 소우주 실험을 수행할 때마다 연구자들은 자연이 제공하는 완전한 분류학적 및 기능적 다양성을 계속 탐구하여 감염 저항성에서 환경 이생물에 대한 보호에 이르기까지 새로운 미생물 분류군과 기능적 능력을 발견할 수 있습니다.
공개
저자는 이 기사의 내용과 관련이 있다고 선언할 이해 상충이 없습니다.
감사의 말
이 원고에 설명 된 작업은 NIH 보조금 R01OD024780 및 R01AG061302의 지원을 받았습니다. KT는 로즈 힐스 재단이 자금을 지원하는 버클리 캘리포니아 대학의 여름 학부 연구 펠로우십의 지원을 받았습니다. 그림 1 의 만화 디자인은 BioRender.com 에서 얻은 것입니다.
자료
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
참고문헌
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유