A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
TACI: البرنامج المساعد ImageJ لتحليل تصوير الكالسيوم 3D
In This Article
Summary
TrackMate Analysis of Calcium Imaging (TACI) هو مكون إضافي ImageJ مفتوح المصدر لتحليل تصوير الكالسيوم 3D الذي يفحص الحركة على المحور z ويحدد القيمة القصوى لكل مكدس z لتمثيل شدة الخلية في النقطة الزمنية المقابلة. يمكنه فصل الخلايا العصبية المتداخلة في الاتجاه الجانبي (x / y) ولكن على مستويات z مختلفة.
Abstract
تطورت الأبحاث في علم الأعصاب لاستخدام أدوات التصوير والحوسبة المعقدة لاستخراج معلومات شاملة من مجموعات البيانات. تصوير الكالسيوم هو تقنية مستخدمة على نطاق واسع تتطلب برامج متطورة للحصول على نتائج موثوقة ، لكن العديد من المختبرات تكافح لاعتماد طرق حسابية عند تحديث البروتوكولات لتلبية المعايير الحديثة. تنشأ صعوبات بسبب نقص المعرفة البرمجية وجدران الدفع للبرمجيات. بالإضافة إلى ذلك ، تعرض الخلايا ذات الأهمية حركات في جميع الاتجاهات أثناء تصوير الكالسيوم. تم تطوير العديد من الأساليب لتصحيح الحركة في الاتجاه الجانبي (x / y).
تصف هذه الورقة سير عمل باستخدام مكون إضافي جديد ل ImageJ ، تحليل TrackMate لتصوير الكالسيوم (TACI) ، لفحص الحركة على المحور z في تصوير الكالسيوم 3D. يحدد هذا البرنامج الحد الأقصى لقيمة التألق من جميع المواضع z التي تظهر فيها الخلايا العصبية ويستخدمها لتمثيل شدة الخلايا العصبية في موضع t المقابل. لذلك ، يمكن لهذه الأداة فصل الخلايا العصبية المتداخلة في الاتجاه الجانبي (x / y) ولكنها تظهر على مستويات z متميزة. كمكون إضافي ل ImageJ ، يعد TACI أداة حسابية سهلة الاستخدام ومفتوحة المصدر لتحليل تصوير الكالسيوم 3D. لقد تحققنا من صحة سير العمل هذا باستخدام الخلايا العصبية الحساسة للحرارة ليرقات الذبابة التي عرضت الحركات في جميع الاتجاهات أثناء تقلب درجة الحرارة ومجموعة بيانات تصوير الكالسيوم 3D التي تم الحصول عليها من دماغ الذبابة.
Introduction
مستوى الكالسيوم داخل الخلايا هو علامة دقيقة على استثارة الخلايا العصبية. يقيس تصوير الكالسيوم التغيرات في الكالسيوم داخل الخلايا لفهم النشاط العصبي1. استخدمت الدراسات في علم الأعصاب هذه الطريقة بشكل متزايد بسبب تطوير تقنيات لقياس تركيز الكالسيوم داخل الخلايا ، بما في ذلك مؤشرات الكالسيوم المشفرة وراثيا (GECIs) ، مثل GCaMP 2,3 ، والتي يمكن التعبير عنها بشكل غير جراحي في مجموعات محددة من الخلايا العصبية من خلال الأساليب الجينية. كما أدى انخفاض تكاليف الليزر ومكونات المجهر إلى زيادة استخدام تصوير الكالسيوم4. الأهم من ذلك ، يسمح تصوير الكالسيوم بتسجيل ودراسة الخلايا العصبية المفردة وكذلك مجموعات الخلايا العصبية الكبيرة في وقت واحد في الحيوانات التي تتحرك بحرية5.
ومع ذلك ، فإن تحليل بيانات تصوير الكالسيوم يمثل تحديا لأنه (1) يتضمن تتبع التغيرات في مضان الخلايا الفردية بمرور الوقت ، (2) تختفي إشارة التألق بشكل متقطع أو تظهر مرة أخرى مع الاستجابات العصبية ، و (3) قد تتحرك الخلايا العصبية في جميع الاتجاهات ، وتحديدا داخل وخارج المستوى البؤري أو تظهر على مستويات متعددة4 ، 6. يستغرق التحليل اليدوي وقتا طويلا ويصبح غير عملي مع زيادة طول التسجيلات وعدد الخلايا العصبية. تم تطوير برامج مختلفة لتسريع عملية تحليل تصوير الكالسيوم. في السابق ، تم تصميم البرامج في سياق تجريبي محدود ، مما يجعل من الصعب على المختبرات الأخرى اعتمادها. أدت الجهود الأخيرة لتلبية المعايير الحديثة لمشاركة البرامج إلى تطوير العديد من الأدوات التي يمكنها تحليل بيانات تصوير الكالسيوم باستمرار عبر مجموعات مختلفة7،8،9،10،11،12،13،14،15،16،17،18،19 . ومع ذلك ، تتطلب معظم هذه الأدوات معرفة برمجية و / أو تعتمد على برامج تجارية. إن الافتقار إلى المعرفة البرمجية وجدران الدفع البرمجية يمنع الباحثين من تبني هذه الأساليب. علاوة على ذلك ، تركز العديد من هذه الأدوات على تصحيح حركة x / y ، على الرغم من أن الحركة على المحور z تحتاج أيضا إلى تشخيص وتصحيح صريح6. هناك حاجة لأداة حسابية لتحليل تصوير الكالسيوم 3D الذي يركز على الخلايا العصبية التي تظهر z-drift وتظهر على طائرات z متعددة. من الناحية المثالية ، يجب أن تستخدم هذه الأداة برامج مفتوحة المصدر ولا تتطلب معرفة برمجية للسماح للمختبرات الأخرى باعتمادها بسهولة.
هنا ، قمنا بتطوير مكون إضافي جديد ل ImageJ ، TACI ، لتحليل بيانات تصوير الكالسيوم 3D. أولا ، يقوم البرنامج بإعادة تسمية ، إذا لزم الأمر ، وينظم بيانات تصوير الكالسيوم 3D حسب مواضع z. يتم تتبع الخلايا ذات الاهتمام في كل موضع z ، ويتم استخراج شدة مضانها بواسطة TrackMate أو أدوات حسابية أخرى. ثم يتم تطبيق TACI لفحص الحركة على المحور z. يحدد القيمة القصوى لمكدس z ويستخدمه لتمثيل كثافة الخلية في النقطة الزمنية المقابلة. يناسب سير العمل هذا تحليل تصوير الكالسيوم 3D مع الحركة في جميع الاتجاهات و / أو مع الخلايا العصبية المتداخلة في الاتجاه الجانبي (x / y) ولكنها تظهر في مواقع z مختلفة. للتحقق من صحة سير العمل هذا ، تم استخدام مجموعات بيانات تصوير الكالسيوم 3D من الخلايا العصبية الحساسة للحرارة ليرقات الذباب والخلايا العصبية للفطر في الدماغ. تجدر الإشارة إلى أن TACI هو مكون إضافي ImageJ مفتوح المصدر ولا يتطلب أي معرفة برمجية.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. تصوير الكالسيوم
- إعداد يرقات الذبابة
ملاحظة: يتم الحفاظ على الذباب واليرقات عند 25 درجة مئوية تحت دورة 12 ساعة: 12 ساعة: مظلمة.- تخدير الذباب مع CO2. فرز 20-45 من الذكور و 20-45 من الإناث في كل قارورة ذبابة ، ومنحهم ما لا يقل عن 24 ساعة إلى 48 ساعة للتعافي من التعرض CO2 .
ملاحظة: يجب أن يستمر التعرض للذبابة لثاني أكسيد الكربون2 لأقصر فترة زمنية ممكنة. - لمزامنة عمر اليرقات ، اضغط على الذباب في قوارير جديدة تحتوي على حبيبات الخميرة ، واسمح لها ب 4-8 ساعات لوضع البيض. قم بإزالة الذباب عن طريق قلبها في قوارير جديدة.
- اجمع اليرقات في 72 ساعة باستخدام 10 مل من محلول السكروز 20٪ وزن.
- تخدير الذباب مع CO2. فرز 20-45 من الذكور و 20-45 من الإناث في كل قارورة ذبابة ، ومنحهم ما لا يقل عن 24 ساعة إلى 48 ساعة للتعافي من التعرض CO2 .
- إعداد المجهر والتحكم في درجة الحرارة
- قم بإجراء التصوير على مجهر متحد البؤر (انظر جدول المواد) ومرحلة بيزو ذات محور z مع إدراج مرحلة باستخدام الإعدادات التالية: الليزر ، الأرجون. وضع المسح ، الإطار ؛ حجم الإطار ، 512 × 512 ؛ السرعة ، الحد الأقصى ؛ عمق القنوات / بت ، 1/8 بت ؛ التكبير ، 1.5 ؛ z-stack ، شريحة = 15 ، احتفظ = شريحة ؛ أجهزة التركيز والاستراتيجية ، التركيز المحدد ؛ التركيز، تركيز واضح على.
- قم بتوصيل وحدة تبريد بلتيير بالمشتت الحراري بواسطة مركب نقل الحرارة لبناء مبرد كهربائي حراري. يتم تشغيل بلتيير بواسطة مصدر طاقة 2 أمبير.
- قم بتوصيل مسبار دقيق حراري بجهاز الحصول على البيانات لتسجيل درجة الحرارة.
- تصوير الكالسيوم
- شطف اليرقات 3x في 1x محلول ملحي مخزن بالفوسفات (PBS).
- ماصة 75 ميكرولتر من 1x PBS على وسط شريحة زجاجية.
- ضع يرقة واحدة أو اثنتين في 1x PBS وضع المسبار الدقيق الحراري بالقرب من اليرقات.
ملاحظة: يجب أن تكون المسافة بين اليرقات والمسبار الدقيق ~ 5 مم. قد تتحرك اليرقات إذا تم وضع المسبار الدقيق بالقرب منها. قد تؤدي المسافة الكبيرة إلى قراءات غير دقيقة لدرجة الحرارة. - تغطية اليرقات وmicroprobe الحرارية مع غطاء زجاجي. أغلق الغطاء بطلاء الأظافر.
- ضع الشريحة على مرحلة المجهر ، وابحث عن التركيز باستخدام هدف 25x ، وضع المبرد الكهروحراري على الشريحة.
ملاحظة: يتم وضع بلتيير مباشرة على الشريحة حيث تقوم اليرقات بتوصيل محفزات درجة الحرارة. - في البرامج البؤرية ، ركز على خلايا الفلورسنت ذات الأهمية. اضبط طاقة الليزر لتجنب التشبع الزائد. قم بتعيين مواضع الشريحة الأولى والأخيرة في إعداد z-stack.
ملاحظة: استخدم أقل طاقة ليزر ممكنة قبل التسجيل لمنع التبييض الضوئي. - ابدأ مسح z-stack وتسجيل درجة الحرارة في نفس الوقت.
- تحكم في مصدر الطاقة الذي يشغل بلتيير لتغيير درجة حرارة سطح بلتيير. قم بتشغيل مصدر الطاقة لخفض درجة الحرارة وإيقاف تشغيله لزيادة درجة الحرارة.
- أوقف مسح z-stack وتسجيل درجة الحرارة.
2. تحليل بيانات تصوير الكالسيوم 3D
- قم بتصدير بيانات تصوير الكالسيوم إلى ملفات TIFF واحفظها في مجلد بنفس اسم الاسم الأساسي لملفات TIFF بالداخل.
ملاحظة: يجب ألا يحتوي اسم الملف على أية فواصل.- استخدم المعلمات التالية لتصدير بيانات تصوير الكالسيوم من ZEN (الإصدار الأسود): نوع الملف ، TIFF ؛ ضغط ، LZW ؛ القنوات ، 2 ؛ Z- الموقف ، كل شيء ؛ الوقت ، كل شيء ؛ المرحلة ، 1 ؛ المنطقة ، كاملة.
- تركيب
- قم بتنزيل TACI-Calcium_Imaging.jar من جيثب (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- قم بتثبيت المكون الإضافي في FIJI بالنقر فوق Plugins في شريط القائمة ثم النقر فوق تثبيت في القائمة المنسدلة (Plugins | تثبيت). ثم أعد تشغيل فيجي.
ملاحظة: لا تستخدم الإضافات | تثبيت البرنامج المساعد. - قم بتشغيل المكون الإضافي بالنقر فوق المكونات الإضافية ثم اختيار TACI-Calcium Imaging (Plugins | TACI-تصوير الكالسيوم).
- استخدم وظيفة RENAME لتحويل أسماء ملفات TIFF إلى البنية المطلوبة.
ملاحظة: تستخدم الأداة filename_h#t#z#c#.tif كبنية افتراضية (h# وc# اختيارية؛ #: عدد صحيح موجب). إذا لم تكن أسماء ملفات الصور في البنية الافتراضية ، فيجب تنفيذ الدالة RENAME .- اختر المجلد الذي تحتاج صور TIFF إلى إعادة تسميته بالنقر فوق استعراض المجلدات.
- املأ معلومات المعلمة. يتم سرد خمسة معلمات ، بما في ذلك اسم الملف ، والمرحلة ، والحد الأقصى لموضع T ، وموضع Max Z ، والقناة. تحتوي كل معلمة على ثلاث قيم: النص السابق وقيمة المعلمة والترتيب.
ملاحظة: المعلومات حساسة لحالة الأحرف. إذا كانت أسماء ملفات الصور تحتوي على عبارة قبل معلمة، فقم بتعبئة العبارة كنص سابق للمعلمة المناظرة. إذا كانت أسماء ملفات الصور تحتوي على عبارة بعد كل المعلمات، فقم بتعبئة العبارة كنص مشاركة.- تأكد من إدخال اسم الملف و Max T-Position و Max Z-Position وأن قيم المعلمات ل Max T-Position و Max Z-Position تتضمن جميع الأرقام.
- انتظر حتى يتم ملء اسم الملف تلقائيا باستخدام اسم المجلد.
- إذا كانت أسماء ملفات TIFF لا تتضمن المرحلة والقناة، اترك قيمة المعلمة المقابلة فارغة، واختر Na بالترتيب.
- انقر فوق إعادة تسمية لإنشاء مجلد بنفس الاسم ونفس _r. لاحظ أنه في المجلد ، تمت إعادة هيكلة أسماء ملفات TIFF لتكون متوافقة مع وظيفة ORGAN .
ملاحظة: تمت إضافة _r إلى أسماء ملفات صور TIFF.
- استخدم وظيفة ORGANIZE لحفظ صور TIFF من نفس موضع z في مجلد واحد.
ملاحظة: يجب أن يكون اسم المجلد نفس اسم الاسم الأساسي لملفات TIFF بالداخل ويجب ألا يحتوي على أي فواصل.- اختر المجلد الذي تحتاج صور TIFF إلى التنظيم فيه بالنقر فوق استعراض المجلدات.
- إذا كان ملف المعلمة CSV (param.csv) موجودا ، فانتظر حتى يتم ملء قيم المعلمات تلقائيا.
ملاحظة: يحتوي ملف المعلمة.csv على تنسيق مطلوب. يجب ملء المعلمات ، بما في ذلك اسم الملف والمرحلة position_t position_z والقناة is_gray ، في الصف 1 من اليسار إلى اليمين بدءا من العمود A. يجب ملء القيم المقابلة لكل معلمة في الصف 2. - إذا كان ملف المعلمة CSV (param.csv) غير موجود ، فقم بتعبئة قيم المعلمات يدويا. تأكد من أن قيم المعلمات الخاصة بالطور والقناة تتضمن أحرفا ، بينما يجب أن تكون قيم المعلمات لموضع T وموضع Z أكبر عدد من المواضع t و z. إذا كانت أسماء ملفات الصور لا تتضمن المرحلة أو القناة، قم بإدخال Na.
- قم بإنشاء صور TIFF بتدرج الرمادي عند الحاجة. اترك مربع هل الصور رمادية؟ غير محدد لتدرج الصور باللون الرمادي.
- انقر فوق تنظيم لإنشاء مجلد بنفس الاسم ونفس _gray_stacks ولإنشاء مجلدات بنفس الاسم و _# (#: z-position) في المجلد. لاحظ أن ملفات TIFF يتم فرزها في مجلدات مقابلة حسب مواضع z وأنه يتم إنشاء ملف باسم param.csv ، حيث يمكن العثور على المعلمات وقيمها.
- استخدم TrackMate في فيجي لاستخراج شدة التألق للخلايا ذات الأهمية من كل موضع z.
ملاحظة: يمكن أيضا تنفيذ هذه الخطوة بواسطة برامج تصوير أخرى.- افتح صور TIFF في مجلد z-position بواسطة فيجي.
- قم بتشغيل TrackMate بالنقر فوق المكونات الإضافية | التتبع | TrackMate ، واضبط المعلمات التالية إذا لزم الأمر.
- استخدم كاشفات DoG أو LoG.
- قم بتغيير قطر النقطة والعتبة وعامل التصفية المتوسط. اضبط قطر النقطة ليكون مشابها لقطر الخلايا. إذا كانت الخلايا بيضاوية ، فاضبط قطر النقطة ليكون مشابها للمحور الثانوي.
ملاحظة: تساعد زيادة العتبة على تجنب التقاط ضوضاء الخلفية كإشارات دون التأثير على شدة الإشارة (الشكل التكميلي 1). ومع ذلك، فإن الزيادة في العتبة قد تفوت الإشارات الحقيقية (الشكل التكميلي 1). إذا كانت الإشارات قوية ، فاستخدم المرشح المتوسط لتقليل ضوضاء الملح والفلفل. - اضبط المرشحات لإزالة بعض الإشارات غير ذات الصلة ، إن لم يكن كلها. المرشحات X و Y هي مرشحات مكانية. استخدم المرشحات X و Y لإزالة الإشارات غير ذات الصلة البعيدة عن الإشارات الحقيقية.
ملاحظة: عند تعيين المرشحات على صورة واحدة ، من الضروري التحقق من جميع الصور الأخرى لضمان عدم إزالة الإشارات الحقيقية. - اضبط المسافة القصوى للربط، والمسافة القصوى لإغلاق الفجوة، وفجوة الإطار القصوى لإغلاق الفجوة. اضبط المسافة القصوى للربط والمسافة القصوى لإغلاق الفجوة لتكون 3x-5x قطر النقطة ، خاصة عندما تتحرك العينات بشكل كبير بمرور الوقت ، للمساعدة في تقليل عدد المسارات. اضبط فجوة الإطار الأقصى لإغلاق الفجوة على عدد الصور في الرصة.
- تصدير شدة التألق للمناطق ذات الأهمية (ROIs) إلى ملف CSV. إذا تم استخدام إصدار TrackMate قديم ، فاختر تصدير جميع إحصائيات النقاط في نافذة تحديد إجراء . إذا كان إصدار TrackMate هو 7.6.1 أو أعلى ، فاختر البقع في نافذة خيارات العرض . تصدير أو حفظ كملفات تفاعلية إلى ملفات CSV.
ملاحظة: كلا الملفين تفاعليان مع نافذة الصورة ؛ يؤدي تمييز عائد الاستثمار إلى عرض عائد الاستثمار المقابل في نافذة الصورة. تتضمن هذه الملفات متوسط الكثافة (MEAN_INTENSITY أو MEAN_INTENSITY_CH1) للخلايا ذات الأهمية في النقاط الزمنية المقابلة (POSITION_T). يجب أن يمثل نفس TRACK_IDs نفس عائد الاستثمار في نقاط زمنية مختلفة. ومع ذلك ، هذا ليس صحيحا دائما وقد يحتاج إلى تصحيح يدويا ، عند الضرورة. إذا لم يتعرف TrackMate على عائد الاستثمار في بعض النقاط الزمنية ، فلن يتم عرض هذه النقاط الزمنية.
- استخراج كثافة مضان الخلفية، وإنشاء ملف Background_list.csv.
ملاحظة: في هذه الدراسة ، تم تقدير شدة الخلفية لكل موضع z باستخدام متوسط قيمة ثلاث إلى خمس نقاط متجاورة من نفس الحجم لا تحتوي على إشارات مضان وكانت من نقاط زمنية مختلفة.- يحتوي ملف Background_list.csv على التنسيق المطلوب: يحتوي كل عمود على معلومات خلية عصبية واحدة ، بدءا من Neuron 0. املأ أرقام الخلايا العصبية ، مثل Neuron 0 ، في الصف 1. بعد ذلك ، قم بتوفير كثافة الخلفية لكل موضع z تم تحليله - إذا تم تحليل خمسة مواضع z للخلية العصبية 0 ، فاملأ خمس قيم لشدة الخلفية أسفل Neuron 0.
ملاحظة: ملف Background_list.csv مطلوب لوظيفة TACI EXTRACT . إذا كانت الخلفية لا تذكر ، فيجب توفير Background_list.csv ذات كثافة الخلفية الصفرية لكل خلية عصبية.
- يحتوي ملف Background_list.csv على التنسيق المطلوب: يحتوي كل عمود على معلومات خلية عصبية واحدة ، بدءا من Neuron 0. املأ أرقام الخلايا العصبية ، مثل Neuron 0 ، في الصف 1. بعد ذلك ، قم بتوفير كثافة الخلفية لكل موضع z تم تحليله - إذا تم تحليل خمسة مواضع z للخلية العصبية 0 ، فاملأ خمس قيم لشدة الخلفية أسفل Neuron 0.
- استخدم الدالة EXTRACT لتحديد شدة التألق القصوى عند كل موضع t وحساب ΔF / F0 كما هو موضح في المعادلة (1).
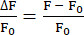 (1)
(1)- قم بإنشاء مجلد وقم بتسميته باستخدام رقم الخلية العصبية ، بدءا من Neuron 0. احفظ ملفات CSV التي تحتوي على معلومات مضان للخلية العصبية المقابلة في المجلد. يحتوي كل ملف CSV على معلومات موضع z واحد ، وبالتالي فإن عدد ملفات CSV يساوي عدد مواضع z التي تم تحليلها. قم بتسمية ملفات CSV ك Mean_Intensity# .csv (# يمثل موضع z) ويتضمن كل ملف CSV عمودين على الأقل: POSITION_T و MEAN_INTENSITY أو MEAN_INTENSITY_CH1.
ملاحظة: إنشاء مجلد لكل خلية مهمة. - احفظ ملف Background_list.csv والمجلدات التي تم إنشاؤها في الخطوة 2.7.1 في مجلد واحد. اختر هذا المجلد بالنقر فوق تصفح الملفات.
ملاحظة: يجب أن يتطابق عدد قيم الخلفية في ملف Background_list.csv مع عدد ملفات Mean_Intensity#.csv لكل خلية عصبية. - يتم ملء ملف الخلفية تلقائيا. املأ أكبر عدد من مواضع t لعدد مواضع T.
- انقر فوق استخراج لإنشاء مجلد نتائج، بما في ذلك ملفات CSV والمؤامرات لكل خلية عصبية. تتضمن ملفات CSV معلومات عن أقصى كثافة مضان و ΔF / F0 في كل موضع t. المخططات عبارة عن مخططات خطية ل ΔF / F0 فوق مواضع t.
ملاحظة: في هذه الدراسة، تم حساب ΔF/F0 باستخدام المعادلة (1). تم استخدام القيمة الأولى لكل موضع z ك F0. إذا كان F0 غير مناسب20 ، فإن المكون الإضافي يوفر ملفات بما في ذلك البيانات الأولية لكل خلية عصبية في مجلد python_files.
- قم بإنشاء مجلد وقم بتسميته باستخدام رقم الخلية العصبية ، بدءا من Neuron 0. احفظ ملفات CSV التي تحتوي على معلومات مضان للخلية العصبية المقابلة في المجلد. يحتوي كل ملف CSV على معلومات موضع z واحد ، وبالتالي فإن عدد ملفات CSV يساوي عدد مواضع z التي تم تحليلها. قم بتسمية ملفات CSV ك Mean_Intensity# .csv (# يمثل موضع z) ويتضمن كل ملف CSV عمودين على الأقل: POSITION_T و MEAN_INTENSITY أو MEAN_INTENSITY_CH1.
- استخدم دالة MERGE لمتوسط ΔF / F0 لكل خلية عصبية ، وحساب SEM ، ورسم متوسط ΔF / F0 على مواضع t.
- اختر مجلد النتائج الذي تم إنشاؤه بواسطة وظيفة EXTRACT بالنقر فوق تصفح الملفات.
- املأ عدد مواضع T بأكبر عدد من مواضع t.
- انقر فوق دمج لإنشاء مجلد merged_data، بما في ذلك ملف merged_data.csv ومخطط Average_dF_F0.png. يتضمن ملف CSV معلومات عن المتوسط و SEM ΔF / F0 في كل موضع t. الرسم عبارة عن مخطط خطي لمتوسط ΔF / F0 على مواضع t.
Access restricted. Please log in or start a trial to view this content.
النتائج
سير عمل تحليل تصوير الكالسيوم 3D
في هذه الدراسة ، قمنا بتطوير مكون إضافي جديد ل ImageJ ، TACI ، ووصفنا سير عمل لتتبع z-drift وتحليل تصوير الكالسيوم 3D الذي يحدد استجابات الخلايا الفردية التي تظهر في مواضع z متعددة (الشكل 1). تحتوي هذه الأداة على أربع وظائف: إعادة تسمية
Access restricted. Please log in or start a trial to view this content.
Discussion
طورت هذه الدراسة مكونا إضافيا جديدا ل ImageJ ، TACI ، ووصفت سير عمل لتحليل تصوير الكالسيوم 3D. تركز العديد من الأدوات المتاحة حاليا على تصحيح حركة x / y ، على الرغم من أن الحركة على المحور z تحتاج أيضا إلى تشخيص أو تصحيح صريح6. أثناء الحصول على الصورة في كائن حي ، لا يمكن تجنب الحركة على ا...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح للكشف عنه.
Acknowledgements
تم استخدام Zeiss LSM 880 في مركز التصوير Fralin لجمع بيانات تصوير الكالسيوم. نحن نقدر الدكتورة ميشيل إل أولسن ويوهانغ بان لمساعدتهما في برنامج IMARIS. نعترف بالدكتور لينوود س. هيث لتعليقاته البناءة على المخطوطة وستيفن جيافاسيس لتعليقاته على ملف GitHub README. تم دعم هذا العمل من قبل NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) و NIH R01GM140130 (https://www.nigms.nih.gov/) إلى L.N. لم يكن للممولين أي دور في تصميم الدراسة أو جمع البيانات وتحليلها أو قرار النشر أو إعداد المخطوطة.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
References
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258(2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517(2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173(2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153(2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432(2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569(2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728(2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869(2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279(2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254(2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252(2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved