このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
TACI: 3D カルシウムイメージング解析のための ImageJ プラグイン
要約
TrackMate カルシウムイメージング分析(TACI)は、z軸の動きを調べ、各zスタックの最大値を特定して、対応する時点での細胞の強度を表す、3Dカルシウムイメージング分析用のオープンソースのImageJプラグインです。横方向(x / y)方向に重なり合うが、異なるz平面上にあるニューロンを分離できます。
要約
神経科学の研究は、データセットから包括的な情報を抽出するために複雑なイメージングおよび計算ツールを使用するように進化しました。カルシウムイメージングは、信頼性の高い結果を得るために高度なソフトウェアを必要とする広く使用されている技術ですが、多くのラボは、最新の基準を満たすようにプロトコルを更新する際に計算方法を採用するのに苦労しています。プログラミングの知識とソフトウェアのペイウォールが不足しているために問題が発生します。さらに、目的の細胞は、カルシウムイメージング中にあらゆる方向の動きを示します。横方向(x / y)方向の動きを補正するために、多くのアプローチが開発されています。
この論文では、新しいImageJプラグインであるTrackMate Analysis of Calcium Imaging (TACI)を使用して、3Dカルシウムイメージングのz軸上の動きを調べるワークフローについて説明します。このソフトウェアは、ニューロンが現れるすべてのz位置から最大蛍光値を識別し、それを使用して対応するt位置でのニューロンの強度を表します。したがって、このツールは、横方向(x / y)方向に重なり合っているが、異なるz平面に現れるニューロンを分離できます。ImageJプラグインとして、TACIは3Dカルシウムイメージング分析のためのユーザーフレンドリーなオープンソースの計算ツールです。このワークフローは、温度変動時にあらゆる方向の動きを表示するハエの幼虫の熱感受性ニューロンと、ハエの脳から取得した3Dカルシウムイメージングデータセットを使用して検証しました。
概要
細胞内カルシウムのレベルは、ニューロンの興奮性の正確なマーカーです。カルシウムイメージングは、細胞内カルシウムの変化を測定して神経活動を理解する1。神経科学の研究は、遺伝的アプローチを通じて特定のニューロンセットで非侵襲的に発現することができるGCaMP 2,3などの遺伝的にコードされたカルシウム指示薬(GECI)を含む細胞内カルシウム濃度を測定するための技術の開発により、この方法をますます使用しています。レーザーと顕微鏡コンポーネントの低コスト化により、カルシウムイメージングの使用も増加しています4。重要なことに、カルシウムイメージングは、自由に動く動物において、単一のニューロンと大きなニューロン集団を同時に記録および研究することを可能にする5。
それにもかかわらず、カルシウム画像化データの分析は、(1)個々の細胞の蛍光の経時的な変化を追跡することを含み、(2)蛍光シグナルが断続的に消失するか、またはニューロン応答とともに再び現れること、および(3)ニューロンがあらゆる方向、特に焦点面に出入りするか、または複数の平面上に現れることがあるため、困難である4、6.手動分析は時間がかかり、記録の長さとニューロンの数が増えるにつれて実用的ではなくなります。カルシウムイメージングの分析プロセスを加速するために、さまざまなソフトウェアプログラムが開発されています。以前は、ソフトウェアは限られた実験コンテキストで設計されていたため、他の研究室がそれを採用することは困難でした。ソフトウェア共有に関する最新の基準を満たすための最近の取り組みにより、異なるグループ間でカルシウムイメージングデータを一貫して分析できるいくつかのツールが開発されました7,8,9,10,11,12,13,14,15,16,17,18,19 .ただし、これらのツールのほとんどはプログラミングの知識を必要とするか、商用ソフトウェアに依存しています。プログラミングの知識とソフトウェアのペイウォールが不足しているため、研究者はこれらの方法を採用することを思いとどまらせています。さらに、これらのツールの多くはx/yモーションの補正に重点を置いていますが、z軸の動きも明示的に診断して修正する必要があります6。zドリフトを示し、複数のz平面に現れるニューロンに焦点を当てた3Dカルシウムイメージングを分析するための計算ツールが必要です。理想的には、このツールはオープンソースソフトウェアを使用し、他のラボが簡単に採用できるようにプログラミングの知識を必要としない必要があります。
ここでは、3Dカルシウムイメージングデータを解析するための新しいImageJプラグインTACIを開発しました。まず、ソフトウェアは必要に応じて名前を変更し、3Dカルシウムイメージングデータをz位置ごとに整理します。目的の細胞は各z位置で追跡され、その蛍光強度はTrackMateまたは他の計算ツールによって抽出されます。次に、TACIを適用してz軸上の動きを調べます。Z スタックの最大値を識別し、それを使用して、対応する時点でのセルの強度を表します。このワークフローは、すべての方向の動きや、横方向(x / y)方向に重なり合っているが異なるz位置に現れるニューロンを持つ3Dカルシウムイメージングの分析に適しています。このワークフローを検証するために、ハエの幼虫の熱感受性ニューロンと脳内のキノコニューロンからの3Dカルシウムイメージングデータセットを使用しました。注目すべきことに、TACIはオープンソースのImageJプラグインであり、プログラミングの知識は必要ありません。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. カルシウムイメージング
- ハエ幼虫の準備
注:ハエと幼虫は、12時間:12時間の明暗サイクルの下で25°Cに維持されます。- ハエをCO2で麻酔する。20〜45人の男性と20〜45人の女性を各フライバイアルに分類し、CO2 曝露から回復するために少なくとも24時間〜48時間与えます。
注:CO2 へのハエの暴露は、可能な限り短い時間続く必要があります。 - 幼虫の年齢を同期させるには、ハエを酵母顆粒を含む新しいバイアルにタップし、4〜8時間産卵させます。ハエを新しいバイアルに入れて取り除きます。
- 10 mLの20%w / vショ糖溶液を使用して72時間で幼虫を収集します。
- ハエをCO2で麻酔する。20〜45人の男性と20〜45人の女性を各フライバイアルに分類し、CO2 曝露から回復するために少なくとも24時間〜48時間与えます。
- 顕微鏡と温度制御のセットアップ
- 次の設定を使用して、共焦点顕微鏡( 材料表を参照)とステージインサート付きのz軸ピエゾステージでイメージングを実行します: レーザー、アルゴン; スキャンモード、フレーム; フレームサイズ、512 x 512、速度、最大、チャンネル/ビット深度、1/8ビット、ズーム、1.5、zスタック、スライス=15、保持=スライス、フォーカスデバイスと戦略、明確なフォーカス、フォーカス、 明確な焦点。
- ペルチェ冷却モジュールを熱伝達コンパウンドによってヒートシンクに取り付けて、熱電冷却器を構築します。ペルチェは2Aの電源で駆動されます。
- 熱電対マイクロプローブをデータ収集デバイスに取り付けて、温度を記録します。
- カルシウムイメージング
- 幼虫を1xリン酸緩衝生理食塩水(PBS)で3回すすぎます。
- 75 μLの1x PBSをスライドガラスの中央にピペットで貼り付けます。
- 1匹または2匹の幼虫を1x PBSに入れ、熱電対マイクロプローブを幼虫の近くに置きます。
注:幼虫とマイクロプローブの間の距離は~5mmでなければなりません。マイクロプローブがそれらに近すぎる位置にある場合、幼虫は動くかもしれません。距離が長いと、温度測定値が不正確になる可能性があります。 - 幼虫と熱電対のマイクロプローブをガラスカバーガラスで覆います。カバーガラスをマニキュアで密封します。
- スライドを顕微鏡ステージに置き、25倍の対物レンズを使用して焦点を見つけ、熱電冷却器をスライドに配置します。
注:ペルチェは、幼虫が温度刺激を届けるスライドに直接配置されます。 - 共焦点ソフトウェアでは、目的の蛍光細胞に焦点を合わせます。過飽和を避けるためにレーザー出力を調整します。Z スタック設定で最初と最後のスライス位置を設定します。
注意: 光退色を防ぐために、記録する前に可能な限り低いレーザー出力を使用してください。 - Zスタックスキャンと温度記録を同時に開始します。
- ペルチェに電力を供給する電源を制御して、ペルチェ表面温度を変更します。電源をオンにして温度を下げ、電源をオフにして温度を上げます。
- Zスタックスキャンと温度記録を停止します。
2. 3次元カルシウムイメージングデータの解析
- カルシウムイメージングデータをTIFFファイルにエクスポートし、内部のTIFFファイルのベース名と同じ名前のフォルダーに保存します。
注: ファイル名に カンマを含めることはできません。- ZEN(ブラックエディション)からカルシウムイメージングデータをエクスポートするには、次のパラメータを使用します: ファイルタイプ、TIFF; 圧縮、LZW、チャンネル、2;Zポジション、すべて。時間、すべて。フェーズ、1;地域、いっぱい。
- 取り付け
- Github (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases) から TACI-Calcium_Imaging.jar をダウンロードします。
- メニューバーのプラグインをクリックし、ドロップダウンメニューの[インストール]をクリックして、FIJIにプラグインをインストールします(プラグイン|インストール)。その後、FIJIを再起動します。
注:プラグインを使用しないでください|プラグインをインストールします。 - プラグインをクリックし、TACI-カルシウムイメージングを選択してプラグインを実行します(プラグイン|TACI-カルシウムイメージング)。
- RENAME 関数を使用して、TIFF ファイル名を必要な構造に変換します。
注: このツールは、既定の構造として filename_h#t#z#c#.tif を使用します (h# と c# は省略可能で、#: 正の整数)。イメージ ファイル名が既定の構造にない場合は、 RENAME 関数を実行する必要があります。- TIFF イメージの名前を変更する必要があるフォルダを選択するには、[ フォルダの参照] をクリックします。
- パラメータ情報を入力します。ファイル名、フェーズ、最大T位置、最大Z位置、チャネルを含む5つのパラメータがリストされています。各パラメーターには、[先行するテキスト]、[パラメーター値]、および [順序] の 3 つの値があります。
注: 情報では大文字と小文字が区別されます。画像ファイル名にパラメータの前にフレーズが含まれている場合は、対応するパラメータの先行テキストとしてフレーズを入力します。画像ファイル名にすべてのパラメータの後にフレーズが含まれている場合は、そのフレーズを投稿テキストとして入力します。- ファイル名、最大T位置、最大Z位置を入力し、最大T位置と最大Z位置のパラメータ値にすべての桁が含まれていることを確認してください。
- ファイル名がフォルダ名を使用して自動的に入力されるのを待ちます。
- TIFF ファイル名にフェーズとチャンネルが含まれていない場合は、対応するパラメータ値を空白のままにして、「順番に Na」を選択します。
- [ 名前の変更 ] をクリックして、同じ名前と_rのフォルダーを作成します。フォルダー内の TIFF ファイル名が 、ORGANIZE 関数と互換性があるように再構築されていることに注意してください。
注: TIFF イメージのファイル名に_rが追加されました。
- ORGANIZE 関数を使用して、同じ Z 位置の TIFF イメージを 1 つのフォルダーに保存します。
注: フォルダ名は、内部の TIFF ファイルの基本名と同じ名前で、コンマは使用できません。- TIFF イメージを整理する必要があるフォルダを選択するには、[ フォルダの参照] をクリックします。
- パラメーター CSV ファイル (param.csv) が存在する場合は、パラメーター値が自動的に入力されるのを待ちます。
注: param.csv ファイルには必要な形式があります。ファイル名、フェーズ、position_t、position_z、チャネル、is_grayなどのパラメーターは、列 A から左から右に行 1 に入力する必要があります。各パラメーターに対応する値を行 2 に入力する必要があります。 - パラメーター CSV ファイル (param.csv) が存在しない場合は、パラメーター値を手動で入力します。[位相]と[チャネル]のパラメータ値に文字が含まれ、[T 位置]と[Z 位置]のパラメータ値が T 位置と Z 位置の最大数であることを確認します。イメージ ファイル名にフェーズまたはチャネルが含まれていない場合は、Na と入力します。
- 必要に応じてグレースケールの TIFF 画像を作成します。[ 画像は灰色ですか? ] のボックスをオフのままにしておくと、画像がグレースケールされます。
- [ 整理 ] をクリックして、同じ名前と_gray_stacksのフォルダーを作成し、フォルダー内に同じ名前と _# (#: z 位置) のフォルダーを生成します。TIFF ファイルが z 位置で対応するフォルダーに並べ替えられ、パラメーターとその値が見つかる param.csv という名前のファイルが生成されることを確認します。
- フィジーのTrackMateを使用して、各z位置から目的の細胞の蛍光強度を抽出します。
注意: この手順は、他のイメージングソフトウェアでも実行できます。- FIJIのz位置フォルダーにあるTIFF画像を開きます。
- プラグインをクリックしてトラックメイトを実行する |トラッキング |TrackMateをクリックし、必要に応じて次のパラメータを調整します。
- DoGまたはLoG検出器を使用してください。
- BLOB の直径、しきい値、および中央値フィルターを変更します。ブロブの直径をセルの直径に似るように調整します。セルが楕円形の場合は、短軸に似たブロブの直径を調整します。
メモ: しきい値 を大きくすると、信号強度に影響を与えずにバックグラウンドノイズが信号として拾われるのを防ぐことができます(補足図1)。ただし、 しきい値 を上げると、真の信号を見逃す可能性があります(補足図1)。信号が強い場合は、 メディアンフィルター を使用して塩とコショウのノイズを減らします。 - フィルターを設定して、すべてではないにしても、無関係な信号の一部を削除します。 フィルター X と Y は空間フィルターです。 フィルターX と Y を使用して、実際の信号から離れた無関係な信号を削除します。
注意: 1つの画像にフィルターが設定されている場合は、他のすべての画像をチェックして、実際の信号が削除されていないことを確認することが重要です。 - リンクの最大距離、ギャップを閉じる最大距離、およびギャップを閉じる最大フレームギャップを設定します。リンクの最大距離とギャップを閉じる最大距離をブロブ直径の3倍から5倍に設定し、特にサンプルが時間の経過とともに大幅に移動する場合は、トラックの数を減らすのに役立ちます。ギャップを閉じる最大フレームギャップをスタック内の画像数に設定します。
- 関心領域(ROI)の蛍光強度をCSVファイルにエクスポートします。古いバージョンの TrackMate を使用している場合は、[アクションの選択] ウィンドウで [すべてのスポット統計をエクスポート] を選択します。TrackMate バージョンが 7.6.1 以降の場合は、[表示オプション] ウィンドウで [スポット] を選択します。インタラクティブファイルとしてCSVファイルにエクスポートまたは保存します。
注:どちらのファイルもイメージウィンドウとインタラクティブです。ROI を強調表示すると、対応する ROI が画像ウィンドウに表示されます。これらのファイルには、対応する時点 (POSITION_T) における対象のセルの平均強度 (MEAN_INTENSITY または MEAN_INTENSITY_CH1) が含まれています。同じTRACK_IDsは、異なる時点で同じROIを表す必要があります。ただし、これは常に当てはまるとは限らず、必要に応じて手動で修正する必要がある場合があります。TrackMateが一部の時点でROIを認識しない場合、それらの時点は表示されません。
- バックグラウンド蛍光強度を抽出し、Background_list.csvファイルを作成します。
注:この研究では、各z位置のバックグラウンド強度は、蛍光シグナルを含まず、異なる時点からの3〜5つの隣接する同じサイズのブロブの平均値を使用して推定されました。- Background_list.csv ファイルには必要な形式があり、各列には Neuron 0 から始まる 1 つのニューロンの情報が含まれています。ニューロン 0 などのニューロン番号を行 1 に入力します。そして、分析された各z位置についてバックグラウンド強度を提供し、ニューロン0について5つのz位置が分析された場合、ニューロン0の下に5つのバックグラウンド強度値を入力する。
注: Background_list.csv ファイルは、TACI 抽出 機能に必要です。バックグラウンドが無視できる場合は、すべてのニューロンのバックグラウンド強度がゼロのBackground_list.csvを提供する必要があります。
- Background_list.csv ファイルには必要な形式があり、各列には Neuron 0 から始まる 1 つのニューロンの情報が含まれています。ニューロン 0 などのニューロン番号を行 1 に入力します。そして、分析された各z位置についてバックグラウンド強度を提供し、ニューロン0について5つのz位置が分析された場合、ニューロン0の下に5つのバックグラウンド強度値を入力する。
- EXTRACT 関数を使用して、各 t 位置の最大蛍光強度を特定し、式 (1) に示すように ΔF/F0 を計算します。
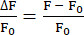 (1)
(1)- フォルダーを作成し、ニューロン番号を使用して Neuron 0 から始まる名前を付けます。対応するニューロンの蛍光情報を含むCSVファイルをフォルダに保存します。各 CSV ファイルには 1 つの Z 位置の情報が含まれているため、CSV ファイルの数は分析された Z 位置の数と等しくなります。CSV ファイルに Mean_Intensity#.csv (# は Z 位置を表す) という名前を付けると、各 CSV ファイルには少なくとも 2 つの列 (POSITION_T と MEAN_INTENSITY または MEAN_INTENSITY_CH1) が含まれます。
注: 目的のセルごとにフォルダを作成します。 - 手順2.7.1で作成したBackground_list.csvファイルとフォルダーを1つのフォルダーに保存します。[ ファイルの参照]をクリックして、このフォルダを選択します。
注: Background_list.csv ファイル内の背景値の数は、各ニューロンの Mean_Intensity#.csv ファイルの数と一致する必要があります。 - バックグラウンドファイルは自動的に入力されます。[T 位置の数] に T 位置の最大数を入力します。
- [抽出] をクリックして、各ニューロンの CSV ファイルとプロットを含む結果フォルダーを作成します。CSVファイルには、各t位置での最大蛍光強度とΔF/F0に関する情報が含まれています。プロットは、t位置に対するΔF/F0の折れ線グラフです。
注:この研究では、ΔF / F0は式(1)を使用して計算されました。各z位置の第1の値をF0とした。このF0が適切でない場合20、プラグインは、python_filesフォルダ内の各ニューロンの生データを含むファイルを提供する。
- フォルダーを作成し、ニューロン番号を使用して Neuron 0 から始まる名前を付けます。対応するニューロンの蛍光情報を含むCSVファイルをフォルダに保存します。各 CSV ファイルには 1 つの Z 位置の情報が含まれているため、CSV ファイルの数は分析された Z 位置の数と等しくなります。CSV ファイルに Mean_Intensity#.csv (# は Z 位置を表す) という名前を付けると、各 CSV ファイルには少なくとも 2 つの列 (POSITION_T と MEAN_INTENSITY または MEAN_INTENSITY_CH1) が含まれます。
- MERGE関数を使用して、各ニューロンのΔF / F 0を平均し、SEMを計算し、t位置の平均ΔF / F0をプロットします。
- [ファイルの参照]をクリックして、EXTRACT関数によって作成された結果フォルダーを選択します。
- Tポジションの数を Tポジション の最大数で入力します。
- マージをクリックして、merged_data.csvファイルとAverage_dF_F0.pngプロットを含むmerged_dataフォルダを作成します。CSVファイルには、各t位置の平均とSEM ΔF / F0に関する情報が含まれています。プロットは、t位置の平均ΔF/F0の折れ線グラフです。
Access restricted. Please log in or start a trial to view this content.
結果
3Dカルシウムイメージング解析のワークフロー
本研究では、新しいImageJプラグインTACIを開発し、zドリフトを追跡し、複数のz位置に現れる個々の細胞の応答を特定する3Dカルシウムイメージングを解析するワークフローについて説明しました(図1)。このツールには、名前の変更、整理、抽出、マージの 4 ?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
本研究では、新しいImageJプラグインTACIを開発し、3Dカルシウムイメージングを解析するワークフローを記述した。現在利用可能な多くのツールは、x / yモーションの補正に重点を置いていますが、z軸の動きも明示的に診断または修正する必要があります6。生きている生物の画像取得中、生物が固定化されていてもz軸上の動きは避けられず、温度変化などの何らかの刺激が?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は開示する利益相反を持っていません。
謝辞
フラリンイメージングセンターのツァイスLSM 880を使用して、カルシウムイメージングデータを収集しました。ミシェル・L・オルセン博士とユハン・パン博士のIMARISソフトウェアの支援に感謝します。原稿に対する建設的なコメントをしてくれたLenwood S. Heath博士と、GitHub READMEファイルへのコメントをくださったSteven Giavasisに感謝します。この作業は、NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) および NIH R01GM140130 (https://www.nigms.nih.gov/) から L.N. までサポートされました。資金提供者は、研究デザイン、データ収集と分析、出版の決定、または原稿の準備において何の役割も果たしていませんでした。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
参考文献
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258(2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517(2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173(2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153(2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432(2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569(2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728(2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869(2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279(2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254(2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252(2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved