É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
TACI: Um plugin ImageJ para análise de imagem de cálcio 3D
Neste Artigo
Resumo
TrackMate Analysis of Calcium Imaging (TACI) é um plugin ImageJ de código aberto para análise de imagem de cálcio 3D que examina o movimento no eixo z e identifica o valor máximo de cada z-stack para representar a intensidade de uma célula no ponto de tempo correspondente. Pode separar neurônios sobrepostos na direção lateral (x / y), mas em diferentes planos z.
Resumo
A pesquisa em neurociência evoluiu para usar imagens complexas e ferramentas computacionais para extrair informações abrangentes de conjuntos de dados. A imagem de cálcio é uma técnica amplamente utilizada que requer software sofisticado para obter resultados confiáveis, mas muitos laboratórios lutam para adotar métodos computacionais ao atualizar protocolos para atender aos padrões modernos. As dificuldades surgem devido à falta de conhecimento de programação e paywalls para software. Além disso, as células de interesse exibem movimentos em todas as direções durante a imagem de cálcio. Muitas abordagens foram desenvolvidas para corrigir o movimento na direção lateral (x/y).
Este artigo descreve um fluxo de trabalho usando um novo plug-in ImageJ, TrackMate Analysis of Calcium Imaging (TACI), para examinar o movimento no eixo z em imagens de cálcio 3D. Este software identifica o valor máximo de fluorescência de todas as posições z em que um neurônio aparece e o usa para representar a intensidade do neurônio na posição t correspondente. Portanto, esta ferramenta pode separar neurônios sobrepostos na direção lateral (x / y), mas aparecendo em planos z distintos. Como um plugin ImageJ, o TACI é uma ferramenta computacional de código aberto e fácil de usar para análise de imagens de cálcio 3D. Validamos esse fluxo de trabalho usando neurônios termossensíveis de larvas de moscas que exibiam movimentos em todas as direções durante a flutuação da temperatura e um conjunto de dados de imagem de cálcio 3D adquirido do cérebro da mosca.
Introdução
O nível de cálcio intracelular é um marcador preciso de excitabilidade neuronal. A imagem de cálcio mede as alterações no cálcio intracelular para entender a atividade neuronal1. Estudos em neurociência têm utilizado cada vez mais esse método devido ao desenvolvimento de técnicas para medir a concentração intracelular de cálcio, incluindo indicadores de cálcio geneticamente codificados (GECIs), como o GCaMP2,3, que podem ser expressos de forma não invasiva em conjuntos específicos de neurônios por meio de abordagens genéticas. Os menores custos de lasers e componentes do microscópio também aumentaram o uso de imagens de cálcio4. É importante ressaltar que a imagem de cálcio permite registrar e estudar neurônios únicos, bem como grandes populações de neurônios simultaneamente em animais em movimento livre5.
No entanto, a análise de dados de imagem de cálcio é desafiadora porque (1) envolve o rastreamento das mudanças na fluorescência de células individuais ao longo do tempo, (2) o sinal de fluorescência desaparece intermitentemente ou reaparece com respostas neuronais e (3) os neurônios podem se mover em todas as direções, especificamente dentro e fora de um plano focal ou aparecendo em vários planos4, 6. A análise manual é demorada e torna-se impraticável à medida que a duração das gravações e o número de neurônios aumentam. Vários programas de software foram desenvolvidos para acelerar o processo de análise de imagens de cálcio. Anteriormente, o software era projetado em um contexto experimental limitado, dificultando a adoção por outros laboratórios. Esforços recentes para atender aos padrões modernos de compartilhamento de software levaram ao desenvolvimento de várias ferramentas que podem analisar consistentemente dados de imagem de cálcio em diferentes grupos 7,8,9,10,11,12,13,14,15,16,17,18,19 . No entanto, a maioria dessas ferramentas requer conhecimento de programação e / ou depende de software comercial. A falta de conhecimento de programação e paywalls de software dissuadem os pesquisadores de adotar esses métodos. Além disso, muitas dessas ferramentas se concentram na correção do movimento x/y, embora o movimento no eixo z também precise ser explicitamente diagnosticado e corrigido6. Há uma necessidade de uma ferramenta computacional para analisar imagens de cálcio 3D que se concentram em neurônios que exibem deriva z e aparecem em vários planos z. Idealmente, essa ferramenta deve usar software de código aberto e não exigir conhecimento de programação para permitir que outros laboratórios a adotem prontamente.
Aqui, desenvolvemos um novo plugin ImageJ, TACI, para analisar dados de imagem de cálcio 3D. Primeiro, o software renomeia, se necessário, e organiza os dados de imagem de cálcio 3D por posições z. As células de interesse são rastreadas em cada posição z, e suas intensidades de fluorescência são extraídas pelo TrackMate ou outras ferramentas computacionais. O TACI é então aplicado para examinar o movimento no eixo z. Ele identifica o valor máximo de uma pilha z e o usa para representar a intensidade de uma célula no ponto de tempo correspondente. Este fluxo de trabalho é adequado para analisar imagens de cálcio 3D com movimento em todas as direções e / ou com neurônios sobrepostos na direção lateral (x / y), mas aparecendo em diferentes posições z. Para validar esse fluxo de trabalho, foram usados conjuntos de dados de imagem de cálcio 3D de neurônios termossensíveis de larvas de moscas e neurônios de cogumelos no cérebro. De notar, TACI é um plugin ImageJ de código aberto e não requer nenhum conhecimento de programação.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Imagem de cálcio
- Preparação de larvas de moscas
NOTA: As moscas e larvas são mantidas a 25 °C sob um ciclo claro:escuro de 12 h:12 h.- Anestesiar as moscas com CO2. Separe 20-45 machos e 20-45 fêmeas em cada frasco para moscas e dê-lhes pelo menos 24 h a 48 h para se recuperarem da exposição ao CO2 .
NOTA: A exposição da mosca ao CO2 deve durar o menor período de tempo possível. - Para sincronizar a idade das larvas, toque sobre as moscas em novos frascos contendo grânulos de levedura e deixe-as colocar ovos de 4 a 8 h. Remova as moscas virando-as em novos frascos para injetáveis.
- Recolher as larvas às 72 h utilizando 10 ml de solução de sacarose a 20% p/v.
- Anestesiar as moscas com CO2. Separe 20-45 machos e 20-45 fêmeas em cada frasco para moscas e dê-lhes pelo menos 24 h a 48 h para se recuperarem da exposição ao CO2 .
- Configuração do microscópio e do controle de temperatura
- Realizar a imagem em um microscópio confocal (consulte a Tabela de Materiais) e um estágio piezo do eixo z com uma inserção de estágio usando as seguintes configurações: laser, argônio; modo de digitalização, quadro; tamanho do quadro, 512 x 512; velocidade, máximo; canais / profundidade de bits, 1/8 Bit; zoom, 1,5; z-stack, fatia = 15, Keep = fatia; dispositivos de foco e estratégia, foco definido; foco, Foco definitivo em.
- Conecte um módulo de resfriamento Peltier a um dissipador de calor pelo composto de transferência de calor para construir um resfriador termoelétrico. O Peltier é alimentado por uma fonte de alimentação de 2 A.
- Conecte uma microssonda de termopar a um dispositivo de aquisição de dados para registrar a temperatura.
- Imagem de cálcio
- Lave as larvas 3x em 1x solução salina tamponada com fosfato (PBS).
- Pipeta 75 μL de 1x PBS no centro de uma lâmina de vidro.
- Coloque uma ou duas larvas em 1x PBS e coloque a microssonda termopar perto das larvas.
NOTA: A distância entre as larvas e a microssonda deve ser de ~5 mm. As larvas podem se mover se a microssonda estiver posicionada muito perto delas. Uma grande distância pode resultar em leituras de temperatura imprecisas. - Cubra as larvas e a microssonda termopar com uma tampa de vidro. Sele a tampa com esmalte.
- Coloque a lâmina no palco do microscópio, encontre o foco usando uma objetiva de 25x e coloque o resfriador termoelétrico na lâmina.
NOTA: O Peltier é colocado diretamente na lâmina onde as larvas devem fornecer os estímulos de temperatura. - Em software confocal, concentre-se nas células fluorescentes de interesse. Ajuste a potência do laser para evitar a saturação excessiva. Defina a primeira e a última posição de fatia na configuração z-stack.
NOTA: Use a menor potência de laser possível antes de gravar para evitar o fotobranqueamento. - Inicie a varredura z-stack e o registro de temperatura ao mesmo tempo.
- Controle a fonte de alimentação que alimenta o Peltier para alterar a temperatura da superfície do Peltier. Ligue a fonte de alimentação para diminuir a temperatura e desligue-a para aumentar a temperatura.
- Pare a varredura z-stack e o registro de temperatura.
2. Análise dos dados de imagem de cálcio 3D
- Exporte os dados de imagem de cálcio para arquivos TIFF e salve-os em uma pasta com o mesmo nome que o nome base dos arquivos TIFF dentro.
Observação : O nome do arquivo não deve conter vírgulas.- Use os seguintes parâmetros para exportar os dados de imagem de cálcio do ZEN (edição preta): tipo de arquivo, TIFF; compressão, LZW; canais, 2; Posição Z, todas; o tempo, todos; fase, 1; região, cheia.
- Instalação
- Baixe o TACI-Calcium_Imaging.jar do Github (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- Instale o plugin no FIJI clicando em Plugins na barra de menus e, em seguida, clicando em Instalar no menu suspenso (Plugins | Instalar). Em seguida, reinicie o FIJI.
NOTA: NÃO use Plugins | Instale o plug-in. - Execute o plugin clicando em Plugins e, em seguida, escolhendo TACI-Calcium Imaging (Plugins | TACI-Imagem de Cálcio).
- Use a função RENAME para converter os nomes de arquivo TIFF para a estrutura necessária.
Observação : A ferramenta usa filename_h#t#z#c#.tif como a estrutura padrão (h# e c# são opcionais; #: um inteiro positivo). Se os nomes de arquivo de imagem não estiverem na estrutura padrão, a função RENAME deverá ser executada.- Escolha a pasta na qual as imagens TIFF precisam ser renomeadas clicando em Procurar Pastas.
- Preencha as informações do parâmetro. Cinco parâmetros são listados, incluindo Filename, Phase, Max T-Position, Max Z-Position e Channel. Cada parâmetro tem três valores: Texto Precedente, Valor do Parâmetro e Ordem.
Observação : as informações diferenciam maiúsculas de minúsculas. Se os nomes de arquivo de imagem contiverem uma frase antes de um parâmetro, preencha a frase como o Texto Anterior do parâmetro correspondente. Se os nomes de arquivo da imagem contiverem uma frase depois de todos os parâmetros, preencha a frase como o Post Text.- Certifique-se de inserir Nome do arquivo, Posição T Máxima e Posição Z Máxima e que os valores de parâmetro para Posição T Máxima e Posição Z Máxima incluem todos os dígitos.
- Aguarde até que o Nome do arquivo seja preenchido automaticamente usando o nome da pasta.
- Se os nomes de arquivo TIFF não incluírem a fase e o canal, deixe o valor do parâmetro correspondente em branco e escolha Na em Ordem.
- Clique em Renomear para criar uma pasta com o mesmo nome e _r. Observe que, na pasta, os nomes de arquivo TIFF foram reestruturados para serem compatíveis com a função ORGANIZAR .
Observação : _r foi adicionado aos nomes de arquivo das imagens TIFF.
- Use a função ORGANIZE para salvar as imagens TIFF da mesma posição z em uma pasta.
Observação : O nome da pasta deve ter o mesmo nome que o nome base dos arquivos TIFF dentro e não deve ter vírgulas.- Escolha a pasta na qual as imagens TIFF precisam ser organizadas clicando em Procurar Pastas.
- Se o arquivo CSV do parâmetro (param.csv) existir, aguarde até que os valores do parâmetro sejam preenchidos automaticamente.
Observação : O arquivo param.csv tem um formato necessário. Os parâmetros, incluindo nome do arquivo, fase, position_t, position_z, canal e is_gray, devem ser preenchidos na linha 1 da esquerda para a direita, começando pela coluna A. Os valores correspondentes para cada parâmetro devem ser preenchidos na linha 2. - Se o arquivo CSV do parâmetro (param.csv) não existir, preencha manualmente os valores do parâmetro. Certifique-se de que os valores dos parâmetros de Fase e Canal incluam letras, enquanto os valores dos parâmetros de Posição T e Posição Z devem ser os maiores números das posições t e z. Se os nomes de arquivo de imagem não incluírem a fase ou o canal, digite Na.
- Crie imagens TIFF em tons de cinza quando necessário. Deixe a caixa Are images gray? desmarcada para colocar as imagens em tons de cinza.
- Clique em Organizar para criar uma pasta com o mesmo nome e _gray_stacks e para gerar pastas com o mesmo nome e _# (#: z-positions) na pasta. Observe que os arquivos TIFF são classificados em pastas correspondentes por z-positions e que um arquivo chamado param.csv é gerado, no qual os parâmetros e seus valores podem ser encontrados.
- Use o TrackMate na FIJI para extrair as intensidades de fluorescência das células de interesse de cada posição z.
NOTA: Esta etapa também pode ser realizada por outro software de imagem.- Abra as imagens TIFF em uma pasta de posição z por FIJI.
- Execute o TrackMate clicando em Plugins | Rastreamento | TrackMate e ajuste os seguintes parâmetros, se necessário.
- Use detectores DoG ou LoG.
- Altere o diâmetro, o limite e o filtro mediano do blob. Ajuste o diâmetro da bolha para ser semelhante ao diâmetro das células. Se as células forem ovais, ajuste o diâmetro da bolha para ser semelhante ao eixo menor.
NOTA: Aumentar o limite ajuda a evitar que o ruído de fundo seja captado como sinais sem afetar as intensidades do sinal (Figura 1 Suplementar). No entanto, um aumento no limiar pode perder sinais verdadeiros (Figura 1 Suplementar). Se os sinais forem fortes, use o filtro mediano para diminuir o ruído de Sal e Pimenta. - Defina os filtros para remover alguns, se não todos, os sinais irrelevantes. Os filtros X e Y são filtros espaciais. Use os filtros X e Y para remover os sinais irrelevantes que estão distantes dos sinais reais.
NOTA: Quando os filtros são definidos em uma imagem, é crucial verificar todas as outras imagens para garantir que os sinais reais não sejam removidos. - Defina a distância máxima de ligação, a distância máxima de fechamento de lacunas e a distância máxima de fechamento de lacunas. Defina a distância máxima de ligação e a distância máxima de fechamento de lacunas para 3x-5x o diâmetro da bolha, especialmente quando as amostras se movem significativamente ao longo do tempo, para ajudar a diminuir o número de faixas. Defina o intervalo máximo de quadros de fechamento de lacunas para o número de imagens na pilha.
- Exporte as intensidades de fluorescência das regiões de interesse (ROIs) para um arquivo CSV. Se uma versão antiga do TrackMate for usada, escolha Exportar todas as estatísticas de pontos na janela Selecionar uma ação. Se a versão do TrackMate for 7.6.1 ou superior, escolha os Pontos na janela Opções de exibição . Exporte ou salve como os arquivos interativos em arquivos CSV.
Observação : ambos os arquivos são interativos com a janela de imagem; o realce de um ROI exibe o ROI correspondente na janela de imagem. Esses arquivos incluem as intensidades médias (MEAN_INTENSITY ou MEAN_INTENSITY_CH1) das células de interesse em pontos de tempo correspondentes (POSITION_T). O mesmo TRACK_IDs deve representar os mesmos ROIs em diferentes pontos de tempo. No entanto, isso nem sempre é verdade e pode precisar ser corrigido manualmente, quando necessário. Se o TrackMate não reconhecer os ROIs em alguns momentos, esses pontos de tempo não serão exibidos.
- Extraia a intensidade de fluorescência de plano de fundo e crie o arquivo Background_list.csv.
NOTA: Neste estudo, a intensidade de fundo para cada posição z foi estimada usando o valor médio de três a cinco bolhas vizinhas do mesmo tamanho que não continham sinais de fluorescência e eram de diferentes pontos de tempo.- O arquivo Background_list.csv tem um formato obrigatório: cada coluna contém as informações de um neurônio, a partir do Neurônio 0. Preencha os números dos neurônios, como o Neurônio 0, na linha 1. Em seguida, forneça a intensidade de fundo para cada posição z analisada - se cinco posições z forem analisadas para o Neurônio 0, preencha cinco valores de intensidade de fundo abaixo do Neurônio 0.
Observação : O arquivo Background_list.csv é necessário para a função TACI EXTRACT . Se o fundo for insignificante, a Background_list.csv com a intensidade de fundo zero de cada neurônio deve ser fornecida.
- O arquivo Background_list.csv tem um formato obrigatório: cada coluna contém as informações de um neurônio, a partir do Neurônio 0. Preencha os números dos neurônios, como o Neurônio 0, na linha 1. Em seguida, forneça a intensidade de fundo para cada posição z analisada - se cinco posições z forem analisadas para o Neurônio 0, preencha cinco valores de intensidade de fundo abaixo do Neurônio 0.
- Use a função EXTRACT para identificar as intensidades máximas de fluorescência em cada posição t e calcular ΔF/F0 como mostrado na equação (1).
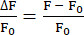 (1)
(1)- Crie uma pasta e nomeie-a usando o número do neurônio, a partir do Neurônio 0. Salve os arquivos CSV que contêm as informações de fluorescência do neurônio correspondente na pasta. Cada arquivo CSV contém as informações de uma posição z, de modo que o número de arquivos CSV é igual ao número de posições z analisadas. Nomeie os arquivos CSV como Mean_Intensity#.csv (# representa a posição z) e cada arquivo CSV inclui pelo menos duas colunas: POSITION_T e MEAN_INTENSITY ou MEAN_INTENSITY_CH1.
Observação : crie uma pasta para cada célula de interesse. - Salve o arquivo Background_list.csv e as pastas criadas na etapa 2.7.1 em uma pasta. Escolha esta pasta clicando em Procurar arquivos.
Observação : O número de valores de plano de fundo no arquivo Background_list.csv deve corresponder ao número de arquivos Mean_Intensity#.csv para cada neurônio. - O arquivo em segundo plano é preenchido automaticamente. Preencha o maior número de posições t para o número de posições T.
- Clique em Extrair para criar uma pasta de resultados, incluindo os arquivos CSV e gráficos para cada neurônio. Os arquivos CSV incluem informações sobre a intensidade máxima de fluorescência e ΔF/F0 em cada posição t. Os gráficos são gráficos de linhas de ΔF/F0 sobre as posições t.
NOTA: Neste estudo, o ΔF/F0 foi calculado por meio da equação (1). O primeiro valor de cada posição z foi utilizado como F0. Se este F0 não for apropriado20, o plugin fornece arquivos incluindo dados brutos para cada neurônio na pasta python_files.
- Crie uma pasta e nomeie-a usando o número do neurônio, a partir do Neurônio 0. Salve os arquivos CSV que contêm as informações de fluorescência do neurônio correspondente na pasta. Cada arquivo CSV contém as informações de uma posição z, de modo que o número de arquivos CSV é igual ao número de posições z analisadas. Nomeie os arquivos CSV como Mean_Intensity#.csv (# representa a posição z) e cada arquivo CSV inclui pelo menos duas colunas: POSITION_T e MEAN_INTENSITY ou MEAN_INTENSITY_CH1.
- Use a função MERGE para calcular a média do ΔF/F 0 de cada neurônio, calcular o MEV e plotar a média do ΔF/F0 sobre as posições t.
- Escolha a pasta de resultados criada pela função EXTRACT clicando em Procurar arquivos.
- Preencha o Número de Posições T com o maior número de posições t.
- Clique em Mesclar para criar uma pasta merged_data, incluindo um arquivo merged_data.csv e um gráfico de Average_dF_F0.png. O arquivo CSV inclui as informações sobre a média e o SEM ΔF/F0 em cada posição t. O gráfico é um gráfico de linhas da média ΔF/F0 sobre as posições t.
Access restricted. Please log in or start a trial to view this content.
Resultados
Fluxo de trabalho de análise de imagem de cálcio 3D
Neste estudo, desenvolvemos um novo plugin ImageJ, TACI, e descrevemos um fluxo de trabalho para rastrear a deriva z e analisar imagens de cálcio 3D que identificam as respostas de células individuais que aparecem em múltiplas posições z (Figura 1). Essa ferramenta tem quatro funções: RENOMEAR, ORGANIZAR, EXTRAIR e MESCLAR. Primeiro, se os nomes...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este estudo desenvolveu um novo plugin ImageJ, TACI, e descreveu um fluxo de trabalho analisando imagens de cálcio 3D. Muitas ferramentas atualmente disponíveis se concentram na correção do movimento x/y, embora o movimento no eixo z também precise ser explicitamente diagnosticado ou corrigido6. Durante a aquisição de imagens em um organismo vivo, o movimento no eixo z é inevitável mesmo quando o organismo está imobilizado, e alguns estímulos, como a mudança de temperatura, muitas veze...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Um Zeiss LSM 880 no Fralin Imaging Center foi utilizado para coletar os dados de imagem de cálcio. Agradecemos à Dra. Michelle L Olsen e Yuhang Pan por sua assistência com o software IMARIS. Agradecemos ao Dr. Lenwood S. Heath por comentários construtivos sobre o manuscrito e Steven Giavasis por comentários sobre o arquivo README do GitHub. Este trabalho foi apoiado pelo NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) e NIH R01GM140130 (https://www.nigms.nih.gov/) para L.N. Os financiadores não tiveram nenhum papel no desenho do estudo, coleta e análise de dados, decisão de publicação ou preparação do manuscrito.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
Referências
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258(2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517(2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173(2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153(2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432(2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569(2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728(2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869(2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279(2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254(2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252(2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados