Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
TACI : un plugin ImageJ pour l’analyse d’imagerie calcique 3D
Dans cet article
Résumé
TrackMate Analysis of Calcium Imaging (TACI) est un plugin ImageJ open-source pour l’analyse 3D de l’imagerie calcique qui examine le mouvement sur l’axe z et identifie la valeur maximale de chaque pile z pour représenter l’intensité d’une cellule au point de temps correspondant. Il peut séparer les neurones qui se chevauchent dans la direction latérale (x / y) mais sur des plans z différents.
Résumé
La recherche en neurosciences a évolué pour utiliser des outils complexes d’imagerie et de calcul pour extraire des informations complètes à partir d’ensembles de données. L’imagerie calcique est une technique largement utilisée qui nécessite un logiciel sophistiqué pour obtenir des résultats fiables, mais de nombreux laboratoires ont du mal à adopter des méthodes de calcul lors de la mise à jour des protocoles pour répondre aux normes modernes. Des difficultés surviennent en raison d’un manque de connaissances en programmation et de paywalls pour les logiciels. De plus, les cellules d’intérêt affichent des mouvements dans toutes les directions pendant l’imagerie calcique. De nombreuses approches ont été développées pour corriger le mouvement dans la direction latérale (x/y).
Ce document décrit un flux de travail utilisant un nouveau plug-in ImageJ, TrackMate Analysis of Calcium Imaging (TACI), pour examiner le mouvement sur l’axe z en imagerie calcique 3D. Ce logiciel identifie la valeur de fluorescence maximale de toutes les positions z dans lesquelles un neurone apparaît et l’utilise pour représenter l’intensité du neurone à la position t correspondante. Par conséquent, cet outil peut séparer les neurones qui se chevauchent dans la direction latérale (x / y) mais apparaissent sur des plans z distincts. En tant que plugin ImageJ, TACI est un outil de calcul convivial et open-source pour l’analyse d’imagerie calcique 3D. Nous avons validé ce flux de travail en utilisant des neurones thermosensibles aux larves de mouches qui affichaient des mouvements dans toutes les directions pendant la fluctuation de température et un ensemble de données d’imagerie calcique 3D acquises à partir du cerveau de la mouche.
Introduction
Le taux de calcium intracellulaire est un marqueur précis de l’excitabilité neuronale. L’imagerie calcique mesure les changements dans le calcium intracellulaire pour comprendre l’activité neuronale1. Les études en neurosciences ont de plus en plus utilisé cette méthode en raison du développement de techniques de mesure de la concentration intracellulaire de calcium, y compris des indicateurs de calcium génétiquement codés (GECI), tels que GCaMP2,3, qui peuvent être exprimés de manière non invasive dans des ensembles spécifiques de neurones par des approches génétiques. La baisse des coûts des lasers et des composants de microscope a également augmenté l’utilisation de l’imagerie calcique4. Il est important de noter que l’imagerie calcique permet d’enregistrer et d’étudier simultanément des neurones uniques ainsi que de grandes populations de neurones chez des animaux en mouvement libre5.
Néanmoins, l’analyse des données d’imagerie calcique est difficile car (1) elle implique de suivre les changements de fluorescence des cellules individuelles au fil du temps, (2) le signal de fluorescence disparaît ou réapparaît par intermittence avec les réponses neuronales, et (3) les neurones peuvent se déplacer dans toutes les directions, en particulier dans et hors d’un plan focal ou apparaissant sur plusieurs plans4, 6. L’analyse manuelle prend beaucoup de temps et devient impraticable à mesure que la longueur des enregistrements et le nombre de neurones augmentent. Divers logiciels ont été développés pour accélérer le processus d’analyse de l’imagerie calcique. Auparavant, le logiciel était conçu dans un contexte expérimental limité, ce qui rendait difficile son adoption par d’autres laboratoires. Les efforts récents pour répondre aux normes modernes de partage de logiciels ont conduit au développement de plusieurs outils capables d’analyser de manière cohérente les données d’imagerie calcique dans différents groupes 7,8,9,10,11,12,13,14,15,16,17,18,19 . Cependant, la plupart de ces outils nécessitent des connaissances en programmation et / ou dépendent de logiciels commerciaux. Un manque de connaissances en programmation et de logiciels payants dissuade les chercheurs d’adopter ces méthodes. De plus, beaucoup de ces outils se concentrent sur la correction du mouvement x/y, bien que le mouvement sur l’axe z doive également être explicitement diagnostiqué et corrigé6. Il existe un besoin d’un outil informatique pour analyser l’imagerie calcique 3D qui se concentre sur les neurones présentant une dérive z et apparaissant sur plusieurs plans z. Idéalement, cet outil devrait utiliser un logiciel open source et ne pas nécessiter de connaissances en programmation pour permettre à d’autres laboratoires de l’adopter facilement.
Ici, nous avons développé un nouveau plugin ImageJ, TACI, pour analyser les données d’imagerie calcique 3D. Tout d’abord, le logiciel renomme, si nécessaire, et organise les données d’imagerie calcique 3D par positions z. Les cellules d’intérêt sont suivies dans chaque position z, et leurs intensités de fluorescence sont extraites par TrackMate ou d’autres outils de calcul. TACI est ensuite appliqué pour examiner le mouvement sur l’axe z. Il identifie la valeur maximale d’une pile z et l’utilise pour représenter l’intensité d’une cellule au point de temps correspondant. Ce flux de travail est adapté à l’analyse de l’imagerie calcique 3D avec un mouvement dans toutes les directions et / ou avec des neurones se chevauchant dans la direction latérale (x / y) mais apparaissant dans différentes positions z. Pour valider ce flux de travail, des ensembles de données d’imagerie calcique 3D provenant de neurones thermosensibles aux larves de mouches et de neurones champignons dans le cerveau ont été utilisés. Il est à noter que TACI est un plugin ImageJ open-source et ne nécessite aucune connaissance en programmation.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Imagerie calcique
- Préparation des larves de mouches
NOTE: Les mouches et les larves sont maintenues à 25 ° C sous un cycle lumière:obscurité de 12 h:12 h.- Anesthésiez les mouches avec du CO2. Trier 20 à 45 mâles et 20 à 45 femelles dans chaque flacon de mouches et leur donner au moins 24 h à 48 h pour se remettre de l’exposition au CO2 .
REMARQUE : L’exposition des mouches au CO2 devrait durer le moins longtemps possible. - Pour synchroniser l’âge des larves, tapotez les mouches dans de nouveaux flacons contenant des granules de levure et laissez-leur pondre 4 à 8 heures pour pondre des œufs. Retirez les mouches en les retournant dans de nouveaux flacons.
- Prélever les larves à 72 h en utilisant 10 mL de solution de saccharose à 20 % p/v.
- Anesthésiez les mouches avec du CO2. Trier 20 à 45 mâles et 20 à 45 femelles dans chaque flacon de mouches et leur donner au moins 24 h à 48 h pour se remettre de l’exposition au CO2 .
- Configuration du microscope et du contrôle de la température
- Effectuer l’imagerie sur un microscope confocal (voir le tableau des matériaux) et un étage piézoélectrique de l’axe z avec un insert de scène en utilisant les paramètres suivants: laser, argon; mode de balayage, cadre; taille de l’image, 512 x 512; vitesse, maximum; canaux/profondeur de bits, 1/8 bit; zoom, 1,5; pile z, tranche = 15, conserver = tranche; dispositifs de mise au point et stratégie, mise au point définie; mise au point, Accent mis sur.
- Fixez un module de refroidissement Peltier à un dissipateur thermique par le composé de transfert de chaleur pour construire un refroidisseur thermoélectrique. Le Peltier est alimenté par une alimentation 2 A.
- Fixez une microsonde thermocouple à un dispositif d’acquisition de données pour enregistrer la température.
- Imagerie calcique
- Rincer les larves 3x dans 1x solution saline tamponnée au phosphate (PBS).
- Pipeter 75 μL de 1x PBS sur le centre d’une lame de verre.
- Mettez une ou deux larves dans 1x PBS et placez la microsonde thermocouple près des larves.
REMARQUE: La distance entre les larves et la microsonde doit être de ~5 mm. Les larves peuvent se déplacer si la microsonde est positionnée trop près d’elles. Une grande distance pourrait entraîner des lectures de température inexactes. - Couvrir les larves et la microsonde thermocouple avec une lamelle de verre couvercle. Scellez le couvercle avec du vernis à ongles.
- Placez la lame sur la platine du microscope, trouvez la mise au point à l’aide d’un objectif 25x et placez le refroidisseur thermoélectrique sur la lame.
REMARQUE: Le Peltier est placé directement sur la lame où les larves doivent délivrer les stimuli de température. - Dans les logiciels confocaux, concentrez-vous sur les cellules fluorescentes d’intérêt. Ajustez la puissance du laser pour éviter la sursaturation. Définissez les positions de la première et de la dernière tranche dans le paramètre z-stack.
REMARQUE: Utilisez la puissance laser la plus faible possible avant d’enregistrer pour éviter le photoblanchiment. - Démarrez le balayage z-stack et l’enregistrement de la température en même temps.
- Contrôlez l’alimentation qui alimente le Peltier pour modifier la température de surface du Peltier. Allumez le bloc d’alimentation pour diminuer la température et éteignez-le pour augmenter la température.
- Arrêtez le balayage z-stack et l’enregistrement de la température.
2. Analyse des données d’imagerie calcique 3D
- Exportez les données d’imagerie calcique vers des fichiers TIFF et enregistrez-les dans un dossier portant le même nom que le nom de base des fichiers TIFF qu’ils contiennent.
Remarque : Le nom de fichier ne doit pas contenir de virgules.- Utilisez les paramètres suivants pour exporter les données d’imagerie calcique de ZEN (édition noire) : type de fichier, TIFF ; compression, LZW ; canaux, 2 ; Position Z, toutes; le temps, tous; phase, 1; région, pleine.
- Installation
- Téléchargez TACI-Calcium_Imaging.jar depuis Github (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- Installez le plugin dans FIJI en cliquant sur Plugins dans la barre de menus, puis en cliquant sur Installer dans le menu déroulant (Plugins | Installer). Ensuite, redémarrez FIJI.
REMARQUE: N’utilisez PAS de plugins | Installez le plugin. - Exécutez le plugin en cliquant sur Plugins , puis en choisissant TACI-Calcium Imaging (Plugins | TACI-Imagerie calcique).
- Utilisez la fonction RENAME pour convertir les noms de fichiers TIFF en la structure requise.
Remarque : L’outil utilise filename_h#t#z#c#.tif comme structure par défaut (h# et c# sont facultatifs ; # : entier positif). Si les noms de fichiers image ne figurent pas dans la structure par défaut, la fonction RENAME doit être exécutée.- Choisissez le dossier dans lequel les images TIFF doivent être renommées en cliquant sur Parcourir les dossiers.
- Renseignez les informations sur les paramètres. Cinq paramètres sont répertoriés, notamment Filename, Phase, Max T-Position, Max Z-Position et Channel. Chaque paramètre possède trois valeurs : Texte précédent, Valeur du paramètre et Ordre.
Remarque : Les informations sont sensibles à la casse. Si les noms de fichiers image contiennent une phrase avant un paramètre, renseignez l’expression en tant que texte précédent du paramètre correspondant. Si les noms de fichiers image contiennent une phrase après tous les paramètres, renseignez-la en tant que texte de publication.- Assurez-vous d’entrer Filename, Max T-Position et Max Z-Position et que les valeurs de paramètre pour Max T-Position et Max Z-Position incluent tous les chiffres.
- Attendez que le nom de fichier soit automatiquement rempli à l’aide du nom du dossier.
- Si les noms de fichiers TIFF n’incluent pas la phase et le canal, laissez la valeur de paramètre correspondante vide et choisissez Na dans l’ordre.
- Cliquez sur Renommer pour créer un dossier portant le même nom et le même _r. Notez que dans le dossier, les noms de fichiers TIFF ont été restructurés pour être compatibles avec la fonction ORGANIZE .
Remarque : _r a été ajouté aux noms de fichiers des images TIFF.
- Utilisez la fonction ORGANIZE pour enregistrer les images TIFF de la même position z dans un dossier.
Remarque : Le nom du dossier doit avoir le même nom que le nom de base des fichiers TIFF à l’intérieur et ne doit pas avoir de virgule.- Choisissez le dossier dans lequel les images TIFF doivent être organisées en cliquant sur Parcourir les dossiers.
- Si le fichier CSV de paramètre (param.csv) existe, attendez que les valeurs des paramètres soient remplies automatiquement.
Remarque : Le fichier param.csv a un format requis. Les paramètres, y compris le nom de fichier, la phase, position_t, position_z, le canal et is_gray, doivent être remplis à la ligne 1 de gauche à droite à partir de la colonne A. Les valeurs correspondantes pour chaque paramètre doivent être remplies à la ligne 2. - Si le fichier CSV de paramètre (param.csv) n’existe pas, renseignez manuellement les valeurs des paramètres. Assurez-vous que les valeurs de paramètre de Phase et Canal incluent des lettres, tandis que les valeurs de paramètre de Position T et Position Z doivent être les plus grands nombres des positions t et z. Si les noms des fichiers image n’incluent pas la phase ou le canal, entrez Na.
- Créez des images TIFF en niveaux de gris si nécessaire. Laissez la case Les images sont-elles grises ? décochée pour mettre les images en niveaux de gris.
- Cliquez sur Organiser pour créer un dossier portant le même nom et le même _gray_stacks et pour générer des dossiers portant le même nom et _# (#: z-positions) dans le dossier. Notez que les fichiers TIFF sont triés dans les dossiers correspondants par z-positions et qu’un fichier nommé param.csv est généré, dans lequel les paramètres et leurs valeurs peuvent être trouvés.
- Utilisez TrackMate dans FIJI pour extraire les intensités de fluorescence des cellules d’intérêt de chaque position z.
Remarque : Cette étape peut également être accomplie par d’autres logiciels d’imagerie.- Ouvrez les images TIFF dans un dossier z-position de FIJI.
- Exécutez TrackMate en cliquant sur Plugins | Suivi | TrackMate et ajustez les paramètres suivants si nécessaire.
- Utilisez des détecteurs DoG ou LoG.
- Modifiez le diamètre de l’objet blob, le seuil et le filtre médian. Ajustez le diamètre de l’objet blob pour qu’il soit similaire au diamètre des cellules. Si les cellules sont ovales, ajustez le diamètre de l’objet blob pour qu’il soit similaire à l’axe mineur.
REMARQUE : L’augmentation du seuil permet d’éviter que le bruit de fond ne soit capté sous forme de signaux sans affecter l’intensité du signal (Figure supplémentaire 1). Cependant, une augmentation du seuil peut manquer les signaux réels (figure supplémentaire 1). Si les signaux sont forts, utilisez le filtre médian pour diminuer le bruit poivre et sel. - Réglez les filtres pour supprimer certains, sinon tous, des signaux non pertinents. Les filtres X et Y sont des filtres spatiaux. Utilisez les filtres X et Y pour supprimer les signaux non pertinents qui sont éloignés des signaux réels.
REMARQUE: Lorsque des filtres sont définis sur une image, il est crucial de vérifier toutes les autres images pour s’assurer que les signaux réels ne sont pas supprimés. - Définissez la distance maximale de liaison, la distance maximale de fermeture d’espace et l’écart d’image maximal de fermeture d’espace. Définissez la distance maximale de liaison et la distance maximale de fermeture de l’espace sur 3x-5x le diamètre de l’objet blob, en particulier lorsque les échantillons se déplacent de manière significative au fil du temps, afin de réduire le nombre de pistes. Définissez l’écart maximal d’images de fermeture de l’espace sur le nombre d’images dans la pile.
- Exportez les intensités de fluorescence des régions d’intérêt (ROI) vers un fichier CSV. Si une ancienne version de TrackMate est utilisée, choisissez Exporter toutes les statistiques de spots dans la fenêtre Sélectionner une action . Si la version de TrackMate est 7.6.1 ou supérieure, choisissez les Spots dans la fenêtre Options d’affichage . Exportez ou enregistrez en tant que fichiers interactifs dans des fichiers CSV.
REMARQUE: Les deux fichiers sont interactifs avec la fenêtre d’image; la mise en surbrillance d’un ROI affiche le ROI correspondant dans la fenêtre d’image. Ces fichiers comprennent les intensités moyennes (MEAN_INTENSITY ou MEAN_INTENSITY_CH1) des cellules d’intérêt aux points temporels correspondants (POSITION_T). Les mêmes TRACK_IDs doivent représenter les mêmes retours sur investissement à différents moments. Cependant, cela n’est pas toujours vrai et peut devoir être corrigé manuellement, si nécessaire. Si TrackMate ne reconnaît pas les retours sur investissement à certains moments, ces points temporels ne sont pas affichés.
- Extrayez l’intensité de fluorescence de fond et créez le fichier Background_list.csv.
REMARQUE: Dans cette étude, l’intensité de fond pour chaque position z a été estimée en utilisant la valeur moyenne de trois à cinq blobs voisins de même taille qui ne contenaient pas de signaux de fluorescence et provenaient de points temporels différents.- Le fichier Background_list.csv a un format requis: chaque colonne contient les informations d’un neurone, à partir de Neuron 0. Renseignez les nombres de neurones, tels que Neurone 0, dans la ligne 1. Ensuite, fournissez l’intensité de fond pour chaque position z analysée - si cinq positions z sont analysées pour le neurone 0, remplissez cinq valeurs d’intensité de fond en dessous du neurone 0.
Remarque : Le fichier Background_list.csv est requis pour la fonction TACI EXTRACT . Si le fond est négligeable, le Background_list.csv avec l’intensité de fond nulle de chaque neurone doit être fourni.
- Le fichier Background_list.csv a un format requis: chaque colonne contient les informations d’un neurone, à partir de Neuron 0. Renseignez les nombres de neurones, tels que Neurone 0, dans la ligne 1. Ensuite, fournissez l’intensité de fond pour chaque position z analysée - si cinq positions z sont analysées pour le neurone 0, remplissez cinq valeurs d’intensité de fond en dessous du neurone 0.
- Utilisez la fonction EXTRACT pour identifier les intensités de fluorescence maximales à chaque position t et calculer ΔF/F0 comme indiqué dans l’équation (1).
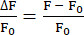 (1)
(1)- Créez un dossier et nommez-le en utilisant le numéro de neurone, en commençant par Neuron 0. Enregistrez les fichiers CSV contenant les informations de fluorescence du neurone correspondant dans le dossier. Chaque fichier CSV contient les informations d’une position z, de sorte que le nombre de fichiers CSV est égal au nombre de positions z analysées. Nommez les fichiers CSV comme Mean_Intensity#.csv (# représente la position z) et chaque fichier CSV comprend au moins deux colonnes : POSITION_T et MEAN_INTENSITY ou MEAN_INTENSITY_CH1.
Remarque : Créez un dossier pour chaque cellule d’intérêt. - Enregistrez le fichier et les dossiers Background_list.csv créés à l’étape 2.7.1 dans un dossier. Choisissez ce dossier en cliquant sur Parcourir les fichiers.
REMARQUE: Le nombre de valeurs d’arrière-plan dans le fichier Background_list.csv doit correspondre au nombre de fichiers Mean_Intensity#.csv pour chaque neurone. - Le fichier d’arrière-plan est automatiquement rempli. Remplissez le plus grand nombre de positions t pour le nombre de positions t.
- Cliquez sur Extraire pour créer un dossier de résultats, y compris les fichiers CSV et les tracés pour chaque neurone. Les fichiers CSV contiennent des informations sur l’intensité de fluorescence maximale et ΔF/F0 à chaque position t. Les tracés sont des graphiques linéaires de ΔF/F0 sur t-positions.
NOTE: Dans cette étude, ΔF / F0 a été calculé à l’aide de l’équation (1). La première valeur de chaque position z a été utilisée comme F0. Si ce F0 n’est pas approprié20, le plugin fournit des fichiers comprenant des données brutes pour chaque neurone dans le dossier python_files.
- Créez un dossier et nommez-le en utilisant le numéro de neurone, en commençant par Neuron 0. Enregistrez les fichiers CSV contenant les informations de fluorescence du neurone correspondant dans le dossier. Chaque fichier CSV contient les informations d’une position z, de sorte que le nombre de fichiers CSV est égal au nombre de positions z analysées. Nommez les fichiers CSV comme Mean_Intensity#.csv (# représente la position z) et chaque fichier CSV comprend au moins deux colonnes : POSITION_T et MEAN_INTENSITY ou MEAN_INTENSITY_CH1.
- Utilisez la fonction MERGE pour faire la moyenne du ΔF/F 0 de chaque neurone, calculer le MEB et tracer la moyenne ΔF/F0 sur les positions t.
- Choisissez le dossier de résultats créé par la fonction EXTRACT en cliquant sur Parcourir les fichiers.
- Remplissez le nombre de positions T avec le plus grand nombre de positions t.
- Cliquez sur Fusionner pour créer un dossier merged_data, y compris un fichier merged_data.csv et un tracé Average_dF_F0.png. Le fichier CSV comprend les informations sur la moyenne et SEM ΔF/F0 à chaque position t. Le graphique est un graphique linéaire de la moyenne ΔF/F0 sur t-positions.
Access restricted. Please log in or start a trial to view this content.
Résultats
Flux de travail d’analyse d’imagerie calcique 3D
Dans cette étude, nous avons développé un nouveau plugin ImageJ, TACI, et décrit un flux de travail pour suivre la dérive z et analyser l’imagerie calcique 3D qui identifie les réponses des cellules individuelles apparaissant dans plusieurs positions z (Figure 1). Cet outil a quatre fonctions : RENOMMER, ORGANISER, EXTRAIRE et FUSIONNER. Tout d?...
Access restricted. Please log in or start a trial to view this content.
Discussion
Cette étude a développé un nouveau plugin ImageJ, TACI, et décrit un flux de travail analysant l’imagerie calcique 3D. De nombreux outils actuellement disponibles se concentrent sur la correction du mouvement x/y, bien que le mouvement sur l’axe z doive également être explicitement diagnostiqué ou corrigé6. Lors de l’acquisition d’images dans un organisme vivant, le mouvement sur l’axe z est inévitable même lorsque l’organisme est immobilisé, et certains stimuli, tels que le...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Un Zeiss LSM 880 dans le Fralin Imaging Center a été utilisé pour collecter les données d’imagerie calcique. Nous remercions le Dr Michelle L Olsen et Yuhang Pan pour leur aide avec le logiciel IMARIS. Nous remercions le Dr Lenwood S. Heath pour ses commentaires constructifs sur le manuscrit et Steven Giavasis pour ses commentaires sur le fichier GitHub README. Ce travail a été soutenu par NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) et NIH R01GM140130 (https://www.nigms.nih.gov/) à L.N. Les bailleurs de fonds n’ont joué aucun rôle dans la conception de l’étude, la collecte et l’analyse des données, la décision de publier ou la préparation du manuscrit.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
Références
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258(2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517(2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173(2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153(2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432(2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569(2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728(2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869(2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279(2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254(2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252(2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon