A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
TACI: תוסף ImageJ לניתוח הדמיית סידן תלת-ממדית
In This Article
Summary
TrackMate Analysis of Calcium Imaging (TACI) הוא תוסף קוד פתוח ImageJ לניתוח הדמיית סידן תלת-ממדית הבוחן תנועה על ציר z ומזהה את הערך המרבי של כל ערימת z כדי לייצג את עוצמת התא בנקודת הזמן המתאימה. הוא יכול להפריד תאי עצב החופפים בכיוון הצידי (x/y) אך במישורי z שונים.
Abstract
המחקר במדעי המוח התפתח לשימוש בכלים מורכבים של הדמיה וחישוב כדי לחלץ מידע מקיף ממערכי נתונים. הדמיית סידן היא טכניקה נפוצה הדורשת תוכנה מתוחכמת כדי להשיג תוצאות אמינות, אך מעבדות רבות מתקשות לאמץ שיטות חישוביות בעת עדכון פרוטוקולים כדי לעמוד בתקנים מודרניים. קשיים מתעוררים עקב חוסר ידע בתכנות וחומות תשלום עבור תוכנה. בנוסף, תאים מעניינים מציגים תנועות לכל הכיוונים במהלך הדמיית סידן. גישות רבות פותחו כדי לתקן את התנועה בכיוון הצידי (x/y).
מאמר זה מתאר זרימת עבודה באמצעות תוסף ImageJ חדש, TrackMate Analysis of Calcium Imaging (TACI), כדי לבחון תנועה על ציר z בדימות סידן תלת-ממדי. תוכנה זו מזהה את ערך הפלואורסצנטיות המרבי מכל מיקומי z שבהם תא עצב מופיע ומשתמשת בו כדי לייצג את עוצמת תא העצב במיקום t המתאים. לכן, הכלי הזה יכול להפריד תאי עצב החופפים בכיוון הצידי (x/y) אך מופיעים במישורי z נפרדים. כתוסף ImageJ, TACI הוא כלי חישובי ידידותי למשתמש בקוד פתוח לניתוח הדמיית סידן תלת-ממדית. תיקפנו את זרימת העבודה הזו באמצעות תאי עצב רגישים לחום של זחלי זבובים שהציגו תנועות לכל הכיוונים במהלך תנודות טמפרטורה ומערך נתונים תלת-ממדי של דימות סידן שנרכש ממוח הזבוב.
Introduction
רמת הסידן התוך-תאי היא סמן מדויק של עוררות עצבית. הדמיית סידן מודדת את השינויים בסידן התוך-תאי כדי להבין את הפעילות העצבית1. מחקרים במדעי המוח השתמשו יותר ויותר בשיטה זו בשל פיתוח טכניקות למדידת ריכוז סידן תוך תאי, כולל אינדיקטורים סידן מקודדים גנטית (GECIs), כגון GCaMP2,3, אשר יכול לבוא לידי ביטוי לא פולשני בקבוצות ספציפיות של נוירונים באמצעות גישות גנטיות. העלויות הנמוכות יותר של לייזרים ורכיבי מיקרוסקופ הגדילו גם את השימוש בדימות סידן4. חשוב לציין, דימות סידן מאפשר להקליט ולחקור נוירונים בודדים, כמו גם אוכלוסיות נוירונים גדולות בו זמנית בבעלי חיים הנעים בחופשיות5.
אף על פי כן, הניתוח של נתוני דימות סידן הוא מאתגר מכיוון ש-(1) הוא כרוך במעקב אחר השינויים בפלואורסצנטיות של תאים בודדים לאורך זמן, (2) האות הפלואורסצנטי נעלם לסירוגין או מופיע שוב עם תגובות עצביות, ו-(3) הנוירונים עשויים לנוע לכל הכיוונים, במיוחד פנימה והחוצה ממישור מוקד או להופיע במספר מישורים4, 6. ניתוח ידני גוזל זמן והופך ללא מעשי ככל שאורך ההקלטות ומספר תאי העצב גדל. תוכנות שונות פותחו כדי להאיץ את תהליך ניתוח הדמיית הסידן. בעבר, התוכנה תוכננה בהקשר ניסיוני מוגבל, מה שהקשה על מעבדות אחרות לאמץ אותה. מאמצים אחרונים לעמוד בסטנדרטים מודרניים לשיתוף תוכנה הובילו לפיתוח מספר כלים שיכולים לנתח באופן עקבי נתוני דימות סידן בקבוצות שונות 7,8,9,10,11,12,13,14,15,16,17,18,19 . עם זאת, רוב הכלים הללו דורשים ידע בתכנות ו/או תלויים בתוכנות מסחריות. מחסור בידע בתכנות וחומות תשלום בתוכנה מרתיעות חוקרים מלאמץ שיטות אלה. יתר על כן, רבים מהכלים הללו מתמקדים בתיקון תנועת x/y, אם כי גם תנועה על ציר z צריכה להיות מאובחנת ומתוקנת במפורש6. יש צורך בכלי חישובי לניתוח דימות סידן תלת-ממדי המתמקד בתאי עצב המציגים סחף Z ומופיעים במספר מישורי z. באופן אידיאלי, כלי זה צריך להשתמש בתוכנת קוד פתוח ולא לדרוש ידע בתכנות כדי לאפשר למעבדות אחרות לאמץ אותו בקלות.
כאן, פיתחנו תוסף ImageJ חדש, TACI, לניתוח נתוני הדמיה תלת ממדית של סידן. ראשית, התוכנה משנה את שמה, במידת הצורך, ומארגנת את נתוני הדמיית הסידן התלת-ממדית לפי מיקומי z. התאים המעניינים נמצאים במעקב בכל מיקום z, ועוצמות הפלואורסצנטיות שלהם מופקות על ידי TrackMate או כלים חישוביים אחרים. לאחר מכן מופעל TACI כדי לבחון את התנועה על ציר z. הוא מזהה את הערך המרבי של ערימת z ומשתמש בו כדי לייצג את עוצמת התא בנקודת הזמן המתאימה. תהליך עבודה זה מתאים לניתוח דימות סידן תלת-ממדי עם תנועה לכל הכיוונים ו/או עם תאי עצב החופפים בכיוון הצידי (x/y) אך מופיעים במיקומי z שונים. כדי לאמת את זרימת העבודה הזו, נעשה שימוש במערכי נתונים תלת-ממדיים של דימות סידן מתאי עצב רגישים לחום של זחלי זבובים ונוירונים של פטריות במוח. שימו לב, TACI הוא תוסף קוד פתוח ImageJ ואינו דורש ידע בתכנות.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. הדמיית סידן
- הכנת זחלי זבובים
הערה: זבובים וזחלים נשמרים ב 25 °C (75 °F) תחת 12 שעות: 12 שעות אור: מחזור כהה.- מרדימים את הזבובים עם CO2. מיין 20-45 זכרים ו 20-45 נקבות לתוך כל בקבוקון זבוב, ולתת להם לפחות 24 שעות עד 48 שעות כדי להתאושש מהחשיפה CO2 .
הערה: חשיפה לזבובים ל-CO2 אמורה להימשך פרק הזמן הקצר ביותר האפשרי. - כדי לסנכרן את גיל הזחלים, טפחו על הזבובים לבקבוקונים חדשים המכילים גרגרי שמרים, ואפשרו להם 4-8 שעות להטיל ביצים. הסר את הזבובים על ידי הפיכתם לבקבוקונים חדשים.
- לאסוף את הזחלים ב 72 שעות באמצעות 10 מ"ל של 20% w/v תמיסת סוכרוז.
- מרדימים את הזבובים עם CO2. מיין 20-45 זכרים ו 20-45 נקבות לתוך כל בקבוקון זבוב, ולתת להם לפחות 24 שעות עד 48 שעות כדי להתאושש מהחשיפה CO2 .
- הגדרת מיקרוסקופ ובקרת טמפרטורה
- בצע את ההדמיה במיקרוסקופ קונפוקלי (ראה טבלת חומרים) ובשלב piezo ציר z עם תוספת במה באמצעות ההגדרות הבאות: לייזר, ארגון; מצב סריקה, מסגרת; גודל מסגרת, 512 x 512; מהירות, מקסימום; ערוצים/עומק סיביות, 1/8 סיביות; זום, 1.5; z-stack, פרוסה = 15, שמור = פרוסה; מכשירי מיקוד ואסטרטגיה, מיקוד מוגדר; מיקוד, דגש מובהק על.
- חבר מודול קירור פלטייה למפזר חום על ידי תרכובת העברת החום כדי לבנות מצנן תרמואלקטרי. הפלטייה מופעל על ידי ספק כוח 2 A.
- חבר מיקרו-גשושית תרמית למכשיר לרכישת נתונים כדי להקליט את הטמפרטורה.
- הדמיית סידן
- שטפו את הזחלים 3x במי מלח חוצצים פוספט (PBS).
- פיפטה 75 μL של 1x PBS על מרכז מגלשת זכוכית.
- שים זחל אחד או שניים ב 1x PBS ומניחים את microprobe thermocouple ליד הזחלים.
הערה: המרחק בין הזחלים לבין microprobe צריך להיות ~ 5 מ"מ. הזחלים עשויים לנוע אם המיקרו-גשושית ממוקמת קרוב מדי אליהם. מרחק גדול עלול לגרום לקריאות טמפרטורה לא מדויקות. - כסו את הזחלים ואת המיקרו-פרוב התרמו-זוגי בכיסוי זכוכית. יש לאטום את הכיסוי בלק.
- הניחו את השקופית על במת המיקרוסקופ, מצאו את הפוקוס באמצעות מטרה של 25x, והניחו את הצידנית התרמואלקטרית על השקופית.
הערה: הפלטייה ממוקמת ישירות על המגלשה שבה הזחלים אמורים לספק את גירויי הטמפרטורה. - בתוכנות קונפוקליות, התמקדו בתאים הפלואורסצנטיים המעניינים. התאם את עוצמת הלייזר כדי למנוע רוויית יתר. קבעו את מיקום הפרוסה הראשונה והאחרונה בקביעת z-stack.
הערה: השתמש בעוצמת הלייזר הנמוכה ביותר האפשרית לפני ההקלטה כדי למנוע הלבנה. - התחל את סריקת z-stack והקלטת הטמפרטורה בו-זמנית.
- שלוט בספק הכוח המניע את Peltier לשנות את טמפרטורת פני השטח של Peltier. הפעל את ספק הכוח כדי להקטין את הטמפרטורה וכבה אותו כדי להעלות את הטמפרטורה.
- עצור את סריקת z-stack ואת רישום הטמפרטורה.
2. ניתוח נתוני הדמיה תלת ממדית של סידן
- יצא את נתוני דימות הסידן לקובצי TIFF ושמור אותם בתיקייה בעלת שם זהה לשם הבסיס של קובצי TIFF שבתוכו.
הערה: שם הקובץ אינו יכול להכיל פסיקים.- השתמש בפרמטרים הבאים כדי לייצא את נתוני הדמיית הסידן מ-ZEN (מהדורה שחורה): סוג קובץ, TIFF; דחיסה, LZW; ערוצים, 2; מיקום Z, כל; זמן, הכל; שלב, 1; אזור, מלא.
- התקנה
- הורד את TACI-Calcium_Imaging.jar מ- Github (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- התקן את התוסף בפיג'י על ידי לחיצה על תוספים בשורת התפריטים ולאחר מכן לחיצה על התקן בתפריט הנפתח (תוספים | התקן). לאחר מכן, הפעל מחדש את FIJI.
הערה: אל תשתמש בתוספים | התקן תוסף. - הפעל את התוסף על ידי לחיצה על תוספים ולאחר מכן בחירה ב- TACI-Calcium Imaging (תוספים | TACI-Calcium Imaging).
- השתמש בפונקציה RENAME כדי להמיר את שמות הקבצים של TIFF למבנה הנדרש.
הערה: הכלי משתמש ב- filename_h#t#z#c#.tif כמבנה ברירת המחדל (h# ו- c# הם אופציונליים; #: מספר שלם חיובי). אם שמות קובצי התמונות אינם במבנה ברירת המחדל, יש להפעיל את הפונקציה RENAME .- בחר את התיקייה שבה יש לשנות את שמן של תמונות TIFF על-ידי לחיצה על עיון בתיקיות.
- מלא את פרטי הפרמטר. חמישה פרמטרים מפורטים, כולל שם קובץ, פאזה, מיקום T מקסימלי, מיקום Z מרבי וערוץ. לכל פרמטר יש שלושה ערכים: טקסט קודם, ערך פרמטר וסדר.
הערה: המידע תלוי רישיות. אם שמות הקבצים של התמונות כוללים צירוף מילים לפני פרמטר, מלא את צירוף המילים כטקסט הקודם של הפרמטר המתאים. אם שמות הקבצים של התמונות מכילים ביטוי אחרי כל הפרמטרים, מלא את צירוף המילים כטקסט הפרסום.- הקפד להזין Filename, Max T-Position ו- Max Z-Position ושערכי הפרמטרים של Max T-Position ו- Max Z-Position כוללים את כל הספרות.
- המתן עד ששם הקובץ ימולא באופן אוטומטי באמצעות שם התיקיה.
- אם שמות הקבצים של TIFF אינם כוללים את הפאזה והערוץ, השאר את ערך הפרמטר המתאים ריק ובחר Na לפי הסדר.
- לחץ על שנה שם כדי ליצור תיקיה באותו שם ובאותו _r. שים לב שבתיקייה, שמות הקבצים של TIFF נבנו מחדש כך שיהיו תואמים לפונקציה ORGAN .
הערה: _r נוסף לשמות הקבצים של תמונות TIFF.
- השתמש בפונקציה ORGANIZE כדי לשמור את תמונות TIFF מאותו מיקום z בתיקייה אחת.
הערה: שם התיקיה חייב להיות זהה לשם הבסיס של קבצי TIFF שבתוכו ואסור שיכלול פסיקים.- בחר את התיקיה שבה יש לארגן את תמונות TIFF על-ידי לחיצה על עיון בתיקיות.
- אם קובץ CSV של הפרמטר (param.csv) קיים, המתן עד שערכי הפרמטרים ימולאו אוטומטית.
הערה: לקובץ param.csv יש תבנית נדרשת. יש למלא את הפרמטרים, כולל שם קובץ, שלב, position_t, position_z, ערוץ ו- is_gray, בשורה 1 משמאל לימין החל מעמודה A. יש למלא את הערכים המתאימים עבור כל פרמטר בשורה 2. - אם קובץ ה- CSV של הפרמטר (param.csv) אינו קיים, מלא ידנית את ערכי הפרמטרים. ודא שערכי הפרמטרים של Phase ו- Channel כוללים אותיות, בעוד שערכי הפרמטרים של T Position ו- Z Position צריכים להיות המספרים הגדולים ביותר של מיקומי t ו- z. אם שמות הקבצים של התמונות אינם כוללים את הפאזה או הערוץ, הזינו Na.
- צרו תמונות TIFF בגווני אפור בעת הצורך. השאר את התיבה האם תמונות אפורות? לא מסומנות בגווני אפור של התמונות.
- לחץ על ארגן כדי ליצור תיקיה בעלת שם _gray_stacks זהים וכדי ליצור תיקיות באותו שם ו- _# (#: z-positions) בתיקיה. שים לב שקבצי TIFF ממוינים לתיקיות מתאימות לפי מיקומי z וכי נוצר קובץ בשם param.csv, שבו ניתן למצוא את הפרמטרים ואת ערכיהם.
- השתמש ב- TrackMate ב- FIJI כדי לחלץ את עוצמות הפלואורסצנטיות של התאים המעניינים מכל מיקום z.
הערה: שלב זה יכול להתבצע גם על-ידי תוכנות הדמיה אחרות.- פתח את תמונות TIFF בתיקייה z-position של FIJI.
- הפעל את TrackMate על ידי לחיצה על תוספים | מעקב | TrackMate, והתאם את הפרמטרים הבאים במידת הצורך.
- השתמש בגלאי DoG או LoG.
- שנה את קוטר הכתמים, הסף והמסנן החציוני. התאם את קוטר הכתמים כך שיהיה דומה לקוטר התאים. אם התאים סגלגלים, התאימו את קוטר הכתמים כך שיהיה דומה לציר המינורי.
הערה: הגדלת הסף מסייעת למנוע מרעשי רקע להיקלט כאותות מבלי להשפיע על עוצמות האות (איור משלים 1). עם זאת, עלייה בסף עלולה להחמיץ איתותים אמיתיים (תרשים משלים 1). אם האותות חזקים, השתמש במסנן החציוני כדי להפחית את רעש המלח והפלפל. - הגדר את המסננים כדי להסיר חלק מהאותות הלא רלוונטיים, אם לא את כולם. מסננים X ו- Y הם מסננים מרחביים. השתמש במסננים X ו- Y כדי להסיר את האותות הלא רלוונטיים הרחוקים מהאותות האמיתיים.
הערה: כאשר מסננים מוגדרים על תמונה אחת, חיוני לבדוק את כל התמונות האחרות כדי להבטיח שהאותות האמיתיים לא יוסרו. - הגדר את המרחק המרבי המקשר, המרחק המרבי לסגירת הפער ומרווח המסגרת המרבי לסגירת הפער. הגדר את המרחק המרבי המקשר ואת המרחק המרבי לסגירת פער להיות פי 3-5 מקוטר הכתם, במיוחד כאשר הדגימות נעות באופן משמעותי לאורך זמן, כדי לסייע בהפחתת מספר המסלולים. קבעו את מרווח המסגרות המרבי לסגירת רווח למספר התמונות בערימה.
- יצא את עוצמות הפלואורסצנטיות של אזורי העניין (ROI) לקובץ CSV. אם נעשה שימוש בגרסת TrackMate ישנה, בחרו ' יצא את כל סטטיסטיקות הספוט ' בחלון 'בחר פעולה '. אם גרסת TrackMate היא 7.6.1 ומעלה, בחרו ב'ספוטים' בחלון אפשרויות תצוגה . יצא או שמור כקבצים אינטראקטיביים בקובצי CSV.
הערה: שני הקבצים אינטראקטיביים עם חלון התמונה; הדגשת החזר השקעה מציגה את החזר ההשקעה המתאים בחלון התמונה. קבצים אלה כוללים את העוצמות הממוצעות (MEAN_INTENSITY או MEAN_INTENSITY_CH1) של התאים המעניינים בנקודות הזמן המתאימות (POSITION_T). אותו TRACK_IDs אמור לייצג את אותם ROI בנקודות זמן שונות. עם זאת, זה לא תמיד נכון וייתכן שיהיה צורך לתקן באופן ידני, בעת הצורך. אם TrackMate אינו מזהה את החזר ההשקעה בנקודות זמן מסוימות, נקודות זמן אלה אינן מוצגות.
- חלץ את עוצמת הפלואורסצנטיות ברקע וצור את קובץ Background_list.csv.
הערה: במחקר זה, עוצמת הרקע עבור כל מיקום z הוערכה באמצעות הערך הממוצע של שלושה עד חמישה כתמים שכנים באותו גודל שלא הכילו אותות פלואורסצנטיים והיו מנקודות זמן שונות.- לקובץ Background_list.csv יש תבנית נדרשת: כל עמודה מכילה את המידע של תא עצב אחד, החל מנוירון 0. מלא את מספרי תאי העצב, כגון נוירון 0, בשורה 1. לאחר מכן, ספק את עוצמת הרקע עבור כל מיקום z שנותח - אם מנותחים חמישה מיקומי z עבור נוירון 0, מלא חמישה ערכי עוצמת רקע מתחת לנוירון 0.
הערה: קובץ Background_list.csv נדרש עבור הפונקציה TACI EXTRACT . אם הרקע זניח, יש לספק את Background_list.csv עם עוצמת הרקע האפסית של כל נוירון.
- לקובץ Background_list.csv יש תבנית נדרשת: כל עמודה מכילה את המידע של תא עצב אחד, החל מנוירון 0. מלא את מספרי תאי העצב, כגון נוירון 0, בשורה 1. לאחר מכן, ספק את עוצמת הרקע עבור כל מיקום z שנותח - אם מנותחים חמישה מיקומי z עבור נוירון 0, מלא חמישה ערכי עוצמת רקע מתחת לנוירון 0.
- השתמש בפונקציה EXTRACT כדי לזהות את העוצמות הפלואורסצנטיות המרביות בכל מיקום t ולחשב ΔF/F0 כפי שמוצג במשוואה (1).
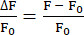 (1)
(1)- צור תיקייה ותן לה שם באמצעות מספר תא העצב, החל מנוירון 0. שמור את קובצי ה- CSV המכילים את המידע הפלואורסצנטי של תא העצב המתאים בתיקיה. כל קובץ CSV מכיל את המידע של מיקום z אחד, כך שמספר קבצי CSV שווה למספר מיקומי z שנותחו. תן לקובצי ה- CSV את השם Mean_Intensity#.csv (# מייצג את מיקום z) וכל קובץ CSV כולל לפחות שתי עמודות: POSITION_T ו- MEAN_INTENSITY או MEAN_INTENSITY_CH1.
הערה: צור תיקיה עבור כל תא מעניין. - שמור את Background_list.csv הקבצים והתיקיות שנוצרו בשלב 2.7.1 בתיקיה אחת. בחר תיקיה זו על ידי לחיצה על עיון בקבצים.
הערה: מספר ערכי הרקע בקובץ Background_list.csv חייב להתאים למספר קבצי Mean_Intensity#.csv עבור כל תא עצב. - קובץ הרקע מתמלא באופן אוטומטי. מלא את המספר הגדול ביותר של עמדות t עבור מספר עמדות T.
- לחץ על Extract כדי ליצור תיקיית תוצאות, כולל קובצי CSV ותרשימים לכל תא עצב. קובצי CSV כוללים מידע על עוצמת הפלואורסצנטיות המרבית ועל ΔF/F0 בכל מיקום t. התרשימים הם תרשימי קו של ΔF/F0 מעל מיקומי t.
הערה: במחקר זה, ΔF/F0 חושב באמצעות משוואה (1). הערך הראשון של כל מיקום z שימש כ-F0. אם F0 זה אינו מתאים20, התוסף מספק קבצים הכוללים נתונים גולמיים עבור כל נוירון בתיקיית python_files.
- צור תיקייה ותן לה שם באמצעות מספר תא העצב, החל מנוירון 0. שמור את קובצי ה- CSV המכילים את המידע הפלואורסצנטי של תא העצב המתאים בתיקיה. כל קובץ CSV מכיל את המידע של מיקום z אחד, כך שמספר קבצי CSV שווה למספר מיקומי z שנותחו. תן לקובצי ה- CSV את השם Mean_Intensity#.csv (# מייצג את מיקום z) וכל קובץ CSV כולל לפחות שתי עמודות: POSITION_T ו- MEAN_INTENSITY או MEAN_INTENSITY_CH1.
- השתמש בפונקציית MERGE כדי לחשב ממוצע ΔF/F 0 של כל נוירון, לחשב את ה- SEM ולשרטט את הממוצע ΔF/F0 על מיקומי t.
- בחר את תיקיית התוצאות שנוצרה על ידי הפונקציה EXTRACT על ידי לחיצה על עיון בקבצים.
- מלא את מספר עמדות T עם המספר הגדול ביותר של עמדות t.
- לחץ על מזג כדי ליצור תיקיית merged_data, כולל קובץ merged_data.csv והתוויית Average_dF_F0.png. קובץ ה- CSV כולל את המידע על הממוצע ו- SEM ΔF/F0 בכל מיקום t. העלילה היא תרשים קו של ממוצע ΔF/F0 מעל מיקומי t.
Access restricted. Please log in or start a trial to view this content.
תוצאות
זרימת עבודה של ניתוח הדמיית סידן תלת-ממדית
במחקר הזה פיתחנו תוסף חדש של ImageJ, TACI, ותיארנו זרימת עבודה למעקב אחר z-drift ולניתוח הדמיית סידן תלת-ממדית שמאתרת את התגובות של תאים בודדים המופיעים במיקומי z מרובים (איור 1). כלי זה כולל ארבע פונקציות: שינוי שם, א?...
Access restricted. Please log in or start a trial to view this content.
Discussion
מחקר זה פיתח תוסף חדש של ImageJ, TACI, ותיאר זרימת עבודה המנתחת הדמיית סידן תלת-ממדית. כלים רבים הזמינים כיום מתמקדים בתיקון תנועת x/y, אם כי גם תנועה על ציר z צריכה להיות מאובחנת במפורש או מתוקנת6. במהלך רכישת תמונה באורגניזם חי, תנועה על ציר z היא בלתי נמנעת גם כאשר האורגניזם משותק, וגי...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
Zeiss LSM 880 במרכז ההדמיה Fralin שימש לאיסוף נתוני הדמיית הסידן. אנו מודים לד"ר מישל ל. אולסן ויוהנג פאן על עזרתם בתוכנת IMARIS. אנו מודים לד"ר לנווד ס. הית' על הערות בונות על כתב היד ולסטיבן ג'יאווסיס על הערות על קובץ ה-README של GitHub. עבודה זו נתמכה על ידי NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) ו- NIH R01GM140130 (https://www.nigms.nih.gov/) ל- L.N. למממנים לא היה כל תפקיד בעיצוב המחקר, באיסוף הנתונים ובניתוחם, בהחלטה על פרסומם או בהכנת כתב היד.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
References
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258(2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517(2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173(2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153(2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25(2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432(2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569(2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728(2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869(2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279(2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254(2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252(2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved