Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
TACI: плагин ImageJ для 3D-анализа кальциевых изображений
В этой статье
Резюме
TrackMate Analysis of Calcium Imaging (TACI) — это плагин ImageJ с открытым исходным кодом для 3D-анализа кальциевых изображений, который исследует движение по оси Z и определяет максимальное значение каждого z-стека для представления интенсивности ячейки в соответствующий момент времени. Он может разделять нейроны, перекрывающиеся в латеральном (x/y) направлении, но в разных z-плоскостях.
Аннотация
Исследования в области нейробиологии эволюционировали, чтобы использовать сложные инструменты визуализации и вычислений для извлечения исчерпывающей информации из наборов данных. Кальциевая визуализация является широко используемым методом, который требует сложного программного обеспечения для получения надежных результатов, но многие лаборатории изо всех сил пытаются использовать вычислительные методы при обновлении протоколов в соответствии с современными стандартами. Трудности возникают из-за отсутствия знаний в области программирования и платного доступа к программному обеспечению. Кроме того, интересующие клетки демонстрируют движения во всех направлениях во время визуализации кальция. Было разработано множество подходов для коррекции движения в боковом (x/y) направлении.
В этой статье описывается рабочий процесс с использованием нового плагина ImageJ, TrackMate Analysis of Calcium Imaging (TACI), для изучения движения по оси z в 3D-визуализации кальция. Это программное обеспечение определяет максимальное значение флуоресценции из всех z-позиций, в которых появляется нейрон, и использует его для представления интенсивности нейрона в соответствующем t-положении. Следовательно, этот инструмент может разделять нейроны, перекрывающиеся в латеральном направлении (x/y), но появляющиеся на разных z-плоскостях. Как плагин ImageJ, TACI представляет собой удобный вычислительный инструмент с открытым исходным кодом для 3D-анализа кальциевых изображений. Мы проверили этот рабочий процесс, используя термочувствительные нейроны личинок мух, которые отображали движения во всех направлениях во время колебаний температуры, и набор данных 3D-визуализации кальция, полученный из мозга мухи.
Введение
Уровень внутриклеточного кальция является точным маркером возбудимости нейронов. Визуализация кальция измеряет изменения внутриклеточного кальция, чтобы понять активность нейронов1. Исследования в области нейробиологии все чаще используют этот метод в связи с разработкой методов измерения внутриклеточной концентрации кальция, включая генетически кодируемые индикаторы кальция (GECI), такие как GCaMP2,3, которые могут быть неинвазивно экспрессированы в определенных наборах нейронов с помощью генетических подходов. Более низкие затраты на лазеры и компоненты микроскопов также увеличили использование кальциевой визуализации4. Важно отметить, что визуализация кальция позволяет одновременно регистрировать и изучать отдельные нейроны, а также большие популяции нейронов у свободно перемещающихся животных5.
Тем не менее, анализ данных визуализации кальция является сложной задачей, потому что (1) он включает в себя отслеживание изменений флуоресценции отдельных клеток с течением времени, (2) сигнал флуоресценции периодически исчезает или снова появляется с реакциями нейронов, и (3) нейроны могут двигаться во всех направлениях, особенно в фокальной плоскости и из нее или появляться в нескольких плоскостях4, 6. Ручной анализ отнимает много времени и становится непрактичным по мере увеличения длины записей и количества нейронов. Для ускорения процесса анализа кальциевых изображений были разработаны различные программы. Ранее программное обеспечение разрабатывалось в ограниченном экспериментальном контексте, что затрудняло его внедрение другими лабораториями. Недавние усилия по соблюдению современных стандартов совместного использования программного обеспечения привели к разработке нескольких инструментов, которые могут последовательно анализировать данные визуализации кальция в разных группах 7,8,9,10,11,12,13,14,15,16,17,18,19 . Однако большинство из этих инструментов требуют знаний в области программирования и/или зависят от коммерческого программного обеспечения. Отсутствие знаний в области программирования и платный доступ к программному обеспечению удерживают исследователей от принятия этих методов. Более того, многие из этих инструментов сосредоточены на коррекции движения x/y, хотя движение по оси z также нуждается в явной диагностике и коррекции6. Существует потребность в вычислительном инструменте для анализа 3D-изображений кальция, который фокусируется на нейронах, демонстрирующих z-дрейф и появляющихся на нескольких z-плоскостях. В идеале этот инструмент должен использовать программное обеспечение с открытым исходным кодом и не требовать знаний в области программирования, чтобы другие лаборатории могли легко его принять.
Здесь мы разработали новый плагин ImageJ, TACI, для анализа данных 3D-визуализации кальция. Во-первых, программное обеспечение переименовывает, если это необходимо, и упорядочивает данные 3D-визуализации кальция по z-позициям. Интересующие ячейки отслеживаются в каждой z-позиции, а их интенсивность флуоресценции извлекается с помощью TrackMate или других вычислительных инструментов. Затем применяется TACI для изучения движения по оси z. Он определяет максимальное значение z-стека и использует его для представления интенсивности ячейки в соответствующий момент времени. Этот рабочий процесс подходит для анализа 3D-визуализации кальция с движением во всех направлениях и/или с нейронами, перекрывающимися в боковом (x/y) направлении, но появляющимися в разных z-положениях. Для проверки этого рабочего процесса были использованы наборы данных 3D-визуализации кальция из термочувствительных нейронов личинок мух и грибовидных нейронов в головном мозге. Следует отметить, что TACI является плагином ImageJ с открытым исходным кодом и не требует каких-либо знаний в области программирования.
протокол
1. Кальциевая визуализация
- Подготовка личинок мух
ПРИМЕЧАНИЕ: Мухи и личинки поддерживаются при температуре 25 ° C в цикле 12 ч: 12 ч свет: темнота.- Обезболивайте мухСО2. Отсортируйте 20-45 самцов и 20-45 самок в каждый флакон с мухой и дайте им не менее 24-48 часов, чтобы оправиться от воздействия CO2 .
ПРИМЕЧАНИЕ: Воздействие CO2 на мух должно длиться как можно более короткий промежуток времени. - Чтобы синхронизировать возраст личинок, постучите мух по новым флаконам, содержащим гранулы дрожжей, и дайте им отложить яйца в течение 4-8 часов. Удалите мух, перевернув их в новые флаконы.
- Соберите личинок через 72 часа, используя 10 мл 20% раствора сахарозы по массе.
- Обезболивайте мухСО2. Отсортируйте 20-45 самцов и 20-45 самок в каждый флакон с мухой и дайте им не менее 24-48 часов, чтобы оправиться от воздействия CO2 .
- Настройка микроскопа и контроля температуры
- Выполните визуализацию на конфокальном микроскопе (см. Таблицу материалов) и пьезостолике по оси Z со вставкой предметного столика, используя следующие настройки: лазер, аргон; режим сканирования, кадр; размер кадра, 512 x 512; скорость, максимальная; каналы/битовая глубина, 1/8 бит; зум, 1,5; z-стек, срез = 15, Keep = срез; фокусировочные устройства и стратегия, Определенная направленность; фокус, Определенная направленность на.
- Прикрепите охлаждающий модуль Пельтье к радиатору с помощью теплопередающего компаунда, чтобы построить термоэлектрический охладитель. Пельтье питается от блока питания 2 А.
- Прикрепите микрозонд термопары к устройству сбора данных для записи температуры.
- Визуализация кальция
- Промойте личинок 3 раза в 1x фосфатно-буферном физиологическом растворе (PBS).
- Пипетка 75 мкл 1x PBS в центр предметного стекла.
- Поместите одну или две личинки в 1x PBS и поместите микрозонд термопары рядом с личинками.
ПРИМЕЧАНИЕ: Расстояние между личинками и микрозондом должно составлять ~5 мм. Личинки могут двигаться, если микрозонд расположен слишком близко к ним. Большое расстояние может привести к неточным показаниям температуры. - Накройте личинки и микрозонд термопары стеклянным покровным стеклом. Заклейте покровное стекло лаком для ногтей.
- Поместите предметное стекло на предметный столик микроскопа, найдите фокус с помощью 25-кратного объектива и поместите термоэлектрический охладитель на предметное стекло.
ПРИМЕЧАНИЕ: Пельтье помещается непосредственно на предметное стекло, где личинки должны доставлять температурные раздражители. - В конфокальном программном обеспечении сосредоточьтесь на интересующих флуоресцентных клетках. Отрегулируйте мощность лазера, чтобы избежать перенасыщения. Установите положение первого и последнего фрагментов в параметре z-stack.
ПРИМЕЧАНИЕ: Перед записью используйте минимально возможную мощность лазера, чтобы предотвратить фотообесцвечивание. - Запустите сканирование z-стека и запись температуры одновременно.
- Управляйте блоком питания, который питает Пельтье, чтобы изменить температуру поверхности Пельтье. Включите источник питания, чтобы снизить температуру, и выключите его, чтобы повысить температуру.
- Остановите сканирование z-стека и запись температуры.
2. Анализ данных 3D-визуализации кальция
- Экспортируйте данные кальциевых изображений в файлы TIFF и сохраните их в папке с тем же именем, что и базовое имя файлов TIFF внутри.
ПРИМЕЧАНИЕ: Имя файла не должно содержать запятых.- Используйте следующие параметры для экспорта данных кальциевых изображений из ZEN (черная версия): тип файла, TIFF; сжатие, LZW; каналы, 2; Z-позиция, все; время, все; фаза, 1; региональный, полный.
- Установка
- Загрузите TACI-Calcium_Imaging.jar с Github (https://github.com/niflylab/TACI_CalciumImagingPlugin/releases).
- Установите плагин на Фиджи, щелкнув «Плагины» в строке меню, а затем нажав «Установить» в раскрывающемся меню (Плагины | Установить). Затем перезапустите FIJI.
ПРИМЕЧАНИЕ: НЕ используйте плагины | Установите плагин. - Запустите плагин, нажав на Плагины, а затем выбрав TACI-Calcium Imaging (Плагины | TACI-кальциевая визуализация).
- Используйте функцию RENAME для преобразования имен файлов TIFF в требуемую структуру.
ПРИМЕЧАНИЕ: Инструмент использует filename_h#t#z#c#.tif в качестве структуры по умолчанию (h# и c# являются необязательными; #: положительное целое число). Если имена файлов изображений не имеют структуры по умолчанию, необходимо выполнить функцию RENAME .- Выберите папку, в которой необходимо переименовать изображения TIFF, нажав « Обзор папок».
- Заполните информацию о параметрах. Перечислены пять параметров, включая имя файла, фазу, максимальную T-позицию, максимальную Z-позицию и канал. Каждый параметр имеет три значения: «Предшествующий текст», «Значение параметра» и «Порядок».
ПРИМЕЧАНИЕ: Информация чувствительна к регистру. Если имена файлов изображений содержат фразу перед параметром, заполните фразу в виде предшествующего текста соответствующего параметра. Если имена файлов изображений содержат фразу после всех параметров, заполните фразу как текст сообщения.- Убедитесь, что вы ввели Filename, Max T-Position и Max Z-Position, а значения параметров для Max T-Position и Max Z-Position включают все цифры.
- Подождите, пока имя файла будет автоматически заполнено с использованием имени папки.
- Если имена файлов TIFF не включают фазу и канал, оставьте соответствующее значение параметра пустым и выберите «Na по порядку».
- Нажмите кнопку Переименовать , чтобы создать папку с таким же именем и _r. Обратите внимание, что в папке имена файлов TIFF были реструктурированы для совместимости с функцией ORGANIZE .
ПРИМЕЧАНИЕ: _r был добавлен к именам файлов изображений TIFF.
- Используйте функцию ORGANIZE для сохранения изображений TIFF из одной и той же z-позиции в одной папке.
ПРИМЕЧАНИЕ: Имя папки должно совпадать с базовым именем файлов TIFF внутри и НЕ должно содержать запятых.- Выберите папку, в которой необходимо упорядочить изображения TIFF, нажав «Обзор папок».
- Если CSV-файл параметра (param.csv) существует, дождитесь автоматического заполнения значений параметров.
ПРИМЕЧАНИЕ: Файл param.csv имеет требуемый формат. Параметры, включая имя файла, фазу, position_t, position_z, канал и is_gray, должны быть заполнены в строке 1 слева направо, начиная со столбца A. Соответствующие значения для каждого параметра должны быть заполнены в строке 2. - Если CSV-файл параметра (param.csv) не существует, заполните значения параметров вручную. Убедитесь, что значения параметров фазы и канала включают буквы, в то время как значения параметров положения T и положения Z должны быть наибольшими числами позиций t и z. Если имена файлов изображений не включают фазу или канал, введите Na.
- При необходимости создавайте изображения TIFF в оттенках серого. Не устанавливайте флажок Являются ли изображения серыми?, чтобы оттенки серого отображались.
- Нажмите кнопку Упорядочить, чтобы создать папку с тем же именем и _gray_stacks, а также создать папки с тем же именем и _# (#: z-позиции) в папке. Обратите внимание, что файлы TIFF сортируются по соответствующим папкам по z-позициям и что генерируется файл с именем param.csv, в котором можно найти параметры и их значения.
- Используйте TrackMate на Фиджи для извлечения интенсивности флуоресценции интересующих клеток из каждой z-позиции.
ПРИМЕЧАНИЕ: Этот шаг также может быть выполнен другим программным обеспечением для обработки изображений.- Откройте изображения TIFF в папке Z-position от FIJI.
- Запустите TrackMate, нажав « Плагины» | Отслеживание | TrackMate и при необходимости отрегулируйте следующие параметры.
- Используйте детекторы DoG или LoG.
- Измените диаметр большого двоичного объекта, пороговое значение и медианный фильтр. Отрегулируйте диаметр большого двоичного объекта так, чтобы он соответствовал диаметру ячеек. Если ячейки овальные, отрегулируйте диаметр большого двоичного объекта так, чтобы он был похож на малую ось.
ПРИМЕЧАНИЕ: Увеличение порогового значения помогает избежать восприятия фонового шума как сигнала, не влияя на интенсивность сигнала (дополнительный рисунок 1). Однако при увеличении порога истинные сигналы могут быть пропущены (дополнительный рисунок 1). Если сигналы сильные, используйте медианный фильтр , чтобы уменьшить шум соли и перца. - Установите фильтры, чтобы удалить некоторые, если не все, нерелевантные сигналы. Фильтры X и Y являются пространственными фильтрами. Используйте фильтры X и Y , чтобы удалить нерелевантные сигналы, которые далеки от реальных сигналов.
ПРИМЕЧАНИЕ: Когда фильтры установлены на одном изображении, очень важно проверить все остальные изображения, чтобы убедиться, что реальные сигналы не удаляются. - Установите максимальное расстояние связывания, максимальное расстояние закрытия зазора и максимальное расстояние закрытия зазора кадра. Установите максимальное расстояние связывания и максимальное расстояние закрытия зазора в 3–5 раз больше диаметра большого двоичного объекта, особенно когда образцы значительно перемещаются с течением времени, чтобы уменьшить количество дорожек. Установите максимальный зазор между кадрами и задайте количество изображений в стопке.
- Экспортируйте интенсивность флуоресценции интересующих областей (ROI) в CSV-файл. Если используется старая версия TrackMate, выберите « Экспортировать статистику всех пятен» в окне «Выберите действие ». Если версия TrackMate — 7.6.1 или выше, выберите « Пятна» в окне «Параметры отображения ». Экспортируйте или сохраняйте как интерактивные файлы в файлы CSV.
ПРИМЕЧАНИЕ: Оба файла взаимодействуют с окном изображения; выделение ROI отображает соответствующий ROI в окне изображения. Эти файлы включают среднюю интенсивность (MEAN_INTENSITY или MEAN_INTENSITY_CH1) интересующих ячеек в соответствующие моменты времени (POSITION_T). Одни и те же TRACK_IDs должны представлять одни и те же показатели рентабельности инвестиций в разные моменты времени. Однако это не всегда так, и, возможно, при необходимости потребуется исправить вручную. Если TrackMate не распознает рентабельность инвестиций в некоторые моменты времени, эти моменты времени не отображаются.
- Извлеките интенсивность фоновой флуоресценции и создайте файл Background_list.csv.
ПРИМЕЧАНИЕ: В этом исследовании интенсивность фона для каждой z-позиции оценивалась с использованием среднего значения трех-пяти соседних пятен одинакового размера, которые не содержали сигналов флуоресценции и находились в разных временных точках.- Файл Background_list.csv имеет обязательный формат: каждый столбец содержит информацию об одном нейроне, начиная с Neuron 0. Заполните номера нейронов, например Нейрон 0, в строке 1. Затем укажите интенсивность фона для каждой проанализированной z-позиции - если пять z-позиций анализируются для нейрона 0, заполните пять значений интенсивности фона под нейроном 0.
ПРИМЕЧАНИЕ: Файл Background_list.csv необходим для функции TACI EXTRACT . Если фон пренебрежимо мал, необходимо обеспечить Background_list.csv с нулевой интенсивностью фона каждого нейрона.
- Файл Background_list.csv имеет обязательный формат: каждый столбец содержит информацию об одном нейроне, начиная с Neuron 0. Заполните номера нейронов, например Нейрон 0, в строке 1. Затем укажите интенсивность фона для каждой проанализированной z-позиции - если пять z-позиций анализируются для нейрона 0, заполните пять значений интенсивности фона под нейроном 0.
- Используйте функцию EXTRACT , чтобы определить максимальную интенсивность флуоресценции в каждом t-положении и вычислить ΔF/F0, как показано в уравнении (1).
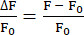 (1)
(1)- Создайте папку и назовите ее, используя номер нейрона, начиная с Neuron 0. Сохраните CSV-файлы, содержащие информацию о флуоресценции соответствующего нейрона, в папке. Каждый CSV-файл содержит информацию об одной z-позиции, поэтому количество CSV-файлов равно количеству проанализированных z-позиций. Назовите CSV-файлы Mean_Intensity#.csv (# обозначает z-позицию), и каждый CSV-файл будет содержать как минимум два столбца: POSITION_T и MEAN_INTENSITY или MEAN_INTENSITY_CH1.
ПРИМЕЧАНИЕ: Создайте папку для каждой интересующей ячейки. - Сохраните файл Background_list.csv и папки, созданные на шаге 2.7.1, в одной папке. Выберите эту папку, нажав « Обзор файлов».
ПРИМЕЧАНИЕ: Количество фоновых значений в файле Background_list.csv должно совпадать с количеством файлов Mean_Intensity#.csv для каждого нейрона. - Фоновый файл заполняется автоматически. Заполните наибольшее количество t-позиций для количества t-позиций.
- Нажмите кнопку «Извлечь», чтобы создать папку результатов, включающую CSV-файлы и графики для каждого нейрона. Файлы CSV содержат информацию о максимальной интенсивности флуоресценции и ΔF/F0 в каждом t-положении. Графики представляют собой линейные диаграммы ΔF/F0 над t-позициями.
ПРИМЕЧАНИЕ: В этом исследовании ΔF/F0 был рассчитан с использованием уравнения (1). Первое значение каждой z-позиции использовалось как F0. Если этот F0 не подходит20, плагин предоставляет файлы, включающие необработанные данные для каждого нейрона в папке python_files.
- Создайте папку и назовите ее, используя номер нейрона, начиная с Neuron 0. Сохраните CSV-файлы, содержащие информацию о флуоресценции соответствующего нейрона, в папке. Каждый CSV-файл содержит информацию об одной z-позиции, поэтому количество CSV-файлов равно количеству проанализированных z-позиций. Назовите CSV-файлы Mean_Intensity#.csv (# обозначает z-позицию), и каждый CSV-файл будет содержать как минимум два столбца: POSITION_T и MEAN_INTENSITY или MEAN_INTENSITY_CH1.
- Используйте функцию MERGE для усреднения ΔF/F 0 каждого нейрона, расчета SEM и построения среднего значения ΔF/F0 по t-позициям.
- Выберите папку результатов, созданную функцией EXTRACT , нажав « Обзор файлов».
- Заполните количество t-позиций наибольшим количеством t-позиций.
- Нажмите кнопку «Объединить», чтобы создать папку merged_data, включающую файл merged_data.csv и Average_dF_F0.png график. Файл CSV содержит информацию о среднем значении и SEM ΔF/F0 в каждой t-позиции. График представляет собой линейный график среднего значения ΔF/F0 по t-позициям.
Результаты
Рабочий процесс 3D-анализа кальциевых изображений
В этом исследовании мы разработали новый плагин ImageJ, TACI, и описали рабочий процесс для отслеживания z-дрейфа и анализа 3D-визуализации кальция, который точно определяет реакцию отдельных клеток, появляющихся в нескольких z-пози...
Обсуждение
В этом исследовании был разработан новый плагин ImageJ, TACI, и описан рабочий процесс, анализирующий 3D-визуализацию кальция. Многие доступные в настоящее время инструменты сосредоточены на коррекции движения x/y, хотя движение по оси z также нуждается в явной диагностике или коррекции
Раскрытие информации
У авторов нет конфликтов интересов, которые необходимо раскрывать.
Благодарности
Zeiss LSM 880 в Центре визуализации Фралина использовался для сбора данных визуализации кальция. Мы выражаем благодарность доктору Мишель Л. Олсен и Юхану Пану за помощь в разработке программного обеспечения IMARIS. Выражаем благодарность д-ру Ленвуду С. Хиту (Lenwood S. Heath) за конструктивные комментарии к рукописи и Стивену Джавасису (Steven Giavasis) за комментарии к файлу сведений GitHub. Эта работа была поддержана NIH R21MH122987 (https://www.nimh.nih.gov/index.shtml) и NIH R01GM140130 (https://www.nigms.nih.gov/) в L.N. Спонсоры не играли никакой роли в дизайне исследования, сборе и анализе данных, принятии решения о публикации или подготовке рукописи.
Материалы
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
Ссылки
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258 (2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517 (2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173 (2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153 (2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25 (2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432 (2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569 (2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728 (2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279 (2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254 (2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252 (2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены