A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تصوير الخلايا الحية لذبابة الفاكهة الميلانوجاستر أدمغة اليرقات الثالثة
In This Article
Summary
هنا ، نناقش سير العمل لإعداد وتشريح وتركيب وتصوير أدمغة الزرع الحية من يرقات ذبابة الفاكهة الميلانوجاستر الثالثة لمراقبة الديناميكيات الخلوية ودون الخلوية في ظل الظروف الفسيولوجية.
Abstract
تخضع الخلايا الجذعية العصبية ذبابة الفاكهة (الخلايا العصبية ، NBs فيما بعد) لانقسامات غير متماثلة ، مما يؤدي إلى تجديد الخلايا العصبية ذاتية التجديد ، بينما تشكل أيضا خلية أم متمايزة (GMC) ، والتي ستخضع لتقسيم إضافي واحد ليؤدي إلى ظهور خليتين عصبيتين أو دبقية. كشفت الدراسات في NBs عن الآليات الجزيئية الكامنة وراء قطبية الخلية ، واتجاه المغزل ، والتجديد الذاتي للخلايا الجذعية العصبية ، والتمايز. يمكن ملاحظة هذه الانقسامات الخلوية غير المتماثلة بسهولة عن طريق تصوير الخلايا الحية ، مما يجعل اليرقات NBs مناسبة بشكل مثالي للتحقيق في الديناميات الزمانية المكانية لانقسام الخلايا غير المتماثل في الأنسجة الحية. عند تشريحها وتصويرها بشكل صحيح في وسط مكمل بالمغذيات ، تنقسم NBs في أدمغة الزرع بقوة لمدة 12-20 ساعة. الأساليب الموصوفة سابقا صعبة تقنيا وقد تشكل تحديا لأولئك الجدد في هذا المجال. هنا ، يتم وصف بروتوكول لإعداد وتشريح وتركيب وتصوير نباتات دماغ اليرقات الحية الثالثة باستخدام مكملات الجسم الدهنية. كما تتم مناقشة المشاكل المحتملة ، ويتم تقديم أمثلة لكيفية استخدام هذه التقنية.
Introduction
انقسام الخلايا غير المتماثل (ACD) هو العملية التي يتم من خلالها تقسيم المكونات تحت الخلوية مثل الحمض النووي الريبي والبروتينات والعضيات بشكل غير متساو بين الخلايا الوليدة 1,2. تظهر هذه العملية بشكل شائع في الخلايا الجذعية ، التي تخضع ل ACD لتؤدي إلى ظهور خلايا ابنة ذات مصائر تنموية مختلفة. ذبابة الفاكهة تنقسم NBs بشكل غير متماثل لإنتاج NB واحد ، والذي يحتفظ بجذعه ، وخلية أم واحدة (GMC). يخضع GMC لمزيد من الانقسامات لإنتاج الخلايا العصبية المتمايزة أو الدبقية3. NBs المقسمة بشكل غير متماثل وفيرة في الأدمغة النامية لليرقات الثالثة ، والتي يمكن ملاحظتها بسهولة عن طريق الفحص المجهري. في المرحلة الثالثة من اليرقات الداخلية ، يوجد ما يقرب من 100 NBs في كل فص دماغي مركزي3،4،5،6.
انقسام الخلايا غير المتماثل هو عملية ديناميكية للغاية. تم استخدام بروتوكولات تصوير الخلايا الحية لقياس وقياس ديناميكيات قطبية الخلية7،8،9،10 ، اتجاه المغزل 11،12،13 ، ديناميات قشرة الأكتوميوسين 14،15،16،17،18 ، الأنابيب الدقيقة وبيولوجيا الجسيم المركزي 19،20، 21،22،23،24،25،26،27 ، والغشاء10،28 وديناميات الكروماتين 29. تعتمد الأوصاف النوعية والكمية ل ACD على طرق وبروتوكولات قوية لتقسيم NBs في أدمغة حية سليمة. يحدد البروتوكول التالي طرق تحضير وتشريح وتصوير أدمغة اليرقات الثالثة لتصوير الخلايا الحية في الجسم الحي باستخدام طريقتين مختلفتين للتركيب. هذه الطرق هي الأنسب للباحثين المهتمين بالديناميكيات الزمانية المكانية لانقسامات الخلايا الجذعية ، وكذلك الانقسامات في خلايا الدماغ الأخرى ، لأنها تسمح بمراقبة الأحداث الخلوية على المدى القصير والطويل. بالإضافة إلى ذلك ، يمكن الوصول بسهولة إلى هذه التقنيات للقادمين الجدد إلى هذا المجال. لقد أثبتنا فعالية هذا النهج وقدرته على التكيف مع أدمغة اليرقات التي تعبر عن الأنابيب الدقيقة الموسومة بالفلورسنت وبروتينات الاندماج القشري. بالإضافة إلى ذلك ، نناقش طرق التحليل واعتبارات التطبيق في دراسات أخرى.
Protocol
ملاحظة: يوضح الشكل 1 المواد المطلوبة لإجراء هذه الدراسة.
1. الاعتبارات والاستعدادات للتجربة
- منع اليرقات من الاكتظاظ.
ملاحظة: ترتبط جودة أدمغة اليرقات المزروعة ارتباطا مباشرا بصحة وجودة اليرقات قبل التشريح. اليرقات التي تعاني من سوء التغذية من الاكتظاظ ستنتج عموما أدمغة أقل جودة30.- تأكد من عدم وجود أكثر من 20-30 يرقة لكل طبق غطاء وجبة لتجنب سوء التغذية. يمكن رؤية أمثلة على ذلك في الشكل 2.
- تصفية و aliquot شنايدر المتوسطة قبل الاستخدام.
- لكل تشريح ، قم بإعداد وسط تصوير وتشريح جديد عن طريق استكمال وسط الحشرات من شنايدر المقتبس بمصل نمو الأبقار بنسبة 1٪ (BGS). عادة ما يكون حجم 5 مل من التشريح ووسط التصوير كافيا لتجربة التصوير.
- قم بتسخين الوسط المكمل إلى درجة حرارة الغرفة (RT) قبل الاستخدام.
- ضع في اعتبارك طول الفيلم المراد جمعه ، واستخدم ذلك لمراعاة مكملات وسيط التصوير ، ونهج التركيب ، وإعدادات اكتساب المجهر.
ملاحظة: في ظل الظروف المثلى ، فإن NBs في الأدمغة المكملة ب BGS فقط سوف تنقسم بقوة لما يزيد عن 3 ساعات.- استكمل وسط التصوير عن طريق إضافة أنسجة الجسم الدهنية اليرقية إلى وسط التصوير لدعم الانقسامات بعد 4 ساعات عند إجراء التجارب التي تتطلب أفلاما أطول.
ملاحظة: تفرز الأجسام الدهنية الميتوجينات التي تدعم انتشار NB31 ، والأجسام الدهنية الكاملة من 10 يرقات كافية لدعم أربعة إلى خمسة أدمغة. علاوة على ذلك ، تبين أن العينات المصورة بشريحة محاطة بغشاء تنقسم لأكثر من 10 ساعات13,32 ، في حين أن العينات المصورة بشريحة متعددة الآبار عادة ما تنقسم بشكل أقل (ملاحظات غير منشورة). - بدلا من ذلك ، قم بتنفيذ وسيط تصوير أكثر تعقيدا للأفلام الطويلة ، كما هو موضح سابقا33. قلل التلف الضوئي عن طريق ضبط وقت التعرض وطاقة الليزر وتكرار أخذ العينات للحصول على أفضل النتائج.
- استكمل وسط التصوير عن طريق إضافة أنسجة الجسم الدهنية اليرقية إلى وسط التصوير لدعم الانقسامات بعد 4 ساعات عند إجراء التجارب التي تتطلب أفلاما أطول.
2. تنظيم اليرقات وجمعها (الشكل 2)
- عبور الذباب البكر البالغ من العمر 1-5 أيام مع الذباب الذكور البالغين البالغ من العمر 1-7 أيام لإنتاج ذرية مع النمط الوراثي المطلوب. للحصول على العائد الأمثل ، عبر 10-15 عذراء الإناث مع 5-10 الذكور. قم بإيداع هذه الذباب في قفص ذباب مع غطاء وجبة (الشكل 2A-C) ، واحتضانها عند 25 درجة مئوية.
- استبدل غطاء الوجبة يوميا. هذا يمنع أغطية الوجبات من أن تصبح مكتظة باليرقات ، مما يقلل من جودة الأدمغة المشرحة.
- إذا كان غطاء الوجبة مغطى بشكل كبير باليرقات (أي >30) ، فقم بتقسيم غطاء الوجبة هذا إلى نصفين ، واستبدل النصف بغطاء وجبة طازج تم قطعه أيضا إلى النصف. بدلا من ذلك ، قم بتبديل أغطية الوجبات على أساس أكثر تكرارا (أي كل 12 ساعة بدلا من كل 24 ساعة). يمكن رؤية أمثلة على أغطية الوجبات المكتظة بالسكان في الشكل 2E ، F.
- احتضان غطاء الوجبة مع اليرقات عند 25 درجة مئوية حتى تصل اليرقات إلى العمر المطلوب.
3. تشريح الجسم الدهني اليرقات (الشكل 3)
ملاحظة: يصف هذا البروتوكول التشريح باستخدام طبق تشريح 3 آبار.
- ماصة ~ 400 ميكرولتر من التشريح والتصوير المتوسط في كل بئر من طبق تشريح 3 آبار.
- اغسل عشرة يرقات من النوع البري عمرها 72-96 ساعة جيدة التغذية عن طريق إمساكها برفق بملقط تشريح وغمسها داخل وخارج محلول التشريح في قاع البئر حتى يتم غسل جميع جسيمات الطعام. بعد الشطف ، انقل اليرقات النظيفة إلى البئر الأوسط.
- باستخدام مجموعة واحدة من الملقط ، أمسك اليرقة بواسطة خطافات الفم. مع المجموعة الأخرى من الملقط ، تمزق جانب واحد من بشرة اليرقة.
- سيؤدي هذا التمزق إلى تسرب الأجسام الدهنية من اليرقة. الأجسام الدهنية بيضاء اللون وشبه شفافة وسيكون لها بنية تشبه الشبكة (الشكل 3I). تميل الأجسام الدهنية أيضا إلى التمسك بنفسها وملاقط التشريح. بمجرد التعرف عليها ، اجمع أكبر قدر ممكن من الجسم الدهني من كل يرقة ، وانقله بالملقط إلى أعلى بئر باستخدام 400 ميكرولتر من وسط تشريح RT.
4. تشريح دماغ اليرقات (الشكل 3)
- اغسل اليرقات التجريبية في وسط التشريح والتصوير على النحو الوارد أعلاه لتحريرها من بقايا الطعام. للحصول على أفضل النتائج ، تجنب تخزين اليرقات غير المشرحة في محلول التشريح. سيؤدي ذلك إلى "غرق" اليرقات وسيؤثر سلبا على جودة الأدمغة المشرحة.
- باستخدام مجموعة واحدة من الملقط ، أمسك اليرقة بواسطة خطافات الفم. باستخدام مجموعة أخرى من الملقط ، قم بقص / تمزيق ما يقرب من 1/3 من اليرقة برفق من الجانب الخلفي (الشكل 3 أ). سيؤدي ذلك إلى "انفجار" عناصر الجهاز الهضمي والأجسام الدهنية والنسيج الضام والجهاز العصبي من الجانب الممزق من اليرقة (الشكل 3 ب).
- باستخدام مجموعة واحدة من الملقط ، أمسك اليرقة بواسطة خطافات الفم. باستخدام المجموعة الأخرى من الملقط ، قم بتنظيف البشرة برفق نحو خطافات الفم أثناء "الدفع" للداخل باستخدام الملقط الذي يمسك خطافات الفم حتى يتم قلب اليرقة بأكملها من الداخل للخارج. تشبه هذه الحركة قلب جورب "من الداخل إلى الخارج" (الشكل 3C ، D).
- اقلب اليرقة بحيث يواجه الجهاز العصبي المركزي والأنسجة الأخرى الخارج بينما لا يزال متصلا بالبشرة. في هذه الخطوة ، حدد موقع الجهاز العصبي المركزي (CNS) لتجنب الإزالة العرضية. باستخدام الملقط ، قم بإزالة جميع الأنسجة غير الموجودة في الجهاز العصبي المركزي برفق ، مع ترك الجهاز العصبي المركزي والدماغ فقط متصلين بالبشرة (الشكل 3E).
- سيتم ربط الدماغ بالبشرة عبر الوصلات المحورية. باستخدام مقص التشريح المجهري ، قم بقطع هذه الوصلات المحورية لتحرير الدماغ من البشرة. للقيام بذلك ، قم أولا بقص بلطف تحت فصوص الدماغ (الشكل 3F). كرر مع الوصلات تحت الحبل العصبي البطني.
ملاحظة: يمكن إجراء هذه الخطوة باستخدام الملقط إذا لم يكن مقص التشريح الدقيق متاحا. توخ الحذر بشكل خاص عند استخدام الملقط للتأكد من أن أنسجة المخ لا تتمدد بشكل مفرط أثناء إزالتها من البشرة لأن الإجهاد الميكانيكي سيؤثر سلبا على صحة الدماغ. - نقل الدماغ تشريح إلى بئر مع تشريح ووسط التصوير. لتجارب التصوير التي تزيد مدتها عن 3 ساعات ، استخدم التشريح ووسط التصوير المكمل بالأجسام الدهنية كما هو موضح أعلاه. تشريح اليرقات على دفعات للحفاظ على وقت التشريح أقل من 20 دقيقة.
5. التركيب والتصوير (الشكل 4)
- للتصوير باستخدام شريحة محاطة بالغشاء34:
- اجمع كل من العقول المشرحة والأجسام الدهنية المعزولة في البئر الأخير من طبق التشريح.
- قم بتجميع نصف الشريحة عن طريق وضع غشاء منفذ للغاز فوق الجزء الخلفي من الشريحة ، واضغط على الحلقة المنقسمة في المنتصف ، وثبتها في مكانها (الشكل 4A-C).
- باستخدام ميكروماصة 200 ميكرولتر ، قم بنقل ما يصل إلى 10 أدمغة تشريح وأكبر قدر ممكن من الجسم الدهني (انظر أعلاه) في ~ 130-140 ميكرولتر من التشريح والتصوير المتوسط إلى الغشاء. تأكد من إيداع الوسط مع العينات في وسط الغشاء المنفذ للغاز (الشكل 4D ، E).
- توجيه الأدمغة لسكان NBs ليتم تصويرهم ونوع المجهر المستخدم (الشكل 4E). ضع العينة بالقرب من هدف المجهر قدر الإمكان. على سبيل المثال ، لتصوير NBs في فصوص الدماغ المركزية ، قم بتوجيه الأدمغة بحيث تكون فصوص الدماغ أقرب إلى الهدف (الشكل 4H).
- بمجرد توجيه الأدمغة ، ضع غطاء زجاجي برفق فوق المحلول على الغشاء. سيؤدي ذلك إلى انتشار المحلول الذي يحتوي على الأدمغة والأجسام الدهنية على كامل الغشاء (الشكل 4F).
- لطخة الحل المفرط عن طريق عقد الأنسجة المختبرية بالقرب من حافة الغطاء. يتم تحقيق الكمية المثلى من المحلول عندما تلمس الأدمغة غطاء الغطاء دون أن يتم سحقها. إذا كانت إعادة التوجيه مطلوبة في هذه الخطوة ، فقم بتحريك غطاء الغطاء بعناية لتحريك الأدمغة.
- شل حركة الغطاء عن طريق وضع هلام البترول المذاب على طول حواف غطاء الغطاء باستخدام فرشاة الرسم. اسمح للهلام بالتصلب (الشكل 4 ز).
- للتصوير باستخدام شريحة تصوير متعددة الآبار (الشكل 4):
- أضف 400 ميكرولتر من وسيط التصوير إلى بئر من شريحة متعددة الآبار (في التجربة التي أجريت هنا ، تم استخدام شريحة صغيرة ذات 8 آبار [μ] ؛ الشكل 4I). نقل الأجسام الدهنية التي تم تشريحها سابقا إلى هذا البئر (انظر الخطوة 3.4).
- قم بإيداع ما يصل إلى 10 أدمغة في مجموعة بالقرب من مركز البئر (الشكل 4J).
- قم بتوجيه الأدمغة لسكان NBs ليتم تصويرهم ولنوع المجهر المستخدم ، كما هو موضح في الخطوة 5.1.4 (الشكل 4K). رتب العينات بحيث تكون قريبة من بعضها البعض. سيؤدي ذلك إلى تقليل المسافة التي يجب أن تقطعها المرحلة بين العينات ، مما يقلل من انحراف العينة أثناء الاستحواذ.
- بمجرد توجيه الأدمغة في البئر ، اسمح للأدمغة بالاستقرار لمدة 2-5 دقائق. هذا يزيد من ثباتها أثناء النقل / التصوير. تحضير المجهر للاستحواذ خلال هذا الوقت.
- قم بتغطية الشريحة μ بغطاء الشريحة ، وانقلها إلى المجهر. ابدأ الاكتساب بأقل طاقة ليزر ووقت تعرض ممكن لتقليل التبييض الضوئي.
6. أفضل ممارسات معالجة البيانات وإدارتها
- معالجة البيانات حسب الحاجة وفقا لبرنامج التحليل المتاح.
- بالنسبة للمثال الموضح هنا، احفظ البيانات التي تم الحصول عليها باستخدام برنامج SlideBook كملف صورة SlideBook (.sld).
- للتحويل إلى نوع الملف الخاص ب Imaris (.ims) باستخدام محول ملفات Imaris ، افتح محول ملفات Imaris في نافذة منفصلة. انقر فوق ملفات .sld واسحبها إلى قسم "الإدخال" في محول ملفات Imaris.
- حدد موقع الإخراج المطلوب للملفات المحولة ، وانقر فوق "بدء الكل".
- بعد التحويل ، قم بعرض البيانات والتعليق عليها في برنامج Imaris.
ملاحظة: يمكن استخدام بدائل لتحليل الصور بدلا من Imaris ، مثل فيجي (https://hpc.nih.gov/apps/Fiji.html) أو Aivia (https://www.aivia-software.com/) أو Volocity (https://www.volocity4d.com/) أو غيرها.
- احتفظ بأكبر قدر ممكن من البيانات الأصلية لحفظ السجلات بشكل صحيح. على سبيل المثال ، إذا تم حفظ برنامج الاستحواذ بتنسيق ملف واحد ولكن تم تحويله إلى تنسيق مختلف للتحليل ، فاحتفظ بالإصدار الذي تم الحصول عليه من البيانات.
- لتحليل البيانات، احتفظ بسجل لأكبر عدد ممكن من التفاصيل حول إعدادات العينة والاستحواذ. تتضمن المعلومات الأساسية التي يجب الاحتفاظ بها النمط الوراثي لليرقات التي تم تشريحها ، وعمر اليرقات قبل التشريح ، وحالة غطاء الوجبة الذي تم تربيتها فيه ، وقوة الليزر المستخدمة أثناء التصوير ، ووقت التعرض ، وطول فترة الاكتساب ، والدقة الزمنية.
7. مثال على القياس الكمي لطول دورة الخلية (الشكل 5)
ملاحظة: في هذا المثال ، تم تصوير اليرقات التي تعبر عن دبابيس علامة القطبية (Pins::EGFP16) وبروتين Jupiter25 المرتبط بالأنابيب الدقيقة (الكرز::Jupiter13). تم إجراء التحليل اللاحق باستخدام برنامج Imaris.
- افتح الفيلم باستخدام برنامج تحليل الصور الذي تختاره. قم بالتمرير خلال طول الفيلم لتحديد تقسيم NBs ، وقم بتسميتها للرجوع إليها في المستقبل. حدد NBs المقسمة بواسطة مغازلها الانقسامية المميزة (الشكل 5C-E).
- تحديد مرحلة دورة الخلية المرجعية لتحديد طول دورة الخلية. في هذا المثال ، يتم استخدام الطور الاستوائي كمرجع.
- حدد يدويا عدد الإطارات بين الأطوار الاستوائية المتتالية، وقم بتحويلها إلى دقائق أو ساعات لتحديد الوقت المستغرق لإكمال دورة خلية واحدة.
- افعل ذلك عن طريق أخذ الدقة الزمنية للفيلم وضربها في عدد الإطارات بين الأطوار. على سبيل المثال ، إذا كانت الدقة الزمنية للفيلم عبارة عن إطار واحد كل 5 دقائق ، وتم ملاحظة الأطوار الاستوائية في الإطار 13 والإطار 35 ، فسيكون الوقت بين هذه الأطوار الاستوائية 110 دقيقة ([35 - 13] × 5).
- ارسم البيانات باستخدام أي برنامج مناسب. تم رسم البيانات الموضحة هنا باستخدام برنامج PRISM.
8. مثال على القياس الكمي لمحاذاة مغزل الخلية (الشكل 5)
ملاحظة: في هذا المثال، يتم إجراء التحليل باستخدام برنامج Imaris.
- افتح ملف الفيلم في Imaris أو برنامج آخر من اختيارك. قم بالتمرير خلال طول الفيلم لتحديد تقسيم NBs ، وقم بتسميتها للرجوع إليها في المستقبل.
- حدد المتجه الذي يتكون من قطبي المغزل باستخدام الجسيمات المركزية القمية والقاعدية (ممثلة ب m) ، على النحو التالي:
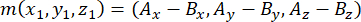
حيث Ax و Ay و Az هي إحداثيات الجسيم المركزي القمي ، و Bx و By و Bz هي إحداثيات الجسيم المركزي القاعدي. وبالمثل ، يتكون محور متجه القسمة (الممثل ب n) من نقطة منتصف الدبابيس القمية::EGFP الهلال والقشرة القاعدية: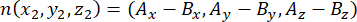
حيث الفأس و Ay و Az هي إحداثيات نقطة منتصف الدبابيس::EGFP الهلال ، و Bx و By و Bz هي إحداثيات نقطة منتصف القشرة القاعدية. - أوجد حجم المتجهين m و n:
حجم م: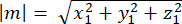
مقدار n: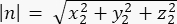
- حدد حاصل الضرب النقطي (الذي يمثله k) ل m و n:
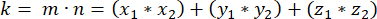
- باستخدام حاصل الضرب النقطي k ومقادير المتجه m و n ، حدد الزاوية بين المتجهات:
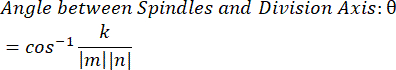
- ارسم البيانات في البرنامج الذي تختاره. تم إعداد البيانات الموضحة هنا في Microsoft Excel وتصورها في PRISM.
النتائج
تشريح وتصوير الفص الدماغي المركزي NBs معبرا عن الدبابيس::EGFP والكرز::كوكب المشتري
لعرض هذا البروتوكول ، اليرقات التي تعبر عن الكرز الذي يحركه UAS :: كوكب المشتري13 والمسمى داخليا الدبابيس :: EGFP16 (w ؛ worGal4 ، UAS-cherry :: jupiter / CyO ؛ تم تصوير الدبابيس :: EGFP / TM6B ، Tb) لمدة 4 س?...
Discussion
يحدد هذا البروتوكول نهجا واحدا لتصوير أدمغة النباتات الحية من يرقات ذبابة الفاكهة الميلانوجاستر . يسمح البروتوكول الموصوف هنا بمراقبة أدمغة الزرع لمدة 12-20 ساعة في ظل الظروف التجريبية المناسبة. يجب إيلاء اهتمام خاص لإعداد العينات وتصميم التجارب المطلوبة. كما ذكر أعلاه ، فإن أحد أهم ا?...
Disclosures
ليس لدى أصحاب البلاغ أي إفصاحات مالية يعلنونها.
Acknowledgements
يتم دعم هذا البحث من قبل R35GM148160 (C. C.) ومنحة تدريب المعاهد الوطنية للصحة (NIH) T32 GM007270 (R. C. S)
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
References
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved