Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie de cellules vivantes du cerveau larvaire du troisième stade de Drosophila melanogaster
Dans cet article
Résumé
Ici, nous discutons d’un flux de travail pour préparer, disséquer, monter et imager des cerveaux d’explants vivants de larves du troisième stade de Drosophila melanogaster afin d’observer la dynamique cellulaire et subcellulaire dans des conditions physiologiques.
Résumé
Les cellules souches neurales de la drosophile (neuroblastes, NBs ci-après) subissent des divisions asymétriques, régénérant le neuroblaste auto-renouvelant, tout en formant une cellule mère ganglionnaire différenciatrice (GMC), qui subira une division supplémentaire pour donner naissance à deux neurones ou glies. Des études sur les NB ont révélé les mécanismes moléculaires sous-jacents à la polarité cellulaire, à l’orientation du fuseau, à l’auto-renouvellement des cellules souches neurales et à la différenciation. Ces divisions cellulaires asymétriques sont facilement observables par imagerie de cellules vivantes, ce qui rend les NB larvaires idéales pour étudier la dynamique spatio-temporelle de la division cellulaire asymétrique dans les tissus vivants. Lorsqu’ils sont correctement disséqués et imagés dans un milieu enrichi en nutriments, les NB dans les cerveaux explantés se divisent vigoureusement pendant 12 à 20 heures. Les méthodes décrites précédemment sont techniquement difficiles et peuvent être difficiles pour ceux qui débutent dans le domaine. Ici, un protocole est décrit pour la préparation, la dissection, le montage et l’imagerie d’explants cérébraux larvaires vivants du troisième stade à l’aide de suppléments de corps gras. Les problèmes potentiels sont également discutés, et des exemples sont fournis sur la façon dont cette technique peut être utilisée.
Introduction
La division cellulaire asymétrique (DCA) est le processus par lequel les composants subcellulaires tels que l’ARN, les protéines et les organites sont répartis de manière inégale entre les cellules filles 1,2. Ce processus est couramment observé dans les cellules souches, qui subissent une DCA pour donner naissance à des cellules filles avec des destins de développement différents. Drosophile Les NB se divisent de manière asymétrique pour produire un NB, qui conserve sa souche, et une cellule mère ganglionnaire (GMC). Le GMC subit d’autres divisions pour produire des neurones différenciateurs ou glies3. Les NB à division asymétrique sont abondants dans le cerveau en développement des larves du troisième stade, qui sont facilement observées par microscopie. Au stade larvaire du troisième stade, il y a environ 100 NB présents dans chaque lobe central du cerveau 3,4,5,6.
La division cellulaire asymétrique est un processus très dynamique. Des protocoles d’imagerie de cellules vivantes ont été utilisés pour mesurer et quantifier la dynamique de la polarité cellulaire 7,8,9,10, l’orientation du fuseau 11,12,13, la dynamique du cortex actomyosine 14,15,16,17,18, la biologie des microtubules et des centrosomes 19,20,21,22,23,24,25,26,27, et membrane 10,28 et dynamique de la chromatine 29. Les descriptions qualitatives et quantitatives de la DCA reposent sur des méthodes et des protocoles robustes pour diviser les NB dans des cerveaux vivants intacts. Le protocole suivant décrit les méthodes de préparation, de dissection et d’imagerie des cerveaux larvaires du troisième stade pour l’imagerie de cellules vivantes in vivo à l’aide de deux approches de montage différentes. Ces méthodes conviennent mieux aux chercheurs qui s’intéressent à la dynamique spatio-temporelle des divisions de cellules souches, ainsi qu’aux divisions dans d’autres cellules du cerveau, car elles permettent d’observer des événements cellulaires à court et à long terme. De plus, ces techniques sont facilement accessibles aux nouveaux arrivants sur le terrain. Nous démontrons l’efficacité et l’adaptabilité de cette approche avec des cerveaux larvaires exprimant des microtubules marqués par fluorescence et des protéines de fusion corticale. Nous discutons également des méthodes d’analyse et des considérations pour l’application dans d’autres études.
Protocole
REMARQUE : La figure 1 montre les matériaux requis pour réaliser cette étude.
1. Considérations et préparatifs de l’expérience
- Empêchez les larves de surpeupler.
REMARQUE : La qualité des cerveaux larvaires d’explantation est directement liée à la santé et à la qualité des larves avant la dissection. Les larves qui souffrent de malnutrition à cause de la surpopulation produiront généralement des cerveaux de qualité inférieure30.- Assurez-vous qu’il n’y a pas plus de 20 à 30 larves par plat de chapeau de repas pour éviter la malnutrition. Des exemples de ceux-ci peuvent être vus dans la figure 2.
- Filtrer et aliquote le milieu de Schneider avant utilisation.
- Pour chaque dissection, préparer un nouveau milieu d’imagerie et de dissection en complétant le milieu insecte aliquote de Schneider avec du sérum de croissance bovine (BGS) à 1 %. Un volume de 5 mL de dissection et de milieu d’imagerie est habituellement suffisant pour une expérience d’imagerie.
- Réchauffer le milieu supplémenté à température ambiante (RT) avant utilisation.
- Tenez compte de la longueur du film à collecter et utilisez-la pour prendre en compte la supplémentation du support d’imagerie, l’approche de montage et les paramètres d’acquisition du microscope.
NOTE: Dans des conditions optimales, les NB dans les cerveaux supplémentés uniquement par BGS se diviseront vigoureusement pendant plus de 3 heures.- Complétez le milieu d’imagerie en ajoutant des tissus adipeux larvaires au milieu d’imagerie pour soutenir les divisions au-delà de 4 heures lors de la réalisation d’expériences nécessitant des films plus longs.
REMARQUE : Les corps adipeux sécrètent des mitogènes qui favorisent la proliférationNB 31, et les corps gras entiers de 10 larves sont suffisants pour soutenir quatre à cinq cerveaux. De plus, il a été démontré que les échantillons imagés avec une lame liée à la membrane se divisent pendant plus de 10 h13,32, tandis que les échantillons imagés avec une lame multipuits se divisent généralement moins souvent (observations non publiées). - Vous pouvez également mettre en œuvre un support d’imagerie plus complexe pour les films plus longs, comme décrit précédemment33. Minimisez les photodommages en ajustant le temps d’exposition, la puissance laser et la fréquence d’échantillonnage pour de meilleurs résultats.
- Complétez le milieu d’imagerie en ajoutant des tissus adipeux larvaires au milieu d’imagerie pour soutenir les divisions au-delà de 4 heures lors de la réalisation d’expériences nécessitant des films plus longs.
2. Mise en scène et collecte des larves (figure 2)
- Croisez des mouches vierges femelles âgées de 1 à 5 jours avec des mouches mâles adultes âgées de 1 à 7 jours pour produire une progéniture avec le génotype souhaité. Pour un rendement optimal, croisez 10-15 vierges femelles avec 5-10 mâles. Déposer ces mouches dans une cage à mouches munie d’un bouchon de repas (figure 2A-C) et incuber à 25 °C.
- Échangez le bouchon de repas tous les jours. Cela empêche les bouchons de repas de devenir surpeuplés de larves, ce qui réduit la qualité des cerveaux disséqués.
- Si le bouchon de farine est recouvert de larves (c.-à-d. >30), divisez-le en deux et remplacez-le par un bouchon de repas frais qui a également été coupé en deux. Sinon, échangez les bouchons de repas plus fréquemment (c.-à-d. toutes les 12 heures au lieu de toutes les 24 heures). Des exemples de bouchons de repas surpeuplés peuvent être vus à la figure 2E, F.
- Incuber le chapeau de farine avec les larves à 25 °C jusqu’à ce que les larves atteignent l’âge désiré.
3. Dissection larvaire du corps adipeux (figure 3)
NOTE: Ce protocole décrit les dissections utilisant une boîte de dissection à 3 puits.
- Pipeter ~400 μL de dissection et de milieu imageur dans chaque puits d’une boîte de dissection à 3 puits.
- Lavez dix larves de type sauvage bien nourries âgées de 72 à 96 heures en les tenant doucement avec des pinces à dissection et en les plongeant dans et hors de la solution de dissection dans le puits le plus bas jusqu’à ce que toutes les particules alimentaires aient été lavées. Après le rinçage, déplacez les larves propres vers le puits du milieu.
- À l’aide d’une pince à épiler, tenez la larve par les crochets buccaux. Avec l’autre pince à épiler, rupture d’un côté de la cuticule de la larve.
- Cette rupture provoquera le débordement des corps graisseux hors de la larve. Les corps gras sont blanc cassé et semi-translucides et auront une structure en forme de réseau (Figure 3I). Les corps gras auront également tendance à coller à eux-mêmes et aux pincettes à dissection. Une fois identifié, prélever autant de graisse que possible sur chaque larve et la transférer avec la pince au puits le plus haut avec 400 μL de milieu de dissection RT.
4. Curage cérébral larvaire (figure 3)
- Laver les larves expérimentales dans un milieu de dissection et d’imagerie comme ci-dessus pour les libérer des résidus alimentaires. Pour de meilleurs résultats, évitez de stocker les larves non disséquées dans la solution de dissection. Cela provoquera la « noyade » des larves et aura un impact négatif sur la qualité des cerveaux disséqués.
- À l’aide d’une pince à épiler, tenez la larve par les crochets buccaux. À l’aide d’une autre pince à épiler, coupez ou arrachez doucement environ 1/3 de la larve du côté postérieur (figure 3A). Cela fera éclater des éléments du tube digestif, des corps adipeux, du tissu conjonctif et du système nerveux du côté rompu de la larve (Figure 3B).
- À l’aide d’une pince à épiler, tenez la larve par les crochets buccaux. Avec l’autre pince à épiler, brossez doucement la cuticule vers les crochets buccaux tout en « poussant » vers l’intérieur avec la pince à épiler qui tient les crochets buccaux jusqu’à ce que toute la larve soit retournée à l’envers. Ce mouvement est similaire à tourner une chaussette « à l’envers » (Figure 3C, D).
- Inversez la larve de sorte que le système nerveux central et les autres tissus soient tournés vers l’extérieur tout en étant connectés à la cuticule. À cette étape, localisez le système nerveux central (SNC) pour éviter une ablation accidentelle. À l’aide d’une pince à épiler, retirez délicatement tout le tissu autre que le SNC, en ne laissant que le SNC et le cerveau attachés à la cuticule (figure 3E).
- Le cerveau sera attaché à la cuticule via des connexions axonales. À l’aide de ciseaux de microdissection, coupez ces connexions axonales pour libérer le cerveau de la cuticule. Pour ce faire, coupez d’abord doucement sous les lobes du cerveau (Figure 3F). Répétez avec les connexions sous le cordon nerveux ventral.
REMARQUE: Cette étape peut être effectuée avec une pince à épiler si les ciseaux de microdissection ne sont pas disponibles. Prenez des précautions particulières lors de l’utilisation d’une pince à épiler pour vous assurer que le tissu cérébral n’est pas trop étiré lors du retrait de la cuticule, car le stress mécanique affectera négativement la santé du cerveau. - Transférer le cerveau disséqué dans un puits avec dissection et milieu d’imagerie. Pour les expériences d’imagerie de plus de 3 heures, utiliser la dissection et le milieu d’imagerie complétés par des corps adipeux comme décrit ci-dessus. Disséquer les larves par lots pour maintenir le temps de dissection en dessous de 20 min.
5. Montage et imagerie (Figure 4)
- Pour l’imagerie avec une lame membranaire34 :
- Rassemblez à la fois les cerveaux disséqués et les corps graisseux isolés dans le dernier puits du plat de dissection.
- Assemblez la moitié de la lame en plaçant une membrane perméable aux gaz à l’arrière de la glissière et appuyez sur l’anneau fendu au centre, en le maintenant en place (Figure 4A-C).
- À l’aide d’une micropipette de 200 μL, transférer jusqu’à 10 cerveaux disséqués et autant de graisse que possible (voir ci-dessus) dans ~130-140 μL de dissection et de milieu d’imagerie à la membrane. Assurez-vous de déposer le milieu avec les échantillons au centre de la membrane perméable aux gaz (Figure 4D, E).
- Orientez les cerveaux en fonction de la population de NB à imager et du type de microscope utilisé (figure 4E). Placez l’échantillon aussi près que possible de l’objectif du microscope. Par exemple, pour imager les NB dans les lobes centraux du cerveau, orientez les cerveaux de manière à ce que les lobes cérébraux soient les plus proches de l’objectif (Figure 4H).
- Une fois que le cerveau est orienté, placez doucement une lamelle de verre sur le dessus de la solution sur la membrane. Cela provoquera la propagation de la solution contenant le cerveau et les corps adipeux sur l’ensemble de la membrane (Figure 4F).
- Éponger une solution excessive en tenant un mouchoir de laboratoire près du bord de la lame. La quantité optimale de solution est obtenue lorsque le cerveau touche la lamelle de couverture sans être écrasé. Si une réorientation est nécessaire à cette étape, déplacez délicatement la lamelle de couverture pour déplacer le cerveau.
- Immobiliser le bordereau de couverture en appliquant de la vaseline fondue le long des bords du bordereau de couverture avec un pinceau. Laisser la gelée se solidifier (Figure 4G).
- Pour l’imagerie avec une lame d’imagerie multipuits (Figure 4) :
- Ajouter 400 μL de milieu d’imagerie à un puits d’une lame multipuits (dans l’expérience réalisée ici, une micro[μ]-lame chambrée à 8 puits a été utilisée; Figure 4I). Transférer les corps graisseux précédemment disséqués dans ce puits (voir étape 3.4).
- Déposez jusqu’à 10 cerveaux dans un groupe près du centre du puits (Figure 4J).
- Orientez les cerveaux en fonction de la population de NB à imager et du type de microscope utilisé, tel que décrit à l’étape 5.1.4 (figure 4K). Disposez les échantillons de manière à ce qu’ils soient proches les uns des autres. Cela minimisera la distance que l’étage doit parcourir entre les échantillons, ce qui réduit la dérive de l’échantillon lors de l’acquisition.
- Une fois que les cerveaux ont été orientés dans le puits, laissez-les s’installer pendant 2 à 5 minutes. Cela augmente leur stabilité pendant le transport/l’imagerie. Préparez le microscope pour l’acquisition pendant ce temps.
- Couvrez la lame de μ avec le couvercle de la lame et transférez-la au microscope. Commencez l’acquisition avec la puissance laser et le temps d’exposition les plus faibles possibles pour minimiser le photoblanchiment.
6. Meilleures pratiques de traitement et de gestion des données
- Traiter les données selon les besoins selon le logiciel d’analyse disponible.
- Pour l’exemple présenté ici, enregistrez les données acquises avec le logiciel SlideBook en tant que fichier image SlideBook (.sld).
- Pour convertir en type de fichier propriétaire d’Imaris (.ims) à l’aide du convertisseur de fichiers Imaris, ouvrez le convertisseur de fichiers Imaris dans une fenêtre séparée. Cliquez sur et faites glisser les fichiers .sld dans la section " entrée " du convertisseur de fichiers Imaris.
- Déterminez l’emplacement de sortie souhaité pour les fichiers convertis, puis cliquez sur « Tout commencer ».
- Après la conversion, visualisez et annotez les données dans le logiciel Imaris.
REMARQUE: Des alternatives pour l’analyse d’images peuvent être utilisées à la place d’Imaris, telles que Fidji (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/) ou autres.
- Conservez autant de données originales que possible pour une bonne tenue des dossiers. Par exemple, si le logiciel d’acquisition est enregistré dans un format de fichier, mais est converti dans un format différent pour analyse, conservez la version acquise des données.
- Pour l’analyse des données, conservez un enregistrement d’autant de détails que possible sur les paramètres d’échantillonnage et d’acquisition. Les informations clés à retenir comprennent le génotype des larves disséquées, l’âge des larves avant la dissection, l’état du chapeau de farine dans lequel elles ont été élevées, la puissance laser utilisée pendant l’imagerie, le temps d’exposition, la durée d’acquisition et la résolution temporelle.
7. Exemple de quantification de la longueur du cycle cellulaire (Figure 5)
REMARQUE: dans cet exemple, les larves exprimant le marqueur de polarité Pins (Pins::EGFP16) et la protéine de liaison aux microtubules Jupiter25 (cherry::Jupiter13) ont été imagées. L’analyse ultérieure a été réalisée à l’aide du logiciel Imaris.
- Ouvrez le film à l’aide du logiciel d’analyse d’image de votre choix. Faites défiler la longueur du film pour identifier les NB qui se divisent et étiquetez-les pour référence future. Identifier les NB divisés par leurs fuseaux mitotiques distincts (figure 5C-E).
- Identifier une étape de cycle cellulaire de référence pour déterminer la durée du cycle cellulaire. Dans cet exemple, la métaphase est utilisée comme référence.
- Déterminez manuellement le nombre d’images entre les métaphases successives et convertissez-le en minutes ou en heures pour déterminer le temps nécessaire pour terminer un cycle cellulaire.
- Pour ce faire, prenez la résolution temporelle du film et multipliez-la par le nombre d’images entre les métaphases. Par exemple, si la résolution temporelle du film est d’une image toutes les 5 minutes et que des métaphases sont observées dans l’image 13 et l’image 35, le temps entre ces métaphases serait de 110 minutes ([35 − 13] × 5).
- Tracez les données avec n’importe quel logiciel approprié. Les données présentées ici ont été tracées à l’aide du logiciel PRISM.
8. Exemple de quantification de l’alignement de la broche cellulaire (Figure 5)
REMARQUE : Dans cet exemple, l’analyse est effectuée à l’aide du logiciel Imaris.
- Ouvrez le fichier vidéo dans Imaris ou un autre logiciel de votre choix. Faites défiler la longueur du film pour identifier les NB qui se divisent et étiquetez-les pour référence future.
- Déterminer le vecteur formé par les pôles fusiformes à l’aide des centrosomes apical et basal (représentés par m), comme suit:
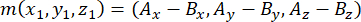
où Ax, Ay et Az sont les coordonnées du centrosome apical, et Bx, By et Bz sont les coordonnées du centrosome basal. De même, l’axe du vecteur division (représenté par n) est formé par le point médian du croissant apical Pins::EGFP et le cortex basal: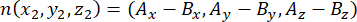
où Ax, Ay et Az sont les coordonnées du point médian du croissant Pins::EGFP, et Bx, By et Bz sont les coordonnées du point médian du cortex basal. - Déterminer l’amplitude des vecteurs m et n :
Magnitude de m: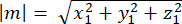
Magnitude de n :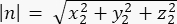
- Déterminez le produit ponctuel (représenté par k) de m et n :
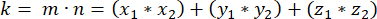
- À l’aide du produit ponctuel k et des magnitudes vectorielles m et n, déterminer l’angle entre les vecteurs:
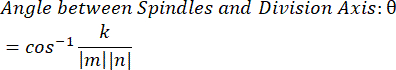
- Tracez les données dans le logiciel de votre choix. Les données présentées ici ont été préparées dans Microsoft Excel et visualisées dans PRISM.
Résultats
Dissection et imagerie du lobe central du cerveau NBs exprimant Pins::EGFP et Cherry::Jupiter
Pour présenter ce protocole, les larves exprimant Cherry::Jupiter13 pilotées par UAS et marquées de manière endogène Pins::EGFP16 (w; worGal4, UAS-cherry::jupiter/CyO; Pins::EGFP/TM6B, Tb) ont été imagées pendant 4 heures à l’aide du protocole décrit à l’aide de lames d’imagerie multipuits (Figure 5C,D).
Discussion
Ce protocole décrit une approche pour l’imagerie de cerveaux d’explants vivants provenant de larves de Drosophila melanogaster . Le protocole décrit ici permet d’observer les cerveaux d’explant pendant 12 à 20 h dans les bonnes conditions expérimentales. Une attention particulière doit être accordée à la préparation des échantillons et à la conception des expériences souhaitées. Comme mentionné ci-dessus, l’un des facteurs les plus critiques qui détermine la qualité du tissu disséqué ...
Déclarations de divulgation
Les auteurs n’ont aucune divulgation financière à déclarer.
Remerciements
Cette recherche est soutenue par R35GM148160 (C. C.) et une subvention de formation T32 GM007270 (R. C. S) des National Institutes of Health (NIH)
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
Références
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon