このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ショウジョウバエメラノガスター第3齢幼虫脳の生細胞イメージング
要約
本稿では,ショウ ジョウバエ・メラノガスター 第3齢幼虫から生きた外植片脳を作製,解剖,マウント,画像化し,生理条件下での細胞内・細胞内動態を観察するワークフローについて述べる.
要約
ショウジョウバエ 神経幹細胞(神経芽細胞、以下NB)は非対称分裂を起こし、自己複製神経芽細胞を再生すると同時に、分化神経節母細胞(GMC)を形成し、さらに1つの分裂を経て2つのニューロンまたはグリアを生成します。NBの研究により、細胞の極性、紡錘体の配向、神経幹細胞の自己複製、および分化の根底にある分子メカニズムが明らかになりました。これらの非対称細胞分裂は生細胞イメージング で 容易に観察できるため、幼虫NBは生体組織における非対称細胞分裂の時空間ダイナミクスを調べるのに理想的です。栄養補給培地で適切に解剖および画像化すると、外植片の脳のNBは12〜20時間にわたって堅牢に分裂します。前述の方法は技術的に難しく、この分野に不慣れな人にとっては難しいかもしれません。ここでは、脂肪体サプリメントを使用した生きた3齢幼虫の脳外植片の調製、解剖、マウント、およびイメージングのためのプロトコルについて説明します。潜在的な問題についても説明し、この手法の使用方法の例を提供します。
概要
不斉細胞分裂(ACD)は、RNA、タンパク質、細胞小器官などの細胞内成分が娘細胞間で不均等に分配されるプロセスです1,2。このプロセスは、ACDを受けて異なる発生運命を持つ娘細胞を生じさせる幹細胞で一般的に見られます。ショウジョウバエNBは非対称に分裂して、そのステム性を保持する1つのNBと1つの神経節母細胞(GMC)を生成します。GMCは、分化ニューロンまたはグリア3を生成するためにさらなる分裂を受ける。非対称分裂NBは、3齢幼虫の発達中の脳に豊富にあり、顕微鏡で容易に観察できます。3齢幼虫期では、各中枢脳葉3,4,5,6に約100個のNBが存在する。
非対称細胞分裂は非常に動的なプロセスです。生細胞イメージングプロトコルは、細胞極性7,8,9,10、紡錘体配向11,12,13、アクトミオシン皮質のダイナミクス14,15,16,17,18、微小管および中心体生物学19,20の測定および定量化に使用されています、21、22、23、24、25、26、27、および膜10、28およびクロマチン動態29。ACDの定性的および定量的記述は、無傷の生きている脳における分割NBを画像化するための堅牢な方法とプロトコルに依存しています。以下のプロトコルは、2つの異なるマウントアプローチを使用して、in vivoで生細胞イメージングのために3齢幼虫の脳を調製、解剖、およびイメージングする方法の概要を示しています。これらの方法は、幹細胞分裂の時空間ダイナミクスや他の脳細胞の分裂に関心のある研究者に最適であり、細胞イベントの短期および長期観察を可能にします。さらに、これらの手法は、この分野の初心者でも簡単にアクセスできます。蛍光タグ付き微小管と皮質融合タンパク質を発現する幼虫の脳に対するこのアプローチの有効性と適応性を実証します。さらに、分析の方法と他の研究への応用のための考慮事項についても議論します。
プロトコル
注: 図1 は、この調査を実行するために必要な資料を示しています。
1. 実験の検討と準備
- 幼虫が過密になるのを防ぎます。
注:外植幼虫の脳の質は、解剖前の幼虫の健康と質に直接関係しています。過密から栄養失調の幼虫は、一般的に低品質の脳を生み出します30。- 栄養失調を避けるために、ミールキャップディッシュごとに20〜30匹以下の幼虫が存在することを確認してください。これらの例を 図 2 に示します。
- 使用前にシュナイダー培地をろ過して分注します。
- 各解剖について、分注したシュナイダー昆虫培地に1%ウシ成長血清(BGS)を添加して、新鮮なイメージングおよび解剖培地を調製します。イメージング実験には、通常、5 mLの容量の解剖およびイメージング媒体で十分です。
- 使用前に、添加した培地を室温(RT)まで温めてください。
- 収集するフィルムの長さを考慮し、それを使用して、イメージング媒体の補充、実装アプローチ、および顕微鏡の取得設定を考慮します。
注:最適な条件下では、BGSのみを補給した脳のNBは、3時間以上にわたって堅牢に分裂します。- より長い動画を必要とする実験を行う際に、幼虫の脂肪体組織をイメージング媒体に追加してイメージング媒体を補完し、4時間以降の分割をサポートします。
注:脂肪体はNB増殖をサポートするマイトジェンを分泌し31、10匹の幼虫の全脂肪体は4〜5匹の脳を支えるのに十分です。さらに、膜結合スライドで画像化されたサンプルは10時間以上分裂することが示されています13,32が、マルチウェルスライドで画像化されたサンプルは通常、それほど頻繁に分裂しません(未発表の観察)。 - 代替的に、より長い映画のためのより複雑な画像化媒体を、先に説明したように33。最良の結果を得るには、露光時間、レーザー出力、サンプリング周波数を調整することで、光損傷を最小限に抑えます。
- より長い動画を必要とする実験を行う際に、幼虫の脂肪体組織をイメージング媒体に追加してイメージング媒体を補完し、4時間以降の分割をサポートします。
2. 幼虫の病期分類と採集(図2)
- 1〜5日齢の雌の処女ハエを1〜7日齢の成人雄ハエと交配して、目的の遺伝子型を持つ子孫を産生します。最適な収量を得るには、10〜15人の女性の処女と5〜10人の男性を交配します。これらのハエをミールキャップ付きのフライケージに入れ(図2A-C)、25°Cでインキュベートします。
- ミールキャップを毎日交換してください。これにより、ミールキャップが幼虫で過密になり、解剖された脳の質が低下するのを防ぎます。
- ミールキャップが幼虫でかなり覆われている場合(つまり、>30)、このミールキャップを半分に分割し、半分も半分にカットされた新しいミールキャップと交換します。または、より頻繁に(つまり、24時間ごとではなく12時間ごと)ミールキャップを交換します。人口過多のミールキャップの例は、 図2E、Fに見ることができます。
- 幼虫が希望の年齢に達するまで、幼虫と一緒に食事キャップを25°Cでインキュベートします。
3.幼虫の脂肪体の解剖(図3)
注:このプロトコルは、3ウェル解剖皿を使用した解剖について説明します。
- ~400 μLの解剖およびイメージング培地を3ウェル解剖皿の各ウェルにピペットで入れます。
- 72〜96時間齢の十分に給餌された野生型の幼虫10匹を、解剖鉗子でそっと持ち、すべての食物粒子が洗い流されるまで、一番下の一番下の解剖液に浸して洗います。すすいだ後、きれいな幼虫を真ん中の井戸に移動します。
- 1セットのピンセットを使用して、マウスフックで幼虫を保持します。もう一方のピンセットで、幼虫のキューティクルの片側を破裂させます。
- この破裂により、脂肪体が幼虫からこぼれます。脂肪体はオフホワイトで半透明で、格子状の構造になります(図3I)。脂肪体はまた、自分自身と解剖ピンセットにくっつく傾向があります。同定されたら、各幼虫からできるだけ多くの脂肪体を採取し、400μLのRT解剖培地で鉗子で一番上のウェルに移します。
4. 幼虫の脳解剖(図3)
- 上記のように解剖およびイメージング媒体で実験幼虫を洗浄して、食物残留物を取り除きます。最良の結果を得るには、解剖されていない幼虫を解剖溶液に保管しないでください。これは幼虫を「溺死」させ、解剖された脳の質に悪影響を及ぼすでしょう。
- 1セットのピンセットを使用して、マウスフックで幼虫を保持します。別のピンセットを使用して、幼虫の約1/3を後側からそっと切断/はぎ取ります(図3A)。これにより、消化管、脂肪体、結合組織、および神経系の要素が幼虫の破裂した側から「破裂」します(図3B)。
- 1セットのピンセットを使用して、マウスフックで幼虫を保持します。もう一方のピンセットで、幼虫全体が裏返しになるまで、ピンセットで口のフックを保持して内側に「押し」ながら、キューティクルをマウスフックに向かってそっと磨きます。この動きは、靴下を「裏返し」にすることに似ています(図3C、D)。
- 中枢神経系や他の組織がキューティクルに接続されたまま外側を向くように幼虫を反転させます。このステップでは、誤って除去しないように中枢神経系(CNS)を見つけます。ピンセットを使用して、CNS以外の組織をすべてそっと取り除き、CNSと脳のみをキューティクルに付着させます(図3E)。
- 脳は軸索接続 を介して キューティクルに付着します。マイクロダイセクションハサミを使用して、これらの軸索接続を切断し、キューティクルから脳を解放します。これを行うには、まず脳葉の下をそっと切ります(図3F)。腹側神経索の下の接続で繰り返します。
注意: この手順は、マイクロダイセクションハサミが利用できない場合は、ピンセットで実行できます。ピンセットを使用するときは、機械的ストレスが脳の健康に悪影響を与えるため、キューティクルからの取り外し中に脳組織が過度に伸びないように特に注意してください。 - 解剖した脳を解剖およびイメージング媒体を備えたウェルに移します。3時間を超えるイメージング実験には、上記のように脂肪体を添加した解剖およびイメージング媒体を使用してください。幼虫をバッチで解剖して、解剖時間を20分未満に保ちます。
5. 実装とイメージング(図4)
- 膜結合スライド34によるイメージングの場合:
- 解剖された脳と単離された脂肪体の両方を解剖皿の最後のウェルに集めます。
- スライドの背面に気体透過膜を置いてスライドの半分を組み立て、分割リングを中央に押し付けて所定の位置に保持します(図4A-C)。
- 200 μLのマイクロピペットを使用して、解剖した脳を~130-140 μLの解剖およびイメージング媒体で最大10個の解剖した脳とできるだけ多くの脂肪体(上記参照)を膜に移します。気体透過膜の中央にサンプルとともに培地を堆積させてください(図4D、E)。
- 画像化するNBの集団と使用する顕微鏡の種類に合わせて脳の向きを調整します(図4E)。サンプルを顕微鏡の対物レンズにできるだけ近づけます。たとえば、中枢脳葉のNBを画像化するには、脳葉が目的に最も近いように脳を向けます(図4H)。
- 脳の向きが決まったら、膜上の溶液の上にガラスのカバーガラスをそっと置きます。これにより、脳と脂肪体を含む溶液が膜全体に広がります(図4F)。
- ラボ組織をカバーガラスの端に近づけて、過剰な溶液を吸い取ります。最適な量の解決策は、脳が押しつぶされることなくカバーガラスに触れたときに達成されます。このステップで方向転換が必要な場合は、カバーガラスを慎重に動かして脳を動かします。
- 絵筆でカバーガラスの端に沿って溶かしたワセリンを塗って、カバーガラスを固定します。ゼリーを固めます(図4G)。
- マルチウェルイメージングスライドによるイメージングの場合(図4):
- 400 μLのイメージング培地をマルチウェルスライドのウェルに加えます(ここで行われた実験では、チャンバー付き8ウェルマイクロ[μ]スライドを使用しました。 図4I)。以前に解剖した脂肪体をこのウェルに移します(ステップ3.4を参照)。
- ウェルの中心近くのクラスターに最大10個の脳を沈着させます(図4J)。
- 手順5.1.4(図4K)で説明されているように、画像化するNBの集団と使用する顕微鏡のタイプに合わせて脳の向きを調整します。サンプルが互いに接近するように配置します。これにより、ステージがサンプル間を移動する距離が最小限に抑えられ、取得中のサンプルドリフトが減少します。
- 脳が井戸に向けられたら、脳を2〜5分間落ち着かせます。これにより、輸送/イメージング中の安定性が向上します。この間に顕微鏡を取得できるように準備します。
- スライドμカバーをスライドカバーで覆い、顕微鏡に移します。光退色を最小限に抑えるために、可能な限り低いレーザー出力と露光時間で取得を開始します。
6. データ処理と管理のベストプラクティス
- 利用可能な分析ソフトウェアに従って、必要に応じてデータを処理します。
- この例では、SlideBookソフトウェアで取得したデータをSlideBook画像ファイル(.sld)として保存します。
- Imarisファイルコンバータを使用してImaris独自のファイルタイプ(.ims)に変換するには、別のウィンドウでImarisファイルコンバータを開きます。.sldファイルをクリックして、Imarisファイルコンバーターの「入力」セクションにドラッグします。
- 変換されたファイルの目的の出力場所を決定し、[すべて開始]をクリックします。
- 変換後、Imarisソフトウェアでデータを表示して注釈を付けます。
注:画像分析の代替手段は、フィジー(https://hpc.nih.gov/apps/Fiji.html)、アイビア(https://www.aivia-software.com/)、ボロシティ(https://www.volocity4d.com/)などのImarisの代わりに使用できます。
- 適切な記録保持のために、元のデータをできるだけ多く保持します。たとえば、集録ソフトウェアを1つのファイル形式で保存し、解析用に別の形式に変換する場合は、集録したバージョンのデータを保持します。
- データ分析では、サンプルと取得の設定についてできるだけ多くの詳細を記録してください。保持する重要な情報には、解剖された幼虫の遺伝子型、解剖前の幼虫の年齢、飼育されたミールキャップの状態、イメージング中に使用されたレーザー出力、露光時間、取得の長さ、および時間分解能が含まれます。
7. 細胞周期長の定量例(図5)
注:この例では、極性マーカーPins(ピン::EGFP16)と微小管結合タンパク質Jupiter25 (チェリー::Jupiter13)を発現する幼虫を画像化しました。その後の分析は、Imarisソフトウェアを使用して実行されました。
- 選択した画像解析ソフトウェアを使用してムービーを開きます。ムービーの長さをスクロールして分割NBを特定し、後で参照できるようにラベルを付けます。分割NBをそれらの異なる有糸分裂紡錘体によって識別する(図5C-E)。
- 参照細胞周期ステージを同定して、細胞周期の長さを決定する。この例では、中期が参照として使用されます。
- 連続する中期間のフレーム数を手動で決定し、それを分または時間に変換して、1つの細胞周期を完了するのにかかる時間を決定します。
- これを行うには、ムービーの時間分解能を取得し、メタフェーズ間のフレーム数を掛けます。たとえば、動画の時間解像度が 5 分ごとに 1 フレームで、フレーム 13 とフレーム 35 で中相が観察される場合、これらの中相間の時間は 110 分になります ([35 − 13] × 5)。
- 適切なソフトウェアでデータをプロットします。ここに示すデータは、PRISMソフトウェアを使用してプロットされました。
8. 細胞紡錘体アライメントの定量例(図5)
注: この例では、分析は Imaris ソフトウェアを使用して実行されます。
- Imarisまたは選択した別のソフトウェアでムービーファイルを開きます。ムービーの長さをスクロールして分割NBを特定し、後で参照できるようにラベルを付けます。
- 次のように、頂端および基底中心体( mで表される)を使用して紡錘体極によって形成されるベクトルを決定します。
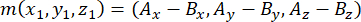
ここで、Ax、Ay、および Az は頂端中心体の座標であり、Bx、By、および Bz は基底中心体の座標です。同様に、分割ベクトルの軸(nで表される)は、頂端ピン::EGFP三日月と基底皮質の中点によって形成されます。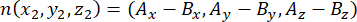
ここで、Ax、Ay、および Az は Pins::EGFP 三日月の中点の座標であり、Bx、By、および Bz は基底皮質の中点の座標です。 - ベクトル m と nの大きさを決定します。
mの大きさ: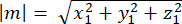
nの大きさ: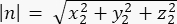
- mとnの内積(kで表される)を決定します。
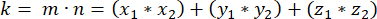
- 内積 k とベクトルの大きさ m および nを使用して、ベクトル間の角度を決定します。
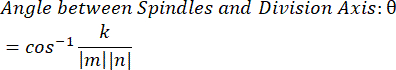
- 選択したソフトウェアでデータをプロットします。ここに示すデータは、Microsoft Excelで作成し、PRISMで可視化したものです。
結果
ピン::EGFPとチェリー::木星を発現する中枢脳葉NBの解剖とイメージング
このプロトコルを紹介するために、UAS駆動のCherry::Jupiter13 と内因的にタグ付けされたPins::EGFP16 (w; worGal4、UAS-cherry::jupiter/CyO;ピン::EGFP/TM6B、Tb)を、マルチウェルイメージングスライドを用いて記載されたプロトコルを用いて4時間画像化した(図5C、
ディスカッション
このプロトコルは、 ショウジョウバエメラノガスター 幼虫からの生きた外植片脳のイメージングのための1つのアプローチを概説します。ここで説明するプロトコルは、適切な実験条件下で外植片脳を12〜20時間観察することを可能にする。サンプルの調製と目的の実験の設計には特別な考慮が必要です。上記のように、解剖された組織の質を決定する最も重要な要素の1つは幼虫の健?...
開示事項
著者には、宣言する財務開示はありません。
謝辞
この研究は、R35GM148160(C.C.)および国立衛生研究所(NIH)トレーニンググラントT32 GM007270(RCS)によってサポートされています
資料
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
参考文献
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved