Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Lebendzell-Bildgebung von Drosophila melanogaster im dritten Stadium des Larvengehirns
In diesem Artikel
Zusammenfassung
Hier diskutieren wir einen Arbeitsablauf zur Präparation, Sektion, Montage und Bildgebung von lebenden Explantatgehirnen aus Drosophila melanogaster-Larven des dritten Stadiums, um die zelluläre und subzelluläre Dynamik unter physiologischen Bedingungen zu beobachten.
Zusammenfassung
Drosophila-neurale Stammzellen (Neuroblasten, im Folgenden NBs) durchlaufen asymmetrische Teilungen, die den sich selbst erneuernden Neuroblasten regenerieren und gleichzeitig eine differenzierende Ganglienmutterzelle (GMC) bilden, die eine zusätzliche Teilung durchläuft, um zwei Neuronen oder Gliazellen entstehen zu lassen. Studien an NBs haben die molekularen Mechanismen aufgedeckt, die der Zellpolarität, der Spindelorientierung, der Selbsterneuerung neuraler Stammzellen und der Differenzierung zugrunde liegen. Diese asymmetrischen Zellteilungen sind durch Lebendzell-Bildgebung leicht zu beobachten, wodurch sich Larven-NBs ideal für die Untersuchung der räumlich-zeitlichen Dynamik der asymmetrischen Zellteilung in lebendem Gewebe eignen. Bei richtiger Präparation und Abbildung in nährstoffangereichertem Medium teilen sich NBs im Explantatgehirn 12-20 Stunden lang robust. Die zuvor beschriebenen Methoden sind technisch schwierig und können für Neulinge auf diesem Gebiet eine Herausforderung darstellen. Hier wird ein Protokoll für die Präparation, Präparation, Montage und Bildgebung von lebenden Larvenexplantaten des dritten Stadiums unter Verwendung von Fettkörperpräparaten beschrieben. Es werden auch mögliche Probleme diskutiert und Beispiele für den Einsatz dieser Technik gegeben.
Einleitung
Asymmetrische Zellteilung (ACD) ist der Prozess, bei dem subzelluläre Komponenten wie RNA, Proteine und Organellen ungleichmäßig auf die Tochterzellen verteilt werden 1,2. Dieser Prozess ist häufig bei Stammzellen zu beobachten, die ACD durchlaufen, um Tochterzellen mit unterschiedlichen Entwicklungsschicksalen hervorzubringen. Drosophila NBs teilen sich asymmetrisch und erzeugen eine NB, die ihre Stammform beibehält, und eine Ganglienmutterzelle (GMC). Das GMC durchläuft weitere Teilungen, um differenzierende Neuronen oder Gliazellenzu erzeugen 3. Asymmetrisch teilende NBs sind in den sich entwickelnden Gehirnen von Larven des dritten Stadiums reichlich vorhanden, die mit Hilfe der Mikroskopie leicht beobachtet werden können. Im Larvenstadium des dritten Stadiums sind in jedem zentralen Hirnlappen etwa 100 NBsvorhanden 3,4,5,6.
Die asymmetrische Zellteilung ist ein hochdynamischer Prozess. Lebendzell-Bildgebungsprotokolle wurden verwendet, um die Dynamik der Zellpolarität 7,8,9,10, der Spindelorientierung 11,12,13, der Dynamik des Aktomyosin-Kortex14,15,16,17,18, der Mikrotubuli- und Zentrosombiologie 19,20 zu messen und zu quantifizieren,21,22,23,24,25,26,27 und Membran 10,28 und Chromatindynamik 29. Qualitative und quantitative Beschreibungen von ACD basieren auf robusten Methoden und Protokollen, um NBs in intakten lebenden Gehirnen abzubilden. Das folgende Protokoll beschreibt Methoden zur Präparation, Sektion und Bildgebung von Larvengehirnen im dritten Stadium für die Bildgebung lebender Zellen in vivo unter Verwendung von zwei verschiedenen Montageansätzen. Diese Methoden eignen sich am besten für Forscher, die sich für die raumzeitliche Dynamik von Stammzellteilungen sowie für Teilungen in anderen Gehirnzellen interessieren, da sie kurz- und langfristige Beobachtungen zellulärer Ereignisse ermöglichen. Darüber hinaus sind diese Techniken für Neulinge auf diesem Gebiet leicht zugänglich. Wir demonstrieren die Effektivität und Anpassungsfähigkeit dieses Ansatzes mit Larvenhirnen, die fluoreszenzmarkierte Mikrotubuli und kortikale Fusionsproteine exprimieren. Darüber hinaus diskutieren wir Analysemethoden und Überlegungen zur Anwendung in anderen Studien.
Protokoll
HINWEIS: Abbildung 1 zeigt die Materialien, die für die Durchführung dieser Studie erforderlich sind.
1. Überlegungen und Vorbereitungen für das Experiment
- Verhindern Sie, dass sich die Larven überfüllen.
HINWEIS: Die Qualität der Larvengehirne steht in direktem Zusammenhang mit der Gesundheit und Qualität der Larven vor der Sektion. Larven, die durch Überfüllung unterernährt sind, bringen im Allgemeinen Gehirne von geringerer Qualitäthervor 30.- Stellen Sie sicher, dass nicht mehr als 20-30 Larven pro Mahlzeit vorhanden sind, um eine Mangelernährung zu vermeiden. Beispiele hierfür sind in Abbildung 2 dargestellt.
- Filtern und aliquotieren Sie das Schneider'sche Medium vor Gebrauch.
- Bereiten Sie für jede Dissektion ein frisches Bildgebungs- und Dissektionsmedium vor, indem Sie aliquotiertes Schneider's Insect Medium mit 1% Rinderwachstumsserum (BGS) ergänzen. Ein Volumen von 5 mL Präparier- und Bildgebungsmedium ist in der Regel für ein bildgebendes Experiment ausreichend.
- Erwärmen Sie das supplementierte Medium vor Gebrauch auf Raumtemperatur (RT).
- Berücksichtigen Sie die Länge des zu erfassenden Films und verwenden Sie diese, um die Ergänzung des Bildgebungsmediums, den Montageansatz und die Aufnahmeeinstellungen des Mikroskops zu berücksichtigen.
HINWEIS: Unter optimalen Bedingungen teilen sich NBs in Gehirnen, die nur mit BGS supplementiert wurden, über 3 Stunden lang robust.- Ergänzen Sie das Bildgebungsmedium, indem Sie dem Bildgebungsmedium Larvenfettgewebe hinzufügen, um bei der Durchführung von Experimenten, die längere Filme erfordern, Teilung nach 4 Stunden zu unterstützen.
HINWEIS: Fettkörper scheiden Mitogene aus, die die NB-Proliferation unterstützen31, und ganze Fettkörper von 10 Larven reichen aus, um vier bis fünf Gehirne zu unterstützen. Darüber hinaus wurde gezeigt, dass Proben, die mit einem membrangebundenen Objektträger aufgenommen wurden, sich über 10 h teilen13,32, während Proben, die mit einem Multiwell-Objektträger aufgenommen wurden, sich in der Regel seltener teilen (unveröffentlichte Beobachtungen). - Implementieren Sie alternativ ein komplexeres Bildmedium für längere Filme, wie zuvor beschrieben33. Minimieren Sie Lichtschäden, indem Sie die Belichtungszeit, die Laserleistung und die Abtastfrequenz anpassen, um beste Ergebnisse zu erzielen.
- Ergänzen Sie das Bildgebungsmedium, indem Sie dem Bildgebungsmedium Larvenfettgewebe hinzufügen, um bei der Durchführung von Experimenten, die längere Filme erfordern, Teilung nach 4 Stunden zu unterstützen.
2. Stadien und Sammlung der Larven (Abbildung 2)
- Kreuzen Sie 1-5 Tage alte weibliche jungfräuliche Fliegen mit 1-7 Tage alten erwachsenen männlichen Fliegen, um Nachkommen mit dem gewünschten Genotyp zu erzeugen. Um einen optimalen Ertrag zu erzielen, kreuzen Sie 10-15 weibliche Jungfrauen mit 5-10 männlichen Sorten. Legen Sie diese Fliegen in einen Fliegenkäfig mit einer Mahlzeitkappe (Abbildung 2A-C) und inkubieren Sie sie bei 25 °C.
- Tauschen Sie die Mahlzeitkappe täglich aus. Dadurch wird verhindert, dass die Mehlkappen mit Larven überfüllt werden, was die Qualität der präparierten Gehirne verringert.
- Wenn der Mehlverschluss deutlich mit Larven bedeckt ist (z. B. >30), teilen Sie diesen Mehlverschluss in zwei Hälften und ersetzen Sie eine Hälfte durch einen frischen Mehlverschluss, der ebenfalls in zwei Hälften geschnitten wurde. Alternativ können Sie die Mahlzeitkappen häufiger austauschen (d. h. alle 12 Stunden statt alle 24 Stunden). Beispiele für überbevölkerte Mehlkappen sind in Abbildung 2E, F zu sehen.
- Inkubieren Sie den Mehldeckel mit Larven bei 25 °C, bis die Larven das gewünschte Alter erreicht haben.
3. Sektion des Larvenfettkörpers (Abbildung 3)
HINWEIS: Dieses Protokoll beschreibt die Präparation mit einer 3-Well-Dissektionsschale.
- Pipettieren Sie ~400 μl Dissektions- und Bildgebungsmedium in jede Vertiefung einer 3-Well-Dissektionsschale.
- Waschen Sie zehn 72-96 h alte, gut genährte Wildtyp-Larven, indem Sie sie vorsichtig mit einer Sezierzange halten und in die Sezierlösung in der untersten Vertiefung tauchen und wieder heraustauchen, bis alle Speisereste abgewaschen sind. Bewegen Sie die sauberen Larven nach dem Spülen in die mittlere Mulde.
- Halten Sie die Larve mit einer Pinzette an den Mundhaken fest. Reißen Sie mit der anderen Pinzette eine Seite der Nagelhaut der Larve auf.
- Dieser Riss führt dazu, dass die Fettkörper aus der Larve herauslaufen. Die Fettkörper sind cremefarben und halbdurchscheinend und haben eine gitterartige Struktur (Abbildung 3I). Die fetten Körper neigen auch dazu, an sich selbst und der Sezierpinzette zu kleben. Sobald Sie identifiziert sind, sammeln Sie so viel Fettkörper wie möglich von jeder Larve und übertragen Sie ihn mit der Pinzette in die oberste Vertiefung mit 400 μl RT-Dissektionsmedium.
4. Dissektion des Larvengehirns (Abbildung 3)
- Waschen Sie die Versuchslarven wie oben beschrieben in Präparieren und Bildgebungsmedium, um sie von Speiseresten zu befreien. Um optimale Ergebnisse zu erzielen, vermeiden Sie es, unpräparierte Larven in der Präparierlösung zu lagern. Dies führt dazu, dass die Larven "ertrinken" und sich negativ auf die Qualität der präparierten Gehirne auswirken.
- Halten Sie die Larve mit einer Pinzette an den Mundhaken fest. Schneiden Sie mit einer anderen Pinzette vorsichtig etwa 1/3 der Larve von der hinteren Seite ab (Abbildung 3A). Dies führt dazu, dass Elemente des Verdauungstrakts, der Fettkörper, des Bindegewebes und des Nervensystems aus der geplatzten Seite der Larve "platzen" (Abbildung 3B).
- Halten Sie die Larve mit einer Pinzette an den Mundhaken fest. Bürsten Sie mit der anderen Pinzette vorsichtig die Nagelhaut in Richtung der Mundhaken, während Sie mit der Pinzette, die die Mundhaken hält, nach innen "drücken", bis die gesamte Larve von innen nach außen gedreht ist. Diese Bewegung ähnelt dem Drehen einer Socke "von innen nach außen" (Abbildung 3C, D).
- Drehen Sie die Larve so um, dass das zentrale Nervensystem und andere Gewebe nach außen zeigen, während sie noch mit der Kutikula verbunden sind. Lokalisieren Sie in diesem Schritt das zentrale Nervensystem (ZNS), um eine versehentliche Entfernung zu vermeiden. Entfernen Sie mit einer Pinzette vorsichtig das gesamte Nicht-ZNS-Gewebe, so dass nur das ZNS und das Gehirn an der Nagelhaut befestigt sind (Abbildung 3E).
- Das Gehirn wird über axonale Verbindungen mit der Kutikula verbunden. Schneiden Sie diese axonalen Verbindungen mit einer Mikrodissektionsschere ab, um das Gehirn von der Nagelhaut zu befreien. Schneiden Sie dazu zunächst vorsichtig unter die Hirnlappen (Abbildung 3F). Wiederholen Sie dies mit den Verbindungen unter dem ventralen Nervenstrang.
HINWEIS: Dieser Schritt kann mit einer Pinzette durchgeführt werden, wenn keine Mikrodissektionsschere verfügbar ist. Achten Sie bei der Verwendung einer Pinzette besonders darauf, dass das Hirngewebe bei der Entfernung aus der Nagelhaut nicht überdehnt wird, da sich mechanischer Stress negativ auf die Gesundheit des Gehirns auswirkt. - Überführen Sie das präparierte Gehirn in eine Vertiefung mit Präparations- und Bildgebungsmedium. Für bildgebende Experimente, die länger als 3 h dauern, verwenden Sie wie oben beschrieben ein mit Fettkörpern angereichertes Präparier- und Bildgebungsmedium. Sezieren Sie die Larven in Chargen, um die Sezierzeit unter 20 Minuten zu halten.
5. Montage und Belichtung (Abbildung 4)
- Für die Bildgebung mit einem membrangebundenen Objektträger34:
- Sammeln Sie sowohl präparierte Gehirne als auch isolierte Fettkörper in der letzten Vertiefung der Sezierschale.
- Montieren Sie die Hälfte des Objektträgers, indem Sie eine gasdurchlässige Membran über die Rückseite des Objektträgers legen, und drücken Sie den Spaltring in die Mitte, um ihn an Ort und Stelle zu halten (Abbildung 4A-C).
- Übertragen Sie mit einer 200-μl-Mikropipette bis zu 10 präparierte Gehirne und so viel Fettkörper wie möglich (siehe oben) in ~130-140 μl Präparier- und Bildgebungsmedium auf die Membran. Achten Sie darauf, das Medium mit den Proben in der Mitte der gasdurchlässigen Membran abzuscheiden (Abbildung 4D, E).
- Richten Sie die Gehirne für die Population der abzubildenden NBs und für die Art des verwendeten Mikroskops aus (Abbildung 4E). Positionieren Sie die Probe so nah wie möglich am Objektiv des Mikroskops. Um beispielsweise NBs in den zentralen Hirnlappen abzubilden, richten Sie die Gehirne so aus, dass die Hirnlappen dem Ziel am nächsten sind (Abbildung 4H).
- Sobald die Gehirne ausgerichtet sind, legen Sie vorsichtig ein Glasdeckglas auf die Lösung auf der Membran. Dies führt dazu, dass sich die Lösung, die das Gehirn und die Fettkörper enthält, über die gesamte Membran verteilt (Abbildung 4F).
- Tupfen Sie überschüssige Lösung ab, indem Sie ein Labortuch nahe an den Rand des Deckglases halten. Die optimale Menge an Lösung wird erreicht, wenn das Gehirn das Deckglas berührt, ohne gequetscht zu werden. Wenn in diesem Schritt eine Neuorientierung erforderlich ist, bewegen Sie das Deckglas vorsichtig, um das Gehirn zu bewegen.
- Immobilisieren Sie das Deckglas, indem Sie geschmolzene Vaseline mit einem Pinsel entlang der Ränder des Deckglases auftragen. Lassen Sie das Gelee fest werden (Abbildung 4G).
- Für die Bildgebung mit einem Multi-Well-Objektträger (Abbildung 4):
- 400 μl Bildgebungsmedium werden in eine Vertiefung eines Multi-Well-Objektträgers gegeben (in dem hier durchgeführten Experiment wurde ein gekammerter 8-Well-Mikro[μ]-Objektträger verwendet; Abbildung 4I). Übertragen Sie die zuvor präparierten Fettkörper in diese Vertiefung (siehe Schritt 3.4).
- Legen Sie bis zu 10 Gehirne in einem Cluster in der Nähe der Mitte des Bohrlochs ab (Abbildung 4J).
- Richten Sie die Gehirne für die Population der abzubildenden NBs und für den verwendeten Mikroskoptyp aus, wie in Schritt 5.1.4 beschrieben (Abbildung 4K). Ordnen Sie die Proben so an, dass sie nahe beieinander liegen. Dadurch wird der Abstand minimiert, den der Tisch zwischen den Proben zurücklegen muss, wodurch die Probendrift während der Erfassung verringert wird.
- Sobald die Gehirne im Brunnen ausgerichtet sind, lassen Sie die Gehirne 2-5 Minuten ruhen. Dies erhöht ihre Stabilität während des Transports/der Bildgebung. Bereiten Sie das Mikroskop während dieser Zeit auf die Aufnahme vor.
- Decken Sie den μ-Objektträger mit der Objektträgerabdeckung ab und legen Sie ihn in das Mikroskop ein. Beginnen Sie die Aufnahme mit der geringstmöglichen Laserleistung und Belichtungszeit, um das Ausbleichen zu minimieren.
6. Best Practices für Datenverarbeitung und -verwaltung
- Verarbeiten Sie die Daten nach Bedarf gemäß der verfügbaren Analysesoftware.
- Speichern Sie für das hier gezeigte Beispiel die erfassten Daten mit der SlideBook-Software als SlideBook-Bilddatei (.sld).
- Um mit dem Imaris File Converter in den proprietären Dateityp Imaris (.ims) zu konvertieren, öffnen Sie den Imaris File Converter in einem separaten Fenster. Klicken Sie auf und ziehen Sie die .sld-Dateien in den Abschnitt "Eingabe" des Imaris File Converters.
- Bestimmen Sie den gewünschten Ausgabeort für die konvertierten Dateien und klicken Sie auf "Alle starten".
- Zeigen Sie die Daten nach der Konvertierung in der Imaris-Software an und kommentieren Sie sie.
HINWEIS: Anstelle von Imaris können Alternativen für die Bildanalyse verwendet werden, z. B. Fidschi (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/) oder andere.
- Bewahren Sie so viele Originaldaten wie möglich auf, um eine ordnungsgemäße Aufzeichnung zu gewährleisten. Wenn die Erfassungssoftware beispielsweise in einem Dateiformat gespeichert, aber für die Analyse in ein anderes Format konvertiert wird, behalten Sie die erfasste Version der Daten bei.
- Notieren Sie für die Datenanalyse so viele Details wie möglich über die Proben- und Erfassungseinstellungen. Zu den wichtigsten Informationen, die aufbewahrt werden müssen, gehören der Genotyp der präparierten Larven, das Alter der Larven vor der Sektion, der Zustand des Mehlverschlusses, in dem sie aufgezogen wurden, die während der Bildgebung verwendete Laserleistung, die Belichtungszeit, die Dauer der Aufnahme und die zeitliche Auflösung.
7. Beispielhafte Quantifizierung der Zellzykluslänge (Abbildung 5)
HINWEIS: In diesem Beispiel wurden Larven abgebildet, die den Polaritätsmarker Pins (Pins::EGFP16) und das Mikrotubuli-bindende Protein Jupiter25 (cherry::Jupiter13) exprimieren. Die anschließende Analyse erfolgte mit der Software Imaris.
- Öffnen Sie den Film mit der Bildanalysesoftware Ihrer Wahl. Scrollen Sie durch die Länge des Films, um trennende NBs zu identifizieren, und beschriften Sie sie zum späteren Nachschlagen. Identifizieren Sie die teilenden NBs anhand ihrer unterschiedlichen mitotischen Spindeln (Abbildung 5C-E).
- Identifizieren Sie ein Referenzzellzyklusstadium, um die Zellzykluslänge zu bestimmen. In diesem Beispiel wird die Metaphase als Referenz verwendet.
- Bestimmen Sie manuell die Anzahl der Frames zwischen aufeinanderfolgenden Metaphasen und konvertieren Sie sie in Minuten oder Stunden, um die Zeit zu bestimmen, die zum Abschließen eines Zellzyklus benötigt wird.
- Nehmen Sie dazu die zeitliche Auflösung des Films und multiplizieren Sie sie mit der Anzahl der Bilder zwischen den Metaphasen. Wenn z. B. die zeitliche Auflösung des Films alle 5 Minuten ein Bild beträgt und Metaphasen in Bild 13 und Bild 35 beobachtet werden, beträgt die Zeit zwischen diesen Metaphasen 110 Minuten ([35 − 13] × 5).
- Zeichnen Sie die Daten mit einer geeigneten Software auf. Die hier gezeigten Daten wurden mit der PRISM-Software grafisch dargestellt.
8. Beispielhafte Quantifizierung der Ausrichtung der Zellspindel (Abbildung 5)
HINWEIS: In diesem Beispiel wird die Analyse mit der Imaris-Software durchgeführt.
- Öffnen Sie die Filmdatei in Imaris oder einer anderen Software Ihrer Wahl. Scrollen Sie durch die Länge des Films, um trennende NBs zu identifizieren, und beschriften Sie sie zum späteren Nachschlagen.
- Bestimmen Sie den von den Spindelpolen gebildeten Vektor mit den apikalen und basalen Zentrosomen (dargestellt durch m) wie folgt:
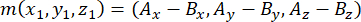
wobei Ax, Ay und Az die Koordinaten des apikalen Zentrosoms und Bx, By und Bz die Koordinaten des basalen Zentrosoms sind. In ähnlicher Weise wird die Achse des Teilungsvektors (dargestellt durch n) durch den Mittelpunkt der apikalen Pins::EGFP-Sichel und des basalen Kortex gebildet: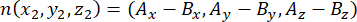
wobei Ax, Ay und Az die Koordinaten des Mittelpunkts der Pins::EGFP-Sichel und Bx, By und Bz die Koordinaten des Mittelpunkts des basalen Kortex sind. - Bestimmen Sie den Betrag der Vektoren m und n:
Magnitude von m: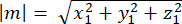
Magnitude von n: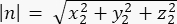
- Bestimmen Sie das Skalarprodukt (dargestellt durch k) von m und n:
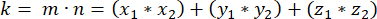
- Bestimmen Sie mit dem Skalarprodukt k und den Vektorgrößen m und n den Winkel zwischen den Vektoren:
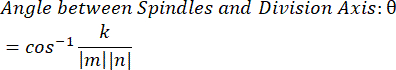
- Zeichnen Sie die Daten in der Software Ihrer Wahl. Die hier gezeigten Daten wurden in Microsoft Excel aufbereitet und in PRISM visualisiert.
Ergebnisse
Dissektion und Bildgebung von zentralen Hirnlappen-NBs, die Pins::EGFP und Cherry::Jupiter exprimieren
Um dieses Protokoll zu demonstrieren, wurden Larven, die UAS-getriebenes Cherry::Jupiter13 exprimieren und endogen mit Pins::EGFP16 markiert sind (w; worGal4, UAS-cherry::jupiter/CyO; Die Pins::EGFP/TM6B, Tb) wurden 4 h lang mit dem beschriebenen Protokoll unter Verwendung von Multi-Well-Imaging-Objektträgern abgebildet (
Diskussion
Dieses Protokoll beschreibt einen Ansatz für die Bildgebung von lebenden Explantatgehirnen aus Drosophila melanogaster-Larven . Das hier beschriebene Protokoll erlaubt es, Explantatgehirne für 12-20 Stunden unter den richtigen experimentellen Bedingungen zu beobachten. Besonderes Augenmerk muss auf die Vorbereitung der Proben und das Design der gewünschten Experimente gelegt werden. Wie bereits erwähnt, ist einer der kritischsten Faktoren, der die Qualität des präparierten Gewebes bestimmt, die Gesundheit ...
Offenlegungen
Die Autoren haben keine finanziellen Angaben zu machen.
Danksagungen
Diese Forschung wird durch R35GM148160 (C. C.) und einen National Institutes of Health (NIH) Training Grant T32 GM007270 (R. C. S) unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
Referenzen
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten