JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Drosophila melanogaster Third Instar 애벌레 뇌의 생세포 이미징
요약
여기에서 우리는 생리학적 조건에서 세포 및 세포 내 역학을 관찰하기 위해 Drosophila melanogaster 세 번째 instar 유충에서 살아있는 외식편 뇌를 준비, 해부, 장착 및 이미지화하는 워크플로에 대해 논의합니다.
초록
초파리 신경 줄기 세포(신경아세포, 이하 NB)는 비대칭 분열을 거쳐 자가 재생 신경아세포를 재생하는 동시에 분화 신경절 모세포(GMC)를 형성하며, 이 세포는 두 개의 뉴런 또는 신경교를 생성하기 위해 한 번의 추가 분열을 겪습니다. NB에 대한 연구는 세포 극성, 방추 방향, 신경 줄기 세포 자가 재생 및 분화의 기초가 되는 분자 메커니즘을 밝혀냈습니다. 이러한 비대칭 세포 분열은 생세포 이미징을 통해 쉽게 관찰할 수 있으므로 유충 NB는 생체 조직에서 비대칭 세포 분열의 시공간 역학을 조사하는 데 이상적입니다. 영양이 보충된 배지에서 적절하게 해부되고 이미지화되면 외식편 뇌의 NB는 12-20시간 동안 강력하게 분열합니다. 이전에 설명한 방법은 기술적으로 어렵고 현장을 처음 접하는 사람들에게는 어려울 수 있습니다. 여기에서는 지방 체내 보충제를 사용하여 살아있는 제3성 유충 뇌 외식편의 준비, 해부, 장착 및 이미징에 대한 프로토콜이 설명됩니다. 잠재적인 문제에 대해서도 논의하고 이 기술을 사용할 수 있는 방법에 대한 예가 제공됩니다.
서문
비대칭 세포 분열(ACD)은 RNA, 단백질 및 세포 소기관과 같은 세포 내 구성 요소가 딸 세포 사이에서 불균등하게 분할되는 과정입니다 1,2. 이 과정은 줄기 세포에서 흔히 볼 수 있으며, 줄기 세포는 ACD를 거쳐 다른 발달 운명을 가진 딸 세포를 생성합니다. 초파리 NB는 비대칭으로 분열하여 줄기를 유지하는 하나의 NB와 하나의 신경절 모세포(GMC)를 생성합니다. GMC는 분화 뉴런 또는 신경교를 생성하기 위해 추가 분열을 겪습니다3. 비대칭적으로 분열하는 NB는 현미경을 통해 쉽게 관찰할 수 있는 제3 유충의 발달 중인 뇌에 풍부합니다. 세 번째 instar 애벌레 단계에는 각 중앙 뇌엽 3,4,5,6에 약 100개의 NB가 존재합니다.
비대칭 세포 분열은 매우 역동적인 과정입니다. 생세포 이미징 프로토콜은 세포 극성 7,8,9,10, 방추 방향11,12,13, 악토미오신 피 질 14,15,16,17,18의 역학, 미세소관 및 중심체 생물학 19,20의 역학을 측정하고 정량화하는 데 사용되었습니다,21,22,23,24,25,26,27, 멤브레인10,28 및 염색질 역학 29. ACD에 대한 정성적 및 정량적 설명은 온전한 살아있는 뇌에서 NB를 분할하는 이미지를 위한 강력한 방법과 프로토콜에 의존합니다. 다음 프로토콜은 두 가지 다른 장착 접근 방식을 사용하여 생체 내 생세포 이미징을 위해 세 번째 instar 유충 뇌를 준비, 해부 및 이미지화하는 방법을 설명합니다. 이러한 방법은 줄기 세포 분열의 시공간 역학과 다른 뇌 세포의 분열에 관심이 있는 연구자에게 가장 적합하며, 세포 사건의 단기 및 장기 관찰이 가능하기 때문입니다. 또한 이러한 기술은 현장에 새로 온 사람들이 쉽게 접근할 수 있습니다. 우리는 형광 표지가 부착된 미세소관 및 피질 융합 단백질을 발현하는 유충 뇌를 통해 이 접근 방식의 효과와 적응성을 입증합니다. 우리는 또한 분석 방법과 다른 연구에 적용하기 위한 고려 사항에 대해 논의합니다.
프로토콜
참고: 그림 1 은 이 연구를 수행하는 데 필요한 자료를 보여줍니다.
1. 실험에 대한 고려 사항 및 준비
- 애벌레가 과밀하는 것을 방지하십시오.
참고: 이식편 유충 뇌의 품질은 해부 전 유충의 건강 및 품질과 직접적인 관련이 있습니다. 과밀로 영양실조에 걸린 유충은 일반적으로 뇌의 질이 낮다(30).- 영양실조를 피하기 위해 식사 뚜껑 접시당 20-30마리 이하의 유충이 존재하도록 하십시오. 이러한 예는 그림 2에서 볼 수 있습니다.
- 사용하기 전에 Schneider의 배지를 필터링하고 분취하십시오.
- 각 해부에 대해 분주된 Schneider의 곤충 배지에 1% 소 성장 혈청(BGS)을 보충하여 신선한 이미징 및 해부 배지를 준비합니다. 5mL의 부피의 해부 및 이미징 배지는 일반적으로 이미징 실험에 충분합니다.
- 사용하기 전에 보충된 매체를 실온(RT)으로 데우십시오.
- 수집할 동영상의 길이를 고려하고 이를 사용하여 이미징 매체의 보충, 장착 방법 및 현미경의 획득 설정을 고려합니다.
참고: 최적의 조건에서 BGS만 보충된 뇌의 NB는 최대 3시간 동안 강력하게 분열합니다.- 더 긴 영화가 필요한 실험을 수행할 때 4시간 이후의 분열을 지원하기 위해 이미징 매체에 애벌레 지방 신체 조직을 추가하여 이미징 매체를 보완합니다.
참고: 지방체는 NB 증식을 지원하는 미토겐을 분비한다31, 10마리의 유충에서 추출한 전지방체는 4-5개의 뇌를 지원하기에 충분하다. 또한, 멤브레인 결합 슬라이드로 이미지화 된 샘플은 10 h13,32 이상 동안 분열하는 것으로 나타 났으며, 다중 웰 슬라이드로 이미징 된 샘플은 일반적으로 덜 자주 분열합니다 (미공개 관찰). - 대안적으로, 이전에33에서 설명한 바와 같이, 더 긴 영화에 대해 더 복잡한 이미징 매체를 구현한다. 최상의 결과를 위해 노출 시간, 레이저 출력 및 샘플링 주파수를 조정하여 광 손상을 최소화합니다.
- 더 긴 영화가 필요한 실험을 수행할 때 4시간 이후의 분열을 지원하기 위해 이미징 매체에 애벌레 지방 신체 조직을 추가하여 이미징 매체를 보완합니다.
2. 유충 병기 결정 및 채취(그림 2)
- 1-5일령 암컷 처녀 파리와 1-7일령 성체 수컷 파리를 교배하여 원하는 유전자형을 가진 자손을 생산한다. 최적의 수확량을 위해 10-15 명의 여성 처녀와 5-10 명의 남성을 교배하십시오. 이 파리를 식사 캡(그림 2A-C)이 있는 플라이 케이지에 넣고 25°C에서 배양합니다.
- 매일 식사 캡을 바꾸십시오. 이것은 식사 뚜껑이 유충으로 과밀화되는 것을 방지하여 해부된 뇌의 품질을 저하시킵니다.
- 식사 캡이 유충으로 상당히 덮인 경우(예: >30) 이 식사 캡을 반으로 나누고 절반도 반으로 자른 신선한 식사 캡으로 교체하십시오. 또는 식사 캡을 더 자주 교체하십시오(즉, 24시간마다 대신 12시간마다). 인구 과잉 식사 캡의 예는 그림 2E, F에서 볼 수 있습니다.
- 유충이 원하는 나이에 도달할 때까지 25°C에서 유충과 함께 식사 캡을 배양합니다.
3. 유충 지방체 해부(그림 3)
참고: 이 프로토콜은 3웰 해부 접시를 사용한 해부를 설명합니다.
- ~400 μL의 해부 및 이미징 배지를 3웰 해부 접시의 각 웰에 피펫팅합니다.
- 72-96시간 된 잘 먹인 야생형 유충 10마리를 해부 집게로 부드럽게 잡고 모든 음식 미립자가 씻겨 나갈 때까지 가장 아래쪽 우물에 해부 용액에 담그고 빼냅니다. 헹구어 낸 후 깨끗한 유충을 중간 우물로 옮깁니다.
- 한 세트의 핀셋을 사용하여 입 고리로 유충을 잡습니다. 다른 핀셋 세트로 유충의 큐티클 한쪽을 파열시킵니다.
- 이 파열로 인해 뚱뚱한 몸이 애벌레에서 쏟아져 나옵니다. 뚱뚱한 몸체는 회백색이고 반투명하며 격자와 같은 구조를 갖습니다(그림 3I). 뚱뚱한 몸은 또한 자신과 해부 핀셋에 달라 붙는 경향이 있습니다. 일단 확인되면 각 유충에서 가능한 한 많은 지방체를 수집하고 400μL의 RT 해부 배지와 함께 집게와 함께 가장 위쪽 우물로 옮깁니다.
4. 유충 뇌 해부 (그림 3)
- 실험용 유충을 위와 같이 해부 및 이미징 배지로 세척하여 음식물 찌꺼기를 제거합니다. 최상의 결과를 얻으려면 해부되지 않은 유충을 해부 용액에 보관하지 마십시오. 이것은 애벌레가 "익사"하게 만들고 해부 된 뇌의 품질에 부정적인 영향을 미칩니다.
- 한 세트의 핀셋을 사용하여 입 고리로 유충을 잡습니다. 다른 핀셋 세트를 사용하여 뒤쪽에서 유충의 약 1/3을 부드럽게 잘라내거나 뜯어냅니다(그림 3A). 이로 인해 소화관, 지방체, 결합 조직 및 신경계의 요소가 유충의 파열된 쪽에서 "파열"됩니다(그림 3B).
- 한 세트의 핀셋을 사용하여 입 고리로 유충을 잡습니다. 다른 핀셋 세트를 사용하여 큐티클을 입 고리 쪽으로 부드럽게 빗질하면서 전체 유충이 뒤집힐 때까지 핀셋으로 입 고리를 잡고 안쪽으로 "밀어"냅니다. 이 동작은 양말을 "뒤집어서" 돌리는 것과 유사합니다(그림 3C, D).
- 중추 신경계와 다른 조직이 큐티클에 연결되어 있는 동안 바깥쪽을 향하도록 유충을 뒤집습니다. 이 단계에서 우발적인 제거를 방지하기 위해 중추신경계(CNS)를 찾습니다. 핀셋을 사용하여 CNS가 아닌 모든 조직을 부드럽게 제거하고 큐티클에 부착된 CNS와 뇌만 남깁니다(그림 3E).
- 뇌는 축삭 연결을 통해 큐티클에 부착됩니다. 미세 해부 가위를 사용하여 이 축삭 연결을 절단하여 큐티클에서 뇌를 분리합니다. 이렇게하려면 먼저 뇌엽 아래를 부드럽게 자릅니다 (그림 3F). 복부 신경줄 아래의 연결로 반복하십시오.
알림: 이 단계는 미세 해부 가위를 사용할 수 없는 경우 핀셋으로 수행할 수 있습니다. 기계적 스트레스는 뇌 건강에 부정적인 영향을 미치므로 핀셋을 사용할 때 큐티클에서 제거하는 동안 뇌 조직이 과도하게 늘어나지 않도록 특히 주의하십시오. - 해부된 뇌를 해부 및 이미징 매체가 있는 우물로 옮깁니다. 3시간보다 긴 이미징 실험의 경우 위에서 설명한 대로 지방체가 보충된 해부 및 이미징 매체를 사용하십시오. 해부 시간을 20분 미만으로 유지하기 위해 유충을 일괄적으로 해부합니다.
5. 장착 및 이미징(그림 4)
- 멤브레인-바운드 슬라이드(34)를 이용한 이미징의 경우:
- 해부된 뇌와 분리된 지방체를 해부 접시의 마지막 우물에 모으십시오.
- 슬라이드 뒷면에 가스 투과성 멤브레인을 배치하여 슬라이드의 절반을 조립하고 분할 링을 중앙으로 눌러 제자리에 고정합니다(그림 4A-C).
- 200μL 마이크로피펫을 사용하여 ~130-140μL의 해부 및 이미징 배지에서 최대 10개의 해부된 뇌와 가능한 한 많은 지방체(위 참조)를 멤브레인으로 옮깁니다. 기체 투과성 멤브레인의 중앙에 샘플이 있는 매체를 증착해야 합니다(그림 4D, E).
- 이미징할 NB의 개체군과 사용 중인 현미경 유형에 맞게 뇌의 방향을 지정합니다(그림 4E). 샘플을 현미경의 대물렌즈에 최대한 가깝게 배치합니다. 예를 들어, 중앙 뇌엽의 NB를 이미지화하려면 뇌엽이 대물렌즈에 가장 가깝도록 뇌의 방향을 지정합니다(그림 4H).
- 뇌의 방향이 정해지면 멤브레인의 용액 위에 유리 커버슬립을 부드럽게 놓습니다. 이렇게 하면 뇌와 지방체를 포함하는 용액이 막 전체로 퍼집니다(그림 4F).
- 실험실 조직을 커버슬립 가장자리 가까이에 대고 과도한 용액을 닦아냅니다. 최적의 용액 양은 뇌가 찌그러지지 않고 커버슬립에 닿을 때 달성됩니다. 이 단계에서 방향 전환이 필요한 경우 커버슬립을 조심스럽게 움직여 뇌를 움직입니다.
- 페인트 브러시로 커버슬립의 가장자리를 따라 녹인 바셀린을 발라 커버슬립을 고정합니다. 젤리가 굳을 때까지 기다립니다(그림 4G).
- 멀티웰 이미징 슬라이드를 사용한 이미징의 경우(그림 4):
- 400 μL의 이미징 배지를 멀티웰 슬라이드의 웰에 추가합니다(여기에서 수행된 실험에서는 챔버가 있는 8웰 마이크로[μ]-슬라이드를 사용했습니다. 그림 4I). 이전에 해부된 지방체를 이 우물로 옮깁니다(3.4단계 참조).
- 우물 중앙 근처의 클러스터에 최대 10개의 뇌를 퇴적합니다(그림 4J).
- 5.1.4단계(그림 4K)에 설명된 대로 이미징할 NB 모집단과 사용 중인 현미경 유형에 맞게 뇌의 방향을 지정합니다. 샘플이 서로 가까이 있도록 배열합니다. 이렇게 하면 스테이지가 샘플 간에 이동해야 하는 거리가 최소화되어 수집 중 샘플 드리프트가 줄어듭니다.
- 뇌가 우물에 방향을 잡으면 뇌가 2-5분 동안 안정되도록 합니다. 이는 운송/이미징 중 안정성을 증가시킵니다. 이 시간 동안 수집을 위해 현미경을 준비합니다.
- 슬라이드 커버로 μ 슬라이드를 덮고 현미경으로 옮깁니다. 광표백을 최소화하기 위해 가능한 가장 낮은 레이저 출력과 노출 시간으로 수집을 시작합니다.
6. 데이터 처리 및 관리 모범 사례
- 사용 가능한 분석 소프트웨어에 따라 필요에 따라 데이터를 처리합니다.
- 여기에 표시된 예의 경우 SlideBook 소프트웨어를 사용하여 수집한 데이터를 SlideBook 이미지 파일(.sld)로 저장합니다.
- Imaris 파일 변환기를 사용하여 Imaris의 독점 파일 형식(.ims)으로 변환하려면 별도의 창에서 Imaris 파일 변환기를 엽니다. .sld 파일을 클릭하여 Imaris 파일 변환기의 "입력" 섹션으로 드래그합니다.
- 변환된 파일의 원하는 출력 위치를 결정하고 "모두 시작"을 클릭합니다.
- 변환 후 Imaris 소프트웨어에서 데이터를 보고 주석을 달 수 있습니다.
참고: Fiji(https://hpc.nih.gov/apps/Fiji.html), Aivia(https://www.aivia-software.com/), Volocity(https://www.volocity4d.com/) 등과 같은 Imaris 대신 이미지 분석을 위한 대안을 사용할 수 있습니다.
- 적절한 기록 보관을 위해 가능한 한 많은 원본 데이터를 보관합니다. 예를 들어, 수집 소프트웨어가 하나의 파일 형식으로 저장되었지만 분석을 위해 다른 형식으로 변환된 경우 수집된 버전의 데이터를 유지합니다.
- 데이터 분석을 위해 샘플 및 수집 설정에 대해 가능한 한 많은 세부 정보를 기록해 두십시오. 보관해야 할 주요 정보에는 해부된 유충의 유전자형, 해부 전 유충의 나이, 사육된 식사 뚜껑의 상태, 이미징 중에 사용된 레이저 출력, 노출 시간, 획득 기간 및 시간 해상도가 포함됩니다.
7. 세포 주기 길이의 정량화 예(그림 5)
참고: 이 예에서는 극성 마커 Pins(Pins::EGFP16)와 미세소관 결합 단백질 Jupiter25 (cherry::Jupiter13)를 발현하는 유충을 이미지화했습니다. 후속 분석은 Imaris 소프트웨어를 사용하여 수행되었습니다.
- 선택한 이미지 분석 소프트웨어를 사용하여 동영상을 엽니다. 동영상의 길이를 스크롤하여 분할 NB를 식별하고 나중에 참조할 수 있도록 레이블을 지정합니다. 별개의 유사분열 방추로 분할하는 NB를 식별합니다(그림 5C-E).
- 세포 주기 길이를 결정하기 위해 참조 세포 주기 단계를 식별합니다. 이 예에서는 메타페이즈가 참조로 사용됩니다.
- 연속적인 중기 사이의 프레임 수를 수동으로 결정하고 분 또는 시간으로 변환하여 하나의 세포 주기를 완료하는 데 걸리는 시간을 결정합니다.
- 영화의 시간적 해상도를 취하고 중상 사이의 프레임 수를 곱하면 됩니다. 예를 들어, 동영상의 시간 해상도가 5분마다 한 프레임이고 프레임 13과 프레임 35에서 메타페이즈가 관찰되는 경우 이러한 메타페이즈 사이의 시간은 110분([35 − 13] × 5)이 됩니다.
- 적절한 소프트웨어를 사용하여 데이터를 플로팅합니다. 여기에 표시된 데이터는 PRISM 소프트웨어를 사용하여 표시되었습니다.
8. 셀 스핀들 정렬의 정량화 예(그림 5)
참고: 이 예에서는 Imaris 소프트웨어를 사용하여 분석을 수행합니다.
- Imaris 또는 선택한 다른 소프트웨어에서 동영상 파일을 엽니다. 동영상의 길이를 스크롤하여 분할 NB를 식별하고 나중에 참조할 수 있도록 레이블을 지정합니다.
- 다음과 같이 정점 및 기저 중심체( m으로 표시)를 사용하여 방추 극에 의해 형성된 벡터를 결정합니다.
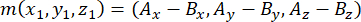
여기서 Ax, Ay 및 Az 는 정점 중심체의 좌표이고 Bx, By 및 Bz 는 기저 중심체의 좌표입니다. 유사하게, 나눗셈 벡터의 축( n으로 표시됨)은 정점 Pins::EGFP 초승달과 기저 피질의 중간점에 의해 형성됩니다.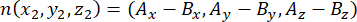
여기서 Ax, Ay 및 Az 는 Pins::EGFP 초승달 중간점의 좌표이고 Bx, By 및 Bz 는 기저 피질의 중간점 좌표입니다. - 벡터 m 과 n의 크기를 결정합니다.
m의 크기 :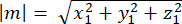
n의 크기: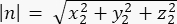
- m과 n의 내적(k로 표시)을 결정합니다.
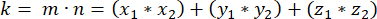
- 내적 k 와 벡터 크기 m 과 n을 사용하여 벡터 사이의 각도를 결정합니다.
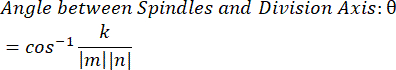
- 선택한 소프트웨어에서 데이터를 플로팅합니다. 여기에 표시된 데이터는 Microsoft Excel에서 작성하여 PRISM으로 시각화한 것입니다.
결과
Pins::EGFP 및 Cherry::Jupiter를 발현하는 중추 뇌엽 NB의 해부 및 이미징
이 프로토콜을 보여주기 위해 UAS 구동 Cherry::Jupiter13 및 내인성 태그가 지정된 Pins::EGFP16(w; worGal4, UAS-cherry::jupiter/CyO; Pins::EGFP/TM6B, Tb)는 멀티웰 이미징 슬라이드를 사용하여 설명된 프로토콜을 사용하여 4시간 동안 이미지화되었습니다(그림 5C,D). U...
토론
이 프로토콜은 Drosophila melanogaster 유충에서 살아있는 외식편 뇌의 이미징을 위한 한 가지 접근 방식을 설명합니다. 여기에 설명된 프로토콜은 올바른 실험 조건에서 체외이식편 뇌를 12-20시간 동안 관찰할 수 있도록 합니다. 샘플 준비와 원하는 실험의 설계에 특별한 고려가 이루어져야합니다. 위에서 언급했듯이 해부 된 조직의 품질을 결정하는 가장 중요한 요소 중 하나는 유충의 건강입?...
공개
저자는 선언할 재무 공개가 없습니다.
감사의 말
이 연구는 R35GM148160(C.C.) 및 국립보건원(NIH) 교육 보조금 T32 GM007270(R.C.S)의 지원을 받습니다
자료
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
참고문헌
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유