È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging delle cellule vive di Drosophila melanogaster Terzo stadio Larvale Cervelli
In questo articolo
Riepilogo
Qui, discutiamo di un flusso di lavoro per preparare, sezionare, montare e visualizzare cervelli di espianti vivi da larve di terzo stadio di Drosophila melanogaster per osservare le dinamiche cellulari e subcellulari in condizioni fisiologiche.
Abstract
Le cellule staminali neurali della Drosophila (neuroblasti, NB in seguito) subiscono divisioni asimmetriche, rigenerando il neuroblasto auto-rinnovante, formando anche una cellula madre gangliare differenziante (GMC), che subirà una divisione aggiuntiva per dare origine a due neuroni o glia. Studi in NB hanno scoperto i meccanismi molecolari alla base della polarità cellulare, dell'orientamento del fuso, dell'auto-rinnovamento delle cellule staminali neurali e della differenziazione. Queste divisioni cellulari asimmetriche sono facilmente osservabili tramite imaging di cellule vive, rendendo le NB larvali ideali per studiare le dinamiche spaziotemporali della divisione cellulare asimmetrica nei tessuti viventi. Se adeguatamente sezionati e ripresi in un mezzo integrato con sostanze nutritive, gli NB nei cervelli espiantati si dividono robustamente per 12-20 ore. I metodi descritti in precedenza sono tecnicamente difficili e possono essere impegnativi per chi è nuovo nel campo. Qui, viene descritto un protocollo per la preparazione, la dissezione, il montaggio e l'imaging di espianti cerebrali larvali vivi di terza stella utilizzando integratori per il corpo grasso. Vengono inoltre discussi i potenziali problemi e vengono forniti esempi su come questa tecnica può essere utilizzata.
Introduzione
La divisione cellulare asimmetrica (ACD) è il processo mediante il quale componenti subcellulari come RNA, proteine e organelli sono partizionati in modo diseguale tra le cellule figlie 1,2. Questo processo è comunemente visto nelle cellule staminali, che subiscono ACD per dare origine a cellule figlie con diversi destini di sviluppo. Drosofila Le NB si dividono asimmetricamente per produrre una NB, che mantiene la sua staminalità, e una cellula madre gangliare (GMC). Il GMC subisce ulteriori divisioni per produrre neuroni differenzianti o glia3. Le NB che si dividono asimmetricamente sono abbondanti nei cervelli in via di sviluppo delle larve di terzo stadio, che sono facilmente osservabili al microscopio. Al terzo stadio larvale instar, ci sono circa 100 NB presenti in ogni lobo cerebrale centrale 3,4,5,6.
La divisione cellulare asimmetrica è un processo altamente dinamico. I protocolli di imaging delle cellule vive sono stati utilizzati per misurare e quantificare la dinamica della polarità cellulare 7,8,9,10, l'orientamento del fuso 11,12,13, la dinamica della corteccia dell'attomiosina14,15,16,17,18, la biologia dei microtubuli e dei centrosomi 19,20,21,22,23,24,25,26,27, e membrana 10,28 e dinamica della cromatina 29. Le descrizioni qualitative e quantitative dell'ACD si basano su metodi e protocolli robusti per la divisione delle immagini NB in cervelli viventi intatti. Il seguente protocollo delinea i metodi per preparare, sezionare e visualizzare cervelli larvali di terzo stadio per l'imaging di cellule vive in vivo utilizzando due diversi approcci di montaggio. Questi metodi sono più adatti per i ricercatori interessati alle dinamiche spaziotemporali delle divisioni delle cellule staminali, nonché alle divisioni in altre cellule cerebrali, in quanto consentono osservazioni a breve e lungo termine di eventi cellulari. Inoltre, queste tecniche sono facilmente accessibili ai nuovi arrivati sul campo. Dimostriamo l'efficacia e l'adattabilità di questo approccio con cervelli larvali che esprimono microtubuli marcati in modo fluorescente e proteine di fusione corticale. Discutiamo inoltre i metodi di analisi e le considerazioni per l'applicazione in altri studi.
Protocollo
NOTA: la Figura 1 mostra i materiali necessari per eseguire questo studio.
1. Considerazioni e preparativi per l'esperimento
- Evitare che le larve si sovraffollano.
NOTA: La qualità del cervello larvale espiantato è direttamente correlata alla salute e alla qualità delle larve prima della dissezione. Le larve che sono malnutrite dal sovraffollamento produrranno generalmente cervelli di qualità inferiore30.- Assicurarsi che non siano presenti più di 20-30 larve per piatto di tappo del pasto per evitare la malnutrizione. Esempi di questi possono essere visti nella Figura 2.
- Filtrare e aliquote il mezzo Schneider prima dell'uso.
- Per ogni dissezione, preparare un nuovo mezzo di imaging e dissezione integrando il mezzo di insetto aliquotato di Schneider con siero di crescita bovina all'1% (BGS). Un volume di 5 ml di dissezione e mezzo di imaging è di solito sufficiente per un esperimento di imaging.
- Riscaldare il mezzo integrato a temperatura ambiente (RT) prima dell'uso.
- Considerare la lunghezza del filmato da raccogliere e utilizzarla per tenere conto dell'integrazione del mezzo di imaging, dell'approccio di montaggio e delle impostazioni di acquisizione del microscopio.
NOTA: In condizioni ottimali, gli NB nel cervello integrati con solo BGS si divideranno robustamente per un massimo di 3 ore.- Integrare il mezzo di imaging aggiungendo tessuti corporei di grasso larvale al mezzo di imaging per supportare le divisioni oltre le 4 ore quando si conducono esperimenti che richiedono filmati più lunghi.
NOTA: I corpi grassi secernono mitogeni che supportano la proliferazione NB31 e i corpi grassi interi di 10 larve sono sufficienti per sostenere da quattro a cinque cervelli. Inoltre, i campioni ripresi con un vetrino legato a membrana hanno dimostrato di dividersi per oltre 10 ore13,32, mentre i campioni ripresi con un vetrino multi-pozzetto di solito si dividono meno spesso (osservazioni non pubblicate). - In alternativa, implementare un supporto di imaging più complesso per i filmati più lunghi, come descritto in precedenza33. Riduci al minimo il fotodanneggiamento regolando il tempo di esposizione, la potenza del laser e la frequenza di campionamento per ottenere i migliori risultati.
- Integrare il mezzo di imaging aggiungendo tessuti corporei di grasso larvale al mezzo di imaging per supportare le divisioni oltre le 4 ore quando si conducono esperimenti che richiedono filmati più lunghi.
2. Stadiazione e raccolta delle larve (Figura 2)
- Incrociare mosche vergini femmine di 1-5 giorni con mosche maschi adulte di 1-7 giorni per produrre progenie con il genotipo desiderato. Per una resa ottimale, incrociare 10-15 vergini femmine con 5-10 maschi. Depositare queste mosche in una gabbia per mosche con un tappo per il pasto (Figura 2A-C) e incubare a 25 °C.
- Scambia il tappo del pasto ogni giorno. Ciò impedisce che i tappi dei pasti diventino sovraffollati di larve, il che riduce la qualità del cervello sezionato.
- Se il tappo del pasto è significativamente coperto di larve (cioè >30), dividere questo tappo del pasto a metà e sostituirne metà con un tappo per pasto fresco che è stato anche tagliato a metà. In alternativa, scambiare i tappi pasto su base più frequente (cioè ogni 12 ore invece di ogni 24 ore). Esempi di tappi pasto sovrappopolati possono essere visti nella Figura 2E, F.
- Incubare il cappuccio del pasto con le larve a 25 °C fino a quando le larve raggiungono l'età desiderata.
3. Dissezione del corpo grasso larvale (Figura 3)
NOTA: questo protocollo descrive le dissezioni utilizzando un piatto di dissezione a 3 pozzetti.
- Pipettare ~400 μL di dissezione e mezzo di imaging in ciascun pozzetto di un piatto di dissezione a 3 pozzetti.
- Lavare dieci larve selvatiche ben nutrite di 72-96 ore tenendole delicatamente con una pinza da dissezione e immergendole dentro e fuori dalla soluzione di dissezione nel pozzo più basso fino a quando tutte le particelle di cibo sono state lavate via. Dopo il risciacquo, spostare le larve pulite nel mezzo del pozzo.
- Usando una serie di pinzette, tieni la larva per i ganci della bocca. Con l'altra serie di pinzette, rompere un lato della cuticola della larva.
- Questa rottura causerà la fuoriuscita dei corpi grassi dalla larva. I corpi grassi sono bianchi sporchi e semi-traslucidi e avranno una struttura reticolare (Figura 3I). I corpi grassi tenderanno anche ad attaccarsi a se stessi e alle pinzette di dissezione. Una volta identificato, raccogliere quanto più grasso possibile da ciascuna larva e trasferirlo con la pinza al pozzo più alto con 400 μL di terreno di dissezione RT.
4. Dissezione cerebrale larvale (Figura 3)
- Lavare le larve sperimentali in dissezione e mezzo di imaging come sopra per liberarle dai residui di cibo. Per ottenere i migliori risultati, evitare di conservare le larve non sezionate nella soluzione di dissezione. Ciò causerà l'"annegamento" delle larve e avrà un impatto negativo sulla qualità dei cervelli sezionati.
- Usando una serie di pinzette, tieni la larva per i ganci della bocca. Utilizzando un'altra serie di pinzette, tagliare/strappare delicatamente circa 1/3 della larva dal lato posteriore (Figura 3A). Ciò farà sì che elementi del tratto digestivo, corpi grassi, tessuto connettivo e sistema nervoso "scoppino" dal lato rotto della larva (Figura 3B).
- Usando una serie di pinzette, tieni la larva per i ganci della bocca. Con l'altra serie di pinzette, spazzolare delicatamente la cuticola verso i ganci della bocca mentre "spingere" verso l'interno con le pinzette che tengono i ganci della bocca fino a quando l'intera larva è capovolta. Questo movimento è simile a girare un calzino "al rovescio" (Figura 3C, D).
- Capovolgere la larva in modo che il sistema nervoso centrale e altri tessuti siano rivolti verso l'esterno pur essendo ancora collegati alla cuticola. A questo punto, individuare il sistema nervoso centrale (SNC) per evitare la rimozione accidentale. Usando una pinzetta, rimuovere delicatamente tutto il tessuto non SNC, lasciando solo il SNC e il cervello attaccati alla cuticola (Figura 3E).
- Il cervello sarà attaccato alla cuticola tramite connessioni assonali. Usando le forbici da microdissezione, taglia queste connessioni assonali per liberare il cervello dalla cuticola. Per fare questo, prima tagliare delicatamente sotto i lobi cerebrali (Figura 3F). Ripetere con le connessioni sotto il cordone nervoso ventrale.
NOTA: Questo passaggio può essere eseguito con una pinzetta se non sono disponibili forbici per microdissezione. Prestare particolare attenzione quando si utilizzano le pinzette per assicurarsi che il tessuto cerebrale non sia eccessivamente teso durante la rimozione dalla cuticola perché lo stress meccanico influenzerà negativamente la salute del cervello. - Trasferire il cervello sezionato in un pozzo con dissezione e mezzo di imaging. Per esperimenti di imaging più lunghi di 3 ore, utilizzare la dissezione e il mezzo di imaging integrati con corpi grassi come descritto sopra. Sezionare le larve in lotti per mantenere il tempo di dissezione inferiore a 20 minuti.
5. Montaggio e imaging (Figura 4)
- Per l'imaging con un vetrino legato a membrana34:
- Raccogli sia i cervelli sezionati che i corpi grassi isolati nell'ultimo pozzetto del piatto di dissezione.
- Assemblare metà della diapositiva posizionando una membrana permeabile al gas sul retro della diapositiva e premere l'anello diviso al centro, tenendolo in posizione (Figura 4A-C).
- Utilizzando una micropipetta da 200 μL, trasferire fino a 10 cervelli sezionati e quanto più corpo grasso possibile (vedi sopra) in ~ 130-140 μL di dissezione e mezzo di imaging alla membrana. Assicurarsi di depositare il mezzo con i campioni al centro della membrana permeabile ai gas (Figura 4D, E).
- Orientare il cervello per la popolazione di NB da visualizzare e per il tipo di microscopio utilizzato (Figura 4E). Posizionare il campione il più vicino possibile all'obiettivo del microscopio. Ad esempio, per visualizzare le NB nei lobi cerebrali centrali, orientare il cervello in modo tale che i lobi cerebrali siano più vicini all'obiettivo (Figura 4H).
- Una volta che il cervello è orientato, posizionare delicatamente un vetrino di vetro sopra la soluzione sulla membrana. Ciò farà sì che la soluzione contenente il cervello e i corpi grassi si diffonda su tutta la membrana (Figura 4F).
- Tamponare la soluzione eccessiva tenendo un fazzoletto di laboratorio vicino al bordo del coprifoglio. La quantità ottimale di soluzione si ottiene quando il cervello tocca il coprislip senza essere schiacciato. Se in questa fase è necessario un riorientamento, spostare con attenzione il coprislip per spostare il cervello.
- Immobilizzare il coprivetrino applicando vaselina fusa lungo i bordi del coprivetrino con un pennello. Lasciare che la gelatina si solidifichi (Figura 4G).
- Per l'imaging con una diapositiva multipozzetto (Figura 4):
- Aggiungere 400 μL di mezzo di imaging a un pozzetto di un vetrino multi-pozzetto (nell'esperimento eseguito qui, è stato utilizzato un micro[μ]-slide a 8 pozzetti camerato; Figura 4I). Trasferire i corpi grassi precedentemente sezionati in questo pozzetto (vedere il passaggio 3.4).
- Deposita fino a 10 cervelli in un cluster vicino al centro del pozzo (Figura 4J).
- Orientare il cervello per la popolazione di NB da visualizzare e per il tipo di microscopio utilizzato, come descritto nella fase 5.1.4 (Figura 4K). Disporre i campioni in modo che siano vicini l'uno all'altro. Ciò ridurrà al minimo la distanza che lo stadio deve spostare tra i campioni, riducendo la deriva del campione durante l'acquisizione.
- Una volta che i cervelli sono stati orientati nel pozzo, lasciare che il cervello si stabilizzi per 2-5 minuti. Ciò aumenta la loro stabilità durante il trasporto/imaging. Preparare il microscopio per l'acquisizione durante questo periodo.
- Coprire il vetrino μ con il coperchio del vetrino e trasferirlo al microscopio. Inizia l'acquisizione con la potenza laser e il tempo di esposizione più bassi possibili per ridurre al minimo il fotosbiancamento.
6. Best practice per l'elaborazione e la gestione dei dati
- Elaborare i dati secondo necessità in base al software di analisi disponibile.
- Per l'esempio mostrato qui, salvare i dati acquisiti con il software SlideBook come file immagine SlideBook (.sld).
- Per convertire nel tipo di file proprietario di Imaris (.ims) utilizzando Imaris File Converter, aprire Imaris File Converter in una finestra separata. Fare clic e trascinare i file .sld nella sezione "input" di Imaris File Converter.
- Determina la posizione di output desiderata per i file convertiti e fai clic su "Inizia tutto".
- Dopo la conversione, visualizzare e annotare i dati nel software Imaris.
NOTA: Le alternative per l'analisi delle immagini possono essere utilizzate al posto di Imaris, come Fiji (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/) o altri.
- Conservare il maggior numero possibile di dati originali per una corretta tenuta dei registri. Ad esempio, se il software di acquisizione viene salvato in un formato di file ma viene convertito in un formato diverso per l'analisi, conservare la versione acquisita dei dati.
- Per l'analisi dei dati, mantenere un record del maggior numero possibile di dettagli sul campione e sulle impostazioni di acquisizione. Le informazioni chiave da conservare includono il genotipo delle larve sezionate, l'età delle larve prima della dissezione, lo stato del cappuccio del pasto in cui sono state allevate, la potenza laser utilizzata durante l'imaging, il tempo di esposizione, la durata dell'acquisizione e la risoluzione temporale.
7. Esempio di quantificazione della lunghezza del ciclo cellulare (Figura 5)
NOTA: in questo esempio, sono state visualizzate larve che esprimono i Pin marcatori di polarità (Pins::EGFP16) e la proteina legante i microtubuli Jupiter25 (cherry::Jupiter13). L'analisi successiva è stata eseguita utilizzando il software Imaris.
- Aprire il filmato utilizzando il software di analisi delle immagini preferito. Scorri la lunghezza del filmato per identificare i NB divisivi ed etichettali per riferimento futuro. Identificare gli NB divisori in base ai loro distinti fusi mitotici (Figura 5C-E).
- Identificare una fase del ciclo cellulare di riferimento per determinare la lunghezza del ciclo cellulare. In questo esempio, la metafase viene utilizzata come riferimento.
- Determinare manualmente il numero di fotogrammi tra le metafasi successive e convertirlo in minuti o ore per determinare il tempo impiegato per completare un ciclo di celle.
- A tale scopo, prendere la risoluzione temporale del filmato e moltiplicarla per il numero di fotogrammi tra le metafasi. Ad esempio, se la risoluzione temporale del filmato è di un fotogramma ogni 5 minuti e le metafasi sono osservate nel fotogramma 13 e nel fotogramma 35, il tempo tra queste metafasi sarebbe di 110 minuti ([35 − 13] × 5).
- Tracciare i dati con qualsiasi software appropriato. I dati mostrati qui sono stati tracciati utilizzando il software PRISM.
8. Esempio di quantificazione dell'allineamento del mandrino cellulare (Figura 5)
NOTA: in questo esempio, l'analisi viene eseguita utilizzando il software Imaris.
- Apri il file del filmato in Imaris o in un altro software di scelta. Scorri la lunghezza del filmato per identificare i NB divisivi ed etichettali per riferimento futuro.
- Determinare il vettore formato dai poli del fuso utilizzando i centrosomi apicali e basali (rappresentati da m), come segue:
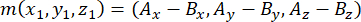
dove Ax, Ay e Az sono le coordinate del centrosoma apicale, e Bx, By e Bz sono le coordinate del centrosoma basale. Allo stesso modo, l'asse del vettore di divisione (rappresentato da n) è formato dal punto medio della mezzaluna apicale::EGFP e dalla corteccia basale: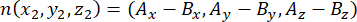
dove Ax, Ay e Az sono le coordinate del punto medio della mezzaluna dei Pini::EGFP, e Bx, By e Bz sono le coordinate del punto medio della corteccia basale. - Determinare la grandezza dei vettori m e n:
Magnitudo di m: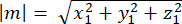
Magnitudine di n: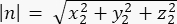
- Determinare il prodotto punto (rappresentato da k) di m e n:
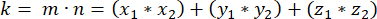
- Utilizzando il prodotto del punto k e le magnitudini vettoriali m e n, determinare l'angolo tra i vettori:
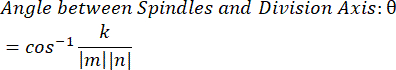
- Tracciare i dati nel software preferito. I dati mostrati qui sono stati preparati in Microsoft Excel e visualizzati in PRISM.
Risultati
Dissezione e imaging del lobo cerebrale centrale NB che esprimono Pins::EGFP e Cherry::Giove
Per mostrare questo protocollo, le larve esprimono Cherry::Jupiter13 guidato da UAS e Pins etichettati in modo endogeno::EGFP16 (w; worGal4, UAS-cherry::jupiter/CyO; I pin::EGFP/TM6B, Tb) sono stati ripresi per 4 ore utilizzando il protocollo descritto utilizzando vetrini di imaging multi-pozzetto (Figura 5C,D). Ulte...
Discussione
Questo protocollo delinea un approccio per l'imaging di cervelli vivi espiantati da larve di Drosophila melanogaster . Il protocollo qui descritto consente di osservare cervelli espiantati per 12-20 ore nelle giuste condizioni sperimentali. Particolare attenzione deve essere data alla preparazione dei campioni e alla progettazione degli esperimenti desiderati. Come accennato in precedenza, uno dei fattori più critici che determina la qualità del tessuto sezionato è la salute delle larve. Per ottenere la massi...
Divulgazioni
Gli autori non hanno informazioni finanziarie da dichiarare.
Riconoscimenti
Questa ricerca è supportata da R35GM148160 (C. C.) e da un National Institutes of Health (NIH) Training Grant T32 GM007270 (R. C. S)
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
Riferimenti
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon