Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de células vivas de cerebros larvales del tercer estadio de Drosophila melanogaster
En este artículo
Resumen
Aquí, discutimos un flujo de trabajo para preparar, diseccionar, montar y obtener imágenes de cerebros de explante vivos de larvas del tercer estadio de Drosophila melanogaster para observar la dinámica celular y subcelular en condiciones fisiológicas.
Resumen
Las células madre neurales de Drosophila (neuroblastos, NB en adelante) experimentan divisiones asimétricas, regenerando el neuroblasto autorrenovador, al tiempo que forman una célula madre ganglionar diferenciadora (GMC), que sufrirá una división adicional para dar lugar a dos neuronas o glía. Los estudios en NB han descubierto los mecanismos moleculares subyacentes a la polaridad celular, la orientación del huso, la autorrenovación de las células madre neurales y la diferenciación. Estas divisiones celulares asimétricas son fácilmente observables a través de imágenes de células vivas, lo que hace que las larvas sean ideales para investigar la dinámica espaciotemporal de la división celular asimétrica en el tejido vivo. Cuando se diseccionan adecuadamente y se obtienen imágenes en un medio suplementado con nutrientes, los NB en los cerebros de explante se dividen robustamente durante 12-20 h. Los métodos descritos anteriormente son técnicamente difíciles y pueden ser desafiantes para aquellos nuevos en el campo. Aquí, se describe un protocolo para la preparación, disección, montaje e imágenes de explantes cerebrales larvales vivos del tercer estadio utilizando suplementos corporales gordos. También se discuten los problemas potenciales y se proporcionan ejemplos de cómo se puede utilizar esta técnica.
Introducción
La división celular asimétrica (ACD) es el proceso por el cual los componentes subcelulares como el ARN, las proteínas y los orgánulos se dividen de manera desigual entre las células hijas 1,2. Este proceso se ve comúnmente en las células madre, que se someten a ACD para dar lugar a células hijas con diferentes destinos de desarrollo. Drosophila Los NB se dividen asimétricamente para producir un NB, que conserva su tallo, y una célula madre ganglionar (GMC). El GMC sufre más divisiones para producir neuronas diferenciadoras o glía3. Los NB que se dividen asimétricamente son abundantes en los cerebros en desarrollo de las larvas del tercer estadio, que se observan fácilmente a través de la microscopía. En la tercera etapa larvaria del estadio, hay aproximadamente 100 NB presentes en cada lóbulo cerebral central 3,4,5,6.
La división celular asimétrica es un proceso altamente dinámico. Se han utilizado protocolos de imagen de células vivas para medir y cuantificar la dinámica de la polaridad celular 7,8,9,10, la orientación del huso 11,12,13, la dinámica de la corteza de actomiosina 14,15,16,17,18, la biología de microtúbulos y centrosomas 19,20,21,22,23,24,25,26,27, y membrana 10,28 y dinámica de la cromatina 29. Las descripciones cualitativas y cuantitativas de ACD se basan en métodos y protocolos robustos para dividir imágenes NB en cerebros vivos intactos. El siguiente protocolo describe métodos para preparar, diseccionar y obtener imágenes de cerebros larvales del tercer estadio para obtener imágenes de células vivas in vivo utilizando dos enfoques de montaje diferentes. Estos métodos son más adecuados para investigadores interesados en la dinámica espaciotemporal de las divisiones de células madre, así como las divisiones en otras células cerebrales, ya que permiten observaciones a corto y largo plazo de eventos celulares. Además, estas técnicas son fácilmente accesibles para los recién llegados al campo. Demostramos la efectividad y adaptabilidad de este enfoque con cerebros larvales que expresan microtúbulos marcados fluorescentemente y proteínas de fusión cortical. Además, discutimos los métodos de análisis y las consideraciones para su aplicación en otros estudios.
Protocolo
NOTA: La figura 1 muestra los materiales necesarios para realizar este estudio.
1. Consideraciones y preparativos para el experimento
- Evite que las larvas se hacinen.
NOTA: La calidad de los cerebros larvales explante está directamente relacionada con la salud y la calidad de las larvas antes de la disección. Las larvas que están desnutridas por el hacinamiento generalmente producirán cerebros de menor calidad30.- Asegúrese de que no haya más de 20-30 larvas presentes por plato de tapa de comida para evitar la desnutrición. Ejemplos de estos se pueden ver en la Figura 2.
- Filtrar y alícuota el medio de Schneider antes de usarlo.
- Para cada disección, prepare nuevas imágenes y medio de disección complementando el medio de insectos de Schneider con suero de crecimiento bovino (BGS) al 1%. Un volumen de 5 ml de disección y medio de imagen suele ser suficiente para un experimento de imagen.
- Caliente el medio suplementado a temperatura ambiente (RT) antes de usarlo.
- Considere la duración de la película que se recopilará y utilícela para factorizar la suplementación del medio de imagen, el enfoque de montaje y los ajustes de adquisición del microscopio.
NOTA: En condiciones óptimas, los NB en cerebros suplementados solo con BGS se dividirán robustamente durante más de 3 h.- Complemente el medio de imagen agregando tejidos corporales grasos larvales al medio de imagen para soportar divisiones pasadas 4 h cuando realice experimentos que requieran películas más largas.
NOTA: Los cuerpos grasos secretan mitógenos que apoyan la proliferación de NB31, y los cuerpos enteros de grasa de 10 larvas son suficientes para soportar de cuatro a cinco cerebros. Además, se ha demostrado que las muestras fotografiadas con un portaobjetos unido a la membrana se dividen durante más de 10 h13,32, mientras que las muestras fotografiadas con un portaobjetos de múltiples pocillos generalmente se dividen con menos frecuencia (observaciones no publicadas). - Alternativamente, implemente un medio de imagen más complejo para películas más largas, como se describió anteriormente33. Minimice el fotodaño ajustando el tiempo de exposición, la potencia del láser y la frecuencia de muestreo para obtener mejores resultados.
- Complemente el medio de imagen agregando tejidos corporales grasos larvales al medio de imagen para soportar divisiones pasadas 4 h cuando realice experimentos que requieran películas más largas.
2. Estadificación y recolección de larvas (Figura 2)
- Cruce moscas vírgenes hembras de 1-5 días de edad con moscas macho adultas de 1-7 días de edad para producir progenie con el genotipo deseado. Para un rendimiento óptimo, cruce 10-15 vírgenes hembras con 5-10 machos. Depositar estas moscas en una jaula para moscas con un gorro de comida (Figura 2A-C) e incubar a 25 °C.
- Cambie la tapa de comida diariamente. Esto evita que las tapas de comida se llenen de larvas, lo que reduce la calidad de los cerebros disecados.
- Si la tapa de comida está cubierta significativamente de larvas (es decir, >30), divida esta tapa de comida por la mitad y reemplace una mitad con una tapa de comida nueva que también se haya cortado por la mitad. Alternativamente, cambie las tapas de comida con mayor frecuencia (es decir, cada 12 h en lugar de cada 24 h). Se pueden ver ejemplos de tapas de comida superpobladas en la Figura 2E, F.
- Incubar el tapón de comida con larvas a 25 °C hasta que las larvas alcancen la edad deseada.
3. Disección del cuerpo graso larval (Figura 3)
NOTA: Este protocolo describe las disecciones utilizando una placa de disección de 3 pocillos.
- Pipetear ~400 μL de disección y medio de imagen en cada pocillo de una placa de disección de 3 pocillos.
- Lave diez larvas de tipo silvestre de 72-96 h de edad bien alimentadas sosteniéndolas suavemente con pinzas de disección y sumergiéndolas dentro y fuera de la solución de disección en el pozo inferior hasta que se hayan lavado todas las partículas de alimentos. Después de enjuagar, mueva las larvas limpias al pozo medio.
- Usando un juego de pinzas, sostenga la larva por los ganchos bucales. Con el otro juego de pinzas, rompe un lado de la cutícula de la larva.
- Esta ruptura hará que los cuerpos gordos se derramen fuera de la larva. Los cuerpos grasos son blanquecinos y semitranslúcidos y tendrán una estructura en forma de celosía (Figura 3I). Los cuerpos gordos también tenderán a adherirse a sí mismos y a las pinzas de disección. Una vez identificado, recolecte la mayor cantidad posible de cuerpo graso de cada larva y transfiéralo con los fórceps al pozo superior con 400 μL de medio de disección RT.
4. Disección larvaria del cerebro (Figura 3)
- Lave las larvas experimentales en disección y medio de imagen como el anterior para liberarlas de residuos de alimentos. Para obtener los mejores resultados, evite almacenar larvas no disecadas en la solución de disección. Esto hará que las larvas se "ahoguen" y tendrá un impacto negativo en la calidad de los cerebros disecados.
- Usando un juego de pinzas, sostenga la larva por los ganchos bucales. Usando otro juego de pinzas, corta/arranca suavemente aproximadamente 1/3 de la larva del lado posterior (Figura 3A). Esto hará que elementos del tracto digestivo, cuerpos grasos, tejido conectivo y sistema nervioso "estallen" del lado roto de la larva (Figura 3B).
- Usando un juego de pinzas, sostenga la larva por los ganchos bucales. Con el otro juego de pinzas, cepille suavemente la cutícula hacia los ganchos bucales mientras "empuja" hacia adentro con las pinzas que sostienen los ganchos bucales hasta que toda la larva esté al revés. Este movimiento es similar a girar un calcetín "de adentro hacia afuera" (Figura 3C, D).
- Invierta la larva para que el sistema nervioso central y otros tejidos miren hacia afuera mientras aún están conectados a la cutícula. En este paso, localice el sistema nervioso central (SNC) para evitar la extirpación accidental. Usando pinzas, retire suavemente todo el tejido que no sea del SNC, dejando solo el SNC y el cerebro unidos a la cutícula (Figura 3E).
- El cerebro se unirá a la cutícula a través de conexiones axonales. Usando tijeras de microdisección, corte estas conexiones axonales para liberar el cerebro de la cutícula. Para hacer esto, primero corte suavemente debajo de los lóbulos cerebrales (Figura 3F). Repita con las conexiones debajo del cordón nervioso ventral.
NOTA: Este paso se puede hacer con pinzas si las tijeras de microdisección no están disponibles. Tenga especial cuidado al usar pinzas para asegurarse de que el tejido cerebral no se estire demasiado durante la extracción de la cutícula porque el estrés mecánico afectará negativamente la salud del cerebro. - Transfiera el cerebro diseccionado a un pozo con disección y medio de imagen. Para experimentos de imágenes de más de 3 h, use disección y medio de imagen suplementado con cuerpos grasos como se describió anteriormente. Diseccionar las larvas en lotes para mantener el tiempo de disección por debajo de 20 min.
5. Montaje e imágenes (Figura 4)
- Para obtener imágenes con una diapositiva34 unida a la membrana:
- Recolecte cerebros disecados y cuerpos grasos aislados en el último pocillo de la placa de disección.
- Ensamble la mitad del portaobjetos colocando una membrana permeable al gas sobre la parte posterior del portaobjetos y presione el anillo dividido en el centro, manteniéndolo en su lugar (Figura 4A-C).
- Usando una micropipeta de 200 μL, transfiera hasta 10 cerebros disecados y la mayor cantidad posible de cuerpo graso (ver arriba) en ~130-140 μL de disección y medio de imagen a la membrana. Asegúrese de depositar el medio con las muestras en el centro de la membrana permeable al gas (Figura 4D, E).
- Orientar los cerebros para la población de RNs a ser fotografiada y para el tipo de microscopio que se utiliza (Figura 4E). Coloque la muestra lo más cerca posible del objetivo del microscopio. Por ejemplo, para obtener imágenes de NB en los lóbulos centrales del cerebro, oriente los cerebros de tal manera que los lóbulos cerebrales estén más cerca del objetivo (Figura 4H).
- Una vez que los cerebros estén orientados, coloque suavemente un cubreobjetos de vidrio sobre la solución en la membrana. Esto hará que la solución que contiene los cerebros y los cuerpos grasos se extienda sobre la totalidad de la membrana (Figura 4F).
- Seque la solución excesiva sosteniendo un tejido de laboratorio cerca del borde del cubreobjetos. La cantidad óptima de solución se logra cuando los cerebros tocan el cubreobjetos sin ser aplastados. Si se requiere reorientación en este paso, mueva con cuidado el cubreobjetos para mover los cerebros.
- Inmovilice el cubreobjetos aplicando vaselina derretida a lo largo de los bordes del cubreobjetos con un pincel. Permita que la gelatina se solidifique (Figura 4G).
- Para obtener imágenes con una diapositiva de imágenes de múltiples pocillos (Figura 4):
- Agregue 400 μL de medio de imagen a un pocillo de un portaobjetos de múltiples pocillos (en el experimento realizado aquí, se utilizó un micro[μ] portaobjetos de 8 pocillos con cámara; Figura 4I). Transfiera los cuerpos grasos previamente diseccionados a este pocillo (ver paso 3.4).
- Depositar hasta 10 cerebros en un grupo cerca del centro del pozo (Figura 4J).
- Orientar los cerebros para la población de RN que se va a obtener imágenes y para el tipo de microscopio que se utiliza, como se describe en el paso 5.1.4 (Figura 4K). Organice las muestras de manera que estén cerca una de la otra. Esto minimizará la distancia que la etapa debe moverse entre las muestras, lo que reduce la deriva de la muestra durante la adquisición.
- Una vez que los cerebros se hayan orientado en el pozo, permita que los cerebros se asienten durante 2-5 minutos. Esto aumenta su estabilidad durante el transporte / imagen. Prepare el microscopio para la adquisición durante este tiempo.
- Cubra el portaobjetos μ con la cubierta del portaobjetos y transfiéralo al microscopio. Comience la adquisición con la menor potencia láser y tiempo de exposición posible para minimizar el fotoblanqueo.
6. Mejores prácticas de procesamiento y gestión de datos
- Procese los datos según sea necesario de acuerdo con el software de análisis disponible.
- Para el ejemplo que se muestra aquí, guarde los datos adquiridos con el software SlideBook como un archivo de imagen de SlideBook (.sld).
- Para convertir al tipo de archivo propietario de Imaris (.ims) utilizando el convertidor de archivos de Imaris, abra el convertidor de archivos de Imaris en una ventana separada. Haga clic y arrastre los archivos .sld a la sección "entrada" del convertidor de archivos Imaris.
- Determine la ubicación de salida deseada para los archivos convertidos y haga clic en "Iniciar todo".
- Después de la conversión, vea y anote los datos en el software Imaris.
NOTA: Se pueden usar alternativas para el análisis de imágenes en lugar de Imaris, como Fiji (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/) u otros.
- Conserve la mayor cantidad posible de datos originales para el mantenimiento adecuado de registros. Por ejemplo, si el software de adquisición se guarda en un formato de archivo pero se convierte a un formato diferente para su análisis, conserve la versión adquirida de los datos.
- Para el análisis de datos, mantenga un registro de tantos detalles como sea posible sobre la muestra y la configuración de adquisición. La información clave para retener incluye el genotipo de las larvas disecadas, la edad de las larvas antes de la disección, el estado de la tapa de comida en la que fueron criadas, la potencia del láser utilizada durante la obtención de imágenes, el tiempo de exposición, la duración de la adquisición y la resolución temporal.
7. Ejemplo de cuantificación de la duración del ciclo celular (Figura 5)
NOTA: en este ejemplo, se obtuvieron imágenes de larvas que expresan el marcador de polaridad Pins (Pins::EGFP16) y la proteína de unión a microtúbulos Júpiter25 (cereza::Júpiter13). El análisis posterior se realizó utilizando el software Imaris.
- Abra la película utilizando el software de análisis de imágenes de su elección. Desplácese por la longitud de la película para identificar los NB divididos y etiquételos para referencia futura. Identifique los RN divisorios por sus distintos husos mitóticos (Figura 5C-E).
- Identifique una etapa del ciclo celular de referencia para determinar la duración del ciclo celular. En este ejemplo, metaphase se utiliza como referencia.
- Determine manualmente el número de fotogramas entre metafases sucesivas y conviértalo en minutos u horas para determinar el tiempo necesario para completar un ciclo celular.
- Haga esto tomando la resolución temporal de la película y multiplicándola por el número de fotogramas entre metafases. Por ejemplo, si la resolución temporal de la película es de un fotograma cada 5 minutos, y las metafases se observan en el fotograma 13 y el fotograma 35, el tiempo entre estas metafases sería de 110 min ([35 − 13] × 5).
- Trazar los datos con cualquier software apropiado. Los datos que se muestran aquí se trazaron utilizando el software PRISM.
8. Ejemplo de cuantificación de la alineación del huso celular (Figura 5)
NOTA: En este ejemplo, el análisis se realiza utilizando el software Imaris.
- Abra el archivo de película en Imaris u otro software de su elección. Desplácese por la longitud de la película para identificar los NB divididos y etiquételos para referencia futura.
- Determine el vector formado por los polos del huso utilizando los centrosomas apical y basal (representados por m), de la siguiente manera:
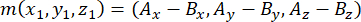
donde Ax, Ay, y Az son las coordenadas del centrosoma apical, y Bx, By y Bz son las coordenadas del centrosoma basal. Del mismo modo, el eje del vector de división (representado por n) está formado por el punto medio de los pines apicales::EGFP media luna y la corteza basal: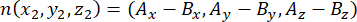
donde Ax, Ay, y Az son las coordenadas del punto medio de los Pines::EGFP media luna, y Bx, By, y Bz son las coordenadas del punto medio de la corteza basal. - Determina la magnitud de los vectores m y n:
Magnitud de m: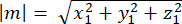
Magnitud de n: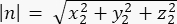
- Determine el producto punto (representado por k) de m y n:
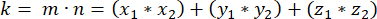
- Usando el producto de puntos k y las magnitudes vectoriales m y n, determina el ángulo entre los vectores:
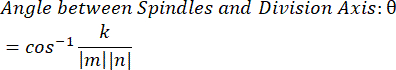
- Trazar los datos en el software de su elección. Los datos que se muestran aquí fueron preparados en Microsoft Excel y visualizados en PRISM.
Resultados
Disección e imágenes del lóbulo central del cerebro NBs que expresan Pins::EGFP y Cherry::Júpiter
Para mostrar este protocolo, las larvas que expresan Cherry::Jupiter13 y Pins endógenamente etiquetados::EGFP16 (w; worGal4, UAS-cherry::jupiter/CyO; Pins::EGFP/TM6B, Tb) se obtuvieron imágenes durante 4 h utilizando el protocolo descrito utilizando diapositivas de imágenes de múltiples pocillos (Figura 5C, D
Discusión
Este protocolo describe un enfoque para la obtención de imágenes de cerebros de explante vivos de larvas de Drosophila melanogaster . El protocolo descrito aquí permite observar cerebros de explante durante 12-20 h en las condiciones experimentales adecuadas. Se debe prestar especial atención a la preparación de muestras y al diseño de los experimentos deseados. Como se mencionó anteriormente, uno de los factores más críticos que determina la calidad del tejido disecado es la salud de las larvas. Para l...
Divulgaciones
Los autores no tienen divulgaciones financieras que declarar.
Agradecimientos
Esta investigación cuenta con el apoyo de R35GM148160 (C. C.) y una subvención de capacitación T32 de los Institutos Nacionales de Salud (NIH) GM007270 (R. C. S)
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
Referencias
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados