A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הדמיה של תאים חיים של Drosophila melanogaster מוח זחל כוכב שלישי
In This Article
Summary
כאן, אנו דנים בתהליך עבודה להכנה, ניתוח, הרכבה ותמונה של מוחות צמחים חיים מזחלי Drosophila melanogaster Third instar כדי לצפות בדינמיקה התאית והתת-תאית בתנאים פיזיולוגיים.
Abstract
תאי גזע עצביים דרוזופילה (נוירובלסטים, NBs להלן) עוברים חלוקות אסימטריות, מחדשות את הנוירובלסט המתחדש מעצמו, תוך יצירת תא אם גנגליון מתמיין (GMC), שיעבור חלוקה אחת נוספת כדי ליצור שני נוירונים או גליה. מחקרים ב- NBs חשפו את המנגנונים המולקולריים העומדים בבסיס קוטביות התא, כיוון הציר, התחדשות עצמית של תאי גזע עצביים והתמיינות. חלוקות תאים אסימטריות אלה ניתנות לצפייה בקלות באמצעות הדמיה של תאים חיים, מה שהופך את NBs הזחלים למתאימים באופן אידיאלי לחקר הדינמיקה המרחבית-זמנית של חלוקת תאים אסימטרית ברקמה חיה. כאשר מנתחים אותם כראוי ומדמיינים אותם במדיום עם תוספי תזונה, NBs במוחות explant מתחלקים בצורה חזקה במשך 12-20 שעות. שיטות שתוארו קודם לכן הן קשות מבחינה טכנית ועשויות להיות מאתגרות לאלה החדשים בתחום. כאן, מתואר פרוטוקול להכנה, נתיחה, הרכבה והדמיה של צמחי מוח חיים של זחל שלישי באמצעות תוספי גוף שמנים. בעיות פוטנציאליות נדונות גם, ודוגמאות מסופקות כיצד טכניקה זו ניתן להשתמש.
Introduction
חלוקת תאים אסימטרית (ACD) היא התהליך שבו רכיבים תת-תאיים כגון רנ"א, חלבונים ואברונים מחולקים באופן לא שווה בין תאי בת 1,2. תהליך זה נפוץ בתאי גזע, אשר עוברים ACD כדי ליצור תאי בת עם גורלות התפתחותיים שונים. דרוזופילה NBs מתחלקים באופן א-סימטרי כדי לייצר NB אחד, ששומר על הגבעול שלו, ותא אם גנגליון אחד (GMC). GMC עובר חלוקות נוספות כדי לייצר נוירונים מתמיינים או גליה3. NBs המתחלקים באופן א-סימטרי נמצאים בשפע במוחות המתפתחים של זחלי כוכב שלישי, אשר נצפים בקלות באמצעות מיקרוסקופיה. בשלב הזחל השלישי של הכוכב, ישנם בערך 100 NBs נוכחים בכל אונת מוח מרכזית 3,4,5,6.
חלוקת תאים אסימטרית היא תהליך דינמי ביותר. פרוטוקולי הדמיה של תאים חיים שימשו למדידה וכימות של הדינמיקה של קוטביות התא 7,8,9,10, כיוון ציר 11,12,13, הדינמיקה של קליפת המוח אקטומיוזין14,15,16,17,18, מיקרוטובול וביולוגיה צנטרוזומית 19,20,21,22,23,24,25,26,27, וממברנה 10,28 ודינמיקה של כרומטין 29. תיאורים איכותיים וכמותיים של ACD מסתמכים על שיטות ופרוטוקולים חזקים כדי לדמיין חלוקת NBs במוחות חיים שלמים. הפרוטוקול הבא מתאר שיטות להכנה, ניתוח ותמונה של מוחות זחלי כוכב שלישי להדמיית תאים חיים in vivo באמצעות שתי גישות הרכבה שונות. שיטות אלה מתאימות ביותר לחוקרים המתעניינים בדינמיקה מרחבית-זמנית של חלוקות תאי גזע, כמו גם חלוקות בתאי מוח אחרים, שכן הן מאפשרות תצפיות קצרות וארוכות טווח של אירועים תאיים. בנוסף, טכניקות אלה נגישות בקלות למצטרפים חדשים לתחום. אנו מדגימים את היעילות ויכולת ההסתגלות של גישה זו עם מוחות זחלים המבטאים מיקרוטובולים מתויגים פלואורסצנטית וחלבוני היתוך קליפת המוח. כמו כן נדון בשיטות ניתוח ובשיקולים ליישום במחקרים אחרים.
Protocol
הערה: איור 1 מציג את החומרים הדרושים לביצוע מחקר זה.
1. שיקולים והכנות לניסוי
- למנוע צפיפות יתר של הזחלים.
הערה: איכות מוחות הזחלים קשורה ישירות לבריאות ואיכות הזחלים לפני הנתיחה. זחלים הסובלים מתת-תזונה כתוצאה מצפיפות יתר יניבו בדרך כלל מוחות באיכות נמוכה יותר30.- ודא כי לא יותר מ 20-30 זחלים נמצאים בכל צלחת כובע ארוחה כדי למנוע תת תזונה. דוגמאות לכך ניתן לראות באיור 2.
- סנן והעלה את המדיום של שניידר לפני השימוש.
- עבור כל דיסקציה, הכינו מדיום הדמיה ודיסקציה טרי על ידי תוספת מדיום החרקים של שניידר עם 1% סרום גידול בקר (BGS). נפח של 5 מ"ל של דיסקציה ואמצעי הדמיה מספיק בדרך כלל לניסוי הדמיה.
- יש לחמם את התוסף לטמפרטורת החדר (RT) לפני השימוש.
- קחו בחשבון את אורך הסרט שיש לאסוף, והשתמשו בו כדי לשקלל את התוספת של מדיום ההדמיה, את גישת ההרכבה ואת הגדרות הרכישה של המיקרוסקופ.
הערה: בתנאים אופטימליים, NBs במוחות בתוספת BGS בלבד יתחלקו בצורה חזקה כלפי מעלה של 3 שעות.- השלם את מדיום ההדמיה על ידי הוספת רקמות גוף שומן הזחל למדיום ההדמיה כדי לתמוך בחלוקות מעבר ל -4 שעות בעת ביצוע ניסויים הדורשים סרטים ארוכים יותר.
הערה: גופי שומן מפרישים מיטוגנים התומכים בהתפשטות NB31, וגופי שומן מלאים מ-10 זחלים מספיקים כדי לתמוך בארבעה עד חמישה מוחות. יתר על כן, דגימות שצולמו עם שקופית קשורה לקרום הוכחו מתחלקות במשך יותר מ-10 שעות13,32, בעוד שדגימות שצולמו עם שקופית מרובת בארות בדרך כלל מתחלקות בתדירות נמוכה יותר (תצפיות שלא פורסמו). - לחלופין, הטמיעו אמצעי הדמיה מורכב יותר עבור סרטים ארוכים יותר, כפי שתואר קודם לכן33. צמצם את נזקי האור על-ידי התאמת זמן החשיפה, עוצמת הלייזר ותדירות הדגימה לקבלת התוצאות הטובות ביותר.
- השלם את מדיום ההדמיה על ידי הוספת רקמות גוף שומן הזחל למדיום ההדמיה כדי לתמוך בחלוקות מעבר ל -4 שעות בעת ביצוע ניסויים הדורשים סרטים ארוכים יותר.
2. היערכות ואיסוף הזחלים (איור 2)
- לחצות זבובים בתולים נקבה בת 1-5 ימים עם זבובים זכרים בוגרים בני 1-7 ימים כדי לייצר צאצאים עם הגנוטיפ הרצוי. לקבלת יבול אופטימלי, לחצות 10-15 בתולות נקבות עם 5-10 זכרים. הכניסו את הזבובים האלה לכלוב זבובים עם פקק ארוחה (איור 2A-C), ודגרו בטמפרטורה של 25°C.
- החליפו את פקק הארוחה מדי יום. זה מונע מכובעי הארוחה להיות צפופים בזחלים, מה שמפחית את איכות המוח המנותח.
- אם מכסה הארוחה מכוסה משמעותית בזחלים (כלומר >30), פצלו את פקק הארוחה לשניים, והחליפו חצי בפקק ארוחה טרי שגם הוא נחתך לשניים. לחלופין, החליפו את מכסות הארוחה על בסיס תכוף יותר (כלומר, כל 12 שעות במקום כל 24 שעות). דוגמאות למכסות ארוחה מאוכלסות יתר על המידה ניתן לראות באיור 2E, F.
- דוגרים על פקק הארוחה עם הזחלים ב 25 מעלות צלזיוס עד שהזחלים מגיעים לגיל הרצוי.
3. דיסקציה של גוף שומן הזחל (איור 3)
הערה: פרוטוקול זה מתאר נתיחות באמצעות צלחת דיסקציה של 3 בארות.
- פיפטה ~ 400 μL של אמצעי דיסקציה והדמיה לכל באר של צלחת דיסקציה 3 בארות.
- שטפו עשרה זחלי בר בני 72-96 שעות הניזונים היטב על ידי החזקתם בעדינות במלקחיים לדיסקציה וטבילתם בתמיסת דיסקציה וממנה בתחתית הבאר עד שכל חלקיקי המזון נשטפו. לאחר השטיפה, מעבירים את הזחלים הנקיים לבאר האמצעית.
- בעזרת סט אחד של פינצטה, החזיקו את הזחל ליד ווי הפה. עם קבוצה אחרת של פינצטה, לקרוע צד אחד של הקוטיקולה של הזחל.
- קרע זה יגרום לגופי השומן להישפך החוצה מהזחל. גופי השומן הם בצבע אוף-ווייט ושקופים למחצה, ויהיה להם מבנה דמוי סריג (איור 3I). גם הגופים השמנים נוטים להיצמד לעצמם ולפינצטה הדיסקציה. לאחר הזיהוי, אספו כמה שיותר שומן מכל זחל, והעבירו אותו עם המלקחיים לבאר העליונה ביותר עם 400 מיקרוליטר של מדיום דיסקציה RT.
4. דיסקציה מוחית של הזחל (איור 3)
- שטפו את זחלי הניסוי בדיסקציה ובאמצעי הדמיה כנ"ל כדי לשחרר אותם משאריות מזון. לקבלת התוצאות הטובות ביותר, הימנעו מאחסון זחלים לא מנותחים בתמיסת הדיסקציה. זה יגרום לזחלים "לטבוע" וישפיע לרעה על איכות המוח המנותח.
- בעזרת סט אחד של פינצטה, החזיקו את הזחל ליד ווי הפה. בעזרת קבוצה אחרת של פינצטה, חתכו/תלשו בעדינות כ-1/3 מהזחל מהצד האחורי (איור 3A). זה יגרום לאלמנטים של מערכת העיכול, גופי השומן, רקמת החיבור ומערכת העצבים "להתפוצץ" החוצה מהצד הקרוע של הזחל (איור 3B).
- בעזרת סט אחד של פינצטה, החזיקו את הזחל ליד ווי הפה. בעזרת הפינצטה השנייה, מברישים בעדינות את הקוטיקולה לכיוון ווי הפה תוך כדי "דחיפה" פנימה כשהפינצטה מחזיקה את ווי הפה עד שכל הזחל הופך מבפנים החוצה. התנועה הזו דומה להפיכת גרב "מבפנים החוצה" (איור 3C, D).
- הפוך את הזחל כך שמערכת העצבים המרכזית ורקמות אחרות פונות החוצה ועדיין מחוברות לקוטיקולה. בשלב זה, אתר את מערכת העצבים המרכזית (CNS) כדי למנוע הסרה בשוגג. באמצעות פינצטה, הסירו בעדינות את כל הרקמה שאינה CNS, והשאירו רק את מערכת העצבים המרכזית ואת המוח מחוברים לקוטיקולה (איור 3E).
- המוח יהיה מחובר לקוטיקולה באמצעות חיבורים אקסונליים. באמצעות מספריים microdissection, לחתוך את הקשרים axonal האלה כדי לשחרר את המוח מן הקוטיקולה. כדי לעשות זאת, תחילה חתכו בעדינות מתחת לאונות המוח (איור 3F). חזור על הפעולה עם החיבורים מתחת לחוט העצבים הגחוני.
הערה: שלב זה עשוי להיעשות עם פינצטה אם מספריים microdissection אינם זמינים. היזהר במיוחד בעת שימוש בפינצטה כדי להבטיח שרקמת המוח לא תימתח יתר על המידה במהלך ההסרה מהציפורן מכיוון שלחץ מכני ישפיע לרעה על בריאות המוח. - מעבירים את המוח המנותח לבאר עם דיסקציה ואמצעי הדמיה. לניסויי הדמיה ארוכים מ-3 שעות, יש להשתמש בדיסקציה ובאמצעי הדמיה בתוספת גופי שומן כמתואר לעיל. נתחו את הזחלים בקבוצות כדי לשמור על זמן הדיסקציה מתחת ל-20 דקות.
5. הרכבה והדמיה (איור 4)
- להדמיה עם שקופית34 הקשורה לקרום:
- אספו גם מוחות מנותחים וגם גופי שומן מבודדים בבאר האחרונה של צלחת הדיסקציה.
- הרכיבו מחצית מהמגלשה על-ידי הנחת קרום חדיר גז על גב המגלשה, ולחצו על הטבעת המפוצלת במרכז, תוך החזקתה במקומה (איור 4A-C).
- באמצעות מיקרופיפטה של 200 μL, העבר עד 10 מוחות מנותחים וכמה שיותר גוף שומן (ראה לעיל) ב~ 130-140 μL של דיסקציה ואמצעי הדמיה לממברנה. הקפידו להפקיד את התווך עם הדגימות במרכז הממברנה החדירה לגז (איור 4D, E).
- כוונו את המוחות לצילום אוכלוסיית NBs ולסוג המיקרוסקופ שבו משתמשים (איור 4E). מקמו את הדגימה קרוב ככל האפשר למטרת המיקרוסקופ. לדוגמה, כדי לדמיין NBs באונות המוח המרכזיות, כוונו את המוחות כך שאונות המוח יהיו הקרובות ביותר למטרה (איור 4H).
- ברגע שהמוח מכוון, הניחו בעדינות כיסוי זכוכית על גבי התמיסה על הממברנה. זה יגרום לתמיסה שמכילה את המוח ואת גופי השומן להתפשט על פני כל הממברנה (איור 4F).
- כתם תמיסה מוגזמת על ידי החזקת רקמת מעבדה קרוב לקצה הכיסוי. הכמות האופטימלית של תמיסה מושגת כאשר המוח נוגע בכיסוי מבלי להימעך. אם נדרשת התמצאות מחדש בשלב זה, הזיזו בזהירות את הכיסוי כדי להזיז את המוח.
- יש לשתק את הכיסוי על ידי מריחת ג'ל פטרולאום מומס לאורך קצוות הכיסוי בעזרת מכחול. אפשרו לג'לי להתמצק (איור 4G).
- להדמיה עם שקופית הדמיה מרובת בארות (איור 4):
- הוסף 400 μL של אמצעי הדמיה לבאר של שקופית מרובת בארות (בניסוי שבוצע כאן, נעשה שימוש בשקופית מיקרו[μ] קאמרית; איור 4I). העבירו את גופי השומן שנותחו בעבר לבאר זו (ראו שלב 3.4).
- הפקדת עד 10 מוחות בצביר ליד מרכז הבאר (איור 4J).
- כוונו את המוחות לצילום אוכלוסיית NBs ולסוג המיקרוסקופ שבו משתמשים, כפי שמתואר בשלב 5.1.4 (איור 4K). סדרו את הדגימות כך שיהיו קרובות זו לזו. פעולה זו תמזער את המרחק שהבמה צריכה לעבור בין דגימות, מה שיפחית את סחף הדגימה במהלך הרכישה.
- לאחר שהמוחות מכוונים לבאר, אפשרו למוח להסתפק במשך 2-5 דקות. זה מגביר את יציבותם במהלך הובלה/הדמיה. הכינו את המיקרוסקופ לרכישה בתקופה זו.
- כסו את המגלשה μ בכיסוי השקופית והעבירו אותה למיקרוסקופ. התחל ברכישה עם עוצמת הלייזר וזמן החשיפה הנמוכים ביותר האפשריים כדי למזער את ההלבנה.
6. שיטות עבודה מומלצות לעיבוד וניהול נתונים
- עבד את הנתונים לפי הצורך בהתאם לתוכנת הניתוח הזמינה.
- עבור הדוגמה המוצגת כאן, שמור את הנתונים שנרכשו באמצעות תוכנת SlideBook כקובץ תמונה של SlideBook ( .sld).
- כדי להמיר לסוג הקובץ הקנייני של Imaris ( .ims) באמצעות Imaris File Converter, פתח את Imaris File Converter בחלון נפרד. לחץ על וגרור את קבצי ה- .sld לקטע "קלט" של ממיר הקבצים Imaris.
- קבע את מיקום הפלט הרצוי עבור הקבצים שהומרו ולחץ על "התחל הכל".
- לאחר ההמרה, הצג והוסף ביאורים לנתונים בתוכנת Imaris.
הערה: ניתן להשתמש בחלופות לניתוח תמונות במקום Imaris, כגון פיג'י (https://hpc.nih.gov/apps/Fiji.html), Aivia (https://www.aivia-software.com/), Volocity (https://www.volocity4d.com/) או אחרות.
- שמור כמה שיותר מהנתונים המקוריים לצורך שמירת רשומות נאותה. לדוגמה, אם תוכנת הרכישה נשמרת בתבנית קובץ אחת אך מומרת לתבנית אחרת לצורך ניתוח, שמור על הגרסה שנרכשה של הנתונים.
- לצורך ניתוח נתונים, שמור תיעוד של פרטים רבים ככל האפשר על הגדרות הדגימה והרכישה. מידע מרכזי שיש לשמור כולל את הגנוטיפ של הזחלים המנותחים, גיל הזחלים לפני הנתיחה, מצב פקק הארוחה שבו גדלו, כוח הלייזר ששימש במהלך ההדמיה, זמן החשיפה, משך הרכישה והרזולוציה הזמנית.
7. כימות לדוגמה של אורך מחזור התא (איור 5)
הערה: בדוגמה זו צולמו זחלים המבטאים את סמן הקוטביות Pins (Pins::EGFP16) ואת החלבון קושר המיקרוטובולים Jupiter25 (דובדבן::יופיטר13). הניתוח הבא בוצע באמצעות תוכנת אימריס.
- פתח את הסרט באמצעות תוכנת ניתוח התמונות הנבחרת. גלול לאורך הסרט כדי לזהות NBs מחולקים, ותייג אותם לעיון עתידי. זהו את ה-NBs המתחלקים לפי הצירים המיטוטיים, הנפרדים שלהם (איור 5C-E).
- זהה שלב מחזור תא התייחסות כדי לקבוע את אורך מחזור התא. בדוגמה זו, metaphase משמש כהפניה.
- קבע ידנית את מספר המסגרות בין מטא-פאזות עוקבות, והמר אותו לדקות או לשעות כדי לקבוע את הזמן הדרוש להשלמת מחזור תא אחד.
- עשו זאת על ידי לקיחת הרזולוציה הזמנית של הסרט והכפלתה במספר הפריימים בין מטאפאזות. לדוגמה, אם הרזולוציה הטמפורלית של הסרט היא פריים אחד בכל 5 דקות, ומטאפאזות נצפות בפריים 13 ובפריים 35, הזמן בין מטאפאזות אלה יהיה 110 דקות ([35 − 13] × 5).
- התווה את הנתונים עם כל תוכנה מתאימה. הנתונים המוצגים כאן שורטטו באמצעות תוכנת PRISM.
8. כימות לדוגמה של יישור ציר התא (איור 5)
הערה: בדוגמה זו, הניתוח מתבצע באמצעות תוכנת Imaris.
- פתח את קובץ הסרט ב- Imaris או בתוכנה אחרת לבחירה. גלול לאורך הסרט כדי לזהות NBs מחולקים, ותייג אותם לעיון עתידי.
- קבע את הווקטור שנוצר על ידי מוטות הציר באמצעות הצנטרוזומים האפיקליים והבזאליים (המיוצגים על ידי m), כדלקמן:
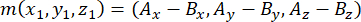
כאשר Ax, Ay ו- Az הן הקואורדינטות של הצנטרוזום האפיקאלי, ו- Bx, By ו- Bz הן הקואורדינטות של הצנטרוזומים הבסיסיים. באופן דומה, ציר וקטור החלוקה (המיוצג על ידי n) נוצר על ידי נקודת האמצע של הסיכות האפיות::EGFP סהר וקליפת המוח הבסיסית: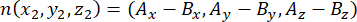
כאשר Ax, Ay ו-Az הן הקואורדינטות של נקודת האמצע של הפינים::EGFP סהר, ו-Bx, By ו-Bz הן הקואורדינטות של נקודת האמצע של קליפת המוח הבסיסית. - קבע את גודל הווקטורים m ו - n:
גודל של m: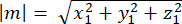
גודל n: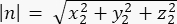
- קבע את מכפלת הנקודות (המיוצגת על ידי k) של m ו - n:
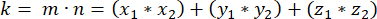
- באמצעות מכפלת הנקודות k והגדלים הווקטוריים m ו-n, לקבוע את הזווית בין הווקטורים:
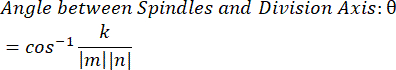
- התווה את הנתונים בתוכנה הנבחרת. הנתונים המוצגים כאן הוכנו ב- Microsoft Excel והוצגו באופן חזותי ב- PRISM.
תוצאות
דיסקציה והדמיה של NBs של אונת המוח המרכזית המבטאים סיכות::EGFP ודובדבן::יופיטר
כדי להציג את הפרוטוקול הזה, הזחלים מבטאים דובדבן מונע כטב"ם::יופיטר13 ומתויגים אנדוגנית סיכות::EGFP16 (w; worGal4, UAS-cherry::jupiter/CyO; פינים::EGFP/TM6B, Tb) צולמו במשך 4 שעות באמצעות הפרוטוקול המתואר ב...
Discussion
פרוטוקול זה מתווה גישה אחת לדימות של מוחות צמחים חיים מזחלי Drosophila melanogaster . הפרוטוקול המתואר כאן מאפשר לצפות במוחות של צמחים במשך 12-20 שעות בתנאי הניסוי המתאימים. יש לתת תשומת לב מיוחדת להכנת הדגימות ולתכנון הניסויים הרצויים. כאמור, אחד הגורמים הקריטיים ביותר הקובעים את איכות הרקמה המנ?...
Disclosures
למחברים אין גילויים כספיים להצהיר.
Acknowledgements
מחקר זה נתמך על ידי R35GM148160 (C. C.) ומענק הכשרה של המכונים הלאומיים לבריאות (NIH) T32 GM007270 (R. C. S)
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 µm polyethersulfone (PES) Membrane | Genesee | 25-231 | Vacuum-driven filters |
| Agar | Genesee | 20-248 | granulated agar |
| Analytical Computer | Dell | NA | Intel Xeon Gold 5222 CPU with two 3.80 GHz processors running Windows 10 on a 64-bit operating system |
| Bovine Growth Serum | HyClone | SH30541.02 | |
| Chambered Imaging Slides | Ibidi | 80826 | |
| Confocal Microscope | Nikon | NA | |
| Custom-machined metal slide | NA | NA | See Cabernard and Doe 2013 (Ref. 34) for specifications |
| Dissection Dishes | Fisher Scientific | 5024343 | 3-well porcelain micro spot plate |
| Dissection Forceps | World Precision Instruments | Dumont #5 | |
| Dissection Microscope | Leica | NA | |
| Dissection Scissors | Fine Science Tools (FST) | 15003-08 | |
| Embryo collection cage | Genesee | 59-100 | |
| Flypad with access to CO2 to anesthetize adult flies | Genesee | 59-172 | |
| Gas-permeable membrane | YSI | 98095 | Gas-permeable membrane |
| Glass Cover Slides | Electron Microscopy Sciences | 72204-03 | # 1.5; 22 mm x 40 mm glass coverslips |
| Imaris | Oxford Instruments | NA | Alternatives: Fiji, Volocity, Aivia |
| Imaris File Converter | Oxford Instruments | NA | |
| Instant Yeast | Saf-Instant | NA | |
| Molasses | Genesee | 62-117 | |
| Petri dish | Greiner Bio-One | 628161 | 60 mm x 15 mm Petri dish |
| Petroleum Jelly | Vaseline | NA | |
| Schneider's Insect Medium with L-glutamine and sodium bicarbonate liquid | Millipore Sigma | S0146 | |
| SlideBook acquisition software | 3i | NA | |
| Vacuum-Driven Filtration Unit with a 0.22 µµm PES membrane filter | Genesee Scientific, GenClone | 25-231 |
References
- Delgado, M. K., Cabernard, C. Mechanical regulation of cell size, fate, and behavior during asymmetric cell division. Current Opinion in Cell Biology. 67, 9-16 (2020).
- Sunchu, B., Cabernard, C. Principles and mechanisms of asymmetric cell division. Development. 147 (13), (2020).
- Homem, C. C. F., Knoblich, J. A. Drosophila neuroblasts: A model for stem cell biology. Development. 139 (23), 4297-4310 (2012).
- Gallaud, E., Pham, T., Cabernard, C. Drosophila melanogaster neuroblasts: A model for asymmetric stem cell divisions. Results and Problems in Cell Differentiation. 61 (1489), 183-210 (2017).
- Loyer, N., Januschke, J. Where does asymmetry come from? Illustrating principles of polarity and asymmetry establishment in Drosophila neuroblasts. Current Opinion in Cell Biology. 62, 70-77 (2020).
- Pollington, H. Q., Seroka, A. Q., Doe, C. Q. From temporal patterning to neuronal connectivity in Drosophila type I neuroblast lineages. Seminars in Cell & Developmental Biology. 142, 4-12 (2023).
- Oon, C. H., Prehoda, K. Asymmetric recruitment and actin dependent cortical flows drive the neuroblast polarity cycle. eLife. 8, e45815 (2019).
- Ramat, A., Hannaford, M., Januschke, J. Maintenance of miranda localization in Drosophila neuroblasts involves interaction with the cognate mRNA. Current Biology. 27 (14), 2101-2111 (2017).
- Oon, C. H., Prehoda, K. E. Phases of cortical actomyosin dynamics coupled to the neuroblast polarity cycle. eLife. 10, e66574 (2021).
- LaFoya, B., Prehoda, K. E. Actin-dependent membrane polarization reveals the mechanical nature of the neuroblast polarity cycle. Cell Reports. 35 (7), 109146 (2021).
- Siller, K. H., Doe, C. Q. Lis1/dynactin regulates metaphase spindle orientation in Drosophila neuroblasts. Developmental Biology. 319 (1), 1-9 (2008).
- Siller, K. H., Cabernard, C., Doe, C. Q. The NuMA-related Mud protein binds Pins and regulates spindle orientation in Drosophila neuroblasts. Nature Cell Biology. 8 (6), 594-600 (2006).
- Cabernard, C., Doe, C. Q. Apical/basal spindle orientation is required for neuroblast homeostasis and neuronal differentiation in Drosophila. Developmental Cell. 17 (1), 134-141 (2009).
- Cabernard, C., Prehoda, K. E., Doe, C. Q. A spindle-independent cleavage furrow positioning pathway. Nature. 467 (7311), 91-94 (2010).
- Connell, M., Cabernard, C., Ricketson, D., Doe, C. Q., Prehoda, K. E. Asymmetric cortical extension shifts cleavage furrow position in Drosophila neuroblasts. Molecular Biology of the Cell. 22 (22), 4220-4226 (2011).
- Tsankova, A., Pham, T. T., Garcia, D. S., Otte, F., Cabernard, C. Cell polarity regulates biased myosin activity and dynamics during asymmetric cell division via Drosophila rho kinase and protein kinase N. Developmental Cell. 42 (2), 143-155 (2017).
- Montembault, E., et al. Myosin efflux promotes cell elongation to coordinate chromosome segregation with cell cleavage. Nature Communications. 8 (1), 326 (2017).
- Roubinet, C., et al. Spatio-temporally separated cortical flows and spindle geometry establish physical asymmetry in fly neural stem cells. Nature Communications. 8 (1), 1383 (2017).
- Januschke, J., et al. Centrobin controls mother-daughter centriole asymmetry in Drosophila neuroblasts. Nature Cell Biology. 15 (3), 241-248 (2013).
- Januschke, J., Llamazares, S., Reina, J., Gonzalez, C. Drosophila neuroblasts retain the daughter centrosome. Nature Communications. 2 (1), 243 (2011).
- Rebollo, E., et al. Functionally unequal centrosomes drive spindle orientation in asymmetrically dividing Drosophila neural stem cells. Developmental Cell. 12 (3), 467-474 (2007).
- Januschke, J., Gonzalez, C. The interphase microtubule aster is a determinant of asymmetric division orientation in Drosophila neuroblasts. The Journal of Cell Biology. 188 (5), 693-706 (2010).
- Rusan, N. M., Peifer, M. A role for a novel centrosome cycle in asymmetric cell division. The Journal of Cell Biology. 177 (1), 13-20 (2007).
- Lerit, D. A., et al. Interphase centrosome organization by the PLP-Cnn scaffold is required for centrosome function. Journal of Cell Biology. 210 (1), 79-97 (2015).
- Gallaud, E., et al. Dynamic centriolar localization of Polo and Centrobin in early mitosis primes centrosome asymmetry. PLoS Biology. 18 (8), e3000762 (2020).
- Ramdas Nair, A., et al. The microcephaly-associated protein Wdr62/CG7337 is required to maintain centrosome asymmetry in Drosophila neuroblasts. Cell Reports. 14 (5), 1100-1113 (2016).
- Singh, P., Nair, A. R., Cabernard, C. The centriolar protein Bld10/Cep135 is required to establish centrosome asymmetry in Drosophila neuroblasts. Current Biology. 24 (13), 1548-1555 (2014).
- LaFoya, B., Prehoda, K. E. Consumption of a polarized membrane reservoir drives asymmetric membrane expansion during the unequal divisions of neural stem cells. Developmental Cell. 1534 (23), 00159 (2023).
- Sunchu, B., et al. Asymmetric chromatin retention and nuclear envelopes separate chromosomes in fused cells in vivo. Communications Biology. 5 (1), 953 (2022).
- Oliveira, A. C., Rebelo, A. R., Homem, C. C. F. Integrating animal development: How hormones and metabolism regulate developmental transitions and brain formation. Developmental Biology. 475, 256-264 (2021).
- Britton, J. S., Edgar, B. A. Environmental control of the cell cycle in Drosophila: nutrition activates mitotic and endoreplicative cells by distinct mechanisms. Development. 125 (11), 2149-2158 (1998).
- Lee, C. -. Y., et al. Drosophila Aurora-A kinase inhibits neuroblast self-renewal by regulating aPKC/Numb cortical polarity and spindle orientation. Genes & Development. 20 (24), 3464-3474 (2006).
- Homem, C. C. F., Reichardt, I., Berger, C., Lendl, T., Knoblich, J. A. Long-term live cell imaging and automated 4D analysis of Drosophila neuroblast lineages. PLoS ONE. 8 (11), e79588 (2013).
- Cabernard, C., Doe, C. Q. Live imaging of neuroblast lineages within intact larval brains in Drosophila. Cold Spring Harbor Protocols. 2013 (10), 970-977 (2013).
- Karpova, N., Bobinnec, Y., Fouix, S., Huitorel, P., Debec, A. Jupiter, a new Drosophila protein associated with microtubules. Cell Motility and the Cytoskeleton. 63 (5), 301-312 (2006).
- Loyer, N., Januschke, J. The last-born daughter cell contributes to division orientation of Drosophila larval neuroblasts. Nature Communications. 9 (1), 3745 (2018).
- Bostock, M. P., et al. An immobilization technique for long-term time-lapse imaging of explanted Drosophila tissues. Frontiers in Cell and Developmental Biology. 8, 590094 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved